hexsha
stringlengths 40
40
| size
int64 6
14.9M
| ext
stringclasses 1
value | lang
stringclasses 1
value | max_stars_repo_path
stringlengths 6
260
| max_stars_repo_name
stringlengths 6
119
| max_stars_repo_head_hexsha
stringlengths 40
41
| max_stars_repo_licenses
sequence | max_stars_count
int64 1
191k
⌀ | max_stars_repo_stars_event_min_datetime
stringlengths 24
24
⌀ | max_stars_repo_stars_event_max_datetime
stringlengths 24
24
⌀ | max_issues_repo_path
stringlengths 6
260
| max_issues_repo_name
stringlengths 6
119
| max_issues_repo_head_hexsha
stringlengths 40
41
| max_issues_repo_licenses
sequence | max_issues_count
int64 1
67k
⌀ | max_issues_repo_issues_event_min_datetime
stringlengths 24
24
⌀ | max_issues_repo_issues_event_max_datetime
stringlengths 24
24
⌀ | max_forks_repo_path
stringlengths 6
260
| max_forks_repo_name
stringlengths 6
119
| max_forks_repo_head_hexsha
stringlengths 40
41
| max_forks_repo_licenses
sequence | max_forks_count
int64 1
105k
⌀ | max_forks_repo_forks_event_min_datetime
stringlengths 24
24
⌀ | max_forks_repo_forks_event_max_datetime
stringlengths 24
24
⌀ | avg_line_length
float64 2
1.04M
| max_line_length
int64 2
11.2M
| alphanum_fraction
float64 0
1
| cells
sequence | cell_types
sequence | cell_type_groups
sequence |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
e7e8319be7deb5116f6796eda2c849c4d661e8b0 | 4,256 | ipynb | Jupyter Notebook | notebooks/Landkreis.ipynb | schnabel/corona-notbremse | 2f60992e30d3c9e8dba26ca76c76aa8d3224ce07 | [
"MIT"
] | null | null | null | notebooks/Landkreis.ipynb | schnabel/corona-notbremse | 2f60992e30d3c9e8dba26ca76c76aa8d3224ce07 | [
"MIT"
] | null | null | null | notebooks/Landkreis.ipynb | schnabel/corona-notbremse | 2f60992e30d3c9e8dba26ca76c76aa8d3224ce07 | [
"MIT"
] | null | null | null | 25.333333 | 134 | 0.417998 | [
[
[
"import pandas as pd",
"_____no_output_____"
],
[
"cols = [\"AdmUnitId\", \"EWZ\", \"GEN\", \"BEZ\"]\ndf = pd.read_csv(\"https://npgeo-corona-npgeo-de.hub.arcgis.com/datasets/917fc37a709542548cc3be077a786c17_0.csv\", usecols=cols)",
"_____no_output_____"
],
[
"df.head()",
"_____no_output_____"
],
[
"df['Region_Prefix'] = \"LK \"\ndf.loc[df['BEZ'] != 'Landkreis', 'Region_Prefix'] = \"SK \"\ndf['Region'] = df['Region_Prefix'] + df['GEN']",
"_____no_output_____"
],
[
"df.to_csv('../Einwohnerzahlen.csv', index=False)",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7e83914ca553ea96ea390d86dde567563dbc69e | 31,373 | ipynb | Jupyter Notebook | SageMaker/Training-Inference/6. BYOC Sklearn/BYOC Sklearn.ipynb | arunprsh/AI-ML-Examples | 3241d658b398e73b94a5cf59ca4b326d1c4219b5 | [
"Apache-2.0"
] | 1 | 2021-05-17T17:37:23.000Z | 2021-05-17T17:37:23.000Z | SageMaker/Training-Inference/6. BYOC Sklearn/BYOC Sklearn.ipynb | arunprsh/AI-ML-Examples | 3241d658b398e73b94a5cf59ca4b326d1c4219b5 | [
"Apache-2.0"
] | null | null | null | SageMaker/Training-Inference/6. BYOC Sklearn/BYOC Sklearn.ipynb | arunprsh/AI-ML-Examples | 3241d658b398e73b94a5cf59ca4b326d1c4219b5 | [
"Apache-2.0"
] | 2 | 2020-10-22T18:37:44.000Z | 2020-10-23T13:55:23.000Z | 28.888582 | 154 | 0.514614 | [
[
[
"### 1. Create Train Script ",
"_____no_output_____"
]
],
[
[
"%%file train\n#!/usr/bin/env python\n\nfrom sklearn.neighbors import KNeighborsClassifier\nimport pandas as pd\nimport numpy as np\nimport pickle\nimport os\n\n\nnp.random.seed(123)\n\n# Define paths for Model Training inside Container.\nINPUT_PATH = '/opt/ml/input/data'\nOUTPUT_PATH = '/opt/ml/output'\nMODEL_PATH = '/opt/ml/model'\nPARAM_PATH = '/opt/ml/input/config/hyperparameters.json'\n\n# Training data sitting in S3 will be copied to this location during training when used with File MODE.\nTRAIN_DATA_PATH = f'{INPUT_PATH}/train'\nTEST_DATA_PATH = f'{INPUT_PATH}/test'\n\ndef train():\n print(\"------- [STARTING TRAINING] -------\")\n train_df = pd.read_csv(os.path.join(TRAIN_DATA_PATH, 'train.csv'), names=['class', 'mass', 'width', 'height', 'color_score'])\n train_df.head()\n X_train = train_df[['mass', 'width', 'height', 'color_score']]\n y_train = train_df['class']\n knn = KNeighborsClassifier()\n knn.fit(X_train, y_train)\n # Save the trained Model inside the Container\n with open(os.path.join(MODEL_PATH, 'model.pkl'), 'wb') as out:\n pickle.dump(knn, out)\n print(\"------- [TRAINING COMPLETE!] -------\")\n \n print(\"------- [STARTING EVALUATION] -------\")\n test_df = pd.read_csv(os.path.join(TEST_DATA_PATH, 'test.csv'), names=['class', 'mass', 'width', 'height', 'color_score'])\n X_test = train_df[['mass', 'width', 'height', 'color_score']]\n y_test = train_df['class']\n acc = knn.score(X_test, y_test)\n print('Accuracy = {:.2f}%'.format(acc * 100))\n print(\"------- [EVALUATION DONE!] -------\")\n\nif __name__ == '__main__':\n train()",
"Overwriting train\n"
]
],
[
[
"### 2. Create Serve Script",
"_____no_output_____"
]
],
[
[
"%%file serve\n#!/usr/bin/env python\n\nfrom flask import Flask, Response, request\nfrom io import StringIO\nimport pandas as pd\nimport logging\nimport pickle\nimport os\n\n\napp = Flask(__name__)\n\nMODEL_PATH = '/opt/ml/model'\n\n# Singleton Class for holding the Model\nclass Predictor:\n model = None\n \n @classmethod\n def load_model(cls):\n print('[LOADING MODEL]')\n if cls.model is None:\n with open(os.path.join(MODEL_PATH, 'model.pkl'), 'rb') as file_:\n cls.model = pickle.load(file_)\n print('MODEL LOADED!')\n return cls.model\n \n @classmethod\n def predict(cls, X):\n clf = cls.load_model()\n return clf.predict(X)\n\[email protected]('/ping', methods=['GET'])\ndef ping():\n print('[HEALTH CHECK]')\n model = Predictor.load_model()\n status = 200\n if model is None:\n status = 404\n return Response(response={\"HEALTH CHECK\": \"OK\"}, status=status, mimetype='application/json')\n\[email protected]('/invocations', methods=['POST'])\ndef invoke():\n data = None\n\n # Transform Payload in CSV to Pandas DataFrame.\n if request.content_type == 'text/csv':\n data = request.data.decode('utf-8')\n data = StringIO(data)\n data = pd.read_csv(data, header=None)\n else:\n return flask.Response(response='This Predictor only supports CSV data', status=415, mimetype='text/plain')\n\n logging.info('Invoked with {} records'.format(data.shape[0]))\n \n predictions = Predictor.predict(data)\n\n # Convert from numpy back to CSV\n out = StringIO()\n pd.DataFrame({'results': predictions}).to_csv(out, header=False, index=False)\n result = out.getvalue()\n\n return Response(response=result, status=200, mimetype='text/csv')\n\nif __name__ == '__main__':\n app.run(host='0.0.0.0', port=8080)",
"Overwriting serve\n"
]
],
[
[
"### 3. Build a Docker Image and Push to ECR",
"_____no_output_____"
]
],
[
[
"%%sh\n\n# Assign a name for your Docker image.\nimage_name=byoc-sklearn\necho \"Image Name: ${image_name}\" \n\n# Retrieve AWS Account.\naccount=$(aws sts get-caller-identity --query Account --output text)\n\n# Get the region defined in the current configuration (default to us-east-1 if none defined).\nregion=$(aws configure get region)\nregion=${region:-us-east-1}\n\necho \"Account: ${account}\" \necho \"Region: ${region}\"\n\nrepository=\"${account}.dkr.ecr.${region}.amazonaws.com\"\necho \"Repository: ${repository}\" \n\nimage=\"${account}.dkr.ecr.${region}.amazonaws.com/${image_name}:latest\"\necho \"Image URI: ${image}\" \n\n# If the repository does not exist in ECR, create it.\naws ecr describe-repositories --repository-names ${image_name} > /dev/null 2>&1\nif [ $? -ne 0 ]\nthen\naws ecr create-repository --repository-name ${image_name} > /dev/null\nfi\n\n# Get the login command from ECR and execute it directly.\naws ecr get-login-password --region ${region} | docker login --username AWS --password-stdin ${repository}\n\n# Build the docker image locally with the image name and tag it.\ndocker build -t ${image_name} .\ndocker tag ${image_name} ${image}\n\n# Finally, push image to ECR with the full image name.\ndocker push ${image}",
"Image Name: byoc-sklearn\nAccount: 892313895307\nRegion: us-east-1\nRepository: 892313895307.dkr.ecr.us-east-1.amazonaws.com\nImage URI: 892313895307.dkr.ecr.us-east-1.amazonaws.com/byoc-sklearn:latest\nLogin Succeeded\nSending build context to Docker daemon 80.38kB\r\r\nStep 1/8 : FROM python:3.7\n ---> 5b86e11778a2\nStep 2/8 : COPY requirements.txt ./\n ---> Using cache\n ---> 8623cb69764a\nStep 3/8 : RUN pip install --no-cache-dir -r requirements.txt\n ---> Using cache\n ---> 00be6a106a8c\nStep 4/8 : COPY train /usr/local/bin\n ---> Using cache\n ---> f55d18c34b89\nStep 5/8 : RUN chmod +x /usr/local/bin/train\n ---> Using cache\n ---> aae62ce0c43b\nStep 6/8 : COPY serve /usr/local/bin\n ---> Using cache\n ---> d9408249ae77\nStep 7/8 : RUN chmod +x /usr/local/bin/serve\n ---> Using cache\n ---> 04fc001c0b7c\nStep 8/8 : EXPOSE 8080\n ---> Using cache\n ---> 6990c97b2383\nSuccessfully built 6990c97b2383\nSuccessfully tagged byoc-sklearn:latest\nThe push refers to repository [892313895307.dkr.ecr.us-east-1.amazonaws.com/byoc-sklearn]\n032f1a03bf08: Preparing\n053f064686a0: Preparing\n59239f9a3c52: Preparing\n34bf625dab71: Preparing\n415a4c435e2d: Preparing\na9066f74cbd8: Preparing\n1b17be258ee0: Preparing\n6522a2852221: Preparing\n56a69ef72608: Preparing\n6f7043721c9b: Preparing\na933681cf349: Preparing\nf49d20b92dc8: Preparing\nfe342cfe5c83: Preparing\n630e4f1da707: Preparing\n9780f6d83e45: Preparing\n56a69ef72608: Waiting\nf49d20b92dc8: Waiting\n6f7043721c9b: Waiting\nfe342cfe5c83: Waiting\n630e4f1da707: Waiting\n9780f6d83e45: Waiting\na933681cf349: Waiting\n1b17be258ee0: Waiting\na9066f74cbd8: Waiting\n6522a2852221: Waiting\n34bf625dab71: Layer already exists\n032f1a03bf08: Layer already exists\n053f064686a0: Layer already exists\n415a4c435e2d: Layer already exists\n59239f9a3c52: Layer already exists\na9066f74cbd8: Layer already exists\n1b17be258ee0: Layer already exists\n6f7043721c9b: Layer already exists\n56a69ef72608: Layer already exists\n6522a2852221: Layer already exists\nf49d20b92dc8: Layer already exists\na933681cf349: Layer already exists\nfe342cfe5c83: Layer already exists\n9780f6d83e45: Layer already exists\n630e4f1da707: Layer already exists\nlatest: digest: sha256:a2ebe6e788d472b87131c8ee6e7ef75d3e2cb27b01d9f1d41d6d4e88274d0e5e size: 3467\n"
]
],
[
[
"### Imports ",
"_____no_output_____"
]
],
[
[
"from sagemaker.predictor import csv_serializer\nimport pandas as pd\nimport sagemaker",
"_____no_output_____"
]
],
[
[
"### Essentials",
"_____no_output_____"
]
],
[
[
"role = sagemaker.get_execution_role()\nsession = sagemaker.Session()\naccount = session.boto_session.client('sts').get_caller_identity()['Account']\nregion = session.boto_session.region_name\nimage_name = 'byoc-sklearn'\nimage_uri = f'{account}.dkr.ecr.{region}.amazonaws.com/{image_name}:latest'",
"_____no_output_____"
]
],
[
[
"### Train (Local Mode)",
"_____no_output_____"
]
],
[
[
"model = sagemaker.estimator.Estimator(\n image_name=image_uri,\n role=role,\n train_instance_count=1,\n train_instance_type='local',\n sagemaker_session=None\n)",
"Parameter image_name will be renamed to image_uri in SageMaker Python SDK v2.\n"
],
[
"model.fit({'train': 'file://.././DATA/train/train.csv', 'test': 'file://.././DATA/test/test.csv'})",
"Creating tmp815252o0_algo-1-n7amc_1 ... \n\u001b[1BAttaching to tmp815252o0_algo-1-n7amc_12mdone\u001b[0m\n\u001b[36malgo-1-n7amc_1 |\u001b[0m ------- [STARTING TRAINING] -------\n\u001b[36malgo-1-n7amc_1 |\u001b[0m ------- [TRAINING COMPLETE!] -------\n\u001b[36malgo-1-n7amc_1 |\u001b[0m ------- [STARTING EVALUATION] -------\n\u001b[36malgo-1-n7amc_1 |\u001b[0m Accuracy = 97.73%\n\u001b[36malgo-1-n7amc_1 |\u001b[0m ------- [EVALUATION DONE!] -------\n\u001b[36mtmp815252o0_algo-1-n7amc_1 exited with code 0\n\u001b[0mAborting on container exit...\n===== Job Complete =====\n"
]
],
[
[
"### Deploy (Locally)",
"_____no_output_____"
]
],
[
[
"predictor = model.deploy(1, 'local', endpoint_name='byoc-sklearn', serializer=csv_serializer)",
"Parameter image will be renamed to image_uri in SageMaker Python SDK v2.\n"
]
],
[
[
"### Evaluate Real Time Inference (Locally)",
"_____no_output_____"
]
],
[
[
"df = pd.read_csv('.././DATA/test/test.csv', header=None)\ntest_df = df.sample(1)",
"_____no_output_____"
],
[
"test_df",
"_____no_output_____"
],
[
"test_df.drop(test_df.columns[[0]], axis=1, inplace=True)\ntest_df",
"_____no_output_____"
],
[
"test_df.values",
"_____no_output_____"
],
[
"prediction = predictor.predict(test_df.values).decode('utf-8').strip()",
"\u001b[36malgo-1-g5u49_1 |\u001b[0m [LOADING MODEL]\n\u001b[36malgo-1-g5u49_1 |\u001b[0m MODEL LOADED!\n\u001b[36malgo-1-g5u49_1 |\u001b[0m 172.18.0.1 - - [04/Nov/2020 00:18:22] \"\u001b[37mPOST /invocations HTTP/1.1\u001b[0m\" 200 -\n\u001b[36malgo-1-g5u49_1 |\u001b[0m INFO:werkzeug:172.18.0.1 - - [04/Nov/2020 00:18:22] \"\u001b[37mPOST /invocations HTTP/1.1\u001b[0m\" 200 -\n"
],
[
"prediction",
"_____no_output_____"
]
],
[
[
"### Train (using SageMaker)",
"_____no_output_____"
]
],
[
[
"WORK_DIRECTORY = '.././DATA'\n\ntrain_data_s3_pointer = session.upload_data(f'{WORK_DIRECTORY}/train', key_prefix='byoc-sklearn/train')\ntest_data_s3_pointer = session.upload_data(f'{WORK_DIRECTORY}/test', key_prefix='byoc-sklearn/test')",
"_____no_output_____"
],
[
"train_data_s3_pointer",
"_____no_output_____"
],
[
"test_data_s3_pointer",
"_____no_output_____"
],
[
"model = sagemaker.estimator.Estimator(\n image_name=image_uri,\n role=role,\n train_instance_count=1,\n train_instance_type='ml.m5.xlarge',\n sagemaker_session=session # ensure the session is set to session\n)",
"Parameter image_name will be renamed to image_uri in SageMaker Python SDK v2.\n"
],
[
"model.fit({'train': train_data_s3_pointer, 'test': test_data_s3_pointer})",
"'s3_input' class will be renamed to 'TrainingInput' in SageMaker Python SDK v2.\n's3_input' class will be renamed to 'TrainingInput' in SageMaker Python SDK v2.\n"
]
],
[
[
"### Deploy Trained Model as SageMaker Endpoint",
"_____no_output_____"
]
],
[
[
"predictor = model.deploy(1, 'ml.m5.xlarge', endpoint_name='byoc-sklearn', serializer=csv_serializer)",
"Parameter image will be renamed to image_uri in SageMaker Python SDK v2.\n"
]
],
[
[
"### Real Time Inference using Deployed Endpoint",
"_____no_output_____"
]
],
[
[
"df = pd.read_csv('.././DATA/test/test.csv', header=None)\ntest_df = df.sample(1)",
"_____no_output_____"
],
[
"test_df.drop(test_df.columns[[0]], axis=1, inplace=True)\ntest_df",
"_____no_output_____"
],
[
"test_df.values",
"_____no_output_____"
],
[
"prediction = predictor.predict(test_df.values).decode('utf-8').strip()",
"_____no_output_____"
],
[
"prediction",
"_____no_output_____"
]
],
[
[
"### Batch Transform (Batch Inference) using Trained SageMaker Model",
"_____no_output_____"
]
],
[
[
"bucket_name = session.default_bucket()\noutput_path = f's3://{bucket_name}/byoc-sklearn/batch_test_out'\n\ntransformer = model.transformer(instance_count=1, \n instance_type='ml.m5.xlarge', \n output_path=output_path, \n assemble_with='Line', \n accept='text/csv')",
"Parameter image will be renamed to image_uri in SageMaker Python SDK v2.\nUsing already existing model: byoc-sklearn-2020-11-04-17-32-19-621\n"
],
[
"WORK_DIRECTORY = '.././DATA'\n\nbatch_input = session.upload_data(f'{WORK_DIRECTORY}/batch_test', key_prefix='byoc-sklearn/batch_test')",
"_____no_output_____"
],
[
"transformer.transform(batch_input, content_type='text/csv', split_type='Line', input_filter='$')\ntransformer.wait()",
".Gracefully stopping... (press Ctrl+C again to force)\n.........................\n\u001b[34m * Serving Flask app \"serve\" (lazy loading)\n * Environment: production\n WARNING: This is a development server. Do not use it in a production deployment.\n Use a production WSGI server instead.\n * Debug mode: off\n * Running on http://0.0.0.0:8080/ (Press CTRL+C to quit)\u001b[0m\n\u001b[34m169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mGET /ping HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[34m169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[33mGET /execution-parameters HTTP/1.1#033[0m\" 404 -\u001b[0m\n\u001b[34m169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mPOST /invocations HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[34mINFO:werkzeug:169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mPOST /invocations HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[34m169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mPOST /invocations HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[34mINFO:werkzeug:169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mPOST /invocations HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[35m * Serving Flask app \"serve\" (lazy loading)\n * Environment: production\n WARNING: This is a development server. Do not use it in a production deployment.\n Use a production WSGI server instead.\n * Debug mode: off\n * Running on http://0.0.0.0:8080/ (Press CTRL+C to quit)\u001b[0m\n\u001b[35m169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mGET /ping HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[35m169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[33mGET /execution-parameters HTTP/1.1#033[0m\" 404 -\u001b[0m\n\u001b[35m169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mPOST /invocations HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[35mINFO:werkzeug:169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mPOST /invocations HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[35m169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mPOST /invocations HTTP/1.1#033[0m\" 200 -\u001b[0m\n\u001b[35mINFO:werkzeug:169.254.255.130 - - [04/Nov/2020 18:01:34] \"#033[37mPOST /invocations HTTP/1.1#033[0m\" 200 -\u001b[0m\n"
]
],
[
[
"#### Inspect Batch Transformed Output",
"_____no_output_____"
]
],
[
[
"s3_client = session.boto_session.client('s3')\ns3_client.download_file(bucket_name, \n 'byoc-sklearn/batch_test_out/batch_test.csv.out', \n '.././DATA/batch_test/batch_test.csv.out')",
"_____no_output_____"
],
[
"with open('.././DATA/batch_test/batch_test.csv.out', 'r') as f:\n results = f.readlines() \n \nprint(\"Transform results: \\n{}\".format(''.join(results)))",
"Transform results: \n1\n3\n0\n1\n1\n3\n1\n3\n0\n0\n0\n3\n0\n0\n2\n\n"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
]
] |
e7e843c6ac9f128219d1dd6b90ad6ab1487ae5a8 | 48,770 | ipynb | Jupyter Notebook | notebooks/dlp11_part01_introduction.ipynb | codingalzi/dlp | aac2df75ab9d51570dad9a17573987c9b4a89af0 | [
"MIT"
] | null | null | null | notebooks/dlp11_part01_introduction.ipynb | codingalzi/dlp | aac2df75ab9d51570dad9a17573987c9b4a89af0 | [
"MIT"
] | null | null | null | notebooks/dlp11_part01_introduction.ipynb | codingalzi/dlp | aac2df75ab9d51570dad9a17573987c9b4a89af0 | [
"MIT"
] | null | null | null | 28.371146 | 2,089 | 0.50691 | [
[
[
"# 11장 자연어처리 1부",
"_____no_output_____"
],
[
"**감사말**: 프랑소와 숄레의 [Deep Learning with Python, Second Edition](https://www.manning.com/books/deep-learning-with-python-second-edition?a_aid=keras&a_bid=76564dff) 10장에 사용된 코드에 대한 설명을 담고 있으며 텐서플로우 2.6 버전에서 작성되었습니다. 소스코드를 공개한 저자에게 감사드립니다.\n\n**tensorflow 버전과 GPU 확인**\n- 구글 코랩 설정: '런타임 -> 런타임 유형 변경' 메뉴에서 GPU 지정 후 아래 명령어 실행 결과 확인\n\n ```\n !nvidia-smi\n ```\n\n- 사용되는 tensorflow 버전 확인\n\n ```python\n import tensorflow as tf\n tf.__version__\n ```\n- tensorflow가 GPU를 사용하는지 여부 확인\n\n ```python\n tf.config.list_physical_devices('GPU')\n ```",
"_____no_output_____"
],
[
"## 주요내용",
"_____no_output_____"
],
[
"- 자연어처리(Natural Language Processing) 소개\n - 단어주머니(bag-of-words) 모델\n - 순차(sequence) 모델\n- 순차 모델 활용\n - 양방향 순환신경망(bidirectional LSTM) 적용\n- 트랜스포머(Transformer) 활용\n- 시퀀스-투-시퀀스(seq2seq) 모델 활용",
"_____no_output_____"
],
[
"## 11.1 자연어처리 소개",
"_____no_output_____"
],
[
"파이썬, 자바, C, C++, C#, 자바스크립트 등 컴퓨터 프로그래밍언어와 구분하기 위해 \n일상에서 사용되는 한국어, 영어 등을 __자연어__(natural language)라 부른다. \n\n자연어의 특성상 정확한 분석을 위한 알고리즘을 구현하는 일은 사실상 매우 어렵다. \n딥러닝 기법이 활용되기 이전깢지 적절한 규칙을 구성하여 자연어를 이해하려는 \n수 많은 시도가 있어왔지만 별로 성공적이지 않았다.\n\n1990년대부터 인터넷으로부터 구해진 엄청난 양의 텍스트 데이터에 머신러닝 기법을\n적용하기 시작했다. 단, 주요 목적이 **언어의 이해**가 아니라 \n아래 예제들처럼 입력 텍스트를 분석하여\n**통계적으로 유용한 정보를 예측**하는 방향으로 수정되었다.\n\n- 텍스트 분류: \"이 문장의 주제는?\"\n- 내용 필터링: \"욕설이 포함되었나?\"\n- 감성 분석: \"내용이 긍정이야 부정이야?\"\n- 언어 모델링: \"이 문장에 이어 어떤 단어가 있어야 하지?\"\n- 번역: \"이거를 한국어로 어떻게 말해?\"\n- 요약: \"이 기사를 한 줄로 요약하면?\"\n\n이와 같은 분석을 **자연어처리**(NLP, Natural Language Processing)이라 하며\n단어(words), 문장(sentences), 문단(paragraphs) 등에서 찾을 수 있는\n패턴(pattern)을 인식하려 시도한다. ",
"_____no_output_____"
],
[
"**머신러닝 활용**",
"_____no_output_____"
],
[
"자연어처리를 위해 1990년대부터 시작된 머신러닝 활용의 변화과정은 다음과 같다.",
"_____no_output_____"
],
[
"- 1990 - 2010년대 초반: \n 결정트리(decision trees), 로지스틱 회귀(logistic regression) 모델이 주로 활용됨.\n\n- 2014-2015: LSTM 등 시퀀스 처리 알고리즘 활용 시작\n\n- 2015-2017: (양방향) 순환신경망이 기본적으로 활용됨.\n\n- 2017-2018: 트랜스포머(Transformer) 모델이 최고의 성능 발휘하며, \n 많은 난제들을 해결함. 현재 가장 많이 활용되는 모델임.",
"_____no_output_____"
],
[
"## 11.2 텍스트 벡터화",
"_____no_output_____"
],
[
"딥러닝 모델은 텍스트 자체를 처리할 수 없다.\n따라서 택스트를 수치형 텐서(numeric tensors)로 변환하는 \n**텍스트 벡터화**(text vectorization) 과정이 요구되며\n보통 다음 세 단계를 따른다.\n\n1. **텍스트 표준화**(text standardization): 소문자화, 마침표 제거 등등\n1. **토큰화**(tokenization): 기본 단위의 **유닛**(units)으로 쪼개기\n - 토큰 예제: 문자, 단어, 단언들의 집합 등등\n1. **어휘 색인화**(vocabulary indexing): 토큰 각각을 하나의 수치형 벡터(numerical vector)로 변환.\n\n아래 그림은 텍스트 벡터화의 기본적인 과정을 잘 보여준다.",
"_____no_output_____"
],
[
"<div align=\"center\"><img src=\"https://drek4537l1klr.cloudfront.net/chollet2/Figures/11-01.png\" style=\"width:60%;\"></div>\n\n그림 출처: [Deep Learning with Python(Manning MEAP)](https://www.manning.com/books/deep-learning-with-python-second-edition)",
"_____no_output_____"
],
[
"**텍스트 표준화**",
"_____no_output_____"
],
[
"다음 두 문장을 표준화를 통해 동일한 문장으로 변환해보자.\n\n- \"sunset came. i was staring at the Mexico sky. Isnt nature splendid??\"\n- \"Sunset came; I stared at the México sky. Isn't nature splendid?\"",
"_____no_output_____"
],
[
"예를 들어 다음 표준화 기법을 사용할 수 있다.\n\n- 모두 소문자화\n- `.`, `;`, `?`, `'` 등 특수 기호 제거\n- 특수 알파벳 변환: \"é\"를 \"e\"로, \"æ\"를 \"ae\"로 등등\n- 동사/명사의 기본형 활용: \"cats\"를 \"[cat]\"로, \"was staring\"과 \"stared\"를 \"[stare]\"로 등등.",
"_____no_output_____"
],
[
"그러면 위 두 문장 모두 아래 문장으로 변환된다.",
"_____no_output_____"
],
[
"- \"sunset came i [stare] at the mexico sky isnt nature splendid\"",
"_____no_output_____"
],
[
"표준화 과정을 통해 어느 정도의 정보를 상실하게 되지만\n학습해야할 내용을 줄여 일반화 성능이 보다 좋은 모델을 훈련시키는 장점이 있다.\n하지만 분석 목적에 따라 표준화 기법은 경우에 따라 달라질 수 있음에 주의해야 한다. \n예를 들어 인터뷰 기사의 경우 물음표(`?`)는 제거하면 안된다.",
"_____no_output_____"
],
[
"**토큰화**",
"_____no_output_____"
],
[
"텍스트 표준화 이후 데이터 분석의 기본 단위인 토큰으로 쪼개야 한다.\n보통 아래 세 가지 방식 중에 하나를 사용한다.\n\n- 단어 기준 토큰화(word-level tokenization)\n - 공백으로 구분된 단어들로 쪼개기. \n - 경우에 따라 동사 어근과 어미를 구분하기도 함: \"star+ing\", \"call+ed\" 등등\n- N-그램 토큰화(N-gram tokenization)\n - N-그램 토큰: 연속으로 위치한 N 개(이하)의 단어 묶음\n - 예제: \"the cat\", \"he was\" 등은 2-그램 토큰이다.\n- 문자 기준 토큰화(character-level tokenization)\n - 하나의 문자가 하나의 토큰임.\n - 문장 생성, 음성 인식 등에서 활용됨.",
"_____no_output_____"
],
[
"일반적으로 문자 기준 토큰화는 잘 사용되지 않는다. \n여기서도 단어 기준 또는 N-그램 토큰화만 이용한다.\n\n- 단어 기준 토큰화: 단어들의 순서를 중요시하는 **순차 모델**(sequence models)을 사용할 경우 활용\n- N-그램 토큰화: 단언들의 순서를 별로 상관하지 않는 **단어주머니(bag-of-words, BOW)** \n 모델을 사용할 경우 활용\n - N-그램: 단어들 사이의 순서에 대한 지역 정보를 어느 정도 유지함.\n - 일종의 특성 공학(feature engineering) 기법이며 따라서 \n 얕은 학습 기반의 언어처리(shallow language-processing) 모델에 활용됨.\n - 1차원 합성곱 신경망, 순환 신경망, 트랜스포머 등은 이 기법을 사용하지 않아도 됨.",
"_____no_output_____"
],
[
"**단어주머니(bag-of-words)**는 N-토큰으로 구성된 집합을 의미하며 \n**N-그램 주머니(bag-of-N-grams)**라고 불리기도 한다.\n예를 들어 \"the cat sat on the mat.\" 문장에 대한 \n2-그램 집합과 3-그램 집합은 각각 다음과 같다.",
"_____no_output_____"
],
[
"- 2-그램 집합\n\n```\n{\"the\", \"the cat\", \"cat\", \"cat sat\", \"sat\",\n \"sat on\", \"on\", \"on the\", \"the mat\", \"mat\"}\n```",
"_____no_output_____"
],
[
"- 3-그램 집합\n\n```\n{\"the\", \"the cat\", \"cat\", \"cat sat\", \"the cat sat\",\n \"sat\", \"sat on\", \"on\", \"cat sat on\", \"on the\",\n \"sat on the\", \"the mat\", \"mat\", \"on the mat\"}\n ```",
"_____no_output_____"
],
[
"**어휘 색인화**",
"_____no_output_____"
],
[
"일반적으로 먼저 훈련셋에 포함된 모든 토큰들의 색인(인덱스)을 작성한다.\n생성된 색인을 각 토큰을 바탕으로 원-핫, 멀티-핫 인코딩 등의 방식을 사용하여\n수치형 텐서로 변환한다.\n\n[4장](https://codingalzi.github.io/dlp/notebooks/dlp04_getting_started_with_neural_networks.html)과 \n[5장](https://codingalzi.github.io/dlp/notebooks/dlp05_fundamentals_of_ml.html)에서 \n설명한 대로 보통 사용 빈도수가 높은 2만 또는 3만 개의 단어만을 대상으로 어휘 색인화를 진행한다.\n당시에 `num_words=10000`을 사용하여 사용 빈도수가 상위 1만 등 안에 드는 단어만을\n대상으로 훈련셋을 구성하였다.\n\n```python\nfrom tensorflow.keras.datasets import imdb\n(train_data, train_labels), (test_data, test_labels) = imdb.load_data(num_words=10000)\n```\n\n케라스의 imdb 데이터셋은 이미 정수들의 시퀀스로 전처리가 되어 있다. \n하지만 여기서는 원본 imdb 데이터셋을 대상으로 전처리를 직접 수행하는 단계부터 살펴볼 것이다.\n이를 위해 아래 사항을 기억해 두어야 한다.\n\n- OOV 인덱스 활용: 어휘 색인에 포함되지 않는 단어는 모두 1로 처리. \n 일반 문장으로 번역되는 경우 \"[UNK]\" 으로 처리됨.\n - OOV = Out Of Vocabulary\n - UNK = Unknown\n- 마스크(mask) 토큰: 무신되어야 하는 토큰을 나타냄. 모두 0으로 처리.\n - 예를 들어, 문장의 길이를 맞추기 위해 사용되는 패딩으로 0으로 채워줄 수 있음.\n \n ```\n [[5, 7, 124, 4, 89]\n [8, 34, 21, 0, 0]]\n ```",
"_____no_output_____"
],
[
"**케라스의 `TextVectorization` 층 활용**",
"_____no_output_____"
],
[
"지금까지 설명한 텍스트 벡터화를 위해 케라스의 `TextVectorization` 층을 활용할 수 있으며\n기본 사용법은 다음과 같다.",
"_____no_output_____"
]
],
[
[
"from tensorflow.keras.layers import TextVectorization\ntext_vectorization = TextVectorization(\n output_mode=\"int\",\n )",
"_____no_output_____"
]
],
[
[
"`TextVectorization` 층 구성에 사용되는 주요 기본 설정은 다음과 같다.\n\n- 표준화: 소문자화와 마침표 등 제거\n - `standardize='lower_and_strip_punctuation'`\n- 토큰화: 단어 기준 쪼개기\n - `ngrams=None`\n - `split='whitespace'`\n- 출력 모드: 출력 텐서의 형식\n - `output_mode=\"int\"`",
"_____no_output_____"
],
[
"표준화와 토큰화 방식을 임의로 지정해서 활용할 수도 있다.\n다만, 파이썬의 기본 문자열 자료형인 `str` 대신에 `tf.string` 텐서를 활용해야 함에 주의해야 한다. \n표준화와 토큰화의 기본값은 아래 두 함수를 활용하는 것과 동일하다.\n\n- `custom_standardization_fn()`\n- `custom_split_fn()`",
"_____no_output_____"
]
],
[
[
"import re\nimport string\nimport tensorflow as tf\n\n# 표준화: 소문자화 및 마침표 제거\ndef custom_standardization_fn(string_tensor):\n lowercase_string = tf.strings.lower(string_tensor)\n return tf.strings.regex_replace(\n lowercase_string, f\"[{re.escape(string.punctuation)}]\", \"\")\n\n# 공백 기준으로 쪼개기\ndef custom_split_fn(string_tensor):\n return tf.strings.split(string_tensor)\n\n# 사용자 정의 표준화 및 쪼개기 활용\ntext_vectorization = TextVectorization(\n output_mode=\"int\",\n standardize=custom_standardization_fn,\n split=custom_split_fn,\n)",
"_____no_output_____"
]
],
[
[
"**예제**",
"_____no_output_____"
],
[
"아래 데이터셋을 대상으로 텍스트 벡터화를 진행해보자.",
"_____no_output_____"
]
],
[
[
"dataset = [\n \"I write, erase, rewrite\",\n \"Erase again, and then\",\n \"A poppy blooms.\",\n]",
"_____no_output_____"
],
[
"text_vectorization.adapt(dataset)",
"_____no_output_____"
]
],
[
[
"생성된 어휘 색인은 다음과 같다.",
"_____no_output_____"
]
],
[
[
"vocabulary = text_vectorization.get_vocabulary()\nvocabulary",
"_____no_output_____"
]
],
[
[
"생성된 어휘 색인을 활용하여 새로운 문장을 벡터화 해보자.",
"_____no_output_____"
]
],
[
[
"test_sentence = \"I write, rewrite, and still rewrite again\"",
"_____no_output_____"
],
[
"encoded_sentence = text_vectorization(test_sentence)\nprint(encoded_sentence)",
"tf.Tensor([ 7 3 5 9 1 5 10], shape=(7,), dtype=int64)\n"
]
],
[
[
"벡터화된 텐서로부터 문장을 복원하면 표준화된 문장이 생성된다.",
"_____no_output_____"
]
],
[
[
"inverse_vocab = dict(enumerate(vocabulary))\n\ndecoded_sentence = \" \".join(inverse_vocab[int(i)] for i in encoded_sentence)\nprint(decoded_sentence)",
"i write rewrite and [UNK] rewrite again\n"
]
],
[
[
"**`TextVectorization` 층 사용법**",
"_____no_output_____"
],
[
"`TextVectorization` 층은 GPU 또는 TPU에서 지원되지 않는다.\n따라서 모델 구성에 직접 사용하는 방식은 모델의 훈련을\n늦출 수 있기에 권장되지 않는다.\n여기서는 대신에 데이터셋 전처리를 모델 구성과 독립적으로 처리하는 방식을 이용한다.\n\n하지만 훈련이 완성된 모델을 실전에 배치할 경우 `TextVectorization` 층을\n완성된 모델에 추가해서 사용하는 게 좋다.\n이에 대한 자세한 설명은 잠시 뒤에 부록에서 설명한다.",
"_____no_output_____"
],
[
"## 11.3 단어 모음 표현법: 집합과 시퀀스",
"_____no_output_____"
],
[
"앞서 언급한 대로 자연어처리 모델에 따라 단어 모음을 다루는 방식이 다르다. \n\n- 단어주머니(bag-of-words) 모델\n - 단어들의 순서를 무시. 단어 모음을 단어들의 집합으로 다룸.\n - 2015년 이전까지 주로 사용됨.\n- 시퀀스(sequence) 모델\n - 순환(recurrent) 모델\n - 단어들의 순서를 시계열 데이터의 스텝처럼 간주.\n - 2015-2016에 주로 사용됨.\n - 트랜스포머(Transformer) 아키텍처\n - 기본적으로 순서를 무시하지만 단어 위치를 학습할 수 있는 능력을 가짐.\n - 2017년 이후 기본적으로 활용됨.",
"_____no_output_____"
],
[
"여기서는 IMDB 영화 리뷰 데이터를 이용하여 두 모델 방식의 \n활용법과 차이점을 소개한다.",
"_____no_output_____"
],
[
"### 11.3.1 IMDB 영화 리뷰 데이터 준비",
"_____no_output_____"
],
[
"이전과는 달리 여기서는 IMDB 데이터셋을 직접 다운로드하여 전처리하는 \n과정을 살펴본다. ",
"_____no_output_____"
],
[
"준비 과정 1: 데이터셋 다운로드 압축 풀기\n\n압축을 풀면 아래 구조의 디렉토리가 생성된다.\n\n```\naclImdb/\n...train/\n......pos/\n......neg/\n...test/\n......pos/\n......neg/\n```\n\n`train`의 `pos`와 `neg` 서브디렉토리에 각각 12,500개의 긍정과 부정 리뷰가\n포함되어 있다. \n\n*주의사항*: 아래 코드는 윈도우의 경우 10 최신 버전 또는 11부터 지원된다.",
"_____no_output_____"
]
],
[
[
"!curl -O https://ai.stanford.edu/~amaas/data/sentiment/aclImdb_v1.tar.gz\n!tar -xf aclImdb_v1.tar.gz",
" % Total % Received % Xferd Average Speed Time Time Time Current\n Dload Upload Total Spent Left Speed\n100 80.2M 100 80.2M 0 0 26.1M 0 0:00:03 0:00:03 --:--:-- 26.1M\n"
]
],
[
[
"`aclImdb/train/unsup` 서브디렉토리는 필요 없기에 삭제한다.",
"_____no_output_____"
]
],
[
[
"if 'google.colab' in str(get_ipython()):\n !rm -r aclImdb/train/unsup\nelse: \n import shutil\n unsup_path = './aclImdb/train/unsup'\n shutil.rmtree(unsup_path)",
"_____no_output_____"
]
],
[
[
"긍정 리뷰 하나의 내용을 살펴보자.\n모델 구성 이전에 훈련 데이터셋을 살펴 보고\n모델에 대한 직관을 갖는 과정이 항상 필요하다.",
"_____no_output_____"
]
],
[
[
"if 'google.colab' in str(get_ipython()):\n !cat aclImdb/train/pos/4077_10.txt\nelse:\n with open('aclImdb/train/pos/4077_10.txt', 'r') as f:\n text = f.read()\n print(text)",
"I first saw this back in the early 90s on UK TV, i did like it then but i missed the chance to tape it, many years passed but the film always stuck with me and i lost hope of seeing it TV again, the main thing that stuck with me was the end, the hole castle part really touched me, its easy to watch, has a great story, great music, the list goes on and on, its OK me saying how good it is but everyone will take there own best bits away with them once they have seen it, yes the animation is top notch and beautiful to watch, it does show its age in a very few parts but that has now become part of it beauty, i am so glad it has came out on DVD as it is one of my top 10 films of all time. Buy it or rent it just see it, best viewing is at night alone with drink and food in reach so you don't have to stop the film.<br /><br />Enjoy"
]
],
[
[
"준비 과정 2: 검증셋 준비\n\n훈련셋의 20%를 검증셋으로 떼어낸다.\n이를 위해 `aclImdb/val` 디렉토리를 생성한 후에\n긍정과 부정 훈련셋 모두 무작위로 섞은 후 그중 20%를 검증셋 디렉토리로 옮긴다.",
"_____no_output_____"
]
],
[
[
"import os, pathlib, shutil, random\n\nbase_dir = pathlib.Path(\"aclImdb\")\nval_dir = base_dir / \"val\"\ntrain_dir = base_dir / \"train\"\n\nfor category in (\"neg\", \"pos\"):\n os.makedirs(val_dir / category) # val 디렉토리 생성\n files = os.listdir(train_dir / category)\n \n random.Random(1337).shuffle(files) # 훈련셋 무작위 섞기\n \n num_val_samples = int(0.2 * len(files)) # 20% 지정 후 검증셋으로 옮기기\n val_files = files[-num_val_samples:]\n \n for fname in val_files:\n shutil.move(train_dir / category / fname,\n val_dir / category / fname)",
"_____no_output_____"
]
],
[
[
"준비 과정 3: 텐서 데이터셋 준비\n\n`text_dataset_from_directory()` 함수를 이용하여 \n훈련셋, 검증셋, 테스트셋을 준비한다. \n자료형은 모두 `Dataset`이며, 배치 크기는 32를 사용한다.",
"_____no_output_____"
]
],
[
[
"from tensorflow import keras\n\nbatch_size = 32\n\ntrain_ds = keras.utils.text_dataset_from_directory(\n \"aclImdb/train\", batch_size=batch_size\n )\n\nval_ds = keras.utils.text_dataset_from_directory(\n \"aclImdb/val\", batch_size=batch_size\n )\n\ntest_ds = keras.utils.text_dataset_from_directory(\n \"aclImdb/test\", batch_size=batch_size\n )",
"Found 20000 files belonging to 2 classes.\nFound 5000 files belonging to 2 classes.\nFound 25000 files belonging to 2 classes.\n"
]
],
[
[
"각 데이터셋은 배치로 구분되며\n입력은 `tf.string` 텐서이고, 타깃은 `int32` 텐서이다.\n크기는 모두 32이며 지정된 배치 크기이다.\n예를 들어, 첫째 배치의 입력과 타깃 데이터의 정보는 다음과 같다.",
"_____no_output_____"
]
],
[
[
"for inputs, targets in train_ds:\n print(\"inputs.shape:\", inputs.shape)\n print(\"inputs.dtype:\", inputs.dtype)\n print(\"targets.shape:\", targets.shape)\n print(\"targets.dtype:\", targets.dtype)\n \n # 예제: 첫째 배치의 첫째 리뷰\n print(\"inputs[0]:\", inputs[0])\n print(\"targets[0]:\", targets[0])\n \n break",
"inputs.shape: (32,)\ninputs.dtype: <dtype: 'string'>\ntargets.shape: (32,)\ntargets.dtype: <dtype: 'int32'>\ninputs[0]: tf.Tensor(b'The film begins with a bunch of kids in reform school and focuses on a kid named \\'Gabe\\', who has apparently worked hard to earn his parole. Gabe and his sister move to a new neighborhood to make a fresh start and soon Gabe meets up with the Dead End Kids. The Kids in this film are little punks, but they are much less antisocial than they\\'d been in other previous films and down deep, they are well-meaning punks. However, in this neighborhood there are also some criminals who are perpetrating insurance fraud through arson and see Gabe as a convenient scapegoat--after all, he\\'d been to reform school and no one would believe he was innocent once he was framed. So, when Gabe is about ready to be sent back to \"The Big House\", it\\'s up to the rest of the gang to save him and expose the real crooks.<br /><br />The \"Dead End Kids\" appeared in several Warner Brothers films in the late 1930s and the films were generally very good (particularly ANGELS WITH DIRTY FACES). However, after the boys\\' contracts expired, they went on to Monogram Studios and the films, to put it charitably, were very weak and formulaic--with Huntz Hall and Leo Gorcey being pretty much the whole show and the group being renamed \"The Bowery Boys\". Because ANGELS WASH THEIR FACES had the excellent writing and production values AND Hall and Gorcey were not constantly mugging for the camera, it\\'s a pretty good film--and almost earns a score of 7 (it\\'s REAL close). In fact, while this isn\\'t a great film aesthetically, it\\'s sure a lot of fun to watch, so I will give it a 7! Sure, it was a tad hokey-particularly towards the end when the kids take the law into their own hands and Reagan ignores the Bill of Rights--but it was also quite entertaining. The Dead End Kids are doing their best performances and Ronald Reagan and Ann Sheridan provided excellent support. Sure, this part of the film was illogical and impossible but somehow it was still funny and rather charming--so if you can suspend disbelief, it works well.', shape=(), dtype=string)\ntargets[0]: tf.Tensor(1, shape=(), dtype=int32)\n"
]
],
[
[
"### 11.3.2 단어주머니 기법",
"_____no_output_____"
],
[
"단어주머니에 채울 토큰으로 어떤 N-그램을 사용할지 먼저 지정해야 한다. \n\n- 유니그램(unigrams): 하나의 단어가 한의 토큰\n- N-그램(N-grams): 최대 N 개의 연속 단어로 이루어진 토큰",
"_____no_output_____"
],
[
"**방식 1: 유니그램 바이너리 인코딩**",
"_____no_output_____"
],
[
"예를 들어 \"the cat sat on the mat\" 문장을 유니그램으로 처리하면 다음 \n단어주머니가 생성된다.\n집합으로 처리되기에 단어들의 순서는 완전히 무시된다.\n\n```\n{\"cat\", \"mat\", \"on\", \"sat\", \"the\"}\n```\n\n이제 모든 문장은 어휘색인에 포함된 단어들의 수만큼 긴 1차원 이진 텐서(binary tensor)로\n처리된다. \n즉, 멀티-핫(multi-hot) 인코딩 방식을 사용해서 텐서로 변환된다.\n[4장](https://codingalzi.github.io/dlp/notebooks/dlp04_getting_started_with_neural_networks.html)과 \n[5장](https://codingalzi.github.io/dlp/notebooks/dlp05_fundamentals_of_ml.html)에서 \n문장을 인코딩 방식과 동일하다. \n\n`TextVectorization` 클래스의 `output_mode=\"multi_hot\"` 옵션을 이용하면\n방금 설명한 내용을 그대로 처리해준다.",
"_____no_output_____"
]
],
[
[
"from tensorflow.keras.layers import TextVectorization\n\ntext_vectorization = TextVectorization(\n max_tokens=20000,\n output_mode=\"multi_hot\",\n)\n\n# 어휘색인 생성\ntext_only_train_ds = train_ds.map(lambda x, y: x)\ntext_vectorization.adapt(text_only_train_ds)",
"_____no_output_____"
]
],
[
[
"생성된 어휘색인을 이용하여 훈련셋, 검증셋, 테스트셋 모두 벡터화한다. ",
"_____no_output_____"
]
],
[
[
"binary_1gram_train_ds = train_ds.map(lambda x, y: (text_vectorization(x), y))\nbinary_1gram_val_ds = val_ds.map(lambda x, y: (text_vectorization(x), y))\nbinary_1gram_test_ds = test_ds.map(lambda x, y: (text_vectorization(x), y))",
"_____no_output_____"
]
],
[
[
"변환된 첫째 배치의 입력과 타깃 데이터의 정보는 다음과 같다.\n`max_tokens=20000`으로 지정하였기에 모든 문장은 길이가 2만인 벡터로 변환되었다.",
"_____no_output_____"
]
],
[
[
"for inputs, targets in binary_1gram_train_ds:\n print(\"inputs.shape:\", inputs.shape)\n print(\"inputs.dtype:\", inputs.dtype)\n print(\"targets.shape:\", targets.shape)\n print(\"targets.dtype:\", targets.dtype)\n print(\"inputs[0]:\", inputs[0])\n print(\"targets[0]:\", targets[0])\n break",
"inputs.shape: (32, 20000)\ninputs.dtype: <dtype: 'float32'>\ntargets.shape: (32,)\ntargets.dtype: <dtype: 'int32'>\ninputs[0]: tf.Tensor([1. 1. 1. ... 0. 0. 0.], shape=(20000,), dtype=float32)\ntargets[0]: tf.Tensor(0, shape=(), dtype=int32)\n"
]
],
[
[
"**밀집 모델 지정**",
"_____no_output_____"
],
[
"단어주머니 모델로 여기서는 밀집 모델을 사용한다. \n`get_model()` 함수가 컴파일 된 단순한 밀집 모델을 반환한다.\n모델의 출력값은 긍정일 확률이며, \n최상위 층의 활성화 함수로 `sigmoid`를 사용한다.",
"_____no_output_____"
]
],
[
[
"from tensorflow import keras\nfrom tensorflow.keras import layers\n\ndef get_model(max_tokens=20000, hidden_dim=16):\n inputs = keras.Input(shape=(max_tokens,))\n x = layers.Dense(hidden_dim, activation=\"relu\")(inputs)\n x = layers.Dropout(0.5)(x)\n outputs = layers.Dense(1, activation=\"sigmoid\")(x) # 긍정일 확률 계산\n \n model = keras.Model(inputs, outputs)\n \n model.compile(optimizer=\"rmsprop\",\n loss=\"binary_crossentropy\",\n metrics=[\"accuracy\"])\n \n return model",
"_____no_output_____"
],
[
"model = get_model()\nmodel.summary()",
"Model: \"model\"\n_________________________________________________________________\n Layer (type) Output Shape Param # \n=================================================================\n input_1 (InputLayer) [(None, 20000)] 0 \n \n dense (Dense) (None, 16) 320016 \n \n dropout (Dropout) (None, 16) 0 \n \n dense_1 (Dense) (None, 1) 17 \n \n=================================================================\nTotal params: 320,033\nTrainable params: 320,033\nNon-trainable params: 0\n_________________________________________________________________\n"
]
],
[
[
"**모델 훈련**",
"_____no_output_____"
],
[
"밀집 모델 훈련과정은 특별한 게 없다.\n훈련 후 테스트셋에 대한 정확도가 89% 보다 조금 낮게 나온다.\n최고 성능의 모델이 테스트셋에 대해 95% 정도 정확도를 내는 것보다는 낮지만\n무작위로 찍는 모델보다는 훨씬 좋은 모델이다.",
"_____no_output_____"
]
],
[
[
"callbacks = [\n keras.callbacks.ModelCheckpoint(\"binary_1gram.keras\",\n save_best_only=True)\n]\n\nmodel.fit(binary_1gram_train_ds.cache(),\n validation_data=binary_1gram_val_ds.cache(),\n epochs=10,\n callbacks=callbacks)\n\nmodel = keras.models.load_model(\"binary_1gram.keras\")\nprint(f\"Test acc: {model.evaluate(binary_1gram_test_ds)[1]:.3f}\")",
"Epoch 1/10\n625/625 [==============================] - 10s 16ms/step - loss: 0.4074 - accuracy: 0.8277 - val_loss: 0.2792 - val_accuracy: 0.8908\nEpoch 2/10\n625/625 [==============================] - 3s 5ms/step - loss: 0.2746 - accuracy: 0.8981 - val_loss: 0.2774 - val_accuracy: 0.8964\nEpoch 3/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2471 - accuracy: 0.9115 - val_loss: 0.2872 - val_accuracy: 0.8976\nEpoch 4/10\n625/625 [==============================] - 3s 5ms/step - loss: 0.2246 - accuracy: 0.9244 - val_loss: 0.3187 - val_accuracy: 0.8936\nEpoch 5/10\n625/625 [==============================] - 3s 6ms/step - loss: 0.2156 - accuracy: 0.9313 - val_loss: 0.3164 - val_accuracy: 0.8960\nEpoch 6/10\n625/625 [==============================] - 3s 6ms/step - loss: 0.2108 - accuracy: 0.9341 - val_loss: 0.3355 - val_accuracy: 0.8934\nEpoch 7/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2052 - accuracy: 0.9366 - val_loss: 0.3354 - val_accuracy: 0.8944\nEpoch 8/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2017 - accuracy: 0.9365 - val_loss: 0.3582 - val_accuracy: 0.8940\nEpoch 9/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2013 - accuracy: 0.9394 - val_loss: 0.3497 - val_accuracy: 0.8938\nEpoch 10/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2043 - accuracy: 0.9394 - val_loss: 0.3631 - val_accuracy: 0.8940\n782/782 [==============================] - 8s 10ms/step - loss: 0.2861 - accuracy: 0.8885\nTest acc: 0.888\n"
]
],
[
[
"**방식 2: 바이그램 바이너리 인코딩**",
"_____no_output_____"
],
[
"바이그램(2-grams)을 유니그램 대신 이용해보자. \n예를 들어 \"the cat sat on the mat\" 문장을 바이그램으로 처리하면 다음 \n단어주머니가 생성된다.\n\n```\n{\"the\", \"the cat\", \"cat\", \"cat sat\", \"sat\",\n \"sat on\", \"on\", \"on the\", \"the mat\", \"mat\"}\n```\n\n`TextVectorization` 클래스의 `ngrams=N` 옵션을 이용하면\nN-그램들로 이루어진 어휘색인을 생성할 수 있다.",
"_____no_output_____"
]
],
[
[
"text_vectorization = TextVectorization(\n ngrams=2,\n max_tokens=20000,\n output_mode=\"multi_hot\",\n)",
"_____no_output_____"
]
],
[
[
"어휘색인 생성과 훈련셋, 검증셋, 테스트셋의 벡터화 과정은 동일하다. ",
"_____no_output_____"
]
],
[
[
"text_vectorization.adapt(text_only_train_ds)\n\nbinary_2gram_train_ds = train_ds.map(lambda x, y: (text_vectorization(x), y))\nbinary_2gram_val_ds = val_ds.map(lambda x, y: (text_vectorization(x), y))\nbinary_2gram_test_ds = test_ds.map(lambda x, y: (text_vectorization(x), y))",
"_____no_output_____"
]
],
[
[
"훈련 후 테스트셋에 대한 정확도가 90%를 조금 웃돌 정도로 많이 향상되었다.",
"_____no_output_____"
]
],
[
[
"model = get_model()\n\ncallbacks = [\n keras.callbacks.ModelCheckpoint(\"binary_2gram.keras\",\n save_best_only=True)\n]\n\nmodel.fit(binary_2gram_train_ds.cache(),\n validation_data=binary_2gram_val_ds.cache(),\n epochs=10,\n callbacks=callbacks)\n\nmodel = keras.models.load_model(\"binary_2gram.keras\")\nprint(f\"Test acc: {model.evaluate(binary_2gram_test_ds)[1]:.3f}\")",
"Epoch 1/10\n625/625 [==============================] - 12s 18ms/step - loss: 0.3857 - accuracy: 0.8347 - val_loss: 0.2791 - val_accuracy: 0.9000\nEpoch 2/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2592 - accuracy: 0.9082 - val_loss: 0.2947 - val_accuracy: 0.8988\nEpoch 3/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2277 - accuracy: 0.9241 - val_loss: 0.3060 - val_accuracy: 0.8978\nEpoch 4/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2074 - accuracy: 0.9333 - val_loss: 0.3417 - val_accuracy: 0.8994\nEpoch 5/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2070 - accuracy: 0.9365 - val_loss: 0.3538 - val_accuracy: 0.8968\nEpoch 6/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.1997 - accuracy: 0.9395 - val_loss: 0.3908 - val_accuracy: 0.8946\nEpoch 7/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.1940 - accuracy: 0.9421 - val_loss: 0.3715 - val_accuracy: 0.8940\nEpoch 8/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.1902 - accuracy: 0.9427 - val_loss: 0.4054 - val_accuracy: 0.8930\nEpoch 9/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.1952 - accuracy: 0.9432 - val_loss: 0.3848 - val_accuracy: 0.8880\nEpoch 10/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.1949 - accuracy: 0.9441 - val_loss: 0.4011 - val_accuracy: 0.8912\n782/782 [==============================] - 9s 11ms/step - loss: 0.2788 - accuracy: 0.8953\nTest acc: 0.895\n"
]
],
[
[
"**방식 3: 바이그램 TF-IDF 인코딩**",
"_____no_output_____"
],
[
"N-그램을 벡터화할 때 사용 빈도를 함께 저장하는 방식을 사용할 수 있다.\n단어의 사용 빈도가 아무래도 문장 평가에 중요한 역할을 수행할 것이기 때문이다.\n아래 코드에서처럼 `output_mode=\"count\"` 옵션을 사용하면 된다.",
"_____no_output_____"
]
],
[
[
"text_vectorization = TextVectorization(\n ngrams=2,\n max_tokens=20000,\n output_mode=\"count\"\n)",
"_____no_output_____"
]
],
[
[
"그런데 이렇게 하면 \"the\", \"a\", \"is\", \"are\" 등의 사용 빈도는 매우 높은 반면에\n\"Chollet\" 등의 단어는 빈도가 거의 0에 가깝게 나온다.\n또한 생성된 벡터의 대부분은 0으로 채워질 것이다. \n`max_tokens=20000`을 사용한 반면에 하나의 문장엔 많아야 몇 십개 정도의 단어만 사용되었기 때문이다. \n\n```python\ninputs[0]: tf.Tensor([1. 1. 1. ... 0. 0. 0.], shape=(20000,), dtype=float32)\n```",
"_____no_output_____"
],
[
"이 점을 고려해서 사용 빈도를 정규화한다. \n평균을 원점으로 만들지는 않고 TF-IDF 값으로 나누기만 실행한다.\n이유는 평균을 옮기면 벡터의 대부분의 값이 0이 아니게 되어\n훈련에 보다 많은 계산이 요구되기 때문이다. \n\n**TF-IDF**의 의미는 다음과 같다.\n\n- `TF`(Term Frequency)\n - 하나의 문장에서 사용되는 단어의 빈도\n - 높을 수록 중요\n - 예를 들어, 하나의 리뷰에 \"terrible\" 이 많이 사용되었다면\n 해당 리뷰는 부정일 가능성 높음.\n- `IDF`(Inverse Document Frequency)\n - 데이터셋 전체 문장에서 사용된 단어의 빈도\n - 낮을 수록 중요. \n - \"the\", \"a\", \"is\" 등의 `IDF` 값은 높지만 별로 중요하지 않음.\n- `TF-IDF = TF / IDF`",
"_____no_output_____"
],
[
"`output_mode=\"tf_idf\"` 옵션을 사용하면 TF-IDF 인코딩을 지원한다.",
"_____no_output_____"
]
],
[
[
"text_vectorization = TextVectorization(\n ngrams=2,\n max_tokens=20000,\n output_mode=\"tf_idf\",\n)",
"_____no_output_____"
]
],
[
[
"훈련 후 테스트셋에 대한 정확도가 다시 89% 아래로 내려간다.\n여기서는 별 도움이 되지 않았지만 많은 텍스트 분류 모델에서는 1% 정도의 성능 향상을 가져온다.\n\n**주의사항**: 아래 코드는 현재(Tensorflow 2.6과 2.7) GPU를 사용하지 않는 경우에만 작동한다. \n이유는 아직 모른다([여기 참조](https://github.com/fchollet/deep-learning-with-python-notebooks/issues/190)).",
"_____no_output_____"
]
],
[
[
"text_vectorization.adapt(text_only_train_ds)\n\ntfidf_2gram_train_ds = train_ds.map(lambda x, y: (text_vectorization(x), y))\ntfidf_2gram_val_ds = val_ds.map(lambda x, y: (text_vectorization(x), y))\ntfidf_2gram_test_ds = test_ds.map(lambda x, y: (text_vectorization(x), y))\n\nmodel = get_model()\nmodel.summary()\ncallbacks = [\n keras.callbacks.ModelCheckpoint(\"tfidf_2gram.keras\",\n save_best_only=True)\n]\nmodel.fit(tfidf_2gram_train_ds.cache(),\n validation_data=tfidf_2gram_val_ds.cache(),\n epochs=10,\n callbacks=callbacks)\n\nmodel = keras.models.load_model(\"tfidf_2gram.keras\")\nprint(f\"Test acc: {model.evaluate(tfidf_2gram_test_ds)[1]:.3f}\")",
"Model: \"model_2\"\n_________________________________________________________________\n Layer (type) Output Shape Param # \n=================================================================\n input_3 (InputLayer) [(None, 20000)] 0 \n \n dense_4 (Dense) (None, 16) 320016 \n \n dropout_2 (Dropout) (None, 16) 0 \n \n dense_5 (Dense) (None, 1) 17 \n \n=================================================================\nTotal params: 320,033\nTrainable params: 320,033\nNon-trainable params: 0\n_________________________________________________________________\nEpoch 1/10\n625/625 [==============================] - 11s 17ms/step - loss: 0.5232 - accuracy: 0.7588 - val_loss: 0.3197 - val_accuracy: 0.8806\nEpoch 2/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.3534 - accuracy: 0.8442 - val_loss: 0.2946 - val_accuracy: 0.8954\nEpoch 3/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.3231 - accuracy: 0.8609 - val_loss: 0.3086 - val_accuracy: 0.8864\nEpoch 4/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.3053 - accuracy: 0.8734 - val_loss: 0.3087 - val_accuracy: 0.8814\nEpoch 5/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2781 - accuracy: 0.8845 - val_loss: 0.3225 - val_accuracy: 0.8878\nEpoch 6/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2703 - accuracy: 0.8870 - val_loss: 0.3472 - val_accuracy: 0.8702\nEpoch 7/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2695 - accuracy: 0.8883 - val_loss: 0.3357 - val_accuracy: 0.8682\nEpoch 8/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2650 - accuracy: 0.8931 - val_loss: 0.3343 - val_accuracy: 0.8664\nEpoch 9/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2606 - accuracy: 0.8901 - val_loss: 0.3546 - val_accuracy: 0.8580\nEpoch 10/10\n625/625 [==============================] - 4s 6ms/step - loss: 0.2575 - accuracy: 0.8924 - val_loss: 0.3318 - val_accuracy: 0.8760\n782/782 [==============================] - 8s 10ms/step - loss: 0.2998 - accuracy: 0.8927\nTest acc: 0.893\n"
]
],
[
[
"**부록: 문자열 벡터화 전처리를 함께 처리하는 모델 내보내기**",
"_____no_output_____"
],
[
"훈련된 모델을 실전에 배치하려면 텍스트 벡터화도 모델과 함께 내보내야 한다.\n이를 위해 `TextVectorization` 층의 결과를 재활용만 하면 된다.",
"_____no_output_____"
]
],
[
[
"inputs = keras.Input(shape=(1,), dtype=\"string\")\n# 텍스트 벡터화 추가\nprocessed_inputs = text_vectorization(inputs)\n# 훈련된 모델에 적용\noutputs = model(processed_inputs)\n\n# 최종 모델\ninference_model = keras.Model(inputs, outputs)",
"_____no_output_____"
]
],
[
[
"`inference_model`은 일반 텍스트 문장을 직접 인자로 받을 수 있다.\n예를 들어 \"That was an excellent movie, I loved it.\"라는 리뷰는\n긍정일 확률이 매우 높다고 예측된다.",
"_____no_output_____"
]
],
[
[
"import tensorflow as tf\n\nraw_text_data = tf.convert_to_tensor([\n [\"That was an excellent movie, I loved it.\"],\n])\n\npredictions = inference_model(raw_text_data)\nprint(f\"{float(predictions[0] * 100):.2f} percent positive\")",
"92.10 percent positive\n"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7e84c292e11280f603f9d3b5381831e4e9e5938 | 917,442 | ipynb | Jupyter Notebook | docs/notebooks/visualization/loading.ipynb | neurodata/brainl | 2de7b5b161000d4d0957de4e836c9e72f7b62ec0 | [
"Apache-2.0"
] | null | null | null | docs/notebooks/visualization/loading.ipynb | neurodata/brainl | 2de7b5b161000d4d0957de4e836c9e72f7b62ec0 | [
"Apache-2.0"
] | 6 | 2020-01-31T22:21:10.000Z | 2020-01-31T22:24:59.000Z | docs/notebooks/visualization/loading.ipynb | neurodata/brainl | 2de7b5b161000d4d0957de4e836c9e72f7b62ec0 | [
"Apache-2.0"
] | null | null | null | 1,196.143416 | 782,304 | 0.951006 | [
[
[
"# Loading neurons from s3",
"_____no_output_____"
]
],
[
[
"import numpy as np\nfrom skimage import io\nfrom pathlib import Path\nfrom brainlit.utils.session import NeuroglancerSession\nfrom brainlit.utils.Neuron_trace import NeuronTrace\nimport napari\nfrom napari.utils import nbscreenshot\n%gui qt",
"_____no_output_____"
]
],
[
[
"## Loading entire neuron from AWS \n",
"_____no_output_____"
],
[
"`s3_trace = NeuronTrace(s3_path,seg_id,mip)` to create a NeuronTrace object with s3 file path\n`swc_trace = NeuronTrace(swc_path)` to create a NeuronTrace object with swc file path\n1. `s3_trace.get_df()` to output the s3 NeuronTrace object as a pd.DataFrame\n2. `swc_trace.get_df()` to output the swc NeuronTrace object as a pd.DataFrame\n3. `swc_trace.generate_df_subset(list_of_voxels)` creates a smaller subset of the original dataframe with coordinates in img space\n4. `swc_trace.get_df_voxel()` to output a DataFrame that converts the coordinates from spatial to voxel coordinates\n5. `swc_trace.get_graph()` to output the s3 NeuronTrace object as a netwrokx.DiGraph\n6. `swc_trace.get_paths()` to output the s3 NeuronTrace object as a list of paths\n7. `ViewerModel.add_shapes` to add the paths as a shape layer into the napari viewer\n8. `swc_trace.get_sub_neuron(bounding_box)` to output NeuronTrace object as a graph cropped by a bounding box\n9. `swc_trace.get_sub_neuron(bounding_box)` to output NeuronTrace object as paths cropped by a bounding box",
"_____no_output_____"
],
[
"### 1. `s3_trace.get_df()`\nThis function outputs the s3 NeuronTrace object as a pd.DataFrame. Each row is a vertex in the swc file with the following information: \n\n`sample number`\n\n`structure identifier`\n\n`x coordinate`\n\n`y coordinate`\n\n`z coordinate`\n\n`radius of dendrite`\n\n`sample number of parent`\n\nThe coordinates are given in spatial units of micrometers ([swc specification](http://www.neuronland.org/NLMorphologyConverter/MorphologyFormats/SWC/Spec.html))",
"_____no_output_____"
]
],
[
[
"\"\"\"\ns3_path = \"s3://open-neurodata/brainlit/brain1_segments\"\nseg_id = 2\nmip = 1\n\ns3_trace = NeuronTrace(s3_path, seg_id, mip)\ndf = s3_trace.get_df()\ndf.head()\n\"\"\"",
"Downloading: 100%|██████████| 1/1 [00:00<00:00, 5.13it/s]\nDownloading: 100%|██████████| 1/1 [00:00<00:00, 5.82it/s]\n"
]
],
[
[
"### 2. `swc_trace.get_df()`\nThis function outputs the swc NeuronTrace object as a pd.DataFrame. Each row is a vertex in the swc file with the following information: \n\n`sample number`\n\n`structure identifier`\n\n`x coordinate`\n\n`y coordinate`\n\n`z coordinate`\n\n`radius of dendrite`\n\n`sample number of parent`\n\nThe coordinates are given in spatial units of micrometers ([swc specification](http://www.neuronland.org/NLMorphologyConverter/MorphologyFormats/SWC/Spec.html))",
"_____no_output_____"
]
],
[
[
"\"\"\"\nswc_path = str(Path().resolve().parents[2] / \"data\" / \"data_octree\" / \"consensus-swcs\" / '2018-08-01_G-002_consensus.swc')\n\nswc_trace = NeuronTrace(path=swc_path)\ndf = swc_trace.get_df()\ndf.head()\n\"\"\"",
"_____no_output_____"
]
],
[
[
"### 3. `swc_trace.generate_df_subset(list_of_voxels)`\nThis function creates a smaller subset of the original dataframe with coordinates in img space. Each row is a vertex in the swc file with the following information: \n\n`sample number`\n\n`structure identifier`\n\n`x coordinate`\n\n`y coordinate`\n\n`z coordinate`\n\n`radius of dendrite`\n\n`sample number of parent`\n\nThe coordinates are given in same spatial units as the image file when using `ngl.pull_vertex_list`",
"_____no_output_____"
]
],
[
[
"\"\"\"# Choose vertices to use for the subneuron\nsubneuron_df = df[0:3] \nvertex_list = subneuron_df['sample'].array \n\n# Define a neuroglancer session\nurl = \"s3://open-neurodata/brainlit/brain1\"\nmip = 1\nngl = NeuroglancerSession(url, mip=mip)\n\n# Get vertices\nseg_id = 2\nbuffer = 10\nimg, bounds, vox_in_img_list = ngl.pull_vertex_list(seg_id=seg_id, v_id_list=vertex_list, buffer = buffer, expand = True)\n\ndf_subneuron = swc_trace.generate_df_subset(vox_in_img_list.tolist(),subneuron_start=0,subneuron_end=3 )\nprint(df_subneuron)\n\"\"\"",
"Downloading: 100%|██████████| 1/1 [00:00<00:00, 6.08it/s]\nDownloading: 100%|██████████| 1/1 [00:00<00:00, 6.95it/s]\nDownloading: 100%|██████████| 1/1 [00:00<00:00, 5.02it/s]\nDownloading: 0%| | 0/4 [00:01<?, ?it/s] sample structure x y z r parent\n0 1 0 106 106 112 1.0 -1\n1 2 0 121 80 61 1.0 1\n2 3 0 61 55 49 1.0 2\n\n"
]
],
[
[
"### 4. `swc_trace.get_df_voxel()` \n\nIf we want to overlay the swc file with a corresponding image, we need to make sure that they are in the same coordinate space. Because an image in an array of voxels, it makes sense to convert the vertices from spatial units into voxel units.\n\nGiven the `spacing` (spatial units/voxel) and `origin` (spatial units) of the image, `swc_to_voxel` does the conversion by using the following equation:\n\n$voxel = \\frac{spatial - origin}{spacing}$",
"_____no_output_____"
]
],
[
[
"# spacing = np.array([0.29875923,0.3044159,0.98840415])\n# origin = np.array([70093.276,15071.596,29306.737])\n\n\n# df_voxel = swc_trace.get_df_voxel(spacing=spacing, origin=origin)\n# df_voxel.head()",
"_____no_output_____"
]
],
[
[
"### 5. `swc_trace.get_graph()`\nA neuron is a graph with no cycles (tree). While napari does not support displaying graph objects, it can display multiple paths. \n\nThe DataFrame already contains all the possible edges in the neurons. Each row in the DataFrame is an edge. For example, from the above we can see that `sample 2` has `parent 1`, which represents edge `(1,2)`. `sample 1` having `parent -1` means that `sample 1` is the root of the tree.\n\n `swc_trace.get_graph()` converts the NeuronTrace object into a networkx directional graph.",
"_____no_output_____"
]
],
[
[
"# G = swc_trace.get_graph()\n# print('Number of nodes:', len(G.nodes))\n# print('Number of edges:', len(G.edges))\n# print('\\n')\n# print('Sample 1 coordinates (x,y,z)')\n# print(G.nodes[1]['x'],G.nodes[1]['y'],G.nodes[1]['z'])",
"Number of nodes: 1650\nNumber of edges: 1649\n\n\nSample 1 coordinates (x,y,z)\n-387 1928 -1846\n"
]
],
[
[
"### 6. `swc_trace.get_paths()` \nThis function returns the NeuronTrace object as a list of non-overlapping paths. The union of the paths forms the graph.\n\nThe algorithm works by:\n\n1. Find longest path in the graph ([networkx.algorithms.dag.dag_longest_path](https://networkx.github.io/documentation/stable/reference/algorithms/generated/networkx.algorithms.dag.dag_longest_path.html))\n2. Remove longest path from graph\n3. Repeat steps 1 and 2 until there are no more edges left in the graph",
"_____no_output_____"
]
],
[
[
"# paths = swc_trace.get_paths()\n# print(f\"The graph was decomposed into {len(paths)} paths\")",
"The graph was decomposed into 179 paths\n"
]
],
[
[
"### 7. `ViewerModel.add_shapes`\nnapari displays \"layers\". The most common layer is the image layer. In order to display the neuron, we use `path` from the [shapes](https://napari.org/tutorials/shapes) layer",
"_____no_output_____"
]
],
[
[
"# viewer = napari.Viewer(ndisplay=3)\n# viewer.add_shapes(data=paths, shape_type='path', edge_color='white', name='Skeleton 2')\n# nbscreenshot(viewer)",
"_____no_output_____"
]
],
[
[
"## Loading sub-neuron\n\nThe image of the entire brain has dimensions of (33792, 25600, 13312) voxels. G-002 spans a sub-image of (7386, 9932, 5383) voxels. Both are too big to load in napari and overlay the neuron.\nTo circumvent this, we can crop out a smaller region of the neuron, load the sub-neuron, and load the corresponding sub-image.\n\nIn order to get a sub-neuron, we need to specify the `bounding_box` that will be used to crop the neuron. `bounding_box` is a length 2 tuple. The first element is one corner of the bounding box (inclusive) and the second element is the opposite corner of the bounding box (exclusive). Both corners are in voxel units.\n\n`add_swc` can do all of this automatically when given `bounding_box` by following these steps:\n\n1. `read_s3` to read the swc file into a pd.DataFrame\n2. `swc_to_voxel` to convert the coordinates from spatial to voxel coordinates\n3. `df_to_graph` to convert the DataFrame into a netwrokx.DiGraph\n**3.1 `swc.get_sub_neuron` to crop the graph by `bounding_box`**\n4. `graph_to_paths` to convert from a graph into a list of paths\n5. `ViewerModel.add_shapes` to add the paths as a shape layer into the napari viewer",
"_____no_output_____"
],
[
"### 8. `swc_trace.get_sub_neuron(bounding_box)` \n### 9. `swc_trace.get_sub_neuron_paths(bounding_box)` \n\nThis function crops a graph by removing edges. It removes edges that do not intersect the bounding box.\n\nEdges that intersect the bounding box will have at least one of its vertices be contained by the bounding box. The algorithm follows this principle by checking the neighborhood of vertices.\n\nFor each vertex *v* in the graph:\n\n1. Find vertices belonging to local neighborhood of *v*\n2. If vertex *v* or any of its local neighborhood vertices are in the bounding box, do nothing. Otherwise, remove vertex *v* and its edges from the graph\n\nWe check the neighborhood of *v* along with *v* because we want the sub-neuron to show all edges that pass through the bounding box, including edges that are only partially contained.\n\n`swc_trace.get_sub_neuron(bounding_box)` returns a sub neuron in graph format\n`swc_trace.get_sub_neuron_paths(bounding_box)` returns a sub neuron in paths format",
"_____no_output_____"
]
],
[
[
"# # Create an NGL session to get the bounding box\n# url = \"s3://open-neurodata/brainlit/brain1\"\n# mip = 1\n# ngl = NeuroglancerSession(url, mip=mip)\n\n# img, bbbox, vox = ngl.pull_chunk(2, 300, 1)\n# bbox = bbbox.to_list()\n# box = (bbox[:3], bbox[3:])\n# print(box)",
"Downloading: 100%|██████████| 1/1 [00:00<00:00, 6.28it/s]\nDownloading: 52it [00:02, 19.61it/s]([7392, 2300, 3120], [7788, 2600, 3276])\n\n"
],
[
"# G_sub = s3_trace.get_sub_neuron(box)\n# paths_sub = s3_trace.get_sub_neuron_paths(box)\n# print(len(G_sub))\n# viewer = napari.Viewer(ndisplay=3)\n# viewer.add_shapes(data=paths_sub, shape_type='path', edge_color='blue', name='sub-neuron')\n\n# # overlay corresponding image (random image but correct should be G-002_15312-4400-6448_15840-4800-6656.tif' )\n# image_path = str(Path().resolve().parents[2] / \"data\" / \"data_octree\" / 'default.0.tif') \n# img_comp = io.imread(image_path)\n# img_comp = np.swapaxes(img_comp,0,2)\n\n# viewer.add_image(img_comp)\n# nbscreenshot(viewer)",
"459\n"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
]
] |
e7e85abb843fad5d9d81e92c116321b7831ba857 | 377,894 | ipynb | Jupyter Notebook | DCGAN_Exercise.ipynb | ng572/DCGAN_SVHN | 2fe076b3b6de70ffadfab2a9e1418a270967e106 | [
"MIT"
] | null | null | null | DCGAN_Exercise.ipynb | ng572/DCGAN_SVHN | 2fe076b3b6de70ffadfab2a9e1418a270967e106 | [
"MIT"
] | null | null | null | DCGAN_Exercise.ipynb | ng572/DCGAN_SVHN | 2fe076b3b6de70ffadfab2a9e1418a270967e106 | [
"MIT"
] | null | null | null | 416.641676 | 230,716 | 0.92844 | [
[
[
"# Deep Convolutional GANs\n\nIn this notebook, you'll build a GAN using convolutional layers in the generator and discriminator. This is called a Deep Convolutional GAN, or DCGAN for short. The DCGAN architecture was first explored in 2016 and has seen impressive results in generating new images; you can read the [original paper, here](https://arxiv.org/pdf/1511.06434.pdf).\n\nYou'll be training DCGAN on the [Street View House Numbers](http://ufldl.stanford.edu/housenumbers/) (SVHN) dataset. These are color images of house numbers collected from Google street view. SVHN images are in color and much more variable than MNIST. \n\n<img src='assets/svhn_dcgan.png' width=80% />\n\nSo, our goal is to create a DCGAN that can generate new, realistic-looking images of house numbers. We'll go through the following steps to do this:\n* Load in and pre-process the house numbers dataset\n* Define discriminator and generator networks\n* Train these adversarial networks\n* Visualize the loss over time and some sample, generated images\n\n#### Deeper Convolutional Networks\n\nSince this dataset is more complex than our MNIST data, we'll need a deeper network to accurately identify patterns in these images and be able to generate new ones. Specifically, we'll use a series of convolutional or transpose convolutional layers in the discriminator and generator. It's also necessary to use batch normalization to get these convolutional networks to train. \n\nBesides these changes in network structure, training the discriminator and generator networks should be the same as before. That is, the discriminator will alternate training on real and fake (generated) images, and the generator will aim to trick the discriminator into thinking that its generated images are real!",
"_____no_output_____"
]
],
[
[
"# import libraries\nimport matplotlib.pyplot as plt\nimport numpy as np\nimport pickle as pkl\n\n%matplotlib inline",
"_____no_output_____"
]
],
[
[
"## Getting the data\n\nHere you can download the SVHN dataset. It's a dataset built-in to the PyTorch datasets library. We can load in training data, transform it into Tensor datatypes, then create dataloaders to batch our data into a desired size.",
"_____no_output_____"
]
],
[
[
"import torch\nfrom torchvision import datasets\nfrom torchvision import transforms\n\n# Tensor transform\ntransform = transforms.ToTensor()\n\n# SVHN training datasets\nsvhn_train = datasets.SVHN(root='data/', split='train', download=True, transform=transform)\n\nbatch_size = 128\nnum_workers = 0\n\n# build DataLoaders for SVHN dataset\ntrain_loader = torch.utils.data.DataLoader(dataset=svhn_train,\n batch_size=batch_size,\n shuffle=True,\n num_workers=num_workers)\n",
"Using downloaded and verified file: data/train_32x32.mat\n"
]
],
[
[
"### Visualize the Data\n\nHere I'm showing a small sample of the images. Each of these is 32x32 with 3 color channels (RGB). These are the real, training images that we'll pass to the discriminator. Notice that each image has _one_ associated, numerical label.",
"_____no_output_____"
]
],
[
[
"# obtain one batch of training images\ndataiter = iter(train_loader)\nimages, labels = dataiter.next()\n\n# plot the images in the batch, along with the corresponding labels\nfig = plt.figure(figsize=(25, 4))\nplot_size=20\nfor idx in np.arange(plot_size):\n ax = fig.add_subplot(2, plot_size/2, idx+1, xticks=[], yticks=[])\n ax.imshow(np.transpose(images[idx], (1, 2, 0)))\n # print out the correct label for each image\n # .item() gets the value contained in a Tensor\n ax.set_title(str(labels[idx].item()))",
"<ipython-input-3-a55faf2ffde6>:9: MatplotlibDeprecationWarning: Passing non-integers as three-element position specification is deprecated since 3.3 and will be removed two minor releases later.\n ax = fig.add_subplot(2, plot_size/2, idx+1, xticks=[], yticks=[])\n"
]
],
[
[
"### Pre-processing: scaling from -1 to 1\n\nWe need to do a bit of pre-processing; we know that the output of our `tanh` activated generator will contain pixel values in a range from -1 to 1, and so, we need to rescale our training images to a range of -1 to 1. (Right now, they are in a range from 0-1.)",
"_____no_output_____"
]
],
[
[
"# current range\nimg = images[0]\n\nprint('Min: ', img.min())\nprint('Max: ', img.max())",
"Min: tensor(0.2902)\nMax: tensor(0.6314)\n"
],
[
"# helper scale function\ndef scale(x, feature_range=(-1, 1)):\n ''' Scale takes in an image x and returns that image, scaled\n with a feature_range of pixel values from -1 to 1. \n This function assumes that the input x is already scaled from 0-1.'''\n # assume x is scaled to (0, 1)\n # scale to feature_range and return scaled x\n range_min, range_max = feature_range\n \n x = x * (range_max - range_min) + range_min\n \n return x\n",
"_____no_output_____"
],
[
"# scaled range\nscaled_img = scale(img)\n\nprint('Scaled min: ', scaled_img.min())\nprint('Scaled max: ', scaled_img.max())",
"Scaled min: tensor(-0.4196)\nScaled max: tensor(0.2627)\n"
]
],
[
[
"---\n# Define the Model\n\nA GAN is comprised of two adversarial networks, a discriminator and a generator.",
"_____no_output_____"
],
[
"## Discriminator\n\nHere you'll build the discriminator. This is a convolutional classifier like you've built before, only without any maxpooling layers. \n* The inputs to the discriminator are 32x32x3 tensor images\n* You'll want a few convolutional, hidden layers\n* Then a fully connected layer for the output; as before, we want a sigmoid output, but we'll add that in the loss function, [BCEWithLogitsLoss](https://pytorch.org/docs/stable/nn.html#bcewithlogitsloss), later\n\n<img src='assets/conv_discriminator.png' width=80%/>\n\nFor the depths of the convolutional layers I suggest starting with 32 filters in the first layer, then double that depth as you add layers (to 64, 128, etc.). Note that in the DCGAN paper, they did all the downsampling using only strided convolutional layers with no maxpooling layers.\n\nYou'll also want to use batch normalization with [nn.BatchNorm2d](https://pytorch.org/docs/stable/nn.html#batchnorm2d) on each layer **except** the first convolutional layer and final, linear output layer. \n\n#### Helper `conv` function \n\nIn general, each layer should look something like convolution > batch norm > leaky ReLU, and so we'll define a function to put these layers together. This function will create a sequential series of a convolutional + an optional batch norm layer. We'll create these using PyTorch's [Sequential container](https://pytorch.org/docs/stable/nn.html#sequential), which takes in a list of layers and creates layers according to the order that they are passed in to the Sequential constructor.\n\nNote: It is also suggested that you use a **kernel_size of 4** and a **stride of 2** for strided convolutions.",
"_____no_output_____"
]
],
[
[
"import torch.nn as nn\nimport torch.nn.functional as F\n\n# helper conv function\ndef conv(in_channels, out_channels, kernel_size, stride=2, padding=1, batch_norm=True):\n \"\"\"Creates a convolutional layer, with optional batch normalization.\n \"\"\"\n layers = []\n conv_layer = nn.Conv2d(in_channels, out_channels, \n kernel_size, stride, padding, bias=False)\n \n # append conv layer\n layers.append(conv_layer)\n\n if batch_norm:\n # append batchnorm layer\n layers.append(nn.BatchNorm2d(out_channels))\n \n # using Sequential container\n return nn.Sequential(*layers)\n",
"_____no_output_____"
],
[
"class Discriminator(nn.Module):\n\n def __init__(self, conv_dim=32):\n super(Discriminator, self).__init__()\n\n # complete init function\n self.conv1 = conv(in_channels=3, out_channels=conv_dim, kernel_size=4, stride=2, batch_norm=False)\n self.conv2 = conv(in_channels=conv_dim, out_channels=conv_dim*2, kernel_size=4, stride=2)\n self.conv3 = conv(in_channels=conv_dim*2, out_channels=conv_dim*4, kernel_size=4, stride=2)\n # 128*4*4\n self.fc = nn.Linear(in_features=128*4*4, out_features=1)\n\n def forward(self, x):\n # complete forward function\n x = self.conv1(x)\n x = F.leaky_relu(x, negative_slope=0.2)\n \n x = self.conv2(x)\n x = F.leaky_relu(x, negative_slope=0.2)\n \n x = self.conv3(x)\n x = F.leaky_relu(x, negative_slope=0.2)\n \n x = x.view(-1, 128*4*4)\n x = self.fc(x)\n return x\n ",
"_____no_output_____"
]
],
[
[
"## Generator\n\nNext, you'll build the generator network. The input will be our noise vector `z`, as before. And, the output will be a $tanh$ output, but this time with size 32x32 which is the size of our SVHN images.\n\n<img src='assets/conv_generator.png' width=80% />\n\nWhat's new here is we'll use transpose convolutional layers to create our new images. \n* The first layer is a fully connected layer which is reshaped into a deep and narrow layer, something like 4x4x512. \n* Then, we use batch normalization and a leaky ReLU activation. \n* Next is a series of [transpose convolutional layers](https://pytorch.org/docs/stable/nn.html#convtranspose2d), where you typically halve the depth and double the width and height of the previous layer. \n* And, we'll apply batch normalization and ReLU to all but the last of these hidden layers. Where we will just apply a `tanh` activation.\n\n#### Helper `deconv` function\n\nFor each of these layers, the general scheme is transpose convolution > batch norm > ReLU, and so we'll define a function to put these layers together. This function will create a sequential series of a transpose convolutional + an optional batch norm layer. We'll create these using PyTorch's Sequential container, which takes in a list of layers and creates layers according to the order that they are passed in to the Sequential constructor.\n\nNote: It is also suggested that you use a **kernel_size of 4** and a **stride of 2** for transpose convolutions.",
"_____no_output_____"
]
],
[
[
"# helper deconv function\ndef deconv(in_channels, out_channels, kernel_size, stride=2, padding=1, batch_norm=True):\n \"\"\"Creates a transposed-convolutional layer, with optional batch normalization.\n \"\"\"\n ## TODO: Complete this function\n ## create a sequence of transpose + optional batch norm layers\n layers = []\n deconv_layer = nn.ConvTranspose2d(in_channels, out_channels, kernel_size, stride, padding, bias=False)\n layers.append(deconv_layer)\n if batch_norm:\n layers.append(nn.BatchNorm2d(out_channels))\n return nn.Sequential(*layers)\n",
"_____no_output_____"
],
[
"class Generator(nn.Module):\n \n def __init__(self, z_size, conv_dim=32):\n super(Generator, self).__init__()\n\n # complete init function\n self.fc = nn.Linear(z_size, 4*4*512)\n \n self.deconv1 = deconv(conv_dim*16, conv_dim*8, kernel_size=4)\n self.deconv2 = deconv(conv_dim*8, conv_dim*4, kernel_size=4)\n self.deconv3 = deconv(conv_dim*4, 3, kernel_size=4, batch_norm=False)\n\n def forward(self, x):\n # complete forward function\n x = self.fc(x)\n x = x.view(-1, 512, 4, 4)\n \n x = self.deconv1(x)\n x = F.relu(x)\n \n x = self.deconv2(x)\n x = F.relu(x)\n \n x = self.deconv3(x)\n x = torch.tanh(x)\n \n return x\n ",
"_____no_output_____"
]
],
[
[
"## Build complete network\n\nDefine your models' hyperparameters and instantiate the discriminator and generator from the classes defined above. Make sure you've passed in the correct input arguments.",
"_____no_output_____"
]
],
[
[
"# define hyperparams\nconv_dim = 32\nz_size = 100\n\n# define discriminator and generator\nD = Discriminator(conv_dim)\nG = Generator(z_size=z_size, conv_dim=conv_dim)\n\nprint(D)\nprint()\nprint(G)",
"Discriminator(\n (conv1): Sequential(\n (0): Conv2d(3, 32, kernel_size=(4, 4), stride=(2, 2), padding=(1, 1), bias=False)\n )\n (conv2): Sequential(\n (0): Conv2d(32, 64, kernel_size=(4, 4), stride=(2, 2), padding=(1, 1), bias=False)\n (1): BatchNorm2d(64, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n )\n (conv3): Sequential(\n (0): Conv2d(64, 128, kernel_size=(4, 4), stride=(2, 2), padding=(1, 1), bias=False)\n (1): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n )\n (fc): Linear(in_features=2048, out_features=1, bias=True)\n)\n\nGenerator(\n (fc): Linear(in_features=100, out_features=8192, bias=True)\n (deconv1): Sequential(\n (0): ConvTranspose2d(512, 256, kernel_size=(4, 4), stride=(2, 2), padding=(1, 1), bias=False)\n (1): BatchNorm2d(256, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n )\n (deconv2): Sequential(\n (0): ConvTranspose2d(256, 128, kernel_size=(4, 4), stride=(2, 2), padding=(1, 1), bias=False)\n (1): BatchNorm2d(128, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n )\n (deconv3): Sequential(\n (0): ConvTranspose2d(128, 3, kernel_size=(4, 4), stride=(2, 2), padding=(1, 1), bias=False)\n )\n)\n"
]
],
[
[
"### Training on GPU\n\nCheck if you can train on GPU. If you can, set this as a variable and move your models to GPU. \n> Later, we'll also move any inputs our models and loss functions see (real_images, z, and ground truth labels) to GPU as well.",
"_____no_output_____"
]
],
[
[
"train_on_gpu = torch.cuda.is_available()\n\nif train_on_gpu:\n # move models to GPU\n G.cuda()\n D.cuda()\n print('GPU available for training. Models moved to GPU')\nelse:\n print('Training on CPU.')\n ",
"GPU available for training. Models moved to GPU\n"
]
],
[
[
"---\n## Discriminator and Generator Losses\n\nNow we need to calculate the losses. And this will be exactly the same as before.\n\n### Discriminator Losses\n\n> * For the discriminator, the total loss is the sum of the losses for real and fake images, `d_loss = d_real_loss + d_fake_loss`. \n* Remember that we want the discriminator to output 1 for real images and 0 for fake images, so we need to set up the losses to reflect that.\n\nThe losses will by binary cross entropy loss with logits, which we can get with [BCEWithLogitsLoss](https://pytorch.org/docs/stable/nn.html#bcewithlogitsloss). This combines a `sigmoid` activation function **and** and binary cross entropy loss in one function.\n\nFor the real images, we want `D(real_images) = 1`. That is, we want the discriminator to classify the real images with a label = 1, indicating that these are real. The discriminator loss for the fake data is similar. We want `D(fake_images) = 0`, where the fake images are the _generator output_, `fake_images = G(z)`. \n\n### Generator Loss\n\nThe generator loss will look similar only with flipped labels. The generator's goal is to get `D(fake_images) = 1`. In this case, the labels are **flipped** to represent that the generator is trying to fool the discriminator into thinking that the images it generates (fakes) are real!",
"_____no_output_____"
]
],
[
[
"def real_loss(D_out, smooth=False):\n batch_size = D_out.size(0)\n # label smoothing\n if smooth:\n # smooth, real labels = 0.9\n labels = torch.ones(batch_size)*0.9\n else:\n labels = torch.ones(batch_size) # real labels = 1\n # move labels to GPU if available \n if train_on_gpu:\n labels = labels.cuda()\n # binary cross entropy with logits loss\n criterion = nn.BCEWithLogitsLoss()\n # calculate loss\n loss = criterion(D_out.squeeze(), labels)\n return loss\n\ndef fake_loss(D_out):\n batch_size = D_out.size(0)\n labels = torch.zeros(batch_size) # fake labels = 0\n if train_on_gpu:\n labels = labels.cuda()\n criterion = nn.BCEWithLogitsLoss()\n # calculate loss\n loss = criterion(D_out.squeeze(), labels)\n return loss",
"_____no_output_____"
]
],
[
[
"## Optimizers\n\nNot much new here, but notice how I am using a small learning rate and custom parameters for the Adam optimizers, This is based on some research into DCGAN model convergence.\n\n### Hyperparameters\n\nGANs are very sensitive to hyperparameters. A lot of experimentation goes into finding the best hyperparameters such that the generator and discriminator don't overpower each other. Try out your own hyperparameters or read [the DCGAN paper](https://arxiv.org/pdf/1511.06434.pdf) to see what worked for them.",
"_____no_output_____"
]
],
[
[
"import torch.optim as optim\n\n# params\nlr = 0.0002\nbeta1=0.5\nbeta2=0.999\n\n# Create optimizers for the discriminator and generator\nd_optimizer = optim.Adam(D.parameters(), lr, [beta1, beta2])\ng_optimizer = optim.Adam(G.parameters(), lr, [beta1, beta2])",
"_____no_output_____"
]
],
[
[
"---\n## Training\n\nTraining will involve alternating between training the discriminator and the generator. We'll use our functions `real_loss` and `fake_loss` to help us calculate the discriminator losses in all of the following cases.\n\n### Discriminator training\n1. Compute the discriminator loss on real, training images \n2. Generate fake images\n3. Compute the discriminator loss on fake, generated images \n4. Add up real and fake loss\n5. Perform backpropagation + an optimization step to update the discriminator's weights\n\n### Generator training\n1. Generate fake images\n2. Compute the discriminator loss on fake images, using **flipped** labels!\n3. Perform backpropagation + an optimization step to update the generator's weights\n\n#### Saving Samples\n\nAs we train, we'll also print out some loss statistics and save some generated \"fake\" samples.\n\n**Evaluation mode**\n\nNotice that, when we call our generator to create the samples to display, we set our model to evaluation mode: `G.eval()`. That's so the batch normalization layers will use the population statistics rather than the batch statistics (as they do during training), *and* so dropout layers will operate in eval() mode; not turning off any nodes for generating samples.",
"_____no_output_____"
]
],
[
[
"import pickle as pkl\n\n# training hyperparams\nnum_epochs = 30\n\n# keep track of loss and generated, \"fake\" samples\nsamples = []\nlosses = []\n\nprint_every = 300\n\n# Get some fixed data for sampling. These are images that are held\n# constant throughout training, and allow us to inspect the model's performance\nsample_size=16\nfixed_z = np.random.uniform(-1, 1, size=(sample_size, z_size))\nfixed_z = torch.from_numpy(fixed_z).float()\n\n# train the network\nfor epoch in range(num_epochs):\n \n for batch_i, (real_images, _) in enumerate(train_loader):\n \n batch_size = real_images.size(0)\n \n # important rescaling step\n real_images = scale(real_images)\n \n # ============================================\n # TRAIN THE DISCRIMINATOR\n # ============================================\n \n d_optimizer.zero_grad()\n \n # 1. Train with real images\n\n # Compute the discriminator losses on real images \n if train_on_gpu:\n real_images = real_images.cuda()\n \n D_real = D(real_images)\n d_real_loss = real_loss(D_real)\n \n # 2. Train with fake images\n \n # Generate fake images\n z = np.random.uniform(-1, 1, size=(batch_size, z_size))\n z = torch.from_numpy(z).float()\n # move x to GPU, if available\n if train_on_gpu:\n z = z.cuda()\n fake_images = G(z)\n \n # Compute the discriminator losses on fake images \n D_fake = D(fake_images)\n d_fake_loss = fake_loss(D_fake)\n \n # add up loss and perform backprop\n d_loss = d_real_loss + d_fake_loss\n d_loss.backward()\n d_optimizer.step()\n \n \n # =========================================\n # TRAIN THE GENERATOR\n # =========================================\n g_optimizer.zero_grad()\n \n # 1. Train with fake images and flipped labels\n \n # Generate fake images\n z = np.random.uniform(-1, 1, size=(batch_size, z_size))\n z = torch.from_numpy(z).float()\n if train_on_gpu:\n z = z.cuda()\n fake_images = G(z)\n \n # Compute the discriminator losses on fake images \n # using flipped labels!\n D_fake = D(fake_images)\n g_loss = real_loss(D_fake) # use real loss to flip labels\n \n # perform backprop\n g_loss.backward()\n g_optimizer.step()\n\n # Print some loss stats\n if batch_i % print_every == 0:\n # append discriminator loss and generator loss\n losses.append((d_loss.item(), g_loss.item()))\n # print discriminator and generator loss\n print('Epoch [{:5d}/{:5d}] | d_loss: {:6.4f} | g_loss: {:6.4f}'.format(\n epoch+1, num_epochs, d_loss.item(), g_loss.item()))\n\n \n ## AFTER EACH EPOCH## \n # generate and save sample, fake images\n G.eval() # for generating samples\n if train_on_gpu:\n fixed_z = fixed_z.cuda()\n samples_z = G(fixed_z)\n samples.append(samples_z)\n G.train() # back to training mode\n\n\n# Save training generator samples\nwith open('train_samples.pkl', 'wb') as f:\n pkl.dump(samples, f)",
"Epoch [ 1/ 30] | d_loss: 1.4085 | g_loss: 0.9993\nEpoch [ 1/ 30] | d_loss: 0.6737 | g_loss: 1.9478\nEpoch [ 2/ 30] | d_loss: 0.7026 | g_loss: 2.6182\nEpoch [ 2/ 30] | d_loss: 0.4292 | g_loss: 2.3596\nEpoch [ 3/ 30] | d_loss: 0.2889 | g_loss: 2.7350\nEpoch [ 3/ 30] | d_loss: 0.1361 | g_loss: 4.5357\nEpoch [ 4/ 30] | d_loss: 0.2069 | g_loss: 4.2325\nEpoch [ 4/ 30] | d_loss: 0.2646 | g_loss: 10.0169\nEpoch [ 5/ 30] | d_loss: 0.1014 | g_loss: 5.4149\nEpoch [ 5/ 30] | d_loss: 0.0929 | g_loss: 4.8199\nEpoch [ 6/ 30] | d_loss: 0.0590 | g_loss: 6.2550\nEpoch [ 6/ 30] | d_loss: 0.3863 | g_loss: 2.4094\nEpoch [ 7/ 30] | d_loss: 0.1023 | g_loss: 4.1469\nEpoch [ 7/ 30] | d_loss: 0.0450 | g_loss: 5.3767\nEpoch [ 8/ 30] | d_loss: 0.1133 | g_loss: 3.1710\nEpoch [ 8/ 30] | d_loss: 0.3909 | g_loss: 2.8371\nEpoch [ 9/ 30] | d_loss: 0.0228 | g_loss: 7.7792\nEpoch [ 9/ 30] | d_loss: 0.5372 | g_loss: 3.8941\nEpoch [ 10/ 30] | d_loss: 0.0888 | g_loss: 4.3109\nEpoch [ 10/ 30] | d_loss: 0.4739 | g_loss: 5.8511\nEpoch [ 11/ 30] | d_loss: 0.1066 | g_loss: 4.5965\nEpoch [ 11/ 30] | d_loss: 0.0896 | g_loss: 8.8515\nEpoch [ 12/ 30] | d_loss: 0.0152 | g_loss: 6.1287\nEpoch [ 12/ 30] | d_loss: 0.0917 | g_loss: 3.1805\nEpoch [ 13/ 30] | d_loss: 0.5349 | g_loss: 6.7379\nEpoch [ 13/ 30] | d_loss: 0.0511 | g_loss: 7.0306\nEpoch [ 14/ 30] | d_loss: 0.0228 | g_loss: 4.7947\nEpoch [ 14/ 30] | d_loss: 0.0280 | g_loss: 6.1609\nEpoch [ 15/ 30] | d_loss: 0.0406 | g_loss: 7.4366\nEpoch [ 15/ 30] | d_loss: 0.0334 | g_loss: 5.7624\nEpoch [ 16/ 30] | d_loss: 0.0413 | g_loss: 6.2405\nEpoch [ 16/ 30] | d_loss: 0.2505 | g_loss: 3.0691\nEpoch [ 17/ 30] | d_loss: 0.1006 | g_loss: 6.3208\nEpoch [ 17/ 30] | d_loss: 0.1634 | g_loss: 3.8810\nEpoch [ 18/ 30] | d_loss: 0.0337 | g_loss: 5.8343\nEpoch [ 18/ 30] | d_loss: 0.2797 | g_loss: 6.6325\nEpoch [ 19/ 30] | d_loss: 0.3332 | g_loss: 4.8526\nEpoch [ 19/ 30] | d_loss: 0.0802 | g_loss: 3.8929\nEpoch [ 20/ 30] | d_loss: 0.3042 | g_loss: 3.1036\nEpoch [ 20/ 30] | d_loss: 0.1205 | g_loss: 1.6828\nEpoch [ 21/ 30] | d_loss: 0.0568 | g_loss: 3.0710\nEpoch [ 21/ 30] | d_loss: 0.1022 | g_loss: 5.4802\nEpoch [ 22/ 30] | d_loss: 0.2595 | g_loss: 8.0963\nEpoch [ 22/ 30] | d_loss: 0.1082 | g_loss: 2.2444\nEpoch [ 23/ 30] | d_loss: 0.0162 | g_loss: 8.6265\nEpoch [ 23/ 30] | d_loss: 0.1178 | g_loss: 5.9417\nEpoch [ 24/ 30] | d_loss: 0.1680 | g_loss: 4.4331\nEpoch [ 24/ 30] | d_loss: 0.5456 | g_loss: 3.4422\nEpoch [ 25/ 30] | d_loss: 0.2071 | g_loss: 2.1712\nEpoch [ 25/ 30] | d_loss: 0.0729 | g_loss: 5.1144\nEpoch [ 26/ 30] | d_loss: 0.0537 | g_loss: 3.7469\nEpoch [ 26/ 30] | d_loss: 0.3997 | g_loss: 6.2408\nEpoch [ 27/ 30] | d_loss: 0.0555 | g_loss: 2.4301\nEpoch [ 27/ 30] | d_loss: 0.1863 | g_loss: 3.7632\nEpoch [ 28/ 30] | d_loss: 0.2211 | g_loss: 3.6232\nEpoch [ 28/ 30] | d_loss: 0.1328 | g_loss: 4.7159\nEpoch [ 29/ 30] | d_loss: 0.1348 | g_loss: 3.1974\nEpoch [ 29/ 30] | d_loss: 0.2392 | g_loss: 2.6332\nEpoch [ 30/ 30] | d_loss: 0.0215 | g_loss: 6.6233\nEpoch [ 30/ 30] | d_loss: 0.2591 | g_loss: 3.3791\n"
]
],
[
[
"## Training loss\n\nHere we'll plot the training losses for the generator and discriminator, recorded after each epoch.",
"_____no_output_____"
]
],
[
[
"fig, ax = plt.subplots()\nlosses = np.array(losses)\nplt.plot(losses.T[0], label='Discriminator', alpha=0.5)\nplt.plot(losses.T[1], label='Generator', alpha=0.5)\nplt.title(\"Training Losses\")\nplt.legend()",
"_____no_output_____"
]
],
[
[
"## Generator samples from training\n\nHere we can view samples of images from the generator. We'll look at the images we saved during training.",
"_____no_output_____"
]
],
[
[
"# helper function for viewing a list of passed in sample images\ndef view_samples(epoch, samples):\n fig, axes = plt.subplots(figsize=(16,4), nrows=2, ncols=8, sharey=True, sharex=True)\n for ax, img in zip(axes.flatten(), samples[epoch]):\n img = img.detach().cpu().numpy()\n img = np.transpose(img, (1, 2, 0))\n img = ((img +1)*255 / (2)).astype(np.uint8) # rescale to pixel range (0-255)\n ax.xaxis.set_visible(False)\n ax.yaxis.set_visible(False)\n im = ax.imshow(img.reshape((32,32,3)))",
"_____no_output_____"
],
[
"_ = view_samples(-1, samples)",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
]
] |
e7e8715ccc2b616bdeec5799166d33268944c427 | 1,473 | ipynb | Jupyter Notebook | 121. Best Time to Buy and Sell Stock.ipynb | SurajpratapsinghSayar/LeetCode-Solutions | 1a4c0a803d54aa77e801eb1052ce92fafc2d1ca8 | [
"MIT"
] | null | null | null | 121. Best Time to Buy and Sell Stock.ipynb | SurajpratapsinghSayar/LeetCode-Solutions | 1a4c0a803d54aa77e801eb1052ce92fafc2d1ca8 | [
"MIT"
] | null | null | null | 121. Best Time to Buy and Sell Stock.ipynb | SurajpratapsinghSayar/LeetCode-Solutions | 1a4c0a803d54aa77e801eb1052ce92fafc2d1ca8 | [
"MIT"
] | null | null | null | 17.746988 | 43 | 0.462322 | [
[
[
"prices = [7,1,5,3,6,4]",
"_____no_output_____"
],
[
"def solution(arr):\n low = min(arr)\n temp = arr[prices.index(low):]\n if len(temp) == 1:\n return 0\n else : \n high = max(temp[1:])\n return high-low",
"_____no_output_____"
],
[
"profit = solution(prices)\nprofit",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code"
]
] |
e7e877f4e687d4bbf2fdf9188de7f0be99d52e76 | 11,447 | ipynb | Jupyter Notebook | Frame_Extraction.ipynb | TeamEpic1/Driver-Drowsiness-Detection | bc40f22314a4da73aa0324ec9b90edb68d055fff | [
"MIT"
] | 2 | 2020-12-10T07:22:41.000Z | 2020-12-21T06:49:01.000Z | Frame_Extraction.ipynb | TeamEpic1/Driver-Drowsiness-Detection | bc40f22314a4da73aa0324ec9b90edb68d055fff | [
"MIT"
] | null | null | null | Frame_Extraction.ipynb | TeamEpic1/Driver-Drowsiness-Detection | bc40f22314a4da73aa0324ec9b90edb68d055fff | [
"MIT"
] | 1 | 2020-12-10T06:14:24.000Z | 2020-12-10T06:14:24.000Z | 32.519886 | 107 | 0.473312 | [
[
[
"!wget http://vlm1.uta.edu/~athitsos/projects/drowsiness/Fold2_part1.zip\n!unzip Fold2_part1.zip",
"--2020-12-01 13:34:20-- http://vlm1.uta.edu/~athitsos/projects/drowsiness/Fold2_part1.zip\nResolving vlm1.uta.edu (vlm1.uta.edu)... 129.107.118.39\nConnecting to vlm1.uta.edu (vlm1.uta.edu)|129.107.118.39|:80... connected.\nHTTP request sent, awaiting response... 200 OK\nLength: 11333579094 (11G) [application/zip]\nSaving to: ‘Fold2_part1.zip’\n\nFold2_part1.zip 100%[===================>] 10.55G 23.1MB/s in 7m 12s \n\n2020-12-01 13:41:32 (25.0 MB/s) - ‘Fold2_part1.zip’ saved [11333579094/11333579094]\n\nArchive: Fold2_part1.zip\n creating: Fold2_part1/\n creating: Fold2_part1/13/\n inflating: Fold2_part1/13/5.mp4 \n inflating: Fold2_part1/13/10.mp4 \n inflating: Fold2_part1/13/0.mp4 \n creating: Fold2_part1/18/\n inflating: Fold2_part1/18/5.mov \n inflating: Fold2_part1/18/0.mov \n inflating: Fold2_part1/18/10.mov \n creating: Fold2_part1/16/\n inflating: Fold2_part1/16/0.MOV \n inflating: Fold2_part1/16/5.MOV \n inflating: Fold2_part1/16/10.MOV \n creating: Fold2_part1/14/\n inflating: Fold2_part1/14/10.mp4 \n inflating: Fold2_part1/14/0.mp4 \n inflating: Fold2_part1/14/5.mp4 \n creating: Fold2_part1/15/\n inflating: Fold2_part1/15/5.mp4 \n inflating: Fold2_part1/15/0.mp4 \n inflating: Fold2_part1/15/10.mp4 \n creating: Fold2_part1/17/\n inflating: Fold2_part1/17/10.mp4 \n inflating: Fold2_part1/17/0.mp4 \n inflating: Fold2_part1/17/5.mp4 \n"
],
[
"from google.colab import drive\ndrive.mount('/content/drive')",
"Mounted at /content/drive\n"
],
[
"!pip install dlib\n!pip install face_recognition\n!pip install opencv-python",
"_____no_output_____"
],
[
"import cv2\nimport matplotlib.pyplot as plt\nimport numpy as np",
"_____no_output_____"
],
[
"def video_to_frames(path):\n # Array of video frames\n video_frames = []\n # Start capturing the feed\n cap = cv2.VideoCapture(path)\n # Find the number of frames\n video_length = int(cap.get(cv2.CAP_PROP_FRAME_COUNT))\n print (\"Number of frames: \", video_length)\n\n # Save frame every 3 seconds\n seconds = 3\n fps = cap.get(cv2.CAP_PROP_FPS)\n print('Frames per second: ', fps)\n\n # number of frames to skip\n multiplier = fps * seconds\n frame_counter = 0\n\n while frame_counter <= video_length:\n cap.set(cv2.CAP_PROP_POS_FRAMES, frame_counter)\n ret, frame = cap.read()\n # Convert frame to rgb\n frame_rgb = cv2.cvtColor(frame, cv2.COLOR_BGR2RGB)\n video_frames.append(frame_rgb)\n frame_counter += multiplier\n\n\n print('Frames captured: ', len(video_frames))\n return video_frames\n\n ",
"_____no_output_____"
],
[
"folder_path = '/content/Fold2_part1'\noutput_path = '/content/drive/MyDrive/Team Epic - Driver Drowsiness Detection'",
"_____no_output_____"
],
[
"for i in range(13, 16):\n for j in range(0,11,5):\n video_path = folder_path + '/' + str(i) + '/' + str(j) + '.mp4'\n captured_frames = video_to_frames(video_path)\n file_path = output_path + '/' + str(i) + '_' + str(j) + '.npy'\n np.save(file_path, np.asarray(captured_frames))",
"Number of frames: 18236\nFrames per second: 29.78902165541211\nFrames captured: 205\nNumber of frames: 20477\nFrames per second: 29.78870102526485\nFrames captured: 230\nNumber of frames: 18486\nFrames per second: 29.789651477514717\nFrames captured: 207\nNumber of frames: 18386\nFrames per second: 30.000032633561005\nFrames captured: 205\nNumber of frames: 19742\nFrames per second: 29.849765353809886\nFrames captured: 221\nNumber of frames: 18692\nFrames per second: 30.000032099328163\nFrames captured: 208\nNumber of frames: 19301\nFrames per second: 30.001013453228925\nFrames captured: 215\nNumber of frames: 18541\nFrames per second: 30.000920690570787\nFrames captured: 207\nNumber of frames: 18483\nFrames per second: 30.000256454066967\nFrames captured: 206\n"
],
[
"# Extract frames for folders 16, 17,18\nfor j in range(0,11,5):\n video_path = folder_path + '/' + str(16) + '/' + str(j) + '.MOV'\n captured_frames = video_to_frames(video_path)\n file_path = output_path + '/' + str(16) + '_' + str(j) + '.npy'\n np.save(file_path, np.asarray(captured_frames))\n\n ",
"Number of frames: 11357\nFrames per second: 24.006425948592412\nFrames captured: 158\nNumber of frames: 14488\nFrames per second: 24.00642911059632\nFrames captured: 202\nNumber of frames: 14491\nFrames per second: 24.00642777761091\nFrames captured: 202\n"
],
[
"for j in range(0,11,5):\n video_path = folder_path + '/' + str(17) + '/' + str(j) + '.mp4'\n captured_frames = video_to_frames(video_path)\n file_path = output_path + '/' + str(17) + '_' + str(j) + '.npy'\n np.save(file_path, np.asarray(captured_frames))",
"Number of frames: 18121\nFrames per second: 29.97002997002997\nFrames captured: 202\nNumber of frames: 18245\nFrames per second: 29.97002997002997\nFrames captured: 203\nNumber of frames: 18212\nFrames per second: 29.9375227636899\nFrames captured: 203\n"
],
[
"for j in range(0,11,5):\n video_path = folder_path + '/' + str(18) + '/' + str(j) + '.mov'\n captured_frames = video_to_frames(video_path)\n file_path = output_path + '/' + str(18) + '_' + str(j) + '.npy'\n np.save(file_path, np.asarray(captured_frames))",
"Number of frames: 11672\nFrames per second: 19.366346161823145\nFrames captured: 201\nNumber of frames: 8975\nFrames per second: 14.858246533765607\nFrames captured: 202\nNumber of frames: 7676\nFrames per second: 12.002220316942426\nFrames captured: 214\n"
],
[
"",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e8789400e4d21728ec13c383252d2b660fb4ea | 147,213 | ipynb | Jupyter Notebook | src/plotting/luminescence/24.11.19/luminescence_plots.ipynb | Switham1/PromoterArchitecture | 0a9021b869ac66cdd622be18cd029950314d111e | [
"MIT"
] | null | null | null | src/plotting/luminescence/24.11.19/luminescence_plots.ipynb | Switham1/PromoterArchitecture | 0a9021b869ac66cdd622be18cd029950314d111e | [
"MIT"
] | null | null | null | src/plotting/luminescence/24.11.19/luminescence_plots.ipynb | Switham1/PromoterArchitecture | 0a9021b869ac66cdd622be18cd029950314d111e | [
"MIT"
] | null | null | null | 76.673438 | 35,576 | 0.680619 | [
[
[
"import pandas as pd\nimport numpy as np\nimport skbio\nfrom collections import Counter\nimport seaborn as sns\nimport matplotlib.pyplot as plt\nfrom scipy import stats\nfrom statsmodels.formula.api import ols\nimport researchpy as rp\nfrom matplotlib import rcParams\nrcParams.update({'figure.autolayout': True})",
"_____no_output_____"
],
[
"luminescence_means = \"../../../../data/luminescence/to_be_sorted/24.11.19/output_means.csv\"\nluminescence_raw = \"../../../../data/luminescence/to_be_sorted/24.11.19/output_raw.csv\"",
"_____no_output_____"
],
[
"luminescence_means_df = pd.read_csv(luminescence_means, header=0)\nluminescence_raw_df = pd.read_csv(luminescence_raw, header=0)",
"_____no_output_____"
],
[
"luminescence_means_df",
"_____no_output_____"
],
[
"luminescence_raw_df",
"_____no_output_____"
],
[
"#add promoter names column\nluminescence_raw_df['Promoter'] = luminescence_raw_df.name ",
"_____no_output_____"
],
[
"luminescence_raw_df.loc[luminescence_raw_df.name == '71 + 72', 'Promoter'] = 'UBQ10'\nluminescence_raw_df.loc[luminescence_raw_df.name == '25+72', 'Promoter'] = 'NIR1'\nluminescence_raw_df.loc[luminescence_raw_df.name == '35+72', 'Promoter'] = 'NOS'\nluminescence_raw_df.loc[luminescence_raw_df.name == '36+72', 'Promoter'] = 'STAP4'\nluminescence_raw_df.loc[luminescence_raw_df.name == '92+72', 'Promoter'] = 'NRP'",
"_____no_output_____"
],
[
"luminescence_raw_df",
"_____no_output_____"
],
[
"#set style to ticks\nsns.set(style=\"ticks\", color_codes=True)",
"_____no_output_____"
],
[
"plot = sns.catplot(x=\"Promoter\", y=\"nluc/fluc\", data=luminescence_raw_df, hue='condition', kind='violin')\n#plot points\nax = sns.swarmplot(x=\"Promoter\", y=\"nluc/fluc\", data=luminescence_raw_df, color=\".25\").get_figure().savefig('../../../../data/plots/luminescence/24.11.19/luminescence_violin.pdf', format='pdf')",
"_____no_output_____"
],
[
"#bar chart, 68% confidence intervals (1 Standard error)\nplot = sns.barplot(x=\"Promoter\", y=\"nluc/fluc\", hue=\"condition\", data=luminescence_raw_df, ci=68, capsize=0.1,errwidth=0.7)\nplt.ylabel(\"Mean_luminescence\").get_figure().savefig('../../../../data/plots/luminescence/24.11.19/promoter_luminescence.pdf', format='pdf')",
"_____no_output_____"
],
[
"#plot raw UBQ10\nplot = sns.barplot(x=\"condition\", y=\"fluc_luminescence\", data=luminescence_raw_df,ci=68,capsize=0.1,errwidth=0.7)\nplot.set_xticklabels(plot.get_xticklabels(), rotation=45)\nplt.ylabel(\"Mean_luminescence\").get_figure().savefig('../../../../data/plots/luminescence/24.11.19/UBQ10fluc_raw.pdf', format='pdf')",
"_____no_output_____"
]
],
[
[
"### get names of each condition for later",
"_____no_output_____"
]
],
[
[
"pd.Categorical(luminescence_raw_df.condition)\nnames = luminescence_raw_df.condition.unique()\nfor name in names:\n print(name)",
"nitrate_free\n100mM nitrate_2hrs_morning\n100mM nitrate_overnight\n"
],
[
"#get list of promoters\npd.Categorical(luminescence_raw_df.Promoter)\nprom_names = luminescence_raw_df.Promoter.unique()\nfor name in prom_names:\n print(name)",
"UBQ10\nNIR1\nNOS\nSTAP4\nNRP\n"
]
],
[
[
"### test normality",
"_____no_output_____"
]
],
[
[
"#returns test statistic, p-value\nfor name1 in prom_names:\n for name in names:\n print('{}: {}'.format(name, stats.shapiro(luminescence_raw_df['nluc/fluc'][luminescence_raw_df.condition == name])))\n",
"nitrate_free: (0.7033216953277588, 0.0002697518502827734)\n100mM nitrate_2hrs_morning: (0.7973607182502747, 0.00463036959990859)\n100mM nitrate_overnight: (0.8101227879524231, 0.004972793627530336)\nnitrate_free: (0.7033216953277588, 0.0002697518502827734)\n100mM nitrate_2hrs_morning: (0.7973607182502747, 0.00463036959990859)\n100mM nitrate_overnight: (0.8101227879524231, 0.004972793627530336)\nnitrate_free: (0.7033216953277588, 0.0002697518502827734)\n100mM nitrate_2hrs_morning: (0.7973607182502747, 0.00463036959990859)\n100mM nitrate_overnight: (0.8101227879524231, 0.004972793627530336)\nnitrate_free: (0.7033216953277588, 0.0002697518502827734)\n100mM nitrate_2hrs_morning: (0.7973607182502747, 0.00463036959990859)\n100mM nitrate_overnight: (0.8101227879524231, 0.004972793627530336)\nnitrate_free: (0.7033216953277588, 0.0002697518502827734)\n100mM nitrate_2hrs_morning: (0.7973607182502747, 0.00463036959990859)\n100mM nitrate_overnight: (0.8101227879524231, 0.004972793627530336)\n"
]
],
[
[
"#### not normal",
"_____no_output_____"
]
],
[
[
"#test variance\nstats.levene(luminescence_raw_df['nluc/fluc'][luminescence_raw_df.condition == names[0]], \n luminescence_raw_df['nluc/fluc'][luminescence_raw_df.condition == names[1]], \n luminescence_raw_df['nluc/fluc'][luminescence_raw_df.condition == names[2]])",
"_____no_output_____"
],
[
"test = luminescence_raw_df.groupby('Promoter')['nluc/fluc'].apply",
"_____no_output_____"
],
[
"test",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
]
] |
e7e87c946b6939f2ca9650f364c33d2174688615 | 46,118 | ipynb | Jupyter Notebook | solution_AIIJC(NLP)/Notebooks/singleAnswering_Aiijc.ipynb | Makual/AIIJC_NLP | 06bca8db0153ff9dd0320fb9b84d43c8d52b8657 | [
"MIT"
] | 1 | 2021-11-13T18:04:55.000Z | 2021-11-13T18:04:55.000Z | solution_AIIJC(NLP)/Notebooks/singleAnswering_Aiijc.ipynb | Makual/AIIJC_NLP | 06bca8db0153ff9dd0320fb9b84d43c8d52b8657 | [
"MIT"
] | null | null | null | solution_AIIJC(NLP)/Notebooks/singleAnswering_Aiijc.ipynb | Makual/AIIJC_NLP | 06bca8db0153ff9dd0320fb9b84d43c8d52b8657 | [
"MIT"
] | null | null | null | 67.325547 | 1,704 | 0.575068 | [
[
[
"Загрузка библиотек и данных",
"_____no_output_____"
]
],
[
[
"!pip install simpletransformers==0.61.13\n!pip uninstall transformers\n!pip install transformers==4.10.0\n!git clone https://github.com/GoldenRMT/WikiSearch.git\n!pip install googledrivedownloader",
"Collecting simpletransformers==0.61.13\n Downloading simpletransformers-0.61.13-py3-none-any.whl (221 kB)\n\u001b[?25l\r\u001b[K |█▌ | 10 kB 20.1 MB/s eta 0:00:01\r\u001b[K |███ | 20 kB 12.5 MB/s eta 0:00:01\r\u001b[K |████▍ | 30 kB 9.6 MB/s eta 0:00:01\r\u001b[K |██████ | 40 kB 8.6 MB/s eta 0:00:01\r\u001b[K |███████▍ | 51 kB 5.3 MB/s eta 0:00:01\r\u001b[K |████████▉ | 61 kB 5.8 MB/s eta 0:00:01\r\u001b[K |██████████▍ | 71 kB 5.7 MB/s eta 0:00:01\r\u001b[K |███████████▉ | 81 kB 6.3 MB/s eta 0:00:01\r\u001b[K |█████████████▎ | 92 kB 6.3 MB/s eta 0:00:01\r\u001b[K |██████████████▉ | 102 kB 5.4 MB/s eta 0:00:01\r\u001b[K |████████████████▎ | 112 kB 5.4 MB/s eta 0:00:01\r\u001b[K |█████████████████▊ | 122 kB 5.4 MB/s eta 0:00:01\r\u001b[K |███████████████████▎ | 133 kB 5.4 MB/s eta 0:00:01\r\u001b[K |████████████████████▊ | 143 kB 5.4 MB/s eta 0:00:01\r\u001b[K |██████████████████████▏ | 153 kB 5.4 MB/s eta 0:00:01\r\u001b[K |███████████████████████▋ | 163 kB 5.4 MB/s eta 0:00:01\r\u001b[K |█████████████████████████▏ | 174 kB 5.4 MB/s eta 0:00:01\r\u001b[K |██████████████████████████▋ | 184 kB 5.4 MB/s eta 0:00:01\r\u001b[K |████████████████████████████ | 194 kB 5.4 MB/s eta 0:00:01\r\u001b[K |█████████████████████████████▋ | 204 kB 5.4 MB/s eta 0:00:01\r\u001b[K |███████████████████████████████ | 215 kB 5.4 MB/s eta 0:00:01\r\u001b[K |████████████████████████████████| 221 kB 5.4 MB/s \n\u001b[?25hRequirement already satisfied: scikit-learn in /usr/local/lib/python3.7/dist-packages (from simpletransformers==0.61.13) (0.22.2.post1)\nRequirement already satisfied: pandas in /usr/local/lib/python3.7/dist-packages (from simpletransformers==0.61.13) (1.1.5)\nCollecting tensorboardx\n Downloading tensorboardX-2.4-py2.py3-none-any.whl (124 kB)\n\u001b[K |████████████████████████████████| 124 kB 33.2 MB/s \n\u001b[?25hCollecting wandb>=0.10.32\n Downloading wandb-0.12.4-py2.py3-none-any.whl (1.7 MB)\n\u001b[K |████████████████████████████████| 1.7 MB 30.1 MB/s \n\u001b[?25hRequirement already satisfied: requests in /usr/local/lib/python3.7/dist-packages (from simpletransformers==0.61.13) (2.23.0)\nCollecting datasets\n Downloading datasets-1.13.3-py3-none-any.whl (287 kB)\n\u001b[K |████████████████████████████████| 287 kB 43.6 MB/s \n\u001b[?25hRequirement already satisfied: numpy in /usr/local/lib/python3.7/dist-packages (from simpletransformers==0.61.13) (1.19.5)\nCollecting streamlit\n Downloading streamlit-1.0.0-py2.py3-none-any.whl (8.3 MB)\n\u001b[K |████████████████████████████████| 8.3 MB 30.8 MB/s \n\u001b[?25hCollecting sentencepiece\n Downloading sentencepiece-0.1.96-cp37-cp37m-manylinux_2_17_x86_64.manylinux2014_x86_64.whl (1.2 MB)\n\u001b[K |████████████████████████████████| 1.2 MB 39.2 MB/s \n\u001b[?25hCollecting seqeval\n Downloading seqeval-1.2.2.tar.gz (43 kB)\n\u001b[K |████████████████████████████████| 43 kB 1.6 MB/s \n\u001b[?25hCollecting tokenizers\n Downloading tokenizers-0.10.3-cp37-cp37m-manylinux_2_5_x86_64.manylinux1_x86_64.manylinux_2_12_x86_64.manylinux2010_x86_64.whl (3.3 MB)\n\u001b[K |████████████████████████████████| 3.3 MB 31.8 MB/s \n\u001b[?25hRequirement already satisfied: tqdm>=4.47.0 in /usr/local/lib/python3.7/dist-packages (from simpletransformers==0.61.13) (4.62.3)\nCollecting transformers>=4.2.0\n Downloading transformers-4.11.3-py3-none-any.whl (2.9 MB)\n\u001b[K |████████████████████████████████| 2.9 MB 35.3 MB/s \n\u001b[?25hRequirement already satisfied: regex in /usr/local/lib/python3.7/dist-packages (from simpletransformers==0.61.13) (2019.12.20)\nRequirement already satisfied: scipy in /usr/local/lib/python3.7/dist-packages (from simpletransformers==0.61.13) (1.4.1)\nCollecting pyyaml>=5.1\n Downloading PyYAML-6.0-cp37-cp37m-manylinux_2_5_x86_64.manylinux1_x86_64.manylinux_2_12_x86_64.manylinux2010_x86_64.whl (596 kB)\n\u001b[K |████████████████████████████████| 596 kB 41.4 MB/s \n\u001b[?25hRequirement already satisfied: importlib-metadata in /usr/local/lib/python3.7/dist-packages (from transformers>=4.2.0->simpletransformers==0.61.13) (4.8.1)\nCollecting huggingface-hub>=0.0.17\n Downloading huggingface_hub-0.0.19-py3-none-any.whl (56 kB)\n\u001b[K |████████████████████████████████| 56 kB 4.5 MB/s \n\u001b[?25hCollecting sacremoses\n Downloading sacremoses-0.0.46-py3-none-any.whl (895 kB)\n\u001b[K |████████████████████████████████| 895 kB 39.0 MB/s \n\u001b[?25hRequirement already satisfied: filelock in /usr/local/lib/python3.7/dist-packages (from transformers>=4.2.0->simpletransformers==0.61.13) (3.3.0)\nRequirement already satisfied: packaging>=20.0 in /usr/local/lib/python3.7/dist-packages (from transformers>=4.2.0->simpletransformers==0.61.13) (21.0)\nRequirement already satisfied: typing-extensions in /usr/local/lib/python3.7/dist-packages (from huggingface-hub>=0.0.17->transformers>=4.2.0->simpletransformers==0.61.13) (3.7.4.3)\nRequirement already satisfied: pyparsing>=2.0.2 in /usr/local/lib/python3.7/dist-packages (from packaging>=20.0->transformers>=4.2.0->simpletransformers==0.61.13) (2.4.7)\nCollecting configparser>=3.8.1\n Downloading configparser-5.0.2-py3-none-any.whl (19 kB)\nRequirement already satisfied: Click!=8.0.0,>=7.0 in /usr/local/lib/python3.7/dist-packages (from wandb>=0.10.32->simpletransformers==0.61.13) (7.1.2)\nCollecting docker-pycreds>=0.4.0\n Downloading docker_pycreds-0.4.0-py2.py3-none-any.whl (9.0 kB)\nCollecting pathtools\n Downloading pathtools-0.1.2.tar.gz (11 kB)\nRequirement already satisfied: python-dateutil>=2.6.1 in /usr/local/lib/python3.7/dist-packages (from wandb>=0.10.32->simpletransformers==0.61.13) (2.8.2)\nCollecting sentry-sdk>=1.0.0\n Downloading sentry_sdk-1.4.3-py2.py3-none-any.whl (139 kB)\n\u001b[K |████████████████████████████████| 139 kB 45.7 MB/s \n\u001b[?25hCollecting subprocess32>=3.5.3\n Downloading subprocess32-3.5.4.tar.gz (97 kB)\n\u001b[K |████████████████████████████████| 97 kB 6.6 MB/s \n\u001b[?25hCollecting shortuuid>=0.5.0\n Downloading shortuuid-1.0.1-py3-none-any.whl (7.5 kB)\nRequirement already satisfied: six>=1.13.0 in /usr/local/lib/python3.7/dist-packages (from wandb>=0.10.32->simpletransformers==0.61.13) (1.15.0)\nRequirement already satisfied: protobuf>=3.12.0 in /usr/local/lib/python3.7/dist-packages (from wandb>=0.10.32->simpletransformers==0.61.13) (3.17.3)\nCollecting GitPython>=1.0.0\n Downloading GitPython-3.1.24-py3-none-any.whl (180 kB)\n\u001b[K |████████████████████████████████| 180 kB 43.8 MB/s \n\u001b[?25hRequirement already satisfied: promise<3,>=2.0 in /usr/local/lib/python3.7/dist-packages (from wandb>=0.10.32->simpletransformers==0.61.13) (2.3)\nRequirement already satisfied: psutil>=5.0.0 in /usr/local/lib/python3.7/dist-packages (from wandb>=0.10.32->simpletransformers==0.61.13) (5.4.8)\nCollecting yaspin>=1.0.0\n Downloading yaspin-2.1.0-py3-none-any.whl (18 kB)\nCollecting gitdb<5,>=4.0.1\n Downloading gitdb-4.0.7-py3-none-any.whl (63 kB)\n\u001b[K |████████████████████████████████| 63 kB 1.6 MB/s \n\u001b[?25hCollecting smmap<5,>=3.0.1\n Downloading smmap-4.0.0-py2.py3-none-any.whl (24 kB)\nRequirement already satisfied: chardet<4,>=3.0.2 in /usr/local/lib/python3.7/dist-packages (from requests->simpletransformers==0.61.13) (3.0.4)\nRequirement already satisfied: idna<3,>=2.5 in /usr/local/lib/python3.7/dist-packages (from requests->simpletransformers==0.61.13) (2.10)\nRequirement already satisfied: urllib3!=1.25.0,!=1.25.1,<1.26,>=1.21.1 in /usr/local/lib/python3.7/dist-packages (from requests->simpletransformers==0.61.13) (1.24.3)\nRequirement already satisfied: certifi>=2017.4.17 in /usr/local/lib/python3.7/dist-packages (from requests->simpletransformers==0.61.13) (2021.5.30)\nRequirement already satisfied: termcolor<2.0.0,>=1.1.0 in /usr/local/lib/python3.7/dist-packages (from yaspin>=1.0.0->wandb>=0.10.32->simpletransformers==0.61.13) (1.1.0)\nRequirement already satisfied: dill in /usr/local/lib/python3.7/dist-packages (from datasets->simpletransformers==0.61.13) (0.3.4)\nRequirement already satisfied: pyarrow!=4.0.0,>=1.0.0 in /usr/local/lib/python3.7/dist-packages (from datasets->simpletransformers==0.61.13) (3.0.0)\nCollecting fsspec[http]>=2021.05.0\n Downloading fsspec-2021.10.1-py3-none-any.whl (125 kB)\n\u001b[K |████████████████████████████████| 125 kB 43.1 MB/s \n\u001b[?25hRequirement already satisfied: multiprocess in /usr/local/lib/python3.7/dist-packages (from datasets->simpletransformers==0.61.13) (0.70.12.2)\nCollecting aiohttp\n Downloading aiohttp-3.7.4.post0-cp37-cp37m-manylinux2014_x86_64.whl (1.3 MB)\n\u001b[K |████████████████████████████████| 1.3 MB 33.1 MB/s \n\u001b[?25hCollecting xxhash\n Downloading xxhash-2.0.2-cp37-cp37m-manylinux2010_x86_64.whl (243 kB)\n\u001b[K |████████████████████████████████| 243 kB 49.2 MB/s \n\u001b[?25hCollecting async-timeout<4.0,>=3.0\n Downloading async_timeout-3.0.1-py3-none-any.whl (8.2 kB)\nCollecting yarl<2.0,>=1.0\n Downloading yarl-1.7.0-cp37-cp37m-manylinux_2_5_x86_64.manylinux1_x86_64.manylinux_2_12_x86_64.manylinux2010_x86_64.whl (271 kB)\n\u001b[K |████████████████████████████████| 271 kB 46.3 MB/s \n\u001b[?25hCollecting multidict<7.0,>=4.5\n Downloading multidict-5.2.0-cp37-cp37m-manylinux_2_5_x86_64.manylinux1_x86_64.manylinux_2_12_x86_64.manylinux2010_x86_64.whl (160 kB)\n\u001b[K |████████████████████████████████| 160 kB 49.2 MB/s \n\u001b[?25hRequirement already satisfied: attrs>=17.3.0 in /usr/local/lib/python3.7/dist-packages (from aiohttp->datasets->simpletransformers==0.61.13) (21.2.0)\nRequirement already satisfied: zipp>=0.5 in /usr/local/lib/python3.7/dist-packages (from importlib-metadata->transformers>=4.2.0->simpletransformers==0.61.13) (3.6.0)\nRequirement already satisfied: pytz>=2017.2 in /usr/local/lib/python3.7/dist-packages (from pandas->simpletransformers==0.61.13) (2018.9)\nRequirement already satisfied: joblib in /usr/local/lib/python3.7/dist-packages (from sacremoses->transformers>=4.2.0->simpletransformers==0.61.13) (1.0.1)\nRequirement already satisfied: altair>=3.2.0 in /usr/local/lib/python3.7/dist-packages (from streamlit->simpletransformers==0.61.13) (4.1.0)\nCollecting blinker\n Downloading blinker-1.4.tar.gz (111 kB)\n\u001b[K |████████████████████████████████| 111 kB 47.0 MB/s \n\u001b[?25hRequirement already satisfied: tornado>=5.0 in /usr/local/lib/python3.7/dist-packages (from streamlit->simpletransformers==0.61.13) (5.1.1)\nRequirement already satisfied: toml in /usr/local/lib/python3.7/dist-packages (from streamlit->simpletransformers==0.61.13) (0.10.2)\nCollecting validators\n Downloading validators-0.18.2-py3-none-any.whl (19 kB)\nRequirement already satisfied: astor in /usr/local/lib/python3.7/dist-packages (from streamlit->simpletransformers==0.61.13) (0.8.1)\nCollecting base58\n Downloading base58-2.1.0-py3-none-any.whl (5.6 kB)\nCollecting pydeck>=0.1.dev5\n Downloading pydeck-0.7.0-py2.py3-none-any.whl (4.3 MB)\n\u001b[K |████████████████████████████████| 4.3 MB 28.5 MB/s \n\u001b[?25hCollecting watchdog\n Downloading watchdog-2.1.6-py3-none-manylinux2014_x86_64.whl (76 kB)\n\u001b[K |████████████████████████████████| 76 kB 4.8 MB/s \n\u001b[?25hRequirement already satisfied: cachetools>=4.0 in /usr/local/lib/python3.7/dist-packages (from streamlit->simpletransformers==0.61.13) (4.2.4)\nRequirement already satisfied: tzlocal in /usr/local/lib/python3.7/dist-packages (from streamlit->simpletransformers==0.61.13) (1.5.1)\nRequirement already satisfied: pillow>=6.2.0 in /usr/local/lib/python3.7/dist-packages (from streamlit->simpletransformers==0.61.13) (7.1.2)\nRequirement already satisfied: jinja2 in /usr/local/lib/python3.7/dist-packages (from altair>=3.2.0->streamlit->simpletransformers==0.61.13) (2.11.3)\nRequirement already satisfied: jsonschema in /usr/local/lib/python3.7/dist-packages (from altair>=3.2.0->streamlit->simpletransformers==0.61.13) (2.6.0)\nRequirement already satisfied: entrypoints in /usr/local/lib/python3.7/dist-packages (from altair>=3.2.0->streamlit->simpletransformers==0.61.13) (0.3)\nRequirement already satisfied: toolz in /usr/local/lib/python3.7/dist-packages (from altair>=3.2.0->streamlit->simpletransformers==0.61.13) (0.11.1)\nRequirement already satisfied: traitlets>=4.3.2 in /usr/local/lib/python3.7/dist-packages (from pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (5.1.0)\nRequirement already satisfied: ipywidgets>=7.0.0 in /usr/local/lib/python3.7/dist-packages (from pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (7.6.5)\nCollecting ipykernel>=5.1.2\n Downloading ipykernel-6.4.1-py3-none-any.whl (124 kB)\n\u001b[K |████████████████████████████████| 124 kB 47.3 MB/s \n\u001b[?25hCollecting ipython<8.0,>=7.23.1\n Downloading ipython-7.28.0-py3-none-any.whl (788 kB)\n\u001b[K |████████████████████████████████| 788 kB 42.5 MB/s \n\u001b[?25hRequirement already satisfied: ipython-genutils in /usr/local/lib/python3.7/dist-packages (from ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.2.0)\nRequirement already satisfied: argcomplete>=1.12.3 in /usr/local/lib/python3.7/dist-packages (from ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (1.12.3)\nRequirement already satisfied: jupyter-client<8.0 in /usr/local/lib/python3.7/dist-packages (from ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (5.3.5)\nRequirement already satisfied: matplotlib-inline<0.2.0,>=0.1.0 in /usr/local/lib/python3.7/dist-packages (from ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.1.3)\nRequirement already satisfied: debugpy<2.0,>=1.0.0 in /usr/local/lib/python3.7/dist-packages (from ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (1.0.0)\nRequirement already satisfied: pexpect>4.3 in /usr/local/lib/python3.7/dist-packages (from ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (4.8.0)\nRequirement already satisfied: pickleshare in /usr/local/lib/python3.7/dist-packages (from ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.7.5)\nRequirement already satisfied: setuptools>=18.5 in /usr/local/lib/python3.7/dist-packages (from ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (57.4.0)\nCollecting prompt-toolkit!=3.0.0,!=3.0.1,<3.1.0,>=2.0.0\n Downloading prompt_toolkit-3.0.20-py3-none-any.whl (370 kB)\n\u001b[K |████████████████████████████████| 370 kB 44.7 MB/s \n\u001b[?25hRequirement already satisfied: backcall in /usr/local/lib/python3.7/dist-packages (from ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.2.0)\nRequirement already satisfied: pygments in /usr/local/lib/python3.7/dist-packages (from ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (2.6.1)\nRequirement already satisfied: decorator in /usr/local/lib/python3.7/dist-packages (from ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (4.4.2)\nRequirement already satisfied: jedi>=0.16 in /usr/local/lib/python3.7/dist-packages (from ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.18.0)\nRequirement already satisfied: jupyterlab-widgets>=1.0.0 in /usr/local/lib/python3.7/dist-packages (from ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (1.0.2)\nRequirement already satisfied: widgetsnbextension~=3.5.0 in /usr/local/lib/python3.7/dist-packages (from ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (3.5.1)\nRequirement already satisfied: nbformat>=4.2.0 in /usr/local/lib/python3.7/dist-packages (from ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (5.1.3)\nRequirement already satisfied: parso<0.9.0,>=0.8.0 in /usr/local/lib/python3.7/dist-packages (from jedi>=0.16->ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.8.2)\nRequirement already satisfied: MarkupSafe>=0.23 in /usr/local/lib/python3.7/dist-packages (from jinja2->altair>=3.2.0->streamlit->simpletransformers==0.61.13) (2.0.1)\nRequirement already satisfied: pyzmq>=13 in /usr/local/lib/python3.7/dist-packages (from jupyter-client<8.0->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (22.3.0)\nRequirement already satisfied: jupyter-core>=4.6.0 in /usr/local/lib/python3.7/dist-packages (from jupyter-client<8.0->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (4.8.1)\nRequirement already satisfied: ptyprocess>=0.5 in /usr/local/lib/python3.7/dist-packages (from pexpect>4.3->ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.7.0)\nRequirement already satisfied: wcwidth in /usr/local/lib/python3.7/dist-packages (from prompt-toolkit!=3.0.0,!=3.0.1,<3.1.0,>=2.0.0->ipython<8.0,>=7.23.1->ipykernel>=5.1.2->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.2.5)\nRequirement already satisfied: notebook>=4.4.1 in /usr/local/lib/python3.7/dist-packages (from widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (5.3.1)\nRequirement already satisfied: Send2Trash in /usr/local/lib/python3.7/dist-packages (from notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (1.8.0)\nRequirement already satisfied: nbconvert in /usr/local/lib/python3.7/dist-packages (from notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (5.6.1)\nRequirement already satisfied: terminado>=0.8.1 in /usr/local/lib/python3.7/dist-packages (from notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.12.1)\nRequirement already satisfied: pandocfilters>=1.4.1 in /usr/local/lib/python3.7/dist-packages (from nbconvert->notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (1.5.0)\nRequirement already satisfied: bleach in /usr/local/lib/python3.7/dist-packages (from nbconvert->notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (4.1.0)\nRequirement already satisfied: mistune<2,>=0.8.1 in /usr/local/lib/python3.7/dist-packages (from nbconvert->notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.8.4)\nRequirement already satisfied: defusedxml in /usr/local/lib/python3.7/dist-packages (from nbconvert->notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.7.1)\nRequirement already satisfied: testpath in /usr/local/lib/python3.7/dist-packages (from nbconvert->notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.5.0)\nRequirement already satisfied: webencodings in /usr/local/lib/python3.7/dist-packages (from bleach->nbconvert->notebook>=4.4.1->widgetsnbextension~=3.5.0->ipywidgets>=7.0.0->pydeck>=0.1.dev5->streamlit->simpletransformers==0.61.13) (0.5.1)\nBuilding wheels for collected packages: subprocess32, pathtools, seqeval, blinker\n Building wheel for subprocess32 (setup.py) ... \u001b[?25l\u001b[?25hdone\n Created wheel for subprocess32: filename=subprocess32-3.5.4-py3-none-any.whl size=6502 sha256=efa28a22a2b7fd34b6548221f8b9ad57f5a923c765a82229aaed72309366f41f\n Stored in directory: /root/.cache/pip/wheels/50/ca/fa/8fca8d246e64f19488d07567547ddec8eb084e8c0d7a59226a\n Building wheel for pathtools (setup.py) ... \u001b[?25l\u001b[?25hdone\n Created wheel for pathtools: filename=pathtools-0.1.2-py3-none-any.whl size=8807 sha256=86603ac541e55e7fa3b422a83aa772722878bb753dc1201bdee5fad78df26c3d\n Stored in directory: /root/.cache/pip/wheels/3e/31/09/fa59cef12cdcfecc627b3d24273699f390e71828921b2cbba2\n Building wheel for seqeval (setup.py) ... \u001b[?25l\u001b[?25hdone\n Created wheel for seqeval: filename=seqeval-1.2.2-py3-none-any.whl size=16181 sha256=35c56849895bc17f3524a520dbc89f65f47efc119ca4d79008f54c6da941ca16\n Stored in directory: /root/.cache/pip/wheels/05/96/ee/7cac4e74f3b19e3158dce26a20a1c86b3533c43ec72a549fd7\n Building wheel for blinker (setup.py) ... \u001b[?25l\u001b[?25hdone\n Created wheel for blinker: filename=blinker-1.4-py3-none-any.whl size=13478 sha256=090fce571dcc5744de99248f9ee8729f7cea68f8b293ec39388c828611d9e980\n Stored in directory: /root/.cache/pip/wheels/22/f5/18/df711b66eb25b21325c132757d4314db9ac5e8dabeaf196eab\nSuccessfully built subprocess32 pathtools seqeval blinker\nInstalling collected packages: prompt-toolkit, ipython, ipykernel, multidict, yarl, smmap, async-timeout, pyyaml, gitdb, fsspec, aiohttp, yaspin, xxhash, watchdog, validators, tokenizers, subprocess32, shortuuid, sentry-sdk, sacremoses, pydeck, pathtools, huggingface-hub, GitPython, docker-pycreds, configparser, blinker, base58, wandb, transformers, tensorboardx, streamlit, seqeval, sentencepiece, datasets, simpletransformers\n Attempting uninstall: prompt-toolkit\n Found existing installation: prompt-toolkit 1.0.18\n Uninstalling prompt-toolkit-1.0.18:\n Successfully uninstalled prompt-toolkit-1.0.18\n Attempting uninstall: ipython\n Found existing installation: ipython 5.5.0\n Uninstalling ipython-5.5.0:\n Successfully uninstalled ipython-5.5.0\n Attempting uninstall: ipykernel\n Found existing installation: ipykernel 4.10.1\n Uninstalling ipykernel-4.10.1:\n Successfully uninstalled ipykernel-4.10.1\n Attempting uninstall: pyyaml\n Found existing installation: PyYAML 3.13\n Uninstalling PyYAML-3.13:\n Successfully uninstalled PyYAML-3.13\n\u001b[31mERROR: pip's dependency resolver does not currently take into account all the packages that are installed. This behaviour is the source of the following dependency conflicts.\njupyter-console 5.2.0 requires prompt-toolkit<2.0.0,>=1.0.0, but you have prompt-toolkit 3.0.20 which is incompatible.\ngoogle-colab 1.0.0 requires ipykernel~=4.10, but you have ipykernel 6.4.1 which is incompatible.\ngoogle-colab 1.0.0 requires ipython~=5.5.0, but you have ipython 7.28.0 which is incompatible.\u001b[0m\nSuccessfully installed GitPython-3.1.24 aiohttp-3.7.4.post0 async-timeout-3.0.1 base58-2.1.0 blinker-1.4 configparser-5.0.2 datasets-1.13.3 docker-pycreds-0.4.0 fsspec-2021.10.1 gitdb-4.0.7 huggingface-hub-0.0.19 ipykernel-6.4.1 ipython-7.28.0 multidict-5.2.0 pathtools-0.1.2 prompt-toolkit-3.0.20 pydeck-0.7.0 pyyaml-6.0 sacremoses-0.0.46 sentencepiece-0.1.96 sentry-sdk-1.4.3 seqeval-1.2.2 shortuuid-1.0.1 simpletransformers-0.61.13 smmap-4.0.0 streamlit-1.0.0 subprocess32-3.5.4 tensorboardx-2.4 tokenizers-0.10.3 transformers-4.11.3 validators-0.18.2 wandb-0.12.4 watchdog-2.1.6 xxhash-2.0.2 yarl-1.7.0 yaspin-2.1.0\n"
],
[
"import nltk\nnltk.download('stopwords')\nnltk.download('punkt')\nfrom nltk.corpus import stopwords\nnltk.download('wordnet')\nstopwords = stopwords.words(\"english\")\nlemmatizer = nltk.stem.WordNetLemmatizer() \n\nfrom nltk.tokenize import RegexpTokenizer, word_tokenize, sent_tokenize\nmain_tokenizer = RegexpTokenizer(r'\\w+',)\nsec_tokenizer = RegexpTokenizer(r'\\S+')\n \nfrom sklearn.externals import joblib\nimport numpy as np\nfrom google.colab import output\nimport urllib\nimport difflib\nimport WikiSearch.wikipedia.wikipedia as wikipedia\nimport pandas as pd\nfrom bs4 import BeautifulSoup",
"[nltk_data] Downloading package stopwords to /root/nltk_data...\n[nltk_data] Unzipping corpora/stopwords.zip.\n[nltk_data] Downloading package punkt to /root/nltk_data...\n[nltk_data] Unzipping tokenizers/punkt.zip.\n[nltk_data] Downloading package wordnet to /root/nltk_data...\n[nltk_data] Unzipping corpora/wordnet.zip.\n"
],
[
"from google_drive_downloader import GoogleDriveDownloader as gdd\n\ngdd.download_file_from_google_drive(file_id='13Nuwm7BV-4RXI9JqjPTDE9rcdupkKqlF',\n dest_path='/Data/AIIJC/aiijc_1578_goodFromTrain_pretrained.model')",
"Downloading 13Nuwm7BV-4RXI9JqjPTDE9rcdupkKqlF into /Data/AIIJC/aiijc_1578_goodFromTrain_pretrained.model... Done.\n"
]
],
[
[
"Функции для предобработки текста",
"_____no_output_____"
]
],
[
[
"def normal_form(word): #Получение нормальной формы слова\n word = word.lower()\n return word\n \ndef clean_html(html): #Очистка html\n soup = BeautifulSoup(BeautifulSoup(html, \"lxml\").text)\n return str(soup.body)\n\ndef get_good_tokens(text): #Выделение ключевых слов\n good_tokens = []\n\n for tokens in tokenizer(text)[1]:\n for token in tokens:\n token = normal_form(token)\n if token not in stopwords:\n good_tokens.append(token)\n return good_tokens\n\ndef tokenizer(text): #Токенизация текста в обработанные и необработанные токены\n raw_tokens = sec_tokenizer.tokenize(text)\n clean_tokens = main_tokenizer.tokenize_sents(raw_tokens)\n \n nClean_tokens = []\n for i in range(len(clean_tokens)):\n nClean_tokens.append([])\n for m in range(len(clean_tokens[i])):\n if normal_form(clean_tokens[i][m]) != 's':\n nClean_tokens[i].append(normal_form(clean_tokens[i][m]))\n\n return (raw_tokens, nClean_tokens)\n \ndef similarity(s1, s2): #Нахождение коэффициента схожести между двумя строками\n normalized1 = s1.lower()\n normalized2 = s2.lower()\n matcher = difflib.SequenceMatcher(None, normalized1, normalized2)\n return matcher.ratio()\n\ndef part_extractor(data,question,step,part_length): #Функция выделения релевантного фрагмента (Текст, вопрос, длинна фрагмента)\n good_tokens = get_good_tokens(question)\n \n tokens = tokenizer(data)\n \n \n for i in range(step-(len(tokens[0]) % step)): #Увеличение количества токенов до кратного длины части\n tokens[0].append('')\n tokens[1].append('')\n \n\n match_counter = 0 #Счетчик точных совпадений токенов\n best_part = '' #Лучшая часть\n max_match_qty = 0 #Максимальное количество совпавших токенов\n \n main_clrTokens = tokens[1]\n main_tokens = tokens[0]\n \n for i in range(0,len(tokens[0])-1,part_length): #Нахождение наиболее релевантной части текста\n tokens = main_tokens[i:i+part_length-1]\n clrTokens = main_clrTokens[i:i+part_length-1]\n\n for good_token in good_tokens:\n if in_tokens(good_token,clrTokens):\n match_counter += 1\n\n if match_counter > max_match_qty:\n max_match_qty = match_counter\n best_part = tokens\n \n \n match_counter = 0\n \n \n\n fin = '' #Восстановление текста\n for i in best_part:\n fin += (i+' ')\n\n \n return fin\n\ndef in_tokens(token,text):\n for i in text:\n for m in i:\n if token == m:\n return True\n return False",
"_____no_output_____"
]
],
[
[
"Загрузка модели и функция для ответов на вопрос",
"_____no_output_____"
]
],
[
[
"model = joblib.load('/Data/AIIJC/aiijc_1578_goodFromTrain_pretrained.model')\n\nmodel.args.max_seq_length = 512\nmodel.args.silent = True",
"_____no_output_____"
],
[
"def answering(question):\n text = question\n\n good_tokens = get_good_tokens(text)\n \n try:\n urls = wikipedia.search(text,results=2)\n except:\n link_1 = '-'\n link_2 = '-'\n\n try:\n link_1 = urls[0]\n except:\n link_1 = '-'\n\n try:\n link_2 = urls[1]\n except:\n link_2 = '-'\n\n #Загрузка статей википедии\n try:\n link_1 = link_1.replace('https://en.wikipedia.org/wiki/','') #Убераем начало ссылки\n link_1 = urllib.parse.unquote(link_1) #Заменяем кривые символы на оригинал\n data_1 = wikipedia.page(link_1,auto_suggest=False).content #Парсим страничку вики\n data_1 = data_1.replace('\\n',' ')\n except:\n pass\n try:\n link_2 = link_2.replace('https://en.wikipedia.org/wiki/','') #Убераем начало ссылки\n link_2 = urllib.parse.unquote(link_2) #Заменяем кривые символы на оригинал\n data_2 = wikipedia.page(link_2,auto_suggest=False).content #Парсим страничку вики\n data_2 = data_2.replace('\\n',' ')\n except:\n pass\n \n\n try: #Поиск релевантного куска длиной 128 токенов с шагом 64 в самой релевантной статье\n context = part_extractor(data_1,question,16,64)\n except:\n pass\n \n try: #Поиск релевантного куска длиной 64 токена с шагом 32 во второй по релевантности статье\n context += ' ' + part_extractor(data_2,question,16,32)\n except:\n pass\n\n try:\n predict = model.predict([{'context': context,'qas': [{'id': 0, 'question': question}]}])[0] #Предсказание ответа\n except:\n predict = [{'answer':['']}]\n predict[0]['answer'][0] = 'empty'\n\n\n if predict[0]['answer'][0] == 'empty':\n try:\n context = part_extractor(data_1,question,16,64)\n predict = model.predict([{'context': context,'qas': [{'id': 0, 'question': question}]}])[0]\n except:\n pass\n\n if predict[0]['answer'][0] == 'empty':\n try:\n context = part_extractor(data_2,question,16,64)\n predict = model.predict([{'context': context,'qas': [{'id': 0, 'question': question}]}])[0]\n except:\n pass\n\n if predict[0]['answer'][0] == 'empty':\n try:\n context = part_extractor(data_1,question,16,128)\n predict = model.predict([{'context': context,'qas': [{'id': 0, 'question': question}]}])[0]\n except:\n pass\n \n if predict[0]['answer'][0] == 'empty':\n try:\n context = part_extractor(data_2,question,16,128)\n predict = model.predict([{'context': context,'qas': [{'id': 0, 'question': question}]}])[0]\n except:\n pass\n \n if predict[0]['answer'][0] == 'empty':\n try:\n context = part_extractor(data_1,question,16,256)\n predict = model.predict([{'context': context,'qas': [{'id': 0, 'question': question}]}])[0]\n except:\n pass\n \n if predict[0]['answer'][0] == 'empty':\n try:\n context = part_extractor(data_2,question,16,256)\n predict = model.predict([{'context': context,'qas': [{'id': 0, 'question': question}]}])[0]\n except:\n pass\n\n return predict[0]['answer'][0]",
"_____no_output_____"
]
],
[
[
"Проверка работоспособности и времени работы функции",
"_____no_output_____"
]
],
[
[
"import time\n\ntime_1 = time.time()\n\nprint(answering(\"What is the name of Trump first daughter?\")) \n\nprint('Время обработки запроса: ' + str(time.time()-time_1))",
"Ivana Marie \"Ivanka\" Trump\nВремя обработки запроса: 1.6687853336334229\n"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7e8852f49fa5d6f8048b8edb5e48bc10ae0c4ef | 21,689 | ipynb | Jupyter Notebook | Week 4/Jupyter Python.ipynb | klxu03/SEAP2020 | 0bcebd4c972be5e6fafcada5eb1f19e8bee671ae | [
"MIT"
] | null | null | null | Week 4/Jupyter Python.ipynb | klxu03/SEAP2020 | 0bcebd4c972be5e6fafcada5eb1f19e8bee671ae | [
"MIT"
] | 1 | 2021-08-23T20:48:21.000Z | 2021-08-23T20:48:21.000Z | Week 4/Jupyter Python.ipynb | klxu03/SEAP2020 | 0bcebd4c972be5e6fafcada5eb1f19e8bee671ae | [
"MIT"
] | 1 | 2020-09-15T01:26:09.000Z | 2020-09-15T01:26:09.000Z | 123.937143 | 17,116 | 0.887593 | [
[
[
"# Importing required classes\nfrom matplotlib import pyplot as plt\nimport numpy as np\n\n# Importing main.py\nfrom main import Main\n\n# Importing helper functions from main\nfrom main import init_weight_array_modified\nfrom main import epitope_distance",
"_____no_output_____"
],
[
"# Importing Python Files from src Directory\n\"\"\" Python File Imports\"\"\"\nfrom src.pngs import PNGS\nfrom src.panel import Panel\nfrom src.blosum import BLOSUM\nfrom src.weights import Weights\nfrom src.epitope_dist import get_epitope_distance\nfrom src.ic50 import IC50\n\n\"\"\" Relative Python Paths \"\"\"\nrel_panel_path = './files/seap2020/136_panel_with_4lts.fa'\nrel_weight_path = './files/seap2020/vrc01_wts.4lts.txt'\nrel_blosum_path = './files/seap2020/BLOSUM62.txt'\nrel_ic50_path = './files/seap2020/vrc01_ic50.txt'\n\n\"\"\" Instantiating Each Class \"\"\"\npanel = Panel(rel_panel_path)\nblosum = BLOSUM(rel_blosum_path)\nweights = Weights(rel_weight_path)\nweight_array_modified = np.zeros(panel.get_seq_length())\nic50 = IC50(rel_ic50_path, (panel.get_number_of_seq() - 2))\n\n# print('5 lowest sequences', ic50.get_lowest_ic50_sequences(5))",
"5 lowest sequences ['001428-2.42' 'JRFL.JB' 'T33-7' 'QH209.14M.A2' 'T280-5']\n"
],
[
"# If I wanted to make modifications to any of the parameters for epitope_distance\n\n# Editing the reference sequence\nreference_sequence = panel.get_reference_sequence(ic50.get_lowest_ic50_sequences(1)) # This panel.get_reference_sequence() function has one parameter, a numpy array of all the of the sequence names you want to read\n\nblosum_dict = Main.get_blosum_dict()\nic50_weights = Main.log_base_10(Main.get_ic50_weights()) # Get the ic50 weight array, and then log it by 10",
"_____no_output_____"
],
[
"# 2D matrix containing epitope distance and its respective IC50 concentration\ndata_2d = epitope_distance(reference_sequence, blosum_dict, ic50_weights)",
"_____no_output_____"
],
[
"# Epitope Distances\nx = data_2d[0]\n# IC50 Concentrations\ny = data_2d[1]\n\n# Making the Scatter Plot\nplt.scatter(x, y)\n\n# Adding Title\nplt.title(\"Epitope Distance vs IC50 Concentration\")\n\n# Adding Labels\nplt.xlabel(\"Epitope Distance\")\nplt.ylabel(\"IC50 Concentration\")\n\nplt.show()",
"_____no_output_____"
],
[
"from scipy.stats import spearmanr\n\n# Calculate Spearman's correlation\ncoef, p = spearmanr(x, y)\nprint(\"Spearman's correlation coefficient: %.3f\" % coef) # Try to improve this value\nprint(\"P Value: %.3f\" % p)",
"Spearman's correlation coefficient: -0.055\nP Value: 0.524\n"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e892e82784a8ee62ac28f51211b0d9967df830 | 93,490 | ipynb | Jupyter Notebook | Tutorials/CNTK_101_LogisticRegression.ipynb | shyamalschandra/CNTK | 0e7a6cd4cc174eab28eaf2ffc660c6380b9e4e2d | [
"MIT"
] | 1 | 2021-11-29T14:56:26.000Z | 2021-11-29T14:56:26.000Z | Tutorials/CNTK_101_LogisticRegression.ipynb | shyamalschandra/CNTK | 0e7a6cd4cc174eab28eaf2ffc660c6380b9e4e2d | [
"MIT"
] | null | null | null | Tutorials/CNTK_101_LogisticRegression.ipynb | shyamalschandra/CNTK | 0e7a6cd4cc174eab28eaf2ffc660c6380b9e4e2d | [
"MIT"
] | null | null | null | 111.830144 | 20,516 | 0.834314 | [
[
[
"from IPython.display import Image",
"_____no_output_____"
]
],
[
[
"# CNTK 101: Logistic Regression and ML Primer\n\nThis tutorial is targeted to individuals who are new to CNTK and to machine learning. In this tutorial, you will train a simple yet powerful machine learning model that is widely used in industry for a variety of applications. The model trained below scales to massive data sets in the most expeditious manner by harnessing computational scalability leveraging the computational resources you may have (one or more CPU cores, one or more GPUs, a cluster of CPUs or a cluster of GPUs), transparently via the CNTK library.\n\nThe following notebook uses Python APIs. If you are looking for this example in BrainScript, please look [here](https://github.com/Microsoft/CNTK/tree/release/2.6/Tutorials/HelloWorld-LogisticRegression). \n\n## Introduction\n\n**Problem**:\nA cancer hospital has provided data and wants us to determine if a patient has a fatal [malignant](https://en.wikipedia.org/wiki/Malignancy) cancer vs. a benign growth. This is known as a classification problem. To help classify each patient, we are given their age and the size of the tumor. Intuitively, one can imagine that younger patients and/or patients with small tumors are less likely to have a malignant cancer. The data set simulates this application: each observation is a patient represented as a dot (in the plot below), where red indicates malignant and blue indicates benign. Note: This is a toy example for learning; in real life many features from different tests/examination sources and the expertise of doctors would play into the diagnosis/treatment decision for a patient. ",
"_____no_output_____"
]
],
[
[
"# Figure 1\nImage(url=\"https://www.cntk.ai/jup/cancer_data_plot.jpg\", width=400, height=400)",
"_____no_output_____"
]
],
[
[
"**Goal**:\nOur goal is to learn a classifier that can automatically label any patient into either the benign or malignant categories given two features (age and tumor size). In this tutorial, we will create a linear classifier, a fundamental building-block in deep networks.",
"_____no_output_____"
]
],
[
[
"# Figure 2\nImage(url= \"https://www.cntk.ai/jup/cancer_classify_plot.jpg\", width=400, height=400)",
"_____no_output_____"
]
],
[
[
"In the figure above, the green line represents the model learned from the data and separates the blue dots from the red dots. In this tutorial, we will walk you through the steps to learn the green line. Note: this classifier does make mistakes, where a couple of blue dots are on the wrong side of the green line. However, there are ways to fix this and we will look into some of the techniques in later tutorials. \n\n**Approach**: \nAny learning algorithm typically has five stages. These are Data reading, Data preprocessing, Creating a model, Learning the model parameters, and Evaluating the model (a.k.a. testing/prediction). \n\n>1. Data reading: We generate simulated data sets with each sample having two features (plotted below) indicative of the age and tumor size.\n>2. Data preprocessing: Often, the individual features such as size or age need to be scaled. Typically, one would scale the data between 0 and 1. To keep things simple, we are not doing any scaling in this tutorial (for details look here: [feature scaling](https://en.wikipedia.org/wiki/Feature_scaling).\n>3. Model creation: We introduce a basic linear model in this tutorial. \n>4. Learning the model: This is also known as training. While fitting a linear model can be done in a variety of ways ([linear regression](https://en.wikipedia.org/wiki/Linear_regression), in CNTK we use Stochastic Gradient Descent a.k.a. [SGD](https://en.wikipedia.org/wiki/Stochastic_gradient_descent).\n>5. Evaluation: This is also known as testing, where one evaluates the model on data sets with known labels (a.k.a. ground-truth) that were never used for training. This allows us to assess how a model would perform in real-world (previously unseen) observations.\n\n## Logistic Regression\n[Logistic regression](https://en.wikipedia.org/wiki/Logistic_regression) is a fundamental machine learning technique that uses a linear weighted combination of features and generates the probability of predicting different classes. In our case, the classifier will generate a probability in [0,1] which can then be compared to a threshold (such as 0.5) to produce a binary label (0 or 1). However, the method shown can easily be extended to multiple classes. \n\n[softmax]: https://en.wikipedia.org/wiki/Multinomial_logistic_regression",
"_____no_output_____"
]
],
[
[
"# Figure 3\nImage(url= \"https://www.cntk.ai/jup/logistic_neuron.jpg\", width=300, height=200)",
"_____no_output_____"
]
],
[
[
"In the above figure, contributions from different input features are linearly weighted and aggregated. The resulting sum is mapped to a (0, 1) range via a [sigmoid]( https://en.wikipedia.org/wiki/Sigmoid_function) function. For classifiers with more than two output labels, one can use a [softmax](https://en.wikipedia.org/wiki/Softmax_function) function.",
"_____no_output_____"
]
],
[
[
"# Import the relevant components\nfrom __future__ import print_function\nimport numpy as np\nimport sys\nimport os\n\nimport cntk as C\nimport cntk.tests.test_utils\ncntk.tests.test_utils.set_device_from_pytest_env() # (only needed for our build system)\nC.cntk_py.set_fixed_random_seed(1) # fix the random seed so that LR examples are repeatable",
"_____no_output_____"
]
],
[
[
"## Data Generation\nLet us generate some synthetic data emulating the cancer example using the `numpy` library. We have two input features (represented in two-dimensions) and two output classes (benign/blue or malignant/red). \n\nIn our example, each observation (a single 2-tuple of features - age and size) in the training data has a label (blue or red). Because we have two output labels, we call this a binary classification task. ",
"_____no_output_____"
]
],
[
[
"# Define the network\ninput_dim = 2\nnum_output_classes = 2",
"_____no_output_____"
]
],
[
[
"### Input and Labels\n\nIn this tutorial we are generating synthetic data using the `numpy` library. In real-world problems, one would use a [reader](https://docs.microsoft.com/en-us/cognitive-toolkit/brainscript-and-python---understanding-and-extending-readers), that would read feature values (`features`: *age* and *tumor size*) corresponding to each observation (patient). The simulated *age* variable is scaled down to have a similar range to that of the other variable. This is a key aspect of data pre-processing that we will learn more about in later tutorials. Note: in general, observations and labels can reside in higher dimensional spaces (when more features or classifications are available) and are then represented as [tensors](https://en.wikipedia.org/wiki/Tensor) in CNTK. More advanced tutorials introduce the handling of high dimensional data. ",
"_____no_output_____"
]
],
[
[
"# Ensure that we always get the same results\nnp.random.seed(0)\n\n# Helper function to generate a random data sample\ndef generate_random_data_sample(sample_size, feature_dim, num_classes):\n # Create synthetic data using NumPy. \n Y = np.random.randint(size=(sample_size, 1), low=0, high=num_classes)\n\n # Make sure that the data is separable \n X = (np.random.randn(sample_size, feature_dim)+3) * (Y+1)\n \n # Specify the data type to match the input variable used later in the tutorial \n # (default type is double)\n X = X.astype(np.float32) \n \n # convert class 0 into the vector \"1 0 0\", \n # class 1 into the vector \"0 1 0\", ...\n class_ind = [Y==class_number for class_number in range(num_classes)]\n Y = np.asarray(np.hstack(class_ind), dtype=np.float32)\n return X, Y",
"_____no_output_____"
],
[
"# Create the input variables denoting the features and the label data. Note: the input \n# does not need additional info on the number of observations (Samples) since CNTK creates only \n# the network topology first \nmysamplesize = 32\nfeatures, labels = generate_random_data_sample(mysamplesize, input_dim, num_output_classes)",
"_____no_output_____"
]
],
[
[
"Let us visualize the input data.\n\n**Note**: If the import of `matplotlib.pyplot` fails, please run `conda install matplotlib`, which will fix the `pyplot` version dependencies. If you are on a python environment different from Anaconda, then use `pip install matplotlib`.",
"_____no_output_____"
]
],
[
[
"# Plot the data \nimport matplotlib.pyplot as plt\n%matplotlib inline\n\n# let 0 represent malignant/red and 1 represent benign/blue \ncolors = ['r' if label == 0 else 'b' for label in labels[:,0]]\n\nplt.scatter(features[:,0], features[:,1], c=colors)\nplt.xlabel(\"Age (scaled)\")\nplt.ylabel(\"Tumor size (in cm)\")\nplt.show()",
"_____no_output_____"
]
],
[
[
"## Model Creation\n\nA logistic regression (a.k.a. LR) network is a simple building block, but has powered many ML \napplications in the past decade. LR is a simple linear model that takes as input a vector of numbers describing the properties of what we are classifying (also known as a feature vector, $\\bf{x}$, the blue nodes in the figure below) and emits the *evidence* ($z$) (output of the green node, also known as \"activation\"). Each feature in the input layer is connected to an output node by a corresponding weight $w$ (indicated by the black lines of varying thickness). ",
"_____no_output_____"
]
],
[
[
"# Figure 4\nImage(url= \"https://www.cntk.ai/jup/logistic_neuron2.jpg\", width=300, height=200)",
"_____no_output_____"
]
],
[
[
"The first step is to compute the evidence for an observation. \n\n$$z = \\sum_{i=1}^n w_i \\times x_i + b = \\textbf{w} \\cdot \\textbf{x} + b$$ \n\nwhere $\\bf{w}$ is the weight vector of length $n$ and $b$ is known as the [bias](https://www.quora.com/What-does-the-bias-term-represent-in-logistic-regression) term. Note: we use **bold** notation to denote vectors. \n\nThe computed evidence is mapped to a (0, 1) range using a `sigmoid` (when the outcome can be in one of two possible classes) or a `softmax` function (when the outcome can be in one of more than two possible classes).\n\nNetwork input and output: \n- **input** variable (a key CNTK concept): \n>An **input** variable is a user-code-facing container where user-provided code fills in different observations (a data point or sample of data points, equivalent to (age, size) tuples in our example) as inputs to the model function during model learning (a.k.a.training) and model evaluation (a.k.a. testing). Thus, the shape of the `input` must match the shape of the data that will be provided. For example, if each data point was a grayscale image of height 10 pixels and width 5 pixels, the input feature would be a vector of 50 floating-point values representing the intensity of each of the 50 pixels, and could be written as `C.input_variable(10*5, np.float32)`. Similarly, in our example the dimensions are age and tumor size, thus `input_dim` = 2. More on data and their dimensions to appear in separate tutorials. ",
"_____no_output_____"
]
],
[
[
"feature = C.input_variable(input_dim, np.float32)",
"_____no_output_____"
]
],
[
[
"## Network setup\n\nThe `linear_layer` function is a straightforward implementation of the equation above. We perform two operations:\n0. multiply the weights ($\\bf{w}$) with the features ($\\bf{x}$) using the CNTK `times` operator,\n1. add the bias term ($b$).\n\nThese CNTK operations are optimized for execution on the available hardware and the implementation hides the complexity away from the user. ",
"_____no_output_____"
]
],
[
[
"# Define a dictionary to store the model parameters\nmydict = {}\n\ndef linear_layer(input_var, output_dim):\n \n input_dim = input_var.shape[0]\n weight_param = C.parameter(shape=(input_dim, output_dim))\n bias_param = C.parameter(shape=(output_dim))\n \n mydict['w'], mydict['b'] = weight_param, bias_param\n\n return C.times(input_var, weight_param) + bias_param",
"_____no_output_____"
]
],
[
[
"`z` will be used to represent the output of the network.",
"_____no_output_____"
]
],
[
[
"output_dim = num_output_classes\nz = linear_layer(feature, output_dim)",
"_____no_output_____"
]
],
[
[
"### Learning model parameters\n\nNow that the network is set up, we would like to learn the parameters $\\bf w$ and $b$ for our simple linear layer. To do so we convert, the computed evidence ($z$) into a set of predicted probabilities ($\\textbf p$) using a `softmax` function.\n\n$$ \\textbf{p} = \\mathrm{softmax}(z)$$ \n\nThe `softmax` is an activation function that normalizes the accumulated evidence into a probability distribution over the classes (Details of [softmax](https://www.cntk.ai/pythondocs/cntk.ops.html#cntk.ops.softmax)). Other choices of activation function can be [here](https://cntk.ai/pythondocs/cntk.layers.layers.html#cntk.layers.layers.Activation).",
"_____no_output_____"
],
[
"## Training\nThe output of the `softmax` is the probabilities of an observation belonging each of the respective classes. For training the classifier, we need to determine what behavior the model needs to mimic. In other words, we want the generated probabilities to be as close as possible to the observed labels. We can accomplish this by minimizing the difference between our output and the ground-truth labels. This difference is calculated by the *cost* or *loss* function.\n\n[Cross entropy](http://cntk.ai/pythondocs/cntk.ops.html#cntk.ops.cross_entropy_with_softmax) is a popular loss function. It is defined as:\n\n$$ H(p) = - \\sum_{j=1}^{| \\textbf y |} y_j \\log (p_j) $$ \n\nwhere $p$ is our predicted probability from `softmax` function and $y$ is the ground-truth label, provided with the training data. In the two-class example, the `label` variable has two dimensions (equal to the `num_output_classes` or $| \\textbf y |$). Generally speaking, the label variable will have $| \\textbf y |$ elements with 0 everywhere except at the index of the true class of the data point, where it will be 1. Understanding the [details](http://colah.github.io/posts/2015-09-Visual-Information/) of the cross-entropy function is highly recommended.",
"_____no_output_____"
]
],
[
[
"label = C.input_variable(num_output_classes, np.float32)\nloss = C.cross_entropy_with_softmax(z, label)",
"_____no_output_____"
]
],
[
[
"### Evaluation\n\nIn order to evaluate the classification, we can compute the [classification_error](https://www.cntk.ai/pythondocs/cntk.metrics.html#cntk.metrics.classification_error), which is 0 if our model was correct (it assigned the true label the most probability), otherwise 1.",
"_____no_output_____"
]
],
[
[
"eval_error = C.classification_error(z, label)",
"_____no_output_____"
]
],
[
[
"### Configure training\n\nThe trainer strives to minimize the `loss` function using an optimization technique. In this tutorial, we will use [Stochastic Gradient Descent](https://en.wikipedia.org/wiki/Stochastic_gradient_descent) (`sgd`), one of the most popular techniques. Typically, one starts with random initialization of the model parameters (the weights and biases, in our case). For each observation, the `sgd` optimizer can calculate the `loss` or error between the predicted label and the corresponding ground-truth label, and apply [gradient descent](http://www.statisticsviews.com/details/feature/5722691/Getting-to-the-Bottom-of-Regression-with-Gradient-Descent.html) to generate a new set of model parameters after each observation. \n\nThe aforementioned process of updating all parameters after each observation is attractive because it does not require the entire data set (all observations) to be loaded in memory and also computes the gradient over fewer datapoints, thus allowing for training on large data sets. However, the updates generated using a single observation at a time can vary wildly between iterations. An intermediate ground is to load a small set of observations into the model and use an average of the `loss` or error from that set to update the model parameters. This subset is called a *minibatch*.\n\nWith minibatches we often sample observations from the larger training dataset. We repeat the process of updating the model parameters using different combinations of training samples, and over a period of time minimize the `loss` (and the error). When the incremental error rates are no longer changing significantly, or after a preset maximum number of minibatches have been processed, we claim that our model is trained.\n\nOne of the key parameters of [optimization](https://en.wikipedia.org/wiki/Category:Convex_optimization) is the `learning_rate`. For now, we can think of it as a scaling factor that modulates how much we change the parameters in any iteration. We will cover more details in later tutorials. \nWith this information, we are ready to create our trainer. ",
"_____no_output_____"
]
],
[
[
"# Instantiate the trainer object to drive the model training\nlearning_rate = 0.5\nlr_schedule = C.learning_rate_schedule(learning_rate, C.UnitType.minibatch) \nlearner = C.sgd(z.parameters, lr_schedule)\ntrainer = C.Trainer(z, (loss, eval_error), [learner])",
"_____no_output_____"
]
],
[
[
"First, let us create some helper functions that will be needed to visualize different functions associated with training. Note: these convenience functions are for understanding what goes on under the hood.",
"_____no_output_____"
]
],
[
[
"# Define a utility function to compute the moving average.\n# A more efficient implementation is possible with np.cumsum() function\ndef moving_average(a, w=10):\n if len(a) < w: \n return a[:] \n return [val if idx < w else sum(a[(idx-w):idx])/w for idx, val in enumerate(a)]\n\n\n# Define a utility that prints the training progress\ndef print_training_progress(trainer, mb, frequency, verbose=1):\n training_loss, eval_error = \"NA\", \"NA\"\n\n if mb % frequency == 0:\n training_loss = trainer.previous_minibatch_loss_average\n eval_error = trainer.previous_minibatch_evaluation_average\n if verbose: \n print (\"Minibatch: {0}, Loss: {1:.4f}, Error: {2:.2f}\".format(mb, training_loss, eval_error))\n \n return mb, training_loss, eval_error",
"_____no_output_____"
]
],
[
[
"### Run the trainer\n\nWe are now ready to train our Logistic Regression model. We want to decide what data we need to feed into the training engine.\n\nIn this example, each iteration of the optimizer will work on 25 samples (25 dots w.r.t. the plot above) a.k.a. `minibatch_size`. We would like to train on 20000 observations. If the number of samples in the data is only 10000, the trainer will make 2 passes through the data. This is represented by `num_minibatches_to_train`. Note: in a real world scenario, we would be given a certain amount of labeled data (in the context of this example, (age, size) observations and their labels (benign / malignant)). We would use a large number of observations for training, say 70%, and set aside the remainder for the evaluation of the trained model.\n\nWith these parameters we can proceed with training our simple feedforward network.",
"_____no_output_____"
]
],
[
[
"# Initialize the parameters for the trainer\nminibatch_size = 25\nnum_samples_to_train = 20000\nnum_minibatches_to_train = int(num_samples_to_train / minibatch_size)",
"_____no_output_____"
],
[
"from collections import defaultdict\n\n# Run the trainer and perform model training\ntraining_progress_output_freq = 50\nplotdata = defaultdict(list)\n\nfor i in range(0, num_minibatches_to_train):\n features, labels = generate_random_data_sample(minibatch_size, input_dim, num_output_classes)\n \n # Assign the minibatch data to the input variables and train the model on the minibatch\n trainer.train_minibatch({feature : features, label : labels})\n batchsize, loss, error = print_training_progress(trainer, i, \n training_progress_output_freq, verbose=1)\n \n if not (loss == \"NA\" or error ==\"NA\"):\n plotdata[\"batchsize\"].append(batchsize)\n plotdata[\"loss\"].append(loss)\n plotdata[\"error\"].append(error)",
"Minibatch: 0, Loss: 0.6931, Error: 0.32\nMinibatch: 50, Loss: 1.9350, Error: 0.36\nMinibatch: 100, Loss: 1.0764, Error: 0.32\nMinibatch: 150, Loss: 0.4856, Error: 0.20\nMinibatch: 200, Loss: 0.1319, Error: 0.08\nMinibatch: 250, Loss: 0.1330, Error: 0.08\nMinibatch: 300, Loss: 0.1012, Error: 0.04\nMinibatch: 350, Loss: 0.1091, Error: 0.04\nMinibatch: 400, Loss: 0.3094, Error: 0.08\nMinibatch: 450, Loss: 0.3230, Error: 0.12\nMinibatch: 500, Loss: 0.3986, Error: 0.20\nMinibatch: 550, Loss: 0.6744, Error: 0.24\nMinibatch: 600, Loss: 0.3004, Error: 0.12\nMinibatch: 650, Loss: 0.1676, Error: 0.12\nMinibatch: 700, Loss: 0.2777, Error: 0.12\nMinibatch: 750, Loss: 0.2311, Error: 0.04\n"
],
[
"# Compute the moving average loss to smooth out the noise in SGD\nplotdata[\"avgloss\"] = moving_average(plotdata[\"loss\"])\nplotdata[\"avgerror\"] = moving_average(plotdata[\"error\"])\n\n# Plot the training loss and the training error\nimport matplotlib.pyplot as plt\n\nplt.figure(1)\nplt.subplot(211)\nplt.plot(plotdata[\"batchsize\"], plotdata[\"avgloss\"], 'b--')\nplt.xlabel('Minibatch number')\nplt.ylabel('Loss')\nplt.title('Minibatch run vs. Training loss')\n\nplt.show()\n\nplt.subplot(212)\nplt.plot(plotdata[\"batchsize\"], plotdata[\"avgerror\"], 'r--')\nplt.xlabel('Minibatch number')\nplt.ylabel('Label Prediction Error')\nplt.title('Minibatch run vs. Label Prediction Error')\nplt.show()",
"_____no_output_____"
]
],
[
[
"### Run evaluation / Testing \n\nNow that we have trained the network, let us evaluate the trained network on data that hasn't been used for training. This is called **testing**. Let us create some new data and evaluate the average error and loss on this set. This is done using `trainer.test_minibatch`. Note the error on this previously unseen data is comparable to the training error. This is a **key** check. Should the error be larger than the training error by a large margin, it indicates that the trained model will not perform well on data that it has not seen during training. This is known as [overfitting](https://en.wikipedia.org/wiki/Overfitting). There are several ways to address overfitting that are beyond the scope of this tutorial, but the Cognitive Toolkit provides the necessary components to address overfitting.\n\nNote: we are testing on a single minibatch for illustrative purposes. In practice, one runs several minibatches of test data and reports the average. \n\n**Question** Why is this suggested? Try plotting the test error over several set of generated data sample and plot using plotting functions used for training. Do you see a pattern?",
"_____no_output_____"
]
],
[
[
"# Run the trained model on a newly generated dataset\ntest_minibatch_size = 25\nfeatures, labels = generate_random_data_sample(test_minibatch_size, input_dim, num_output_classes)\n\ntrainer.test_minibatch({feature : features, label : labels})",
"_____no_output_____"
]
],
[
[
"### Checking prediction / evaluation \nFor evaluation, we softmax the output of the network into a probability distribution over the two classes, the probability of each observation being malignant or benign. ",
"_____no_output_____"
]
],
[
[
"out = C.softmax(z)\nresult = out.eval({feature : features})",
"_____no_output_____"
]
],
[
[
"Let us compare the ground-truth label with the predictions. They should be in agreement.\n\n**Question:** \n- How many predictions were mislabeled? Can you change the code below to identify which observations were misclassified? ",
"_____no_output_____"
]
],
[
[
"print(\"Label :\", [np.argmax(label) for label in labels])\nprint(\"Predicted:\", [np.argmax(x) for x in result])",
"Label : [1, 0, 0, 1, 1, 1, 0, 1, 1, 0, 1, 1, 1, 0, 1, 0, 1, 1, 0, 0, 1, 0, 0, 0, 1]\nPredicted: [1, 0, 0, 0, 0, 0, 0, 1, 1, 0, 1, 1, 1, 0, 1, 0, 1, 1, 0, 0, 1, 0, 0, 0, 1]\n"
]
],
[
[
"### Visualization\nIt is desirable to visualize the results. In this example, the data can be conveniently plotted using two spatial dimensions for the input (patient age on the x-axis and tumor size on the y-axis), and a color dimension for the output (red for malignant and blue for benign). For data with higher dimensions, visualization can be challenging. There are advanced dimensionality reduction techniques, such as [t-sne](https://en.wikipedia.org/wiki/T-distributed_stochastic_neighbor_embedding) that allow for such visualizations.",
"_____no_output_____"
]
],
[
[
"# Model parameters\nprint(mydict['b'].value)\n\nbias_vector = mydict['b'].value\nweight_matrix = mydict['w'].value\n\n# Plot the data \nimport matplotlib.pyplot as plt\n\n# let 0 represent malignant/red, and 1 represent benign/blue\ncolors = ['r' if label == 0 else 'b' for label in labels[:,0]]\nplt.scatter(features[:,0], features[:,1], c=colors)\nplt.plot([0, bias_vector[0]/weight_matrix[0][1]], \n [ bias_vector[1]/weight_matrix[0][0], 0], c = 'g', lw = 3)\nplt.xlabel(\"Patient age (scaled)\")\nplt.ylabel(\"Tumor size (in cm)\")\nplt.show()",
"[ 8.00007153 -8.00006485]\n"
]
],
[
[
"**Exploration Suggestions** \n- Try exploring how the classifier behaves with different data distributions - e.g., changing the `minibatch_size` parameter from 25 to 64. Why is the error increasing?\n- Try exploring different activation functions\n- Try exploring different learners \n- You can explore training a [multiclass logistic regression](https://en.wikipedia.org/wiki/Multinomial_logistic_regression) classifier. ",
"_____no_output_____"
],
[
"### Appendix\n\nMany of the terminologies in machine learning literature can be difficult to map to. Each toolkit / book / paper have their way of referring to the synonymous terminologies. Here are a few CNTK terminologies and their equivalent terms in literature:\n\n- a sequence (in CNTK) is also referred to as an instance\n- a sample (in CNTK) is also referred to as a feature\n- input stream(s) (in CNTK) is also referred to as feature column or a feature\n- the criterion (in CNTK) is also referred to as the loss\n- the evaluation error (in CNTK) is also referred to as a metric",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
]
] |
e7e89508cc0dcc60cf45c133dccc51fe760af120 | 343,150 | ipynb | Jupyter Notebook | notebooks/ch03_first_ml.ipynb | ychoi-kr/pytorch_book_info | c4f4ce4fcceb1012c01a2ac50bf4737647e19840 | [
"Apache-2.0"
] | null | null | null | notebooks/ch03_first_ml.ipynb | ychoi-kr/pytorch_book_info | c4f4ce4fcceb1012c01a2ac50bf4737647e19840 | [
"Apache-2.0"
] | null | null | null | notebooks/ch03_first_ml.ipynb | ychoi-kr/pytorch_book_info | c4f4ce4fcceb1012c01a2ac50bf4737647e19840 | [
"Apache-2.0"
] | null | null | null | 192.348655 | 170,512 | 0.897846 | [
[
[
"# 3장\t처음 시작하는 머신러닝",
"_____no_output_____"
]
],
[
[
"# 必要ライブラリの導入\n\n!pip install japanize_matplotlib | tail -n 1\n!pip install torchviz | tail -n 1",
"'tail'은(는) 내부 또는 외부 명령, 실행할 수 있는 프로그램, 또는\n배치 파일이 아닙니다.\n'tail'은(는) 내부 또는 외부 명령, 실행할 수 있는 프로그램, 또는\n배치 파일이 아닙니다.\n"
],
[
"# 必要ライブラリのインポート\n\n%matplotlib inline\nimport numpy as np\nimport matplotlib.pyplot as plt\n#import japanize_matplotlib\nfrom IPython.display import display",
"_____no_output_____"
],
[
"# PyTorch関連ライブラリ\nimport torch\nfrom torchviz import make_dot",
"_____no_output_____"
],
[
"# デフォルトフォントサイズ変更\nplt.rcParams['font.size'] = 14\n\n# デフォルトグラフサイズ変更\nplt.rcParams['figure.figsize'] = (6,6)\n\n# デフォルトで方眼表示ON\nplt.rcParams['axes.grid'] = True\n\n# numpyの浮動小数点の表示精度\nnp.set_printoptions(suppress=True, precision=4)",
"_____no_output_____"
],
[
"# warning表示off\nimport warnings\nwarnings.simplefilter('ignore')",
"_____no_output_____"
]
],
[
[
"## 3.4\t경사 하강법의 구현 방법",
"_____no_output_____"
]
],
[
[
"def L(u, v):\n return 3 * u**2 + 3 * v**2 - u*v + 7*u - 7*v + 10\ndef Lu(u, v):\n return 6* u - v + 7\ndef Lv(u, v):\n return 6* v - u - 7\n\nu = np.linspace(-5, 5, 501)\nv = np.linspace(-5, 5, 501)\nU, V = np.meshgrid(u, v)\nZ = L(U, V)",
"_____no_output_____"
],
[
"# 勾配降下法のシミュレーション\nW = np.array([4.0, 4.0])\nW1 = [W[0]]\nW2 = [W[1]]\nN = 21\nalpha = 0.05\nfor i in range(N):\n W = W - alpha *np.array([Lu(W[0], W[1]), Lv(W[0], W[1])])\n W1.append(W[0])\n W2.append(W[1])",
"_____no_output_____"
],
[
"n_loop=11\n\nWW1 = np.array(W1[:n_loop])\nWW2 = np.array(W2[:n_loop])\nZZ = L(WW1, WW2)\nfig = plt.figure(figsize=(8,8))\nax = plt.axes(projection='3d')\nax.set_zlim(0,250)\nax.set_xlabel('W')\nax.set_ylabel('B')\nax.set_zlabel('loss')\nax.view_init(50, 240)\nax.xaxis._axinfo[\"grid\"]['linewidth'] = 2.\nax.yaxis._axinfo[\"grid\"]['linewidth'] = 2.\nax.zaxis._axinfo[\"grid\"]['linewidth'] = 2.\nax.contour3D(U, V, Z, 100, cmap='Blues', alpha=0.7)\nax.plot3D(WW1, WW2, ZZ, 'o-', c='k', alpha=1, markersize=7)\nplt.show()\nfig.savefig('fig03-06.tif', format='tif', dpi=300)",
"_____no_output_____"
]
],
[
[
"## 3.5 データ前処理\n5人の人の身長と体重のデータを使う。 \n1次関数で身長から体重を予測する場合、最適な直線を求めることが目的。",
"_____no_output_____"
]
],
[
[
"# サンプルデータの宣言\nsampleData1 = np.array([\n [166, 58.7],\n [176.0, 75.7],\n [171.0, 62.1],\n [173.0, 70.4],\n [169.0,60.1]\n])\nprint(sampleData1)",
"[[166. 58.7]\n [176. 75.7]\n [171. 62.1]\n [173. 70.4]\n [169. 60.1]]\n"
],
[
"# 機械学習モデルで扱うため、身長だけを抜き出した変数xと\n# 体重だけを抜き出した変数yをセットする\n\nx = sampleData1[:,0]\ny = sampleData1[:,1]",
"_____no_output_____"
],
[
"import matplotlib\n# '맑은 고딕'으로 폰트 설정\nmatplotlib.rcParams['font.family'] = 'Malgun Gothic'\n# 한글 폰트에서 마이너스(-) 폰트가 깨지는 것을 방지\nmatplotlib.rcParams['axes.unicode_minus'] = False",
"_____no_output_____"
],
[
"# 散布図表示で状況の確認\n\nfig1 = plt.gcf()\nplt.scatter(x, y, c='k', s=50)\nplt.xlabel('$x$: 신장(cm) ')\nplt.ylabel('$y$: 체중(kg)')\nplt.title('신장과 체중의 관계')\nplt.show()\nplt.draw()\nfig1.savefig('ex03-03.tif', format='tif', dpi=300)",
"_____no_output_____"
]
],
[
[
"### 座標系の変換\n機械学習モデルでは、データは0に近い値を持つことが望ましい。 \nそこで、x, y ともに平均値が0になるように平行移動し、新しい座標系をX, Yとする。",
"_____no_output_____"
]
],
[
[
"X = x - x.mean()\nY = y - y.mean()",
"_____no_output_____"
],
[
"# 散布図表示で結果の確認\n\nfig1 = plt.gcf()\nplt.scatter(X, Y, c='k', s=50)\nplt.xlabel('$X$')\nplt.ylabel('$Y$')\nplt.title('데이터 가공 후 신장과 체중의 관계')\nplt.show()\nplt.draw()\nfig1.savefig('ex03-04.tif', format='tif', dpi=300)",
"_____no_output_____"
]
],
[
[
"## 3.6 予測計算",
"_____no_output_____"
]
],
[
[
"# XとYをテンソル変数化する\n\nX = torch.tensor(X).float()\nY = torch.tensor(Y).float()",
"_____no_output_____"
],
[
"# 結果確認\n\nprint(X)\nprint(Y)",
"tensor([-5., 5., 0., 2., -2.])\ntensor([-6.7000, 10.3000, -3.3000, 5.0000, -5.3000])\n"
],
[
"# 重み変数の定義\n# WとBは勾配計算をするので、requires_grad=Trueとする\n\nW = torch.tensor(1.0, requires_grad=True).float()\nB = torch.tensor(1.0, requires_grad=True).float()",
"_____no_output_____"
],
[
"# 予測関数は一次関数\n\ndef pred(X):\n return W * X + B",
"_____no_output_____"
],
[
"# 予測値の計算\n\nYp = pred(X)",
"_____no_output_____"
],
[
"# 結果標示\n\nprint(Yp)",
"tensor([-4., 6., 1., 3., -1.], grad_fn=<AddBackward0>)\n"
],
[
"# 予測値の計算グラフ可視化\n\nparams = {'W': W, 'B': B}\ng = make_dot(Yp, params=params)\ndisplay(g)\ng.render('ex03-08', format='tif')\n!dot -Ttif -Gdpi=300 ex03-08 -o ex03-08_large.tif",
"_____no_output_____"
]
],
[
[
"## 3.7 損失計算",
"_____no_output_____"
]
],
[
[
"# 損失関数は誤差二乗平均\n\ndef mse(Yp, Y):\n loss = ((Yp - Y) ** 2).mean()\n return loss",
"_____no_output_____"
],
[
"# 損失計算\n\nloss = mse(Yp, Y)",
"_____no_output_____"
],
[
"# 結果標示\n\nprint(loss)",
"tensor(13.3520, grad_fn=<MeanBackward0>)\n"
],
[
"# 損失の計算グラフ可視化\n\nparams = {'W': W, 'B': B}\ng = make_dot(loss, params=params)\ndisplay(g)\ng.render('ex03-11', format='tif')\n!dot -Ttif -Gdpi=300 ex03-11 -o ex03-11_large.tif",
"_____no_output_____"
]
],
[
[
"## 3.8 勾配計算",
"_____no_output_____"
]
],
[
[
"# 勾配計算\n\nloss.backward()",
"_____no_output_____"
],
[
"# 勾配値確認\n\nprint(W.grad)\nprint(B.grad)",
"tensor(-19.0400)\ntensor(2.0000)\n"
]
],
[
[
"## 3.9 パラメータ修正",
"_____no_output_____"
]
],
[
[
"# 学習率の定義\n\nlr = 0.001",
"_____no_output_____"
],
[
"# 경사를 기반으로 파라미터 수정\n\nW -= lr * W.grad\nB -= lr * B.grad",
"_____no_output_____"
]
],
[
[
"WとBは一度計算済みなので、この状態で値の更新ができない \n次の書き方にする必要がある",
"_____no_output_____"
]
],
[
[
"# 勾配を元にパラメータ修正\n# with torch.no_grad() を付ける必要がある\n\nwith torch.no_grad():\n W -= lr * W.grad\n B -= lr * B.grad\n \n # 計算済みの勾配値をリセットする\n W.grad.zero_()\n B.grad.zero_()",
"_____no_output_____"
],
[
"# パラメータと勾配値の確認\n\nprint(W)\nprint(B)\nprint(W.grad)\nprint(B.grad)",
"tensor(1.0190, requires_grad=True)\ntensor(0.9980, requires_grad=True)\ntensor(0.)\ntensor(0.)\n"
]
],
[
[
"元の値はどちらも1.0だったので、Wは微少量増加、Bは微少量減少したことがわかる。 \nこの計算を繰り返すことで、最適なWとBを求めるのが勾配降下法となる。",
"_____no_output_____"
],
[
"## 3.10 繰り返し計算",
"_____no_output_____"
]
],
[
[
"# 初期化\n\n# WとBを変数として扱う\nW = torch.tensor(1.0, requires_grad=True).float()\nB = torch.tensor(1.0, requires_grad=True).float()\n\n# 繰り返し回数\nnum_epochs = 500\n\n# 学習率\nlr = 0.001\n\n# 記録用配列初期化\nhistory = np.zeros((0, 2))",
"_____no_output_____"
],
[
"# ループ処理\n\nfor epoch in range(num_epochs):\n\n # 予測計算\n Yp = pred(X)\n \n # 損失計算\n loss = mse(Yp, Y)\n \n # 勾配計算\n loss.backward()\n \n with torch.no_grad():\n # パラメータ修正\n W -= lr * W.grad\n B -= lr * B.grad\n \n # 勾配値の初期化\n W.grad.zero_()\n B.grad.zero_()\n \n # 損失の記録\n if (epoch %10 == 0):\n item = np.array([epoch, loss.item()])\n history = np.vstack((history, item))\n print(f'epoch = {epoch} loss = {loss:.4f}')\n ",
"epoch = 0 loss = 13.3520\nepoch = 10 loss = 10.3855\nepoch = 20 loss = 8.5173\nepoch = 30 loss = 7.3364\nepoch = 40 loss = 6.5858\nepoch = 50 loss = 6.1047\nepoch = 60 loss = 5.7927\nepoch = 70 loss = 5.5868\nepoch = 80 loss = 5.4476\nepoch = 90 loss = 5.3507\nepoch = 100 loss = 5.2805\nepoch = 110 loss = 5.2275\nepoch = 120 loss = 5.1855\nepoch = 130 loss = 5.1507\nepoch = 140 loss = 5.1208\nepoch = 150 loss = 5.0943\nepoch = 160 loss = 5.0703\nepoch = 170 loss = 5.0480\nepoch = 180 loss = 5.0271\nepoch = 190 loss = 5.0074\nepoch = 200 loss = 4.9887\nepoch = 210 loss = 4.9708\nepoch = 220 loss = 4.9537\nepoch = 230 loss = 4.9373\nepoch = 240 loss = 4.9217\nepoch = 250 loss = 4.9066\nepoch = 260 loss = 4.8922\nepoch = 270 loss = 4.8783\nepoch = 280 loss = 4.8650\nepoch = 290 loss = 4.8522\nepoch = 300 loss = 4.8399\nepoch = 310 loss = 4.8281\nepoch = 320 loss = 4.8167\nepoch = 330 loss = 4.8058\nepoch = 340 loss = 4.7953\nepoch = 350 loss = 4.7853\nepoch = 360 loss = 4.7756\nepoch = 370 loss = 4.7663\nepoch = 380 loss = 4.7574\nepoch = 390 loss = 4.7488\nepoch = 400 loss = 4.7406\nepoch = 410 loss = 4.7327\nepoch = 420 loss = 4.7251\nepoch = 430 loss = 4.7178\nepoch = 440 loss = 4.7108\nepoch = 450 loss = 4.7040\nepoch = 460 loss = 4.6976\nepoch = 470 loss = 4.6913\nepoch = 480 loss = 4.6854\nepoch = 490 loss = 4.6796\n"
]
],
[
[
"## 3.11 結果確認",
"_____no_output_____"
]
],
[
[
"# パラメータの最終値\nprint('W = ', W.data.numpy())\nprint('B = ', B.data.numpy())\n\n#損失の確認\nprint(f'초기상태: 손실:{history[0,1]:.4f}') \nprint(f'최종상태: 손실:{history[-1,1]:.4f}') ",
"W = 1.820683\nB = 0.3675114\n초기상태: 손실:13.3520\n최종상태: 손실:4.6796\n"
],
[
"# 学習曲線の表示 (損失)\n\nfig1 = plt.gcf()\nplt.plot(history[:,0], history[:,1], 'b')\nplt.xlabel('반복 횟수')\nplt.ylabel('손실')\nplt.title('학습 곡선(손실)')\nplt.show()\nplt.draw()\nfig1.savefig('ex03-19.tif', format='tif', dpi=300)",
"_____no_output_____"
]
],
[
[
"### 散布図に回帰直線を重ね書きする",
"_____no_output_____"
]
],
[
[
"# xの範囲を求める(Xrange)\nX_max = X.max()\nX_min = X.min()\nX_range = np.array((X_min, X_max))\nX_range = torch.from_numpy(X_range).float()\nprint(X_range)\n\n# 対応するyの予測値を求める\nY_range = pred(X_range)\nprint(Y_range.data)",
"tensor([-5., 5.])\ntensor([-8.7359, 9.4709])\n"
],
[
"# グラフ描画\n\nfig1 = plt.gcf()\nplt.scatter(X, Y, c='k', s=50)\nplt.xlabel('$X$')\nplt.ylabel('$Y$')\nplt.plot(X_range.data, Y_range.data, lw=2, c='b')\nplt.title('신장과 체중의 상관 직선(가공 후)')\nplt.show()\nplt.draw()\nfig1.savefig('ex03-20.tif', format='tif', dpi=300)",
"_____no_output_____"
]
],
[
[
"### 加工前データへの回帰直線描画",
"_____no_output_____"
]
],
[
[
"# y座標値とx座標値の計算\n\nx_range = X_range + x.mean()\nyp_range = Y_range + y.mean()",
"_____no_output_____"
],
[
"# グラフ描画\n\nfig1 = plt.gcf()\nplt.scatter(x, y, c='k', s=50)\nplt.xlabel('$x$')\nplt.ylabel('$y$')\nplt.plot(x_range, yp_range.data, lw=2, c='b')\nplt.title('신장과 체중의 상관 직선(가공 전)')\nplt.show()\nplt.draw()\nfig1.savefig('ex03-21.tif', format='tif', dpi=300)",
"_____no_output_____"
]
],
[
[
"## 3.12 最適化関数とstep関数の利用",
"_____no_output_____"
]
],
[
[
"# 初期化\n\n# WとBを変数として扱う\nW = torch.tensor(1.0, requires_grad=True).float()\nB = torch.tensor(1.0, requires_grad=True).float()\n\n# 繰り返し回数\nnum_epochs = 500\n\n# 学習率\nlr = 0.001\n\n# optimizerとしてSGD(確率的勾配降下法)を指定する\nimport torch.optim as optim\noptimizer = optim.SGD([W, B], lr=lr)\n\n# 記録用配列初期化\nhistory = np.zeros((0, 2))",
"_____no_output_____"
],
[
"# ループ処理\n\nfor epoch in range(num_epochs):\n\n # 予測計算\n Yp = pred(X)\n \n # 損失計算\n loss = mse(Yp, Y)\n\n # 勾配計算\n loss.backward()\n\n # パラメータ修正\n optimizer.step()\n \n #勾配値初期化\n optimizer.zero_grad()\n \n # 損失値の記録\n if (epoch %10 == 0):\n item = np.array([epoch, loss.item()])\n history = np.vstack((history, item))\n print(f'epoch = {epoch} loss = {loss:.4f}')",
"epoch = 0 loss = 13.3520\nepoch = 10 loss = 10.3855\nepoch = 20 loss = 8.5173\nepoch = 30 loss = 7.3364\nepoch = 40 loss = 6.5858\nepoch = 50 loss = 6.1047\nepoch = 60 loss = 5.7927\nepoch = 70 loss = 5.5868\nepoch = 80 loss = 5.4476\nepoch = 90 loss = 5.3507\nepoch = 100 loss = 5.2805\nepoch = 110 loss = 5.2275\nepoch = 120 loss = 5.1855\nepoch = 130 loss = 5.1507\nepoch = 140 loss = 5.1208\nepoch = 150 loss = 5.0943\nepoch = 160 loss = 5.0703\nepoch = 170 loss = 5.0480\nepoch = 180 loss = 5.0271\nepoch = 190 loss = 5.0074\nepoch = 200 loss = 4.9887\nepoch = 210 loss = 4.9708\nepoch = 220 loss = 4.9537\nepoch = 230 loss = 4.9373\nepoch = 240 loss = 4.9217\nepoch = 250 loss = 4.9066\nepoch = 260 loss = 4.8922\nepoch = 270 loss = 4.8783\nepoch = 280 loss = 4.8650\nepoch = 290 loss = 4.8522\nepoch = 300 loss = 4.8399\nepoch = 310 loss = 4.8281\nepoch = 320 loss = 4.8167\nepoch = 330 loss = 4.8058\nepoch = 340 loss = 4.7953\nepoch = 350 loss = 4.7853\nepoch = 360 loss = 4.7756\nepoch = 370 loss = 4.7663\nepoch = 380 loss = 4.7574\nepoch = 390 loss = 4.7488\nepoch = 400 loss = 4.7406\nepoch = 410 loss = 4.7327\nepoch = 420 loss = 4.7251\nepoch = 430 loss = 4.7178\nepoch = 440 loss = 4.7108\nepoch = 450 loss = 4.7040\nepoch = 460 loss = 4.6976\nepoch = 470 loss = 4.6913\nepoch = 480 loss = 4.6854\nepoch = 490 loss = 4.6796\n"
],
[
"# パラメータの最終値\nprint('W = ', W.data.numpy())\nprint('B = ', B.data.numpy())\n\n#損失の確認\nprint(f'初期状態: 損失:{history[0,1]:.4f}') \nprint(f'最終状態: 損失:{history[-1,1]:.4f}') ",
"W = 1.820683\nB = 0.3675114\n初期状態: 損失:13.3520\n最終状態: 損失:4.6796\n"
],
[
"# 学習曲線の表示 (損失)\n\nplt.plot(history[:,0], history[:,1], 'b')\nplt.xlabel('繰り返し回数')\nplt.ylabel('損失')\nplt.title('学習曲線(損失)')\nplt.show()",
"_____no_output_____"
]
],
[
[
"3.7の結果と見比べるとまったく同じであることがわかる。 \nつまり、step関数でやっていることは、次のコードと同じ。\n\n```py3\n\n with torch.no_grad():\n # パラメータ修正 (フレームワークを使う場合はstep関数)\n W -= lr * W.grad\n B -= lr * B.grad\n```",
"_____no_output_____"
],
[
"### 最適化関数のチューニング",
"_____no_output_____"
]
],
[
[
"# 初期化\n\n# WとBを変数として扱う\nW = torch.tensor(1.0, requires_grad=True).float()\nB = torch.tensor(1.0, requires_grad=True).float()\n\n# 繰り返し回数\nnum_epochs = 500\n\n# 学習率\nlr = 0.001\n\n# optimizerとしてSGD(確率的勾配降下法)を指定する\nimport torch.optim as optim\noptimizer = optim.SGD([W, B], lr=lr, momentum=0.9)\n\n# 記録用配列初期化\nhistory2 = np.zeros((0, 2))",
"_____no_output_____"
],
[
"# ループ処理\n\nfor epoch in range(num_epochs):\n\n # 予測計算\n Yp = pred(X)\n \n # 損失計算\n loss = mse(Yp, Y)\n\n # 勾配計算\n loss.backward()\n\n # パラメータ修正\n optimizer.step()\n \n #勾配値初期化\n optimizer.zero_grad()\n \n # 損失値の記録\n if (epoch %10 == 0):\n item = np.array([epoch, loss.item()])\n history2 = np.vstack((history2, item))\n print(f'epoch = {epoch} loss = {loss:.4f}')\n ",
"epoch = 0 loss = 13.3520\nepoch = 10 loss = 5.7585\nepoch = 20 loss = 5.9541\nepoch = 30 loss = 5.0276\nepoch = 40 loss = 4.8578\nepoch = 50 loss = 4.7052\nepoch = 60 loss = 4.6327\nepoch = 70 loss = 4.5940\nepoch = 80 loss = 4.5698\nepoch = 90 loss = 4.5574\nepoch = 100 loss = 4.5495\nepoch = 110 loss = 4.5452\nepoch = 120 loss = 4.5426\nepoch = 130 loss = 4.5411\nepoch = 140 loss = 4.5403\nepoch = 150 loss = 4.5398\nepoch = 160 loss = 4.5395\nepoch = 170 loss = 4.5393\nepoch = 180 loss = 4.5392\nepoch = 190 loss = 4.5391\nepoch = 200 loss = 4.5391\nepoch = 210 loss = 4.5391\nepoch = 220 loss = 4.5391\nepoch = 230 loss = 4.5390\nepoch = 240 loss = 4.5390\nepoch = 250 loss = 4.5390\nepoch = 260 loss = 4.5390\nepoch = 270 loss = 4.5390\nepoch = 280 loss = 4.5390\nepoch = 290 loss = 4.5390\nepoch = 300 loss = 4.5390\nepoch = 310 loss = 4.5390\nepoch = 320 loss = 4.5390\nepoch = 330 loss = 4.5390\nepoch = 340 loss = 4.5390\nepoch = 350 loss = 4.5390\nepoch = 360 loss = 4.5390\nepoch = 370 loss = 4.5390\nepoch = 380 loss = 4.5390\nepoch = 390 loss = 4.5390\nepoch = 400 loss = 4.5390\nepoch = 410 loss = 4.5390\nepoch = 420 loss = 4.5390\nepoch = 430 loss = 4.5390\nepoch = 440 loss = 4.5390\nepoch = 450 loss = 4.5390\nepoch = 460 loss = 4.5390\nepoch = 470 loss = 4.5390\nepoch = 480 loss = 4.5390\nepoch = 490 loss = 4.5390\n"
],
[
"# 学習曲線の表示 (損失)\n\nfig1 = plt.gcf()\nplt.plot(history[:,0], history[:,1], 'b', label='기본값 설정')\nplt.plot(history2[:,0], history2[:,1], 'k', label='momentum=0.9')\nplt.xlabel('반복 횟수')\nplt.ylabel('손실')\nplt.legend()\nplt.title('학습 곡선(손실)')\nplt.show()\nplt.draw()\nfig1.savefig('ex03-27.tif', format='tif', dpi=300)",
"_____no_output_____"
]
],
[
[
"## コラム 局所最適解",
"_____no_output_____"
]
],
[
[
"def f(x):\n return x * (x+1) * (x+2) * (x-2)",
"_____no_output_____"
],
[
"x = np.arange(-3, 2.7, 0.05)\ny = f(x)\n\nplt.plot(x, y)\nplt.axis('off')\nplt.show()",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
]
] |
e7e89ad070a23cf3b66c19279cadb85bc049f72a | 272,704 | ipynb | Jupyter Notebook | Assignment_06/Assignment_ML_L2_Sankalp_Jain_ipynb_txt.ipynb | Sankalp679/SHALA | 14bc0f84145babcd5ebc35ee309e41cca28a0c6b | [
"MIT"
] | 5 | 2020-06-12T12:04:06.000Z | 2020-08-14T16:23:33.000Z | Assignment_06/Assignment_ML_L2_Sankalp_Jain_ipynb_txt.ipynb | Sankalp679/SHALA | 14bc0f84145babcd5ebc35ee309e41cca28a0c6b | [
"MIT"
] | null | null | null | Assignment_06/Assignment_ML_L2_Sankalp_Jain_ipynb_txt.ipynb | Sankalp679/SHALA | 14bc0f84145babcd5ebc35ee309e41cca28a0c6b | [
"MIT"
] | 1 | 2021-05-22T10:53:53.000Z | 2021-05-22T10:53:53.000Z | 94.198273 | 20,770 | 0.714225 | [
[
[
"# Assignment 2: **Machine learning with tree based models** ",
"_____no_output_____"
],
[
"In this assignment, you will work on the **Titanic** dataset and use machine learning to create a model that predicts which passengers survived the **Titanic** shipwreck. ",
"_____no_output_____"
],
[
"---\n## About the dataset:\n---\n* The column named `Survived` is the label and the remaining columns are features. \n* The features can be described as given below:\n <table>\n <thead>\n <tr>\n <th>Variable</th>\n <th>Definition </th>\n </tr>\n </thead>\n <tbody>\n <tr>\n <td>pclass</td>\n <td>Ticket class\t</td>\n </tr>\n <tr>\n <td>SibSp</td>\n <td>Number of siblings / spouses aboard the Titanic</td>\n </tr>\n <tr>\n <td>Parch</td>\n <td>Number of parents / children aboard the Titanic</td>\n </tr>\n <tr>\n <td>Ticket</td>\n <td>Ticket number</td>\n </tr>\n <tr>\n <td>Embarked</td>\n <td>Port of Embarkation: C = Cherbourg, Q = Queenstown, S = Southampton</td>\n </tr>\n </tbody>\n</table> \t",
"_____no_output_____"
],
[
"---\n## Instructions\n---\n* Apply suitable data pre-processing techniques, if needed. \n* Implement a few classifiers to create your model and compare the performance metrics by plotting the curves like roc_auc, confusion matrix, etc. ",
"_____no_output_____"
]
],
[
[
"import pandas as pd \nimport numpy as np\nimport matplotlib.pyplot as plt\nfrom sklearn.impute import SimpleImputer\nimport seaborn as sns",
"_____no_output_____"
],
[
"from sklearn.tree import DecisionTreeClassifier\nfrom sklearn.model_selection import train_test_split,cross_val_score,GridSearchCV\nfrom sklearn.linear_model import LinearRegression,LogisticRegression\nfrom sklearn.neighbors import KNeighborsClassifier as KNN\nfrom sklearn.ensemble import RandomForestClassifier,VotingClassifier,BaggingClassifier,AdaBoostClassifier,GradientBoostingClassifier\nfrom sklearn.metrics import accuracy_score,mean_squared_error as MSE,roc_auc_score,confusion_matrix,classification_report,roc_curve \nfrom xgboost import XGBClassifier\nimport xgboost as xgb\nSEED=1",
"_____no_output_____"
],
[
"titanic_data = pd.read_csv('https://raw.githubusercontent.com/shala2020/shala2020.github.io/master/Lecture_Materials/Assignments/MachineLearning/L2/titanic.csv')\ntitanic_data.head()",
"_____no_output_____"
],
[
"titanic_data.shape",
"_____no_output_____"
],
[
"print(titanic_data.isna().sum())",
"PassengerId 0\nSurvived 0\nPclass 0\nName 0\nSex 0\nAge 177\nSibSp 0\nParch 0\nTicket 0\nFare 0\nCabin 687\nEmbarked 2\ndtype: int64\n"
],
[
"titanic_data.dtypes",
"_____no_output_____"
],
[
"titanic_data.describe()",
"_____no_output_____"
],
[
"titanic_data.info()",
"<class 'pandas.core.frame.DataFrame'>\nRangeIndex: 891 entries, 0 to 890\nData columns (total 12 columns):\n # Column Non-Null Count Dtype \n--- ------ -------------- ----- \n 0 PassengerId 891 non-null int64 \n 1 Survived 891 non-null int64 \n 2 Pclass 891 non-null int64 \n 3 Name 891 non-null object \n 4 Sex 891 non-null object \n 5 Age 714 non-null float64\n 6 SibSp 891 non-null int64 \n 7 Parch 891 non-null int64 \n 8 Ticket 891 non-null object \n 9 Fare 891 non-null float64\n 10 Cabin 204 non-null object \n 11 Embarked 889 non-null object \ndtypes: float64(2), int64(5), object(5)\nmemory usage: 83.7+ KB\n"
],
[
"titanic_data = titanic_data.drop(['PassengerId','Name','Cabin','Ticket'], axis=1)",
"_____no_output_____"
],
[
"titanic_data",
"_____no_output_____"
],
[
"imp =SimpleImputer(missing_values=np.nan, strategy='mean')\nimp.fit(titanic_data[['Age']]) \ntitanic_data['Age'] = imp.transform(titanic_data[['Age']])",
"_____no_output_____"
],
[
"titanic_data['Embarked'].describe()",
"_____no_output_____"
],
[
"common_value='S'\ntitanic_data['Embarked'] = titanic_data['Embarked'].fillna(common_value)",
"_____no_output_____"
],
[
"titanic_data['Sex'] = titanic_data['Sex'].apply(lambda x: 0 if x==\"male\" else 1)",
"_____no_output_____"
],
[
"titanic_data",
"_____no_output_____"
],
[
"print(titanic_data.isna().sum())",
"Survived 0\nPclass 0\nSex 0\nAge 0\nSibSp 0\nParch 0\nFare 0\nEmbarked 0\ndtype: int64\n"
],
[
"ports = {\"C\":0,\"Q\":1,\"S\":2}\ntitanic_data['Embarked'] = titanic_data['Embarked'].map(ports)\ntitanic_data",
"_____no_output_____"
],
[
"titanic_data['Age']=titanic_data['Age'].astype(int) \ntitanic_data['Fare']=titanic_data['Fare'].astype(int) \n",
"_____no_output_____"
],
[
"titanic_data",
"_____no_output_____"
],
[
"pd.qcut(titanic_data['Fare'],4)",
"_____no_output_____"
],
[
" titanic_data.loc[ titanic_data['Age'] <=19, 'Age'] = 0\n titanic_data.loc[(titanic_data['Age'] > 19 )& (titanic_data['Age'] <= 25), 'Age'] = 1\n titanic_data.loc[(titanic_data['Age'] > 25) & (titanic_data['Age'] <= 29), 'Age'] = 2\n titanic_data.loc[(titanic_data['Age'] > 29) & (titanic_data['Age'] <= 31), 'Age'] = 3\n titanic_data.loc[(titanic_data['Age'] > 31) & (titanic_data['Age'] <= 40), 'Age'] = 4\n titanic_data.loc[(titanic_data['Age'] > 40) & (titanic_data['Age'] <= 80), 'Age'] = 5\n titanic_data['Age'].value_counts()",
"_____no_output_____"
],
[
" titanic_data.loc[ titanic_data['Fare'] <=7, 'Fare'] = 0\n titanic_data.loc[(titanic_data['Fare'] > 7 )& (titanic_data['Fare'] <= 14), 'Fare'] = 1\n titanic_data.loc[(titanic_data['Fare'] > 14) & (titanic_data['Fare'] <= 31), 'Fare'] = 2\n titanic_data.loc[(titanic_data['Fare'] > 31) & (titanic_data['Fare'] <= 512), 'Fare'] = 3\n titanic_data.loc[(titanic_data['Fare'] > 512), 'Fare'] = 4\n titanic_data['Fare'].value_counts()",
"_____no_output_____"
],
[
"titanic_data['Relatives']=titanic_data['SibSp']+titanic_data['Parch']\ntitanic_data",
"_____no_output_____"
],
[
"titanic_data['Fare_Per_Person'] = titanic_data['Fare']/(titanic_data['Relatives']+1)\ntitanic_data['Fare_Per_Person'] = titanic_data['Fare_Per_Person'].astype(int)\ntitanic_data",
"_____no_output_____"
],
[
"titanic_data['Age_Class']= titanic_data['Age']* titanic_data['Pclass']\ntitanic_data",
"_____no_output_____"
],
[
"y=titanic_data['Survived']\nX=titanic_data.drop(['Survived','Parch','Fare_Per_Person'],axis=1)\n",
"_____no_output_____"
],
[
"# Split data into 70% train and 30% test\nX_train, X_test, y_train, y_test = train_test_split(X, y, test_size= 0.3, random_state= SEED)\n# Instantiate individual classifiers\nlr = LogisticRegression(random_state=SEED)\nknn = KNN()\ndt = DecisionTreeClassifier(random_state=SEED)\nrf = RandomForestClassifier(n_estimators=300,random_state=SEED)\nbc = BaggingClassifier(base_estimator=dt, n_estimators=300, n_jobs=-1,random_state=SEED,oob_score=True)\nadb = AdaBoostClassifier(base_estimator=dt, n_estimators=100,random_state=SEED) \ngb= GradientBoostingClassifier(n_estimators=300, max_depth=1, random_state=SEED,subsample=0.8,max_features=0.2) \nxgb = xgb.XGBClassifier(learning_rate=0.01)\n# Define a list called classifier that contains the tuples (classifier_name, classifier)\nclassifiers = [('Logistic Regression', lr),('K Nearest Neighbours', knn),\n ('Classification Tree', dt),('Random Forest',rf),\n ('Bagging Classifier',bc),('Adaboost',adb),('Gradient Boosting',gb),('Xtreme GB',xgb)]",
"_____no_output_____"
],
[
"import warnings\nwarnings.filterwarnings(\"ignore\")\n# Iterate over the defined list of tuples containing the classifiers\nfor clf_name, clf in classifiers:\n #fit clf to the training set\n clf.fit(X_train, y_train)\n # Predict the labels of the test set\n y_pred = clf.predict(X_test)\n # Evaluate the accuracy of clf on the test set\n print('{:s} : {:.3f}'.format(clf_name, accuracy_score(y_test, y_pred)))\n print(confusion_matrix(y_test,y_pred))\n y_pred_proba = clf.predict_proba(X_test)[:,1] \n clf_roc_auc_score = roc_auc_score(y_test, y_pred_proba)\n print('ROC AUC score: {:.2f}'.format(clf_roc_auc_score)) \n fpr, tpr, thresholds = roc_curve(y_test, y_pred_proba) \n plt.plot([0, 1], [0, 1], 'k--') \n plt.plot(fpr, tpr, label='Random Forest Classification') \n plt.xlabel('False Positive Rate') \n plt.ylabel('True Positive Rate') \n plt.title('ROC Curve') \n plt.show();\n print(classification_report(y_test, y_pred))\n print(\"=\"*60)\noob_accuracy = bc.oob_score_\nprint('OOB accuracy of bagging classifier: {:.3f}'.format(oob_accuracy))\n",
"Logistic Regression : 0.761\n[[130 23]\n [ 41 74]]\nROC AUC score: 0.82\n"
],
[
"# Instantiate a VotingClassifier 'vc' \nvc = VotingClassifier(estimators=classifiers) \n# Fit 'vc' to the traing set and predict test set labels \nvc.fit(X_train, y_train) \ny_pred = vc.predict(X_test) \n# Evaluate the test-set accuracy of 'vc' \nprint('Voting Classifier: {:.3f}'.format(accuracy_score(y_test, y_pred)))\n",
"Voting Classifier: 0.769\n"
],
[
"classifiers = [('Logistic Regression', lr),('K Nearest Neighbours', knn),\n ('Classification Tree', dt),('Random Forest',rf),\n ('Bagging Classifier',bc),('Adaboost',adb),('Gradient Boosting',gb)]\nlr = LogisticRegression(random_state=SEED)\nknn = KNN()\ndt = DecisionTreeClassifier(random_state=SEED)\nrf = RandomForestClassifier(n_estimators=300,random_state=SEED)\nbc = BaggingClassifier(base_estimator=dt, n_estimators=300, n_jobs=-1,random_state=SEED,oob_score=True)\nadb = AdaBoostClassifier(base_estimator=dt, n_estimators=100,random_state=SEED) \ngb= GradientBoostingClassifier(n_estimators=300, max_depth=1, random_state=SEED,subsample=0.8,max_features=0.2) \nfor clf_name, clf in classifiers:\n scores = cross_val_score(clf, X_train, y_train, cv=10, scoring = \"accuracy\")\n print('{:s} '.format(clf_name))\n print(\"Scores:\", scores)\n print(\"Mean:\", scores.mean())\n print(\"Standard Deviation:\", scores.std())\n ",
"Logistic Regression \nScores: [0.74603175 0.76190476 0.80952381 0.82258065 0.88709677 0.83870968\n 0.79032258 0.80645161 0.79032258 0.80645161]\nMean: 0.8059395801331284\nStandard Deviation: 0.03737172818289197\nK Nearest Neighbours \nScores: [0.79365079 0.76190476 0.76190476 0.77419355 0.82258065 0.66129032\n 0.77419355 0.75806452 0.72580645 0.80645161]\nMean: 0.7640040962621607\nStandard Deviation: 0.042863565726017516\nClassification Tree \nScores: [0.71428571 0.71428571 0.85714286 0.72580645 0.87096774 0.77419355\n 0.83870968 0.77419355 0.79032258 0.72580645]\nMean: 0.7785714285714285\nStandard Deviation: 0.05686578060636469\nRandom Forest \nScores: [0.76190476 0.74603175 0.85714286 0.74193548 0.87096774 0.80645161\n 0.80645161 0.82258065 0.82258065 0.77419355]\nMean: 0.8010240655401943\nStandard Deviation: 0.04218475753815971\nBagging Classifier \nScores: [0.76190476 0.76190476 0.84126984 0.74193548 0.87096774 0.79032258\n 0.80645161 0.80645161 0.80645161 0.75806452]\nMean: 0.7945724526369687\nStandard Deviation: 0.0383355279531469\nAdaboost \nScores: [0.79365079 0.74603175 0.85714286 0.70967742 0.87096774 0.82258065\n 0.82258065 0.82258065 0.79032258 0.77419355]\nMean: 0.8009728622631848\nStandard Deviation: 0.04657531249278569\nGradient Boosting \nScores: [0.73015873 0.77777778 0.84126984 0.83870968 0.90322581 0.83870968\n 0.85483871 0.80645161 0.83870968 0.79032258]\nMean: 0.8220174091141834\nStandard Deviation: 0.045299681256918044\n"
],
[
"feature_imp = pd.Series(rf.feature_importances_,index=list(X.columns.values.tolist())).sort_values(ascending=False)\nfeature_imp",
"_____no_output_____"
],
[
"plt.figure(figsize=(10,10))\nsns.barplot(x=feature_imp, y=feature_imp.index)\n# Add labels to your graph\nplt.xlabel('Feature Importance Score')\nplt.ylabel('Features')\nplt.title(\"Visualizing Important Features\")\nplt.legend()\nplt.show()",
"No handles with labels found to put in legend.\n"
],
[
"param_grid = { \"criterion\" : [\"gini\", \"entropy\"], \"min_samples_leaf\" : [1, 5, 10, 25, 50, 70], \n \"min_samples_split\" : [2, 4, 10, 12, 16, 18, 25, 35],\n \"n_estimators\": [100, 400, 700, 1000, 1500]}\nraf = RandomForestClassifier(random_state=SEED)\nclfa = GridSearchCV(estimator=raf, param_grid=param_grid, n_jobs=-1)\nclfa.fit(X_train, y_train)\nclfa.best_params_",
"_____no_output_____"
],
[
"So bagging classifier is the classifer with highest accuracy=78% and oob score=80.4% among all the classifers and will be used to train our model.",
"_____no_output_____"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e89c6264ff50a7d79d989454457a4f85e3e38d | 1,597 | ipynb | Jupyter Notebook | The fifth chapter/5-20.ipynb | youxi0011983/PythonFluent | 3d573f1dc42434df2ffef6ca172eb8b81a9231f0 | [
"Apache-2.0"
] | null | null | null | The fifth chapter/5-20.ipynb | youxi0011983/PythonFluent | 3d573f1dc42434df2ffef6ca172eb8b81a9231f0 | [
"Apache-2.0"
] | null | null | null | The fifth chapter/5-20.ipynb | youxi0011983/PythonFluent | 3d573f1dc42434df2ffef6ca172eb8b81a9231f0 | [
"Apache-2.0"
] | null | null | null | 18.356322 | 66 | 0.496556 | [
[
[
"from clip_annot import clip\nfrom inspect import signature\nsig = signature(clip)\nsig.return_annotation",
"_____no_output_____"
],
[
"for name,param in sig.parameters.items():\n print(param.kind,':',name,'=',param.default)\n",
"POSITIONAL_OR_KEYWORD : text = <class 'inspect._empty'>\nPOSITIONAL_OR_KEYWORD : max_len = 80\n"
]
]
] | [
"code"
] | [
[
"code",
"code"
]
] |
e7e8a2f71015bd38d7a006a31d057c30af6585a2 | 57,333 | ipynb | Jupyter Notebook | exoplanet1.ipynb | bshub6/machine-learning-challenge | 5682979d992024970156ca4ff1a36ad26c12cd53 | [
"MIT"
] | null | null | null | exoplanet1.ipynb | bshub6/machine-learning-challenge | 5682979d992024970156ca4ff1a36ad26c12cd53 | [
"MIT"
] | null | null | null | exoplanet1.ipynb | bshub6/machine-learning-challenge | 5682979d992024970156ca4ff1a36ad26c12cd53 | [
"MIT"
] | null | null | null | 38.843496 | 1,599 | 0.381107 | [
[
[
"# Update sklearn to prevent version mismatches\n!pip install sklearn --upgrade",
"_____no_output_____"
],
[
"# install joblib. This will be used to save your model. \n# Restart your kernel after installing \n!pip install joblib",
"_____no_output_____"
],
[
"\nimport pandas as pd\n",
"_____no_output_____"
]
],
[
[
"# Read the CSV and Perform Basic Data Cleaning",
"_____no_output_____"
]
],
[
[
"df = pd.read_csv(\"exoplanet_data.csv\")\n# Drop the null columns where all values are null\ndf = df.dropna(axis='columns', how='all')\n# Drop the null rows\ndf = df.dropna()\ndf.head()",
"_____no_output_____"
],
[
"df.describe()",
"_____no_output_____"
]
],
[
[
"# Select your features (columns)",
"_____no_output_____"
]
],
[
[
"# Set features. This will also be used as your x values.\n\ntarget = df[\"koi_disposition\"]\ndata = df.drop(\"koi_disposition\", axis=1)\nfeature_names = data.columns\ndata.head()",
"_____no_output_____"
]
],
[
[
"# Create a Train Test Split\n\nUse `koi_disposition` for the y values",
"_____no_output_____"
]
],
[
[
"from sklearn.model_selection import train_test_split\nX_train, X_test, y_train, y_test = train_test_split(data, target, random_state=42)",
"_____no_output_____"
],
[
"X_train.head()",
"_____no_output_____"
]
],
[
[
"# Pre-processing\n\nScale the data using the MinMaxScaler and perform some feature selection",
"_____no_output_____"
]
],
[
[
"# Scale your data\nfrom sklearn.preprocessing import MinMaxScaler\nX_minmax = MinMaxScaler().fit(X_train)\n\nX_train_minmax = X_minmax.transform(X_train)\nX_test_minmax = X_minmax.transform(X_test)",
"_____no_output_____"
],
[
"from sklearn.svm import SVC \nmodel = SVC(kernel='linear')\nmodel.fit(X_train_minmax, y_train)",
"_____no_output_____"
]
],
[
[
"# Train the Model\n\n",
"_____no_output_____"
]
],
[
[
"print(f\"Training Data Score: {model.score(X_train_minmax, y_train)}\")\nprint(f\"Testing Data Score: {model.score(X_test_minmax, y_test)}\")",
"Training Data Score: 0.8455082967766546\nTesting Data Score: 0.8415331807780321\n"
]
],
[
[
"# Hyperparameter Tuning\n\nUse `GridSearchCV` to tune the model's parameters",
"_____no_output_____"
]
],
[
[
"# Create the GridSearchCV model\nfrom sklearn.model_selection import GridSearchCV\nparam_grid = {'C': [1, 5, 10, 50],\n 'gamma': [0.0001, 0.0005, 0.001, 0.005]}\ngrid = GridSearchCV(model, param_grid, verbose=3)",
"_____no_output_____"
],
[
"# Train the model with GridSearch\ngrid.fit(X_train_minmax, y_train)",
"C:\\Users\\bdsan\\Anaconda3\\lib\\site-packages\\sklearn\\model_selection\\_split.py:1978: FutureWarning: The default value of cv will change from 3 to 5 in version 0.22. Specify it explicitly to silence this warning.\n warnings.warn(CV_WARNING, FutureWarning)\n[Parallel(n_jobs=1)]: Using backend SequentialBackend with 1 concurrent workers.\n"
],
[
"print(grid.best_params_)\nprint(grid.best_score_)",
"{'C': 50, 'gamma': 0.0001}\n0.8815563608621019\n"
],
[
"# Model Accuracy\nprint('Test Acc: %.3f' % model.score(X_test, y_test))",
"Test Acc: 0.429\n"
],
[
"\n# Make prediction and save to variable for report.\npredictions = grid.predict(X_test_minmax)",
"_____no_output_____"
],
[
"# Print Classification Report.\nfrom sklearn.metrics import classification_report\nprint(classification_report(y_test, predictions))",
" precision recall f1-score support\n\n CANDIDATE 0.81 0.67 0.73 411\n CONFIRMED 0.76 0.85 0.80 484\nFALSE POSITIVE 0.98 1.00 0.99 853\n\n accuracy 0.88 1748\n macro avg 0.85 0.84 0.84 1748\n weighted avg 0.88 0.88 0.88 1748\n\n"
]
],
[
[
"# Save the Model",
"_____no_output_____"
]
],
[
[
"# save your model by updating \"your_name\" with your name\n# and \"your_model\" with your model variable\n# be sure to turn this in to BCS\n# if joblib fails to import, try running the command to install in terminal/git-bash\nimport joblib\nfilename = 'models/bridgette_svm.sav'\njoblib.dump(model, filename)",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7e8acbd1a0daac088ff9bd4b8ac5cea71fb6678 | 429,733 | ipynb | Jupyter Notebook | scripts/watershed/Watershed Transform 3D Fully Convolutional.ipynb | esgomezm/deepcell-tf | 6693c9ed7e76793561e6c2281437acaf3e4fa441 | [
"Apache-2.0"
] | 1 | 2021-09-03T21:06:44.000Z | 2021-09-03T21:06:44.000Z | scripts/watershed/Watershed Transform 3D Fully Convolutional.ipynb | esgomezm/deepcell-tf | 6693c9ed7e76793561e6c2281437acaf3e4fa441 | [
"Apache-2.0"
] | null | null | null | scripts/watershed/Watershed Transform 3D Fully Convolutional.ipynb | esgomezm/deepcell-tf | 6693c9ed7e76793561e6c2281437acaf3e4fa441 | [
"Apache-2.0"
] | 1 | 2020-06-24T23:04:26.000Z | 2020-06-24T23:04:26.000Z | 154.358118 | 217,412 | 0.869114 | [
[
[
"# Watershed Distance Transform for 3D Data\n---\nImplementation of papers:\n\n[Deep Watershed Transform for Instance Segmentation](http://openaccess.thecvf.com/content_cvpr_2017/papers/Bai_Deep_Watershed_Transform_CVPR_2017_paper.pdf)\n\n[Learn to segment single cells with deep distance estimator and deep cell detector](https://arxiv.org/abs/1803.10829)",
"_____no_output_____"
]
],
[
[
"import os\nimport errno\nimport datetime\n\nimport numpy as np\n\nimport deepcell",
"Using TensorFlow backend.\n"
]
],
[
[
"### Load the Training Data",
"_____no_output_____"
]
],
[
[
"# Download the data (saves to ~/.keras/datasets)\nfilename = 'mousebrain.npz'\ntest_size = 0.1 # % of data saved as test\nseed = 0 # seed for random train-test split\n\n(X_train, y_train), (X_test, y_test) = deepcell.datasets.mousebrain.load_data(filename, test_size=test_size, seed=seed)\n\nprint('X.shape: {}\\ny.shape: {}'.format(X_train.shape, y_train.shape))",
"Downloading data from https://deepcell-data.s3.amazonaws.com/nuclei/mousebrain.npz\n1730158592/1730150850 [==============================] - 106s 0us/step\nX.shape: (176, 15, 256, 256, 1)\ny.shape: (176, 15, 256, 256, 1)\n"
]
],
[
[
"### Set up filepath constants",
"_____no_output_____"
]
],
[
[
"# the path to the data file is currently required for `train_model_()` functions\n\n# change DATA_DIR if you are not using `deepcell.datasets`\nDATA_DIR = os.path.expanduser(os.path.join('~', '.keras', 'datasets'))\n\n# DATA_FILE should be a npz file, preferably from `make_training_data`\nDATA_FILE = os.path.join(DATA_DIR, filename)\n\n# confirm the data file is available\nassert os.path.isfile(DATA_FILE)",
"_____no_output_____"
],
[
"# Set up other required filepaths\n\n# If the data file is in a subdirectory, mirror it in MODEL_DIR and LOG_DIR\nPREFIX = os.path.relpath(os.path.dirname(DATA_FILE), DATA_DIR)\n\nROOT_DIR = '/data' # TODO: Change this! Usually a mounted volume\nMODEL_DIR = os.path.abspath(os.path.join(ROOT_DIR, 'models', PREFIX))\nLOG_DIR = os.path.abspath(os.path.join(ROOT_DIR, 'logs', PREFIX))\n\n# create directories if they do not exist\nfor d in (MODEL_DIR, LOG_DIR):\n try:\n os.makedirs(d)\n except OSError as exc: # Guard against race condition\n if exc.errno != errno.EEXIST:\n raise",
"_____no_output_____"
]
],
[
[
"### Set up training parameters",
"_____no_output_____"
]
],
[
[
"from tensorflow.keras.optimizers import SGD\nfrom deepcell.utils.train_utils import rate_scheduler\n\nfgbg_model_name = 'conv_fgbg_3d_model'\nconv_model_name = 'conv_watershed_3d_model'\n\nn_epoch = 10 # Number of training epochs\nnorm_method = 'whole_image' # data normalization - `whole_image` for 3d conv\nreceptive_field = 61 # should be adjusted for the scale of the data\n\noptimizer = SGD(lr=0.01, decay=1e-6, momentum=0.9, nesterov=True)\n\nlr_sched = rate_scheduler(lr=0.01, decay=0.99)\n\n# FC training settings\nn_skips = 3 # number of skip-connections (only for FC training)\nbatch_size = 1 # FC training uses 1 image per batch\n\n# Transformation settings\ntransform = 'watershed'\ndistance_bins = 4 # number of distance classes\nerosion_width = 1 # erode edges, improves segmentation when cells are close\n\n# 3D Settings\nframes_per_batch = 3",
"_____no_output_____"
]
],
[
[
"### First, create a foreground/background separation model\n\n#### Instantiate the fgbg model",
"_____no_output_____"
]
],
[
[
"from deepcell import model_zoo\n\nfgbg_model = model_zoo.bn_feature_net_skip_3D(\n receptive_field=receptive_field,\n n_features=2, # segmentation mask (is_cell, is_not_cell)\n n_frames=frames_per_batch,\n n_skips=n_skips,\n n_conv_filters=32,\n n_dense_filters=128,\n input_shape=tuple([frames_per_batch] + list(X_train.shape[2:])),\n multires=False,\n last_only=False,\n norm_method='whole_image')",
"_____no_output_____"
]
],
[
[
"#### Train the fgbg model",
"_____no_output_____"
]
],
[
[
"from deepcell.training import train_model_conv\n\nfgbg_model = train_model_conv(\n model=fgbg_model,\n dataset=DATA_FILE, # full path to npz file\n model_name=fgbg_model_name,\n test_size=test_size,\n seed=seed,\n transform='fgbg',\n optimizer=optimizer,\n batch_size=batch_size,\n frames_per_batch=frames_per_batch,\n n_epoch=n_epoch,\n model_dir=MODEL_DIR,\n lr_sched=rate_scheduler(lr=0.01, decay=0.95),\n rotation_range=180,\n flip=True,\n shear=False,\n zoom_range=(0.8, 1.2))",
"X_train shape: (198, 15, 256, 256, 1)\ny_train shape: (198, 15, 256, 256, 1)\nX_test shape: (22, 15, 256, 256, 1)\ny_test shape: (22, 15, 256, 256, 1)\nOutput Shape: (None, 3, 256, 256, 2)\nNumber of Classes: 2\nTraining on 1 GPUs\nEpoch 1/10\n197/198 [============================>.] - ETA: 0s - loss: 0.8965 - model_loss: 0.2152 - model_1_loss: 0.2171 - model_2_loss: 0.2121 - model_3_loss: 0.2163 - model_acc: 0.9120 - model_1_acc: 0.9044 - model_2_acc: 0.9113 - model_3_acc: 0.9067\nEpoch 00001: val_loss improved from inf to 1.07529, saving model to /data/models/conv_fgbg_3d_model.h5\n198/198 [==============================] - 133s 673ms/step - loss: 0.8945 - model_loss: 0.2147 - model_1_loss: 0.2166 - model_2_loss: 0.2116 - model_3_loss: 0.2158 - model_acc: 0.9122 - model_1_acc: 0.9046 - model_2_acc: 0.9115 - model_3_acc: 0.9069 - val_loss: 1.0753 - val_model_loss: 0.3084 - val_model_1_loss: 0.2489 - val_model_2_loss: 0.2391 - val_model_3_loss: 0.2430 - val_model_acc: 0.9378 - val_model_1_acc: 0.9172 - val_model_2_acc: 0.9246 - val_model_3_acc: 0.9233\nEpoch 2/10\n197/198 [============================>.] - ETA: 0s - loss: 0.7316 - model_loss: 0.1725 - model_1_loss: 0.1739 - model_2_loss: 0.1755 - model_3_loss: 0.1739 - model_acc: 0.9246 - model_1_acc: 0.9222 - model_2_acc: 0.9219 - model_3_acc: 0.9232\nEpoch 00002: val_loss improved from 1.07529 to 1.02578, saving model to /data/models/conv_fgbg_3d_model.h5\n198/198 [==============================] - 108s 547ms/step - loss: 0.7372 - model_loss: 0.1742 - model_1_loss: 0.1755 - model_2_loss: 0.1767 - model_3_loss: 0.1750 - model_acc: 0.9246 - model_1_acc: 0.9222 - model_2_acc: 0.9219 - model_3_acc: 0.9232 - val_loss: 1.0258 - val_model_loss: 0.2438 - val_model_1_loss: 0.2449 - val_model_2_loss: 0.2461 - val_model_3_loss: 0.2551 - val_model_acc: 0.9296 - val_model_1_acc: 0.9310 - val_model_2_acc: 0.9389 - val_model_3_acc: 0.9429\nEpoch 3/10\n197/198 [============================>.] - ETA: 0s - loss: 0.6888 - model_loss: 0.1632 - model_1_loss: 0.1636 - model_2_loss: 0.1634 - model_3_loss: 0.1626 - model_acc: 0.9282 - model_1_acc: 0.9265 - model_2_acc: 0.9277 - model_3_acc: 0.9277\nEpoch 00003: val_loss improved from 1.02578 to 0.99577, saving model to /data/models/conv_fgbg_3d_model.h5\n198/198 [==============================] - 108s 548ms/step - loss: 0.6880 - model_loss: 0.1630 - model_1_loss: 0.1634 - model_2_loss: 0.1633 - model_3_loss: 0.1624 - model_acc: 0.9282 - model_1_acc: 0.9264 - model_2_acc: 0.9276 - model_3_acc: 0.9277 - val_loss: 0.9958 - val_model_loss: 0.2410 - val_model_1_loss: 0.2488 - val_model_2_loss: 0.2339 - val_model_3_loss: 0.2361 - val_model_acc: 0.9151 - val_model_1_acc: 0.9145 - val_model_2_acc: 0.9202 - val_model_3_acc: 0.9169\nEpoch 4/10\n197/198 [============================>.] - ETA: 0s - loss: 0.6923 - model_loss: 0.1636 - model_1_loss: 0.1646 - model_2_loss: 0.1648 - model_3_loss: 0.1634 - model_acc: 0.9286 - model_1_acc: 0.9271 - model_2_acc: 0.9284 - model_3_acc: 0.9292\nEpoch 00004: val_loss improved from 0.99577 to 0.96332, saving model to /data/models/conv_fgbg_3d_model.h5\n198/198 [==============================] - 108s 547ms/step - loss: 0.6920 - model_loss: 0.1635 - model_1_loss: 0.1646 - model_2_loss: 0.1648 - model_3_loss: 0.1633 - model_acc: 0.9287 - model_1_acc: 0.9271 - model_2_acc: 0.9285 - model_3_acc: 0.9293 - val_loss: 0.9633 - val_model_loss: 0.2329 - val_model_1_loss: 0.2375 - val_model_2_loss: 0.2301 - val_model_3_loss: 0.2270 - val_model_acc: 0.8982 - val_model_1_acc: 0.8952 - val_model_2_acc: 0.8998 - val_model_3_acc: 0.9054\nEpoch 5/10\n197/198 [============================>.] - ETA: 0s - loss: 0.6872 - model_loss: 0.1633 - model_1_loss: 0.1625 - model_2_loss: 0.1638 - model_3_loss: 0.1618 - model_acc: 0.9274 - model_1_acc: 0.9267 - model_2_acc: 0.9262 - model_3_acc: 0.9282\nEpoch 00005: val_loss improved from 0.96332 to 0.96122, saving model to /data/models/conv_fgbg_3d_model.h5\n198/198 [==============================] - 108s 546ms/step - loss: 0.6896 - model_loss: 0.1638 - model_1_loss: 0.1631 - model_2_loss: 0.1645 - model_3_loss: 0.1624 - model_acc: 0.9273 - model_1_acc: 0.9265 - model_2_acc: 0.9260 - model_3_acc: 0.9281 - val_loss: 0.9612 - val_model_loss: 0.2280 - val_model_1_loss: 0.2326 - val_model_2_loss: 0.2325 - val_model_3_loss: 0.2323 - val_model_acc: 0.9260 - val_model_1_acc: 0.9179 - val_model_2_acc: 0.9242 - val_model_3_acc: 0.9140\nEpoch 6/10\n197/198 [============================>.] - ETA: 0s - loss: 0.6726 - model_loss: 0.1590 - model_1_loss: 0.1591 - model_2_loss: 0.1603 - model_3_loss: 0.1583 - model_acc: 0.9290 - model_1_acc: 0.9277 - model_2_acc: 0.9273 - model_3_acc: 0.9286\nEpoch 00006: val_loss did not improve from 0.96122\n198/198 [==============================] - 108s 546ms/step - loss: 0.6717 - model_loss: 0.1588 - model_1_loss: 0.1589 - model_2_loss: 0.1601 - model_3_loss: 0.1581 - model_acc: 0.9290 - model_1_acc: 0.9277 - model_2_acc: 0.9274 - model_3_acc: 0.9286 - val_loss: 1.0302 - val_model_loss: 0.2523 - val_model_1_loss: 0.2546 - val_model_2_loss: 0.2410 - val_model_3_loss: 0.2465 - val_model_acc: 0.8991 - val_model_1_acc: 0.8924 - val_model_2_acc: 0.9154 - val_model_3_acc: 0.9130\nEpoch 7/10\n197/198 [============================>.] - ETA: 0s - loss: 0.6620 - model_loss: 0.1565 - model_1_loss: 0.1566 - model_2_loss: 0.1574 - model_3_loss: 0.1557 - model_acc: 0.9301 - model_1_acc: 0.9281 - model_2_acc: 0.9290 - model_3_acc: 0.9297\nEpoch 00007: val_loss improved from 0.96122 to 0.92732, saving model to /data/models/conv_fgbg_3d_model.h5\n198/198 [==============================] - 108s 547ms/step - loss: 0.6616 - model_loss: 0.1564 - model_1_loss: 0.1565 - model_2_loss: 0.1573 - model_3_loss: 0.1556 - model_acc: 0.9300 - model_1_acc: 0.9281 - model_2_acc: 0.9290 - model_3_acc: 0.9296 - val_loss: 0.9273 - val_model_loss: 0.2280 - val_model_1_loss: 0.2261 - val_model_2_loss: 0.2177 - val_model_3_loss: 0.2197 - val_model_acc: 0.9086 - val_model_1_acc: 0.9049 - val_model_2_acc: 0.9144 - val_model_3_acc: 0.9117\nEpoch 8/10\n197/198 [============================>.] - ETA: 0s - loss: 0.6602 - model_loss: 0.1563 - model_1_loss: 0.1562 - model_2_loss: 0.1564 - model_3_loss: 0.1555 - model_acc: 0.9312 - model_1_acc: 0.9294 - model_2_acc: 0.9296 - model_3_acc: 0.9298\nEpoch 00008: val_loss did not improve from 0.92732\n198/198 [==============================] - 108s 545ms/step - loss: 0.6601 - model_loss: 0.1563 - model_1_loss: 0.1562 - model_2_loss: 0.1564 - model_3_loss: 0.1554 - model_acc: 0.9313 - model_1_acc: 0.9295 - model_2_acc: 0.9297 - model_3_acc: 0.9299 - val_loss: 0.9669 - val_model_loss: 0.2298 - val_model_1_loss: 0.2335 - val_model_2_loss: 0.2339 - val_model_3_loss: 0.2338 - val_model_acc: 0.9224 - val_model_1_acc: 0.9255 - val_model_2_acc: 0.9318 - val_model_3_acc: 0.9229\nEpoch 9/10\n197/198 [============================>.] - ETA: 0s - loss: 0.6534 - model_loss: 0.1554 - model_1_loss: 0.1542 - model_2_loss: 0.1548 - model_3_loss: 0.1532 - model_acc: 0.9312 - model_1_acc: 0.9312 - model_2_acc: 0.9315 - model_3_acc: 0.9314\nEpoch 00009: val_loss improved from 0.92732 to 0.88550, saving model to /data/models/conv_fgbg_3d_model.h5\n198/198 [==============================] - 108s 547ms/step - loss: 0.6536 - model_loss: 0.1554 - model_1_loss: 0.1542 - model_2_loss: 0.1549 - model_3_loss: 0.1533 - model_acc: 0.9310 - model_1_acc: 0.9310 - model_2_acc: 0.9313 - model_3_acc: 0.9312 - val_loss: 0.8855 - val_model_loss: 0.2115 - val_model_1_loss: 0.2154 - val_model_2_loss: 0.2107 - val_model_3_loss: 0.2121 - val_model_acc: 0.9330 - val_model_1_acc: 0.9328 - val_model_2_acc: 0.9316 - val_model_3_acc: 0.9308\nEpoch 10/10\n197/198 [============================>.] - ETA: 0s - loss: 0.6626 - model_loss: 0.1569 - model_1_loss: 0.1567 - model_2_loss: 0.1572 - model_3_loss: 0.1560 - model_acc: 0.9306 - model_1_acc: 0.9295 - model_2_acc: 0.9292 - model_3_acc: 0.9297\nEpoch 00010: val_loss did not improve from 0.88550\n198/198 [==============================] - 108s 545ms/step - loss: 0.6622 - model_loss: 0.1568 - model_1_loss: 0.1566 - model_2_loss: 0.1571 - model_3_loss: 0.1559 - model_acc: 0.9304 - model_1_acc: 0.9293 - model_2_acc: 0.9290 - model_3_acc: 0.9296 - val_loss: 0.9433 - val_model_loss: 0.2337 - val_model_1_loss: 0.2267 - val_model_2_loss: 0.2240 - val_model_3_loss: 0.2230 - val_model_acc: 0.9096 - val_model_1_acc: 0.9157 - val_model_2_acc: 0.9234 - val_model_3_acc: 0.9264\n"
]
],
[
[
"### Next, Create a model for the watershed energy transform\n\n#### Instantiate the distance transform model",
"_____no_output_____"
]
],
[
[
"from deepcell import model_zoo\n\nwatershed_model = model_zoo.bn_feature_net_skip_3D(\n fgbg_model=fgbg_model,\n receptive_field=receptive_field,\n n_skips=n_skips,\n n_features=distance_bins,\n n_frames=frames_per_batch,\n n_conv_filters=32,\n n_dense_filters=128,\n multires=False,\n last_only=False,\n input_shape=tuple([frames_per_batch] + list(X_train.shape[2:])),\n norm_method='whole_image')",
"_____no_output_____"
]
],
[
[
"#### Train the model",
"_____no_output_____"
]
],
[
[
"from deepcell.training import train_model_conv\n\nwatershed_model = train_model_conv(\n model=watershed_model,\n dataset=DATA_FILE, # full path to npz file\n model_name=conv_model_name,\n test_size=test_size,\n seed=seed,\n transform=transform,\n distance_bins=distance_bins,\n erosion_width=erosion_width,\n optimizer=optimizer,\n batch_size=batch_size,\n n_epoch=n_epoch,\n frames_per_batch=frames_per_batch,\n model_dir=MODEL_DIR,\n lr_sched=lr_sched,\n rotation_range=180,\n flip=True,\n shear=False,\n zoom_range=(0.8, 1.2))",
"X_train shape: (198, 15, 256, 256, 1)\ny_train shape: (198, 15, 256, 256, 1)\nX_test shape: (22, 15, 256, 256, 1)\ny_test shape: (22, 15, 256, 256, 1)\nOutput Shape: (None, 3, 256, 256, 4)\nNumber of Classes: 4\nTraining on 1 GPUs\nEpoch 1/10\n197/198 [============================>.] - ETA: 0s - loss: 3.8927 - model_5_loss: 0.9546 - model_6_loss: 0.9520 - model_7_loss: 0.9633 - model_8_loss: 0.9501 - model_5_acc: 0.8515 - model_6_acc: 0.8609 - model_7_acc: 0.8556 - model_8_acc: 0.8664\nEpoch 00001: val_loss improved from inf to 3.58243, saving model to /data/models/conv_watershed_3d_model.h5\n198/198 [==============================] - 171s 862ms/step - loss: 3.8903 - model_5_loss: 0.9541 - model_6_loss: 0.9513 - model_7_loss: 0.9626 - model_8_loss: 0.9497 - model_5_acc: 0.8516 - model_6_acc: 0.8611 - model_7_acc: 0.8557 - model_8_acc: 0.8664 - val_loss: 3.5824 - val_model_5_loss: 0.9999 - val_model_6_loss: 0.8246 - val_model_7_loss: 0.8505 - val_model_8_loss: 0.8347 - val_model_5_acc: 0.8552 - val_model_6_acc: 0.8806 - val_model_7_acc: 0.8899 - val_model_8_acc: 0.8820\nEpoch 2/10\n197/198 [============================>.] - ETA: 0s - loss: 3.2688 - model_5_loss: 0.7954 - model_6_loss: 0.8017 - model_7_loss: 0.8018 - model_8_loss: 0.7971 - model_5_acc: 0.8935 - model_6_acc: 0.8904 - model_7_acc: 0.8868 - model_8_acc: 0.8929\nEpoch 00002: val_loss improved from 3.58243 to 3.26194, saving model to /data/models/conv_watershed_3d_model.h5\n198/198 [==============================] - 144s 727ms/step - loss: 3.2674 - model_5_loss: 0.7951 - model_6_loss: 0.8014 - model_7_loss: 0.8014 - model_8_loss: 0.7967 - model_5_acc: 0.8936 - model_6_acc: 0.8904 - model_7_acc: 0.8869 - model_8_acc: 0.8930 - val_loss: 3.2619 - val_model_5_loss: 0.8245 - val_model_6_loss: 0.8164 - val_model_7_loss: 0.7766 - val_model_8_loss: 0.7716 - val_model_5_acc: 0.9008 - val_model_6_acc: 0.9094 - val_model_7_acc: 0.9056 - val_model_8_acc: 0.9073\nEpoch 3/10\n197/198 [============================>.] - ETA: 0s - loss: 3.2074 - model_5_loss: 0.7824 - model_6_loss: 0.7851 - model_7_loss: 0.7882 - model_8_loss: 0.7788 - model_5_acc: 0.8969 - model_6_acc: 0.8971 - model_7_acc: 0.8925 - model_8_acc: 0.8956\nEpoch 00003: val_loss improved from 3.26194 to 3.08470, saving model to /data/models/conv_watershed_3d_model.h5\n198/198 [==============================] - 143s 725ms/step - loss: 3.2081 - model_5_loss: 0.7827 - model_6_loss: 0.7852 - model_7_loss: 0.7883 - model_8_loss: 0.7790 - model_5_acc: 0.8967 - model_6_acc: 0.8970 - model_7_acc: 0.8924 - model_8_acc: 0.8955 - val_loss: 3.0847 - val_model_5_loss: 0.7503 - val_model_6_loss: 0.7541 - val_model_7_loss: 0.7527 - val_model_8_loss: 0.7546 - val_model_5_acc: 0.9105 - val_model_6_acc: 0.9094 - val_model_7_acc: 0.8988 - val_model_8_acc: 0.9046\nEpoch 4/10\n197/198 [============================>.] - ETA: 0s - loss: 3.2189 - model_5_loss: 0.7860 - model_6_loss: 0.8026 - model_7_loss: 0.7803 - model_8_loss: 0.7770 - model_5_acc: 0.8933 - model_6_acc: 0.8874 - model_7_acc: 0.8916 - model_8_acc: 0.8949\nEpoch 00004: val_loss did not improve from 3.08470\n198/198 [==============================] - 143s 723ms/step - loss: 3.2174 - model_5_loss: 0.7857 - model_6_loss: 0.8021 - model_7_loss: 0.7800 - model_8_loss: 0.7767 - model_5_acc: 0.8933 - model_6_acc: 0.8874 - model_7_acc: 0.8916 - model_8_acc: 0.8950 - val_loss: 3.0984 - val_model_5_loss: 0.7471 - val_model_6_loss: 0.7796 - val_model_7_loss: 0.7491 - val_model_8_loss: 0.7495 - val_model_5_acc: 0.9032 - val_model_6_acc: 0.9134 - val_model_7_acc: 0.8882 - val_model_8_acc: 0.9122\nEpoch 5/10\n197/198 [============================>.] - ETA: 0s - loss: 3.1495 - model_5_loss: 0.7716 - model_6_loss: 0.7765 - model_7_loss: 0.7652 - model_8_loss: 0.7632 - model_5_acc: 0.9012 - model_6_acc: 0.8977 - model_7_acc: 0.8981 - model_8_acc: 0.9031\nEpoch 00005: val_loss improved from 3.08470 to 3.01958, saving model to /data/models/conv_watershed_3d_model.h5\n198/198 [==============================] - 144s 726ms/step - loss: 3.1469 - model_5_loss: 0.7710 - model_6_loss: 0.7758 - model_7_loss: 0.7644 - model_8_loss: 0.7626 - model_5_acc: 0.9012 - model_6_acc: 0.8977 - model_7_acc: 0.8981 - model_8_acc: 0.9031 - val_loss: 3.0196 - val_model_5_loss: 0.7375 - val_model_6_loss: 0.7557 - val_model_7_loss: 0.7238 - val_model_8_loss: 0.7295 - val_model_5_acc: 0.8905 - val_model_6_acc: 0.9011 - val_model_7_acc: 0.8767 - val_model_8_acc: 0.8719\nEpoch 6/10\n197/198 [============================>.] - ETA: 0s - loss: 3.0814 - model_5_loss: 0.7565 - model_6_loss: 0.7578 - model_7_loss: 0.7482 - model_8_loss: 0.7457 - model_5_acc: 0.8979 - model_6_acc: 0.8955 - model_7_acc: 0.8965 - model_8_acc: 0.9008\nEpoch 00006: val_loss improved from 3.01958 to 2.92890, saving model to /data/models/conv_watershed_3d_model.h5\n198/198 [==============================] - 144s 725ms/step - loss: 3.0772 - model_5_loss: 0.7555 - model_6_loss: 0.7567 - model_7_loss: 0.7472 - model_8_loss: 0.7447 - model_5_acc: 0.8981 - model_6_acc: 0.8957 - model_7_acc: 0.8967 - model_8_acc: 0.9009 - val_loss: 2.9289 - val_model_5_loss: 0.7209 - val_model_6_loss: 0.7166 - val_model_7_loss: 0.7114 - val_model_8_loss: 0.7069 - val_model_5_acc: 0.9053 - val_model_6_acc: 0.9081 - val_model_7_acc: 0.8920 - val_model_8_acc: 0.9041\nEpoch 7/10\n197/198 [============================>.] - ETA: 0s - loss: 3.0843 - model_5_loss: 0.7582 - model_6_loss: 0.7587 - model_7_loss: 0.7492 - model_8_loss: 0.7450 - model_5_acc: 0.9002 - model_6_acc: 0.8977 - model_7_acc: 0.8987 - model_8_acc: 0.9020\nEpoch 00007: val_loss did not improve from 2.92890\n198/198 [==============================] - 143s 722ms/step - loss: 3.0841 - model_5_loss: 0.7582 - model_6_loss: 0.7586 - model_7_loss: 0.7492 - model_8_loss: 0.7449 - model_5_acc: 0.9003 - model_6_acc: 0.8979 - model_7_acc: 0.8989 - model_8_acc: 0.9022 - val_loss: 3.0507 - val_model_5_loss: 0.7429 - val_model_6_loss: 0.7490 - val_model_7_loss: 0.7422 - val_model_8_loss: 0.7434 - val_model_5_acc: 0.8986 - val_model_6_acc: 0.8998 - val_model_7_acc: 0.8944 - val_model_8_acc: 0.9109\nEpoch 8/10\n197/198 [============================>.] - ETA: 0s - loss: 3.0380 - model_5_loss: 0.7474 - model_6_loss: 0.7455 - model_7_loss: 0.7375 - model_8_loss: 0.7344 - model_5_acc: 0.8997 - model_6_acc: 0.8984 - model_7_acc: 0.8992 - model_8_acc: 0.9030\nEpoch 00008: val_loss did not improve from 2.92890\n198/198 [==============================] - 143s 724ms/step - loss: 3.0375 - model_5_loss: 0.7472 - model_6_loss: 0.7455 - model_7_loss: 0.7374 - model_8_loss: 0.7342 - model_5_acc: 0.8996 - model_6_acc: 0.8983 - model_7_acc: 0.8991 - model_8_acc: 0.9030 - val_loss: 3.0694 - val_model_5_loss: 0.7353 - val_model_6_loss: 0.7731 - val_model_7_loss: 0.7532 - val_model_8_loss: 0.7347 - val_model_5_acc: 0.8916 - val_model_6_acc: 0.8562 - val_model_7_acc: 0.8855 - val_model_8_acc: 0.8808\nEpoch 9/10\n197/198 [============================>.] - ETA: 0s - loss: 3.0477 - model_5_loss: 0.7486 - model_6_loss: 0.7477 - model_7_loss: 0.7391 - model_8_loss: 0.7390 - model_5_acc: 0.9000 - model_6_acc: 0.8975 - model_7_acc: 0.8999 - model_8_acc: 0.9026\nEpoch 00009: val_loss improved from 2.92890 to 2.91570, saving model to /data/models/conv_watershed_3d_model.h5\n198/198 [==============================] - 144s 726ms/step - loss: 3.0471 - model_5_loss: 0.7485 - model_6_loss: 0.7474 - model_7_loss: 0.7390 - model_8_loss: 0.7390 - model_5_acc: 0.9001 - model_6_acc: 0.8975 - model_7_acc: 0.9000 - model_8_acc: 0.9027 - val_loss: 2.9157 - val_model_5_loss: 0.7177 - val_model_6_loss: 0.7192 - val_model_7_loss: 0.7003 - val_model_8_loss: 0.7052 - val_model_5_acc: 0.8953 - val_model_6_acc: 0.9110 - val_model_7_acc: 0.9087 - val_model_8_acc: 0.8886\nEpoch 10/10\n197/198 [============================>.] - ETA: 0s - loss: 3.0652 - model_5_loss: 0.7553 - model_6_loss: 0.7531 - model_7_loss: 0.7425 - model_8_loss: 0.7411 - model_5_acc: 0.9027 - model_6_acc: 0.9017 - model_7_acc: 0.9013 - model_8_acc: 0.9044\nEpoch 00010: val_loss improved from 2.91570 to 2.90629, saving model to /data/models/conv_watershed_3d_model.h5\n198/198 [==============================] - 144s 726ms/step - loss: 3.0634 - model_5_loss: 0.7548 - model_6_loss: 0.7526 - model_7_loss: 0.7420 - model_8_loss: 0.7407 - model_5_acc: 0.9026 - model_6_acc: 0.9016 - model_7_acc: 0.9012 - model_8_acc: 0.9043 - val_loss: 2.9063 - val_model_5_loss: 0.7080 - val_model_6_loss: 0.7071 - val_model_7_loss: 0.7149 - val_model_8_loss: 0.7030 - val_model_5_acc: 0.9048 - val_model_6_acc: 0.9090 - val_model_7_acc: 0.9118 - val_model_8_acc: 0.9021\n"
]
],
[
[
"### Run the model\n\nThe model was trained on only a `frames_per_batch` frames at a time. In order to run this data on a full set of frames, a new model must be instantiated, which will load the trained weights.\n\n#### Save weights of trained models",
"_____no_output_____"
]
],
[
[
"fgbg_weights_file = os.path.join(MODEL_DIR, '{}.h5'.format(fgbg_model_name))\nfgbg_model.save_weights(fgbg_weights_file)\n\nwatershed_weights_file = os.path.join(MODEL_DIR, '{}.h5'.format(conv_model_name))\nwatershed_model.save_weights(watershed_weights_file)",
"_____no_output_____"
]
],
[
[
"#### Initialize the new models",
"_____no_output_____"
]
],
[
[
"from deepcell import model_zoo\n\n# All training parameters should match except for the `input_shape`\n\nrun_fgbg_model = model_zoo.bn_feature_net_skip_3D(\n receptive_field=receptive_field,\n n_features=2,\n n_frames=frames_per_batch,\n n_skips=n_skips,\n n_conv_filters=32,\n n_dense_filters=128,\n input_shape=tuple(X_test.shape[1:]),\n multires=False,\n last_only=False,\n norm_method=norm_method)\nrun_fgbg_model.load_weights(fgbg_weights_file)\n\nrun_watershed_model = model_zoo.bn_feature_net_skip_3D(\n fgbg_model=run_fgbg_model,\n receptive_field=receptive_field,\n n_skips=n_skips,\n n_features=distance_bins,\n n_frames=frames_per_batch,\n n_conv_filters=32,\n n_dense_filters=128,\n multires=False,\n last_only=False,\n input_shape=tuple(X_test.shape[1:]),\n norm_method=norm_method)\nrun_watershed_model.load_weights(watershed_weights_file)",
"_____no_output_____"
],
[
"# too many batches at once causes OOM\nX_test, y_test = X_test[:4], y_test[:4]\nprint(X_test.shape)",
"(4, 15, 256, 256, 1)\n"
]
],
[
[
"#### Make predictions on test data",
"_____no_output_____"
]
],
[
[
"test_images = run_watershed_model.predict(X_test)[-1]\ntest_images_fgbg = run_fgbg_model.predict(X_test)[-1]\n\nprint('watershed transform shape:', test_images.shape)\nprint('segmentation mask shape:', test_images_fgbg.shape)",
"watershed transform shape: (4, 15, 256, 256, 4)\nsegmentation mask shape: (4, 15, 256, 256, 2)\n"
]
],
[
[
"#### Watershed post-processing",
"_____no_output_____"
]
],
[
[
"argmax_images = []\nfor i in range(test_images.shape[0]):\n max_image = np.argmax(test_images[i], axis=-1)\n argmax_images.append(max_image)\nargmax_images = np.array(argmax_images)\nargmax_images = np.expand_dims(argmax_images, axis=-1)\n\nprint('watershed argmax shape:', argmax_images.shape)",
"watershed argmax shape: (4, 15, 256, 256, 1)\n"
],
[
"# threshold the foreground/background\n# and remove back ground from watershed transform\nthreshold = 0.5\n\nfg_thresh = test_images_fgbg[..., 1] > threshold\nfg_thresh = np.expand_dims(fg_thresh, axis=-1)\n\nargmax_images_post_fgbg = argmax_images * fg_thresh",
"_____no_output_____"
],
[
"# Apply watershed method with the distance transform as seed\nfrom skimage.measure import label\nfrom skimage.morphology import watershed\nfrom skimage.feature import peak_local_max\n\nwatershed_images = []\nfor i in range(argmax_images_post_fgbg.shape[0]):\n image = fg_thresh[i, ..., 0]\n distance = argmax_images_post_fgbg[i, ..., 0]\n\n local_maxi = peak_local_max(\n test_images[i, ..., -1],\n min_distance=10,\n threshold_abs=0.05,\n indices=False,\n labels=image,\n exclude_border=False)\n\n markers = label(local_maxi)\n segments = watershed(-distance, markers, mask=image)\n watershed_images.append(segments)\n\nwatershed_images = np.array(watershed_images)\nwatershed_images = np.expand_dims(watershed_images, axis=-1)",
"_____no_output_____"
]
],
[
[
"### Plot the results",
"_____no_output_____"
]
],
[
[
"import matplotlib.pyplot as plt\nimport matplotlib.animation as animation\n\nindex = np.random.randint(low=0, high=watershed_images.shape[0])\nframe = np.random.randint(low=0, high=watershed_images.shape[1])\n\nprint('Image:', index)\nprint('Frame:', frame)\n\nfig, axes = plt.subplots(ncols=3, nrows=2, figsize=(15, 15), sharex=True, sharey=True)\nax = axes.ravel()\n\nax[0].imshow(X_test[index, frame, ..., 0])\nax[0].set_title('Source Image')\n\nax[1].imshow(test_images_fgbg[index, frame, ..., 1])\nax[1].set_title('FGBG Prediction')\n\nax[2].imshow(fg_thresh[index, frame, ..., 0], cmap='jet')\nax[2].set_title('FGBG {}% Threshold'.format(int(threshold * 100)))\n\nax[3].imshow(argmax_images[index, frame, ..., 0], cmap='jet')\nax[3].set_title('Distance Transform')\n\nax[4].imshow(argmax_images_post_fgbg[index, frame, ..., 0], cmap='jet')\nax[4].set_title('Distance Transform w/o Background')\n\nax[5].imshow(watershed_images[index, frame, ..., 0], cmap='jet')\nax[5].set_title('Watershed Segmentation')\n\nfig.tight_layout()\nplt.show()",
"Image: 2\nFrame: 7\n"
],
[
"# Can also export as a video\n# But this does not render well on GitHub\nfrom IPython.display import HTML\nfrom deepcell.utils.plot_utils import get_js_video\n\nHTML(get_js_video(watershed_images[..., [-1]], batch=index))",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
]
] |
e7e8c9520dac5e603f7cb78436cf9fdf5b14e64d | 173,507 | ipynb | Jupyter Notebook | prep-4-matplotlib/matplot2-figure-objects.ipynb | luiul/statistics-meets-logistics | 44a13558a8fcb4fab5add3616c818470aae2922e | [
"MIT"
] | 4 | 2020-12-31T12:03:03.000Z | 2021-11-09T19:24:33.000Z | prep-4-matplotlib/matplot2-figure-objects.ipynb | luiul/stats-meets-log | 44a13558a8fcb4fab5add3616c818470aae2922e | [
"MIT"
] | null | null | null | prep-4-matplotlib/matplot2-figure-objects.ipynb | luiul/stats-meets-log | 44a13558a8fcb4fab5add3616c818470aae2922e | [
"MIT"
] | 1 | 2021-01-08T11:59:49.000Z | 2021-01-08T11:59:49.000Z | 657.223485 | 38,308 | 0.757825 | [
[
[
"import numpy as np\nimport pandas as pd \nimport matplotlib.pyplot as plt\npd.set_option('display.max_columns', None)",
"_____no_output_____"
],
[
"a = np.linspace(0,10,11)\nb = a**4",
"_____no_output_____"
],
[
"a",
"_____no_output_____"
],
[
"b",
"_____no_output_____"
],
[
"x = np.arange(0,10)",
"_____no_output_____"
],
[
"y = 2*x",
"_____no_output_____"
],
[
"fig = plt.figure()\n# the default figure is size x*y and has zero axes\n# most important parameters are the figsize and dpi\n\naxes = fig.add_axes([0,0,1,1])\n\naxes.plot(a,b);",
"_____no_output_____"
],
[
"fig = plt.figure()\n\n# large axes\naxes1 = fig.add_axes([0,0,1,1])\naxes1.plot(a,b)\n\n# small axes\naxes2 = fig.add_axes([0.2,0.2,1,0.5])",
"_____no_output_____"
],
[
"fig = plt.figure()\n\n# large axes\naxes1 = fig.add_axes([0,0,1,1])\naxes1.plot(a,b)\n\n# small axes\naxes2 = fig.add_axes([0.2,0.5,0.25,0.25])\naxes2.plot(x,y)",
"_____no_output_____"
],
[
"fig = plt.figure()\n\n# large axes\naxes1 = fig.add_axes([0,0,1,1])\naxes1.plot(a,b)\n\naxes1.set_xlim(0,8)\naxes1.set_ylim(0,8000)\naxes1.set_xlabel('A')\naxes1.set_ylabel('B')\naxes1.set_title('Power of Four')\n\n# small axes\naxes2 = fig.add_axes([1,1,2,2])\n# useful for legends outside of the plot\n\naxes2.plot(a,b)\n\naxes2.set_xlim(1,2)\naxes2.set_ylim(0,18)\naxes2.set_xlabel('A')\naxes2.set_ylabel('B')\naxes2.set_title('Zoomed In')",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e8fa300212a9c19466d5b7addaa4c21c990484 | 26,134 | ipynb | Jupyter Notebook | Python Pandas Tutorials 06.ipynb | HenriqueArgentieri/Tutoriais | e15e279223828f3c8b19e884eeb45db1fd2ace18 | [
"MIT"
] | null | null | null | Python Pandas Tutorials 06.ipynb | HenriqueArgentieri/Tutoriais | e15e279223828f3c8b19e884eeb45db1fd2ace18 | [
"MIT"
] | null | null | null | Python Pandas Tutorials 06.ipynb | HenriqueArgentieri/Tutoriais | e15e279223828f3c8b19e884eeb45db1fd2ace18 | [
"MIT"
] | null | null | null | 28.192017 | 113 | 0.317977 | [
[
[
"Tutorial 6 - Handle Missing Data replace function",
"_____no_output_____"
]
],
[
[
"import pandas as pd\nimport numpy as np",
"_____no_output_____"
],
[
"df = pd.read_csv('sample_data_tutorial_06.csv')\ndf",
"_____no_output_____"
],
[
"newdf = df.replace(-99999,np.NaN)\nnewdf",
"_____no_output_____"
],
[
"newdf = df.replace([-99999, -88888],np.NaN)\nnewdf",
"_____no_output_____"
],
[
"newdf = df.replace({\n 'temperature': -99999,\n 'windspeed': [-99999, -88888],\n 'event': 'No event'\n }, np.NaN)\nnewdf",
"_____no_output_____"
],
[
"# Podemos gerar um mapa das alterações que queremos fazer:\nnewdf = df.replace({\n -99999: np.NaN,\n -88888: np.NaN,\n 'No event': 'Sunny'\n})\nnewdf",
"_____no_output_____"
],
[
"# Importando outro csv com algumas unidades que precisam ser limpas!\ndf = pd.read_csv('sample_data_tutorial_06a.csv')\ndf",
"_____no_output_____"
],
[
"# É necessário usar o 'regex' (regular expression)\n# No caso abaixo estamos substituindo todas as letras (de A a Z - maiúscula e minúscula) por vazio (='')\nnewdf = df.replace('[A-Za-z]', '', regex=True)\nnewdf",
"_____no_output_____"
],
[
"# Observe, no caso anterior, que ele removeu o que pedimos mas também removeu toda coluna 'event'\n# Para fazer as substituições somente em determinadas colunas é preciso utilizar o dicionário:\nnewdf = df.replace({\n 'temperature': '[A-Za-z]', \n 'windspeed': '[A-Za-z]'\n}, '', regex=True)\nnewdf",
"_____no_output_____"
]
]
] | [
"raw",
"code"
] | [
[
"raw"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e90027bee46027068c9e105358c156e9f94e46 | 29,841 | ipynb | Jupyter Notebook | file_parse.ipynb | ivanlohvyn/beetroot_parse_pdf | a00c121282e4e9b327a7db856f948d93983facd7 | [
"MIT"
] | null | null | null | file_parse.ipynb | ivanlohvyn/beetroot_parse_pdf | a00c121282e4e9b327a7db856f948d93983facd7 | [
"MIT"
] | null | null | null | file_parse.ipynb | ivanlohvyn/beetroot_parse_pdf | a00c121282e4e9b327a7db856f948d93983facd7 | [
"MIT"
] | null | null | null | 32.121636 | 652 | 0.45843 | [
[
[
"from pdfminer.high_level import extract_text\n\nwith open('/home/azashiro/Desktop/beetroot_task/task.pdf','rb') as file:\n theses = extract_text(file)\n t = \" \".join(theses.split())\n plain_text = \"\".join(t)\n #print(plain_text)",
"_____no_output_____"
],
[
"import re\n\ntext = re.sub(r'Poster abstracts\\s\\d', \"\", plain_text)\ntext = re.sub(r'Poster abstractsActa Derm Venereol 2018\\s\\d+', \"\", text)\ntext = re.sub(r'www.medicaljournals.se/acta5th World Psoriasis & Psoriatic Arthritis Conference 2018', '', text)\ntext = re.sub(r'\\w-\\s', '', text)\ntext = re.sub(r'\\xad', '', text)\n#print(text)",
"_____no_output_____"
],
[
"pattern = re.compile(r\"(P\\d{3})\")\ntext_list = []\ntext_list = re.split(pattern, text)\ntext_list.remove(text_list[0]) # remove page number\n#print(text_list)",
"_____no_output_____"
],
[
"def convert_text_to_dictionary(text):\n it = iter(text)\n create_dict = dict(zip(it, it))\n return create_dict\n\ntext_dict = convert_text_to_dictionary(text_list)\n#print(text_dict)",
"_____no_output_____"
]
],
[
[
"PARSE SINGLE ABSTRACT WITH NON-INDEXED AUTHORS LIST",
"_____no_output_____"
]
],
[
[
"abstract = text_dict['P123']\n#abstract",
"_____no_output_____"
],
[
"abstract_info = re.findall(r\"\\w+[A-Z\\w+]\\w+.*(?=TNF\\stherapy.*)\", abstract)\nabstract_head = str(abstract_info[0])\nabstract_head",
"_____no_output_____"
],
[
"authors_info = re.findall(r\"\\w+[^A-Z\\d)\\W]\\s\\w.*(?=TNF\\stherapy*)\", abstract)\nauthors = str(authors_info[0])\nauthors",
"_____no_output_____"
],
[
"author_name = re.findall(r\"\\w.+(?=Spherix)\", authors)\nauthor_name\nauthor_list = [x for x in author_name[0].split(',')]\nauthor_list",
"_____no_output_____"
],
[
"author_location = re.findall(r\"Spherix[^*]+\", authors)\nauthor_location",
"_____no_output_____"
],
[
"pattern = re.compile(r\"\\w+[^A-Z\\d\\W]\\s\\w.*\")\nabstract_title = [re.sub(pattern, \"\", i) for i in abstract_info]\nabstract_title",
"_____no_output_____"
],
[
"abstract_text = re.findall(r\"(TNF\\stherapy.*)\", abstract)\n#abstract_text",
"_____no_output_____"
],
[
"import pandas as pd\n\ndf = pd.DataFrame({\"About the person\": 'Name (incl. titles if any mentioned)',\n \"Unnamed: 1\": 'Affiliation(s) Name(s)',\n \"Unnamed: 2\": \"Person's Location\",\n \"About the session/topic\": \"Session Name\",\n \"Unnamed: 4\": 'Topic Title',\n \"Unnamed: 5\": 'Presentation Abstract'}, index=[0])",
"_____no_output_____"
],
[
"df1 = pd.DataFrame({\"About the person\": author_list[2],\n \"Unnamed: 1\": author_location,\n \"Unnamed: 2\": \"\",\n \"About the session/topic\": \"P123\",\n \"Unnamed: 4\": abstract_title,\n \"Unnamed: 5\": abstract_text})\ndf1",
"_____no_output_____"
]
],
[
[
"PARSE SINGLE ABSTRACT WITH INDEXED AUTHORS LIST",
"_____no_output_____"
]
],
[
[
"abstract = text_dict['P120']\n#abstract",
"_____no_output_____"
],
[
"abstract_info = re.findall(r\"\\w+[A-Z\\w+]\\w+.*(?=Introduction.*)\", abstract)\nabstract_head = str(abstract_info[0])\nabstract_head",
"_____no_output_____"
],
[
"authors_info = re.findall(r\"\\w+[^A-Z\\d)\\W]\\s\\w.*(?=Introduction.*)\", abstract)\nauthors = str(authors_info[0])\nauthors",
"_____no_output_____"
],
[
"author_name = re.findall(r\"(\\w+.\\s[a-zA-z\\s-]+\\d)\", authors)\nauthor_name",
"_____no_output_____"
],
[
"author_location = re.findall(r\"(\\d\\w+\\W[a-zA-z-'\\s&,\\s.,(A-Z)]+)\", authors)\nauthor_location",
"_____no_output_____"
],
[
"import string\nfrom collections import namedtuple\n\nDigitGroup = namedtuple('DigitGroup', ['keys', 'values'])\n\ndef combine(all_keys, all_values):\n by_digit = {}\n\n for word in all_keys:\n for char in word:\n if char in string.digits:\n group = by_digit.get(char)\n if not group:\n group = DigitGroup(word, [])\n by_digit[char] = group\n break\n\n for word in all_values:\n for char in word:\n if char in string.digits:\n group = by_digit[char]\n group.values.append(word)\n break\n \n return dict(by_digit.values())",
"_____no_output_____"
],
[
"combined_dict = combine(author_location, author_name)\ncombined_dict",
"_____no_output_____"
],
[
"list_of_dict = [{k: v} for k, v in combined_dict.items()]\nlist_of_dict",
"_____no_output_____"
],
[
"import itertools\n\ni = list_of_dict[6]\nget_key = i.keys()\nnames = [] \nfor key, value in ( \n itertools.chain.from_iterable( \n [itertools.product((k, ), v) for k, v in i.items()])): \n names.append(value)\nnames",
"_____no_output_____"
],
[
"name = [re.sub(r'[0-9]', '', i) for i in names]\nprint(name)\nlocation = [re.sub(r'[0-9]', '', i) for i in get_key]\nprint(location)",
"['Kristian Reich']\n['SCIderm Research Institute and Dermatologikum Hamburg, Haburg, Germany ']\n"
],
[
"pattern = re.compile(r\"\\w+[^A-Z\\d\\W]\\s\\w.*\")\nabstract_title = [re.sub(pattern, \"\", i) for i in abstract_info]\nabstract_title",
"_____no_output_____"
],
[
"abstract_text = re.findall(r\"(Introduction.*)\", abstract)\n#abstract_text",
"_____no_output_____"
],
[
"import pandas as pd\ndf2 = pd.DataFrame({\"About the person\": name[0],\n \"Unnamed: 1\": location,\n \"Unnamed: 2\": \"\",\n \"About the session/topic\": \"P120\",\n \"Unnamed: 4\": abstract_title,\n \"Unnamed: 5\": abstract_text})\ndf2",
"_____no_output_____"
],
[
"df = pd.read_excel('/home/azashiro/Desktop/Datas.xlsx')",
"_____no_output_____"
],
[
"df73 = df72.append(df2, ignore_index=True)\ndf73",
"_____no_output_____"
],
[
"'''Import pandas DataFrame into Excel file'''\n\nexcel_file = df73.to_excel(\"/home/azashiro/Desktop/beetroot_task/Datas.xlsx\", index=False)\nexcel_file",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e9167c1d038757749216bd968b6e296ffec7fe | 2,951 | ipynb | Jupyter Notebook | examples/ch06/snippets_ipynb/06.03.02selfcheck.ipynb | germanngc/PythonFundamentals | 14d22baa30d7c3c5404fc11362709669e92474b8 | [
"Apache-2.0"
] | null | null | null | examples/ch06/snippets_ipynb/06.03.02selfcheck.ipynb | germanngc/PythonFundamentals | 14d22baa30d7c3c5404fc11362709669e92474b8 | [
"Apache-2.0"
] | null | null | null | examples/ch06/snippets_ipynb/06.03.02selfcheck.ipynb | germanngc/PythonFundamentals | 14d22baa30d7c3c5404fc11362709669e92474b8 | [
"Apache-2.0"
] | null | null | null | 27.073394 | 156 | 0.468994 | [
[
[
"\n# 6.3.2 Self Check",
"_____no_output_____"
],
[
"**2. _(IPython Session)_** Given the sets `{10, 20, 30}` and `{5, 10, 15, 20}` use the mathematical set operators to produce the following results:\n\n**a.** `{30}`\n \n**b.** `{5, 15, 30}` \n\n**c.** `{5, 10, 15, 20, 30}` \n\n**d.** `{10, 20}`\n\n**Answer:**",
"_____no_output_____"
]
],
[
[
"{10, 20, 30} - {5, 10, 15, 20}",
"_____no_output_____"
],
[
"{10, 20, 30} ^ {5, 10, 15, 20}",
"_____no_output_____"
],
[
"{10, 20, 30} | {5, 10, 15, 20}",
"_____no_output_____"
],
[
"{10, 20, 30} & {5, 10, 15, 20}",
"_____no_output_____"
],
[
"##########################################################################\n# (C) Copyright 2019 by Deitel & Associates, Inc. and #\n# Pearson Education, Inc. All Rights Reserved. #\n# #\n# DISCLAIMER: The authors and publisher of this book have used their #\n# best efforts in preparing the book. These efforts include the #\n# development, research, and testing of the theories and programs #\n# to determine their effectiveness. The authors and publisher make #\n# no warranty of any kind, expressed or implied, with regard to these #\n# programs or to the documentation contained in these books. The authors #\n# and publisher shall not be liable in any event for incidental or #\n# consequential damages in connection with, or arising out of, the #\n# furnishing, performance, or use of these programs. #\n##########################################################################\n",
"_____no_output_____"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7e91ede96d65783cfc085c50d2fe695c66338c3 | 49,606 | ipynb | Jupyter Notebook | notebook/jupyter_youtube_vimeo.ipynb | puyopop/python-snippets | 9d70aa3b2a867dd22f5a5e6178a5c0c5081add73 | [
"MIT"
] | 174 | 2018-05-30T21:14:50.000Z | 2022-03-25T07:59:37.000Z | notebook/jupyter_youtube_vimeo.ipynb | puyopop/python-snippets | 9d70aa3b2a867dd22f5a5e6178a5c0c5081add73 | [
"MIT"
] | 5 | 2019-08-10T03:22:02.000Z | 2021-07-12T20:31:17.000Z | notebook/jupyter_youtube_vimeo.ipynb | puyopop/python-snippets | 9d70aa3b2a867dd22f5a5e6178a5c0c5081add73 | [
"MIT"
] | 53 | 2018-04-27T05:26:35.000Z | 2022-03-25T07:59:37.000Z | 306.209877 | 15,353 | 0.931823 | [
[
[
"import IPython.display",
"_____no_output_____"
],
[
"IPython.display.YouTubeVideo('6XvmhE1J9PY')",
"_____no_output_____"
],
[
"IPython.display.YouTubeVideo('6XvmhE1J9PY', width=480, height=270)",
"_____no_output_____"
],
[
"IPython.display.YouTubeVideo('6XvmhE1J9PY', start=30)",
"_____no_output_____"
],
[
"IPython.display.VimeoVideo('289502328')",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7e9229402bfeb6d51bf990b75e02c85bd95ddbf | 7,511 | ipynb | Jupyter Notebook | 004_Python_Nested_if_statement.ipynb | chen181016/03_Python_Flow_Control | 6b3a3c05235cdb5981db487dbaaf2e9c2a838fb5 | [
"MIT"
] | 3 | 2021-09-23T05:44:55.000Z | 2021-12-26T13:36:52.000Z | 004_Python_Nested_if_statement.ipynb | Dengjinjing04/03_Python_Flow_Control | a6678a73bc591bb58c3b4c4a2585dca2dac67da1 | [
"MIT"
] | null | null | null | 004_Python_Nested_if_statement.ipynb | Dengjinjing04/03_Python_Flow_Control | a6678a73bc591bb58c3b4c4a2585dca2dac67da1 | [
"MIT"
] | null | null | null | 24.62623 | 453 | 0.512582 | [
[
[
"<small><small><i>\nAll the IPython Notebooks in this lecture series by Dr. Milan Parmar are available @ **[GitHub](https://github.com/milaan9/03_Python_Flow_Control)**\n</i></small></small>",
"_____no_output_____"
],
[
"# Python Nested `if` statement\n\nWe can have a nested-**[if-else](https://github.com/milaan9/03_Python_Flow_Control/blob/main/002_Python_if_else_statement.ipynb)** or nested-**[if-elif-else](https://github.com/milaan9/03_Python_Flow_Control/blob/main/003_Python_if_elif_else_statement%20.ipynb)** statement inside another **`if-else`** statement. This is called **nesting** in computer programming. The nested if statements is useful when we want to make a series of decisions.\n\nAny number of these statements can be nested inside one another. Indentation is the only way to figure out the level of nesting. They can get confusing, so they must be avoided unless necessary.\n\nWe can use nested if statements for situations where we want to check for a **secondary condition** if the first condition executes as **`True`**.",
"_____no_output_____"
],
[
"### Syntax:\n\n#### Example 1:\n```python\nif conditon_outer:\n if condition_inner:\n statement of nested if\n else:\n statement of nested if else:\n statement ot outer if\nelse:\n Outer else \nstatement outside if block\n```\n\n#### Example 2:\n```python\nif expression1:\n statement(s)\n if expression2:\n statement(s)\n elif expression3:\n statement(s)\n elif expression4:\n statement(s)\n else:\n statement(s)\nelse:\n statement(s)\n```",
"_____no_output_____"
]
],
[
[
"# Example 1:\n\na=10\nif a>=20: # Condition FALSE\n print (\"Condition is True\")\nelse: # Code will go to ELSE body\n if a>=15: # Condition FALSE\n print (\"Checking second value\")\n else: # Code will go to ELSE body\n print (\"All Conditions are false\")",
"All Conditions are false\n"
],
[
"# Example 2:\n\nx = 10\ny = 12\nif x > y:\n print( \"x>y\")\nelif x < y:\n print( \"x<y\")\n if x==10:\n print (\"x=10\")\n else:\n print (\"invalid\")\nelse:\n print (\"x=y\")",
"x<y\nx=10\n"
],
[
"# Example 3:\n\nnum1 = 0\nif (num1 != 0): # For zero condition is FALSE\n if(num1 > 0): \n print(\"num1 is a positive number\")\n else: \n print(\"num1 is a negative number\")\nelse: # For zero condition is TRUE\n print(\"num1 is neither positive nor negative\")",
"num1 is neither positive nor negative\n"
],
[
"# Example 4:\n\n'''In this program, we input a number check if the number is \npositive or negative or zero and display an appropriate message. \nThis time we use nested if statement'''\n\nnum = float(input(\"Enter a number: \"))\nif num >= 0:\n if num == 0:\n print(\"Zero\")\n else:\n print(\"Positive number\")\nelse:\n print(\"Negative number\")",
"Enter a number: 9\nPositive number\n"
],
[
"# Example 5:\n\ndef number_arithmetic(num1, num2):\n if num1 >= num2:\n if num1 == num2:\n print(f'{num1} and {num2} are equal')\n else:\n print(f'{num1} is greater than {num2}')\n else:\n print(f'{num1} is smaller than {num2}')\n\nnumber_arithmetic(96, 66)\n# Output 96 is greater than 66\nnumber_arithmetic(96, 96)\n# Output 56 and 56 are equal",
"96 is greater than 66\n96 and 96 are equal\n"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7e92b2176d49a80f7178ebd90123f9134162b31 | 70,273 | ipynb | Jupyter Notebook | notebooks/Original_U-Net_PyTorch.ipynb | jimmiemunyi/fastai-experiments | fed54fe547f738ca80556bb27f840a40b8a2c073 | [
"MIT"
] | 2 | 2021-09-09T07:10:50.000Z | 2021-11-18T21:40:11.000Z | notebooks/Original_U-Net_PyTorch.ipynb | jimmiemunyi/fastai-experiments | fed54fe547f738ca80556bb27f840a40b8a2c073 | [
"MIT"
] | null | null | null | notebooks/Original_U-Net_PyTorch.ipynb | jimmiemunyi/fastai-experiments | fed54fe547f738ca80556bb27f840a40b8a2c073 | [
"MIT"
] | null | null | null | 196.843137 | 58,774 | 0.88058 | [
[
[
"<a href=\"https://colab.research.google.com/github/jimmiemunyi/deeplearning-experiments/blob/main/notebooks/Original_U-Net_PyTorch.ipynb\" target=\"_parent\"><img src=\"https://colab.research.google.com/assets/colab-badge.svg\" alt=\"Open In Colab\"/></a>\n",
"_____no_output_____"
]
],
[
[
"import torch\nimport torch.nn as nn\nimport torchvision.transforms.functional as TF",
"_____no_output_____"
]
],
[
[
"## The Original U-Net Architecture",
"_____no_output_____"
],
[
"",
"_____no_output_____"
],
[
"Defining the double convolution block:",
"_____no_output_____"
]
],
[
[
"def conv_block(ni, nf):\n return nn.Sequential(\n nn.Conv2d(ni, nf, kernel_size=3, stride=1),\n nn.ReLU(inplace=True),\n nn.Conv2d(nf, nf, kernel_size=3, stride=1),\n nn.ReLU(inplace=True)\n )",
"_____no_output_____"
]
],
[
[
"Implementing the origal architecture:",
"_____no_output_____"
]
],
[
[
"class UNET(nn.Module):\n def __init__(self, in_channels=1, out_channels=1, \n features = [64, 128, 256, 512]):\n super(UNET, self).__init__()\n\n self.encoder = nn.ModuleList()\n self.pool = nn.MaxPool2d(kernel_size=2, stride=2)\n\n # create the contracting path (encoder + bottleneck)\n for feature in features:\n self.encoder.append(conv_block(in_channels, feature))\n in_channels = feature\n self.bottleneck = conv_block(features[-1], features[-1]*2)\n\n # create the expansive path\n self.decoder = nn.ModuleList()\n # reversed because we want to create from last to first\n for feature in reversed(features):\n self.decoder.append(\n nn.Sequential(\n nn.ConvTranspose2d(feature*2, feature, kernel_size=2, stride=2),\n conv_block(feature*2, feature)\n )\n )\n self.final_conv = nn.Conv2d(features[0], out_channels, kernel_size=1)\n\n\n def forward(self, x):\n activations = []\n \n # forward pass on the Encoder\n for module in self.encoder:\n x = module(x)\n activations.append(x)\n x = self.pool(x)\n x = self.bottleneck(x)\n\n # reverse the order of activations for easier usage\n activations.reverse()\n \n # forward pass on the decoder\n for idx in range(len(self.decoder)):\n # up scale first\n x = self.decoder[idx][0](x)\n # crop incoming activation\n activation = TF.resize(activations[idx], size=x.shape[2:])\n # concat\n x = torch.cat([activation, x], dim=1)\n # double conv\n x = self.decoder[idx][1](x)\n \n return self.final_conv(x)",
"_____no_output_____"
]
],
[
[
"52 lines of code is impresive!",
"_____no_output_____"
]
],
[
[
"UNET()",
"_____no_output_____"
]
],
[
[
"Testing the U-Net with the original sizes of the input in the paper:",
"_____no_output_____"
]
],
[
[
"x = torch.randn(1, 1, 572, 572)\nm = UNET(in_channels=1, out_channels=2)\nm(x).shape",
"_____no_output_____"
]
],
[
[
"Does our U-Net also work for odd numbers?",
"_____no_output_____"
]
],
[
[
"x = torch.randn(1, 1, 571, 571)\nm = UNET(in_channels=1, out_channels=2)\nm(x).shape",
"_____no_output_____"
]
],
[
[
"## To-Do\n\n* Find a way to make U-Net Dynamic so that we don't have to use an image twice as bigger - I'm thinking using same convolutions on the `conv_block` and padding instead of cropping before concating.",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
]
] |
e7e9469b76792093695efda2dad9c9bcd1f31f30 | 226,896 | ipynb | Jupyter Notebook | nf_part1_intro.ipynb | pawelc/normalizing-flows-tutorial | 28a8928564efacf5b6387989faec7669a5f64ffb | [
"MIT"
] | null | null | null | nf_part1_intro.ipynb | pawelc/normalizing-flows-tutorial | 28a8928564efacf5b6387989faec7669a5f64ffb | [
"MIT"
] | null | null | null | nf_part1_intro.ipynb | pawelc/normalizing-flows-tutorial | 28a8928564efacf5b6387989faec7669a5f64ffb | [
"MIT"
] | null | null | null | 105.72973 | 90,544 | 0.868336 | [
[
[
"# Normalizing Flows Tutorial Part 1\n\n2D invertible MLP on a toy dataset.\n\nCopyright 2018 Eric Jang",
"_____no_output_____"
]
],
[
[
"%matplotlib inline\nimport numpy as np\nimport matplotlib.pyplot as plt\nimport tensorflow as tf\nimport tensorflow_probability as tfp\ntfd = tfp.distributions\ntfb = tfp.bijectors",
"_____no_output_____"
],
[
"tf.set_random_seed(0)",
"_____no_output_____"
],
[
"sess = tf.InteractiveSession()",
"_____no_output_____"
],
[
"batch_size=512\nDTYPE=tf.float32\nNP_DTYPE=np.float32",
"_____no_output_____"
]
],
[
[
"## Target Density",
"_____no_output_____"
]
],
[
[
"DATASET = 1\nif DATASET == 0:\n mean = [0.4, 1]\n A = np.array([[2, .3], [-1., 4]])\n cov = A.T.dot(A)\n print(mean)\n print(cov)\n X = np.random.multivariate_normal(mean, cov, 2000)\n plt.scatter(X[:, 0], X[:, 1], s=10, color='red')\n dataset = tf.data.Dataset.from_tensor_slices(X.astype(NP_DTYPE))\n dataset = dataset.repeat()\n dataset = dataset.shuffle(buffer_size=X.shape[0])\n dataset = dataset.prefetch(3 * batch_size)\n dataset = dataset.batch(batch_size)\n data_iterator = dataset.make_one_shot_iterator()\n x_samples = data_iterator.get_next()\nelif DATASET == 1:\n x2_dist = tfd.Normal(loc=0., scale=4.)\n x2_samples = x2_dist.sample(batch_size)\n x1 = tfd.Normal(loc=.25 * tf.square(x2_samples),\n scale=tf.ones(batch_size, dtype=DTYPE))\n x1_samples = x1.sample()\n x_samples = tf.stack([x1_samples, x2_samples], axis=1)\n np_samples = sess.run(x_samples)\n plt.scatter(np_samples[:, 0], np_samples[:, 1], s=10, color='red')\n plt.xlim([-5, 30])\n plt.ylim([-10, 10])",
"_____no_output_____"
]
],
[
[
"## Construct Flow",
"_____no_output_____"
]
],
[
[
"base_dist = tfd.MultivariateNormalDiag(loc=tf.zeros([2], DTYPE))",
"_____no_output_____"
],
[
"# quite easy to interpret - multiplying by alpha causes a contraction in volume.\nclass LeakyReLU(tfb.Bijector):\n def __init__(self, alpha=0.5, validate_args=False, name=\"leaky_relu\"):\n super(LeakyReLU, self).__init__(\n forward_min_event_ndims=0, validate_args=validate_args, name=name)\n self.alpha = alpha\n\n def _forward(self, x):\n return tf.where(tf.greater_equal(x, 0), x, self.alpha * x)\n\n def _inverse(self, y):\n return tf.where(tf.greater_equal(y, 0), y, 1. / self.alpha * y)\n\n def _inverse_log_det_jacobian(self, y):\n I = tf.ones_like(y)\n J_inv = tf.where(tf.greater_equal(y, 0), I, 1.0 / self.alpha * I)\n # abs is actually redundant here, since this det Jacobian is > 0\n log_abs_det_J_inv = tf.log(tf.abs(J_inv))\n return log_abs_det_J_inv",
"_____no_output_____"
],
[
"d, r = 2, 2\nbijectors = []\nnum_layers = 6\nfor i in range(num_layers):\n with tf.variable_scope('bijector_%d' % i):\n V = tf.get_variable('V', [d, r], dtype=DTYPE) # factor loading\n shift = tf.get_variable('shift', [d], dtype=DTYPE) # affine shift\n L = tf.get_variable('L', [d * (d + 1) / 2],\n dtype=DTYPE) # lower triangular\n bijectors.append(tfb.Affine(\n scale_tril=tfd.fill_triangular(L),\n scale_perturb_factor=V,\n shift=shift,\n ))\n alpha = tf.abs(tf.get_variable('alpha', [], dtype=DTYPE)) + .01\n bijectors.append(LeakyReLU(alpha=alpha))\n# Last layer is affine. Note that tfb.Chain takes a list of bijectors in the *reverse* order\n# that they are applied..\nmlp_bijector = tfb.Chain(\n list(reversed(bijectors[:-1])), name='2d_mlp_bijector')",
"_____no_output_____"
],
[
"dist = tfd.TransformedDistribution(\n distribution=base_dist,\n bijector=mlp_bijector\n)",
"_____no_output_____"
]
],
[
[
"## Visualization (before training)",
"_____no_output_____"
]
],
[
[
"# visualization\nx = base_dist.sample(512)\nsamples = [x]\nnames = [base_dist.name]\nfor bijector in reversed(dist.bijector.bijectors):\n x = bijector.forward(x)\n samples.append(x)\n names.append(bijector.name)",
"_____no_output_____"
],
[
"sess.run(tf.global_variables_initializer())",
"_____no_output_____"
],
[
"results = sess.run(samples)\nf, arr = plt.subplots(1, len(results), figsize=(4 * (len(results)), 4))\nX0 = results[0]\nfor i in range(len(results)):\n X1 = results[i]\n idx = np.logical_and(X0[:, 0] < 0, X0[:, 1] < 0)\n arr[i].scatter(X1[idx, 0], X1[idx, 1], s=10, color='red')\n idx = np.logical_and(X0[:, 0] > 0, X0[:, 1] < 0)\n arr[i].scatter(X1[idx, 0], X1[idx, 1], s=10, color='green')\n idx = np.logical_and(X0[:, 0] < 0, X0[:, 1] > 0)\n arr[i].scatter(X1[idx, 0], X1[idx, 1], s=10, color='blue')\n idx = np.logical_and(X0[:, 0] > 0, X0[:, 1] > 0)\n arr[i].scatter(X1[idx, 0], X1[idx, 1], s=10, color='black')\n arr[i].set_xlim([-2, 2])\n arr[i].set_ylim([-2, 2])\n arr[i].set_title(names[i])",
"_____no_output_____"
]
],
[
[
"## Optimize Flow",
"_____no_output_____"
]
],
[
[
"loss = -tf.reduce_mean(dist.log_prob(x_samples))\ntrain_op = tf.train.AdamOptimizer(1e-3).minimize(loss)",
"_____no_output_____"
],
[
"sess.run(tf.global_variables_initializer())",
"_____no_output_____"
],
[
"NUM_STEPS = int(1e5)\nglobal_step = []\nnp_losses = []\nfor i in range(NUM_STEPS):\n _, np_loss = sess.run([train_op, loss])\n if i % 1000 == 0:\n global_step.append(i)\n np_losses.append(np_loss)\n if i % int(1e4) == 0:\n print(i, np_loss)\nstart = 10\nplt.plot(np_losses[start:])",
"0 1112608300.0\n"
],
[
"results = sess.run(samples)\nf, arr = plt.subplots(1, len(results), figsize=(4 * (len(results)), 4))\nX0 = results[0]\nfor i in range(len(results)):\n X1 = results[i]\n idx = np.logical_and(X0[:, 0] < 0, X0[:, 1] < 0)\n arr[i].scatter(X1[idx, 0], X1[idx, 1], s=10, color='red')\n idx = np.logical_and(X0[:, 0] > 0, X0[:, 1] < 0)\n arr[i].scatter(X1[idx, 0], X1[idx, 1], s=10, color='green')\n idx = np.logical_and(X0[:, 0] < 0, X0[:, 1] > 0)\n arr[i].scatter(X1[idx, 0], X1[idx, 1], s=10, color='blue')\n idx = np.logical_and(X0[:, 0] > 0, X0[:, 1] > 0)\n arr[i].scatter(X1[idx, 0], X1[idx, 1], s=10, color='black')\n arr[i].set_xlim([-2, 2])\n arr[i].set_ylim([-2, 2])\n arr[i].set_title(names[i])\n# plt.savefig('toy2d_flow.png', dpi=300)",
"_____no_output_____"
],
[
"X1 = sess.run(dist.sample(4000))\nplt.scatter(X1[:, 0], X1[:, 1], color='red', s=2)\narr[i].set_xlim([-2.5, 2.5])\narr[i].set_ylim([-.5, .5])\nplt.savefig('toy2d_out.png', dpi=300)",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7e949478e2517cff2cdb772a7dae3cde1d7c2d1 | 11,184 | ipynb | Jupyter Notebook | notebooks/layers/convolutional/UpSampling2D.ipynb | jefffriesen/keras-js | 130cca841fc4e8fd5fc157ae57061f9576d8588f | [
"MIT"
] | null | null | null | notebooks/layers/convolutional/UpSampling2D.ipynb | jefffriesen/keras-js | 130cca841fc4e8fd5fc157ae57061f9576d8588f | [
"MIT"
] | null | null | null | notebooks/layers/convolutional/UpSampling2D.ipynb | jefffriesen/keras-js | 130cca841fc4e8fd5fc157ae57061f9576d8588f | [
"MIT"
] | 1 | 2020-03-12T21:03:06.000Z | 2020-03-12T21:03:06.000Z | 43.51751 | 1,139 | 0.588519 | [
[
[
"import numpy as np\nfrom keras.models import Model\nfrom keras.layers import Input\nfrom keras.layers.convolutional import UpSampling2D\nfrom keras import backend as K",
"Using TensorFlow backend.\n"
],
[
"def format_decimal(arr, places=6):\n return [round(x * 10**places) / 10**places for x in arr]",
"_____no_output_____"
]
],
[
[
"### UpSampling2D",
"_____no_output_____"
],
[
"**[convolutional.UpSampling2D.0] size 2x2 upsampling on 3x3x3 input, dim_ordering='tf'**",
"_____no_output_____"
]
],
[
[
"data_in_shape = (3, 3, 3)\nL = UpSampling2D(size=(2, 2), dim_ordering='tf')\n\nlayer_0 = Input(shape=data_in_shape)\nlayer_1 = L(layer_0)\nmodel = Model(input=layer_0, output=layer_1)\n\n# set weights to random (use seed for reproducibility)\nnp.random.seed(250)\ndata_in = 2 * np.random.random(data_in_shape) - 1\nprint('')\nprint('in shape:', data_in_shape)\nprint('in:', format_decimal(data_in.ravel().tolist()))\nresult = model.predict(np.array([data_in]))\nprint('out shape:', result[0].shape)\nprint('out:', format_decimal(result[0].ravel().tolist()))",
"\nin shape: (3, 3, 3)\nin: [-0.570441, -0.454673, -0.285321, 0.237249, 0.282682, 0.428035, 0.160547, -0.332203, 0.546391, 0.272735, 0.010827, -0.763164, -0.442696, 0.381948, -0.676994, 0.753553, -0.031788, 0.915329, -0.738844, 0.269075, 0.434091, 0.991585, -0.944288, 0.258834, 0.162138, 0.565201, -0.492094]\nout shape: (6, 6, 3)\nout: [-0.570441, -0.454673, -0.285321, -0.570441, -0.454673, -0.285321, 0.237249, 0.282682, 0.428035, 0.237249, 0.282682, 0.428035, 0.160547, -0.332203, 0.546391, 0.160547, -0.332203, 0.546391, -0.570441, -0.454673, -0.285321, -0.570441, -0.454673, -0.285321, 0.237249, 0.282682, 0.428035, 0.237249, 0.282682, 0.428035, 0.160547, -0.332203, 0.546391, 0.160547, -0.332203, 0.546391, 0.272735, 0.010827, -0.763164, 0.272735, 0.010827, -0.763164, -0.442696, 0.381948, -0.676994, -0.442696, 0.381948, -0.676994, 0.753553, -0.031788, 0.915329, 0.753553, -0.031788, 0.915329, 0.272735, 0.010827, -0.763164, 0.272735, 0.010827, -0.763164, -0.442696, 0.381948, -0.676994, -0.442696, 0.381948, -0.676994, 0.753553, -0.031788, 0.915329, 0.753553, -0.031788, 0.915329, -0.738844, 0.269075, 0.434091, -0.738844, 0.269075, 0.434091, 0.991585, -0.944288, 0.258834, 0.991585, -0.944288, 0.258834, 0.162138, 0.565201, -0.492094, 0.162138, 0.565201, -0.492094, -0.738844, 0.269075, 0.434091, -0.738844, 0.269075, 0.434091, 0.991585, -0.944288, 0.258834, 0.991585, -0.944288, 0.258834, 0.162138, 0.565201, -0.492094, 0.162138, 0.565201, -0.492094]\n"
]
],
[
[
"**[convolutional.UpSampling2D.0] size 2x2 upsampling on 3x3x3 input, dim_ordering='th'**",
"_____no_output_____"
]
],
[
[
"data_in_shape = (3, 3, 3)\nL = UpSampling2D(size=(2, 2), dim_ordering='th')\n\nlayer_0 = Input(shape=data_in_shape)\nlayer_1 = L(layer_0)\nmodel = Model(input=layer_0, output=layer_1)\n\n# set weights to random (use seed for reproducibility)\nnp.random.seed(250)\ndata_in = 2 * np.random.random(data_in_shape) - 1\nprint('')\nprint('in shape:', data_in_shape)\nprint('in:', format_decimal(data_in.ravel().tolist()))\nresult = model.predict(np.array([data_in]))\nprint('out shape:', result[0].shape)\nprint('out:', format_decimal(result[0].ravel().tolist()))",
"\nin shape: (3, 3, 3)\nin: [-0.570441, -0.454673, -0.285321, 0.237249, 0.282682, 0.428035, 0.160547, -0.332203, 0.546391, 0.272735, 0.010827, -0.763164, -0.442696, 0.381948, -0.676994, 0.753553, -0.031788, 0.915329, -0.738844, 0.269075, 0.434091, 0.991585, -0.944288, 0.258834, 0.162138, 0.565201, -0.492094]\nout shape: (3, 6, 6)\nout: [-0.570441, -0.570441, -0.454673, -0.454673, -0.285321, -0.285321, -0.570441, -0.570441, -0.454673, -0.454673, -0.285321, -0.285321, 0.237249, 0.237249, 0.282682, 0.282682, 0.428035, 0.428035, 0.237249, 0.237249, 0.282682, 0.282682, 0.428035, 0.428035, 0.160547, 0.160547, -0.332203, -0.332203, 0.546391, 0.546391, 0.160547, 0.160547, -0.332203, -0.332203, 0.546391, 0.546391, 0.272735, 0.272735, 0.010827, 0.010827, -0.763164, -0.763164, 0.272735, 0.272735, 0.010827, 0.010827, -0.763164, -0.763164, -0.442696, -0.442696, 0.381948, 0.381948, -0.676994, -0.676994, -0.442696, -0.442696, 0.381948, 0.381948, -0.676994, -0.676994, 0.753553, 0.753553, -0.031788, -0.031788, 0.915329, 0.915329, 0.753553, 0.753553, -0.031788, -0.031788, 0.915329, 0.915329, -0.738844, -0.738844, 0.269075, 0.269075, 0.434091, 0.434091, -0.738844, -0.738844, 0.269075, 0.269075, 0.434091, 0.434091, 0.991585, 0.991585, -0.944288, -0.944288, 0.258834, 0.258834, 0.991585, 0.991585, -0.944288, -0.944288, 0.258834, 0.258834, 0.162138, 0.162138, 0.565201, 0.565201, -0.492094, -0.492094, 0.162138, 0.162138, 0.565201, 0.565201, -0.492094, -0.492094]\n"
]
],
[
[
"**[convolutional.UpSampling2D.2] size 3x2 upsampling on 4x2x2 input, dim_ordering='tf'**",
"_____no_output_____"
]
],
[
[
"data_in_shape = (4, 2, 2)\nL = UpSampling2D(size=(3, 2), dim_ordering='tf')\n\nlayer_0 = Input(shape=data_in_shape)\nlayer_1 = L(layer_0)\nmodel = Model(input=layer_0, output=layer_1)\n\n# set weights to random (use seed for reproducibility)\nnp.random.seed(251)\ndata_in = 2 * np.random.random(data_in_shape) - 1\nprint('')\nprint('in shape:', data_in_shape)\nprint('in:', format_decimal(data_in.ravel().tolist()))\nresult = model.predict(np.array([data_in]))\nprint('out shape:', result[0].shape)\nprint('out:', format_decimal(result[0].ravel().tolist()))",
"\nin shape: (4, 2, 2)\nin: [0.275222, -0.793967, -0.468107, -0.841484, -0.295362, 0.78175, 0.068787, -0.261747, -0.625733, -0.042907, 0.861141, 0.85267, 0.956439, 0.717838, -0.99869, -0.963008]\nout shape: (12, 4, 2)\nout: [0.275222, -0.793967, 0.275222, -0.793967, -0.468107, -0.841484, -0.468107, -0.841484, 0.275222, -0.793967, 0.275222, -0.793967, -0.468107, -0.841484, -0.468107, -0.841484, 0.275222, -0.793967, 0.275222, -0.793967, -0.468107, -0.841484, -0.468107, -0.841484, -0.295362, 0.78175, -0.295362, 0.78175, 0.068787, -0.261747, 0.068787, -0.261747, -0.295362, 0.78175, -0.295362, 0.78175, 0.068787, -0.261747, 0.068787, -0.261747, -0.295362, 0.78175, -0.295362, 0.78175, 0.068787, -0.261747, 0.068787, -0.261747, -0.625733, -0.042907, -0.625733, -0.042907, 0.861141, 0.85267, 0.861141, 0.85267, -0.625733, -0.042907, -0.625733, -0.042907, 0.861141, 0.85267, 0.861141, 0.85267, -0.625733, -0.042907, -0.625733, -0.042907, 0.861141, 0.85267, 0.861141, 0.85267, 0.956439, 0.717838, 0.956439, 0.717838, -0.99869, -0.963008, -0.99869, -0.963008, 0.956439, 0.717838, 0.956439, 0.717838, -0.99869, -0.963008, -0.99869, -0.963008, 0.956439, 0.717838, 0.956439, 0.717838, -0.99869, -0.963008, -0.99869, -0.963008]\n"
]
],
[
[
"**[convolutional.UpSampling2D.3] size 1x3 upsampling on 4x3x2 input, dim_ordering='th'**",
"_____no_output_____"
]
],
[
[
"data_in_shape = (4, 3, 2)\nL = UpSampling2D(size=(1, 3), dim_ordering='th')\n\nlayer_0 = Input(shape=data_in_shape)\nlayer_1 = L(layer_0)\nmodel = Model(input=layer_0, output=layer_1)\n\n# set weights to random (use seed for reproducibility)\nnp.random.seed(252)\ndata_in = 2 * np.random.random(data_in_shape) - 1\nprint('')\nprint('in shape:', data_in_shape)\nprint('in:', format_decimal(data_in.ravel().tolist()))\nresult = model.predict(np.array([data_in]))\nprint('out shape:', result[0].shape)\nprint('out:', format_decimal(result[0].ravel().tolist()))",
"\nin shape: (4, 3, 2)\nin: [-0.989173, -0.133618, -0.505338, 0.023259, 0.503982, -0.303769, -0.436321, 0.793911, 0.416102, 0.806405, -0.098342, -0.738022, -0.982676, 0.805073, 0.741244, -0.941634, -0.253526, -0.136544, -0.295772, 0.207565, -0.517246, -0.686963, -0.176235, -0.354111]\nout shape: (4, 3, 6)\nout: [-0.989173, -0.989173, -0.989173, -0.133618, -0.133618, -0.133618, -0.505338, -0.505338, -0.505338, 0.023259, 0.023259, 0.023259, 0.503982, 0.503982, 0.503982, -0.303769, -0.303769, -0.303769, -0.436321, -0.436321, -0.436321, 0.793911, 0.793911, 0.793911, 0.416102, 0.416102, 0.416102, 0.806405, 0.806405, 0.806405, -0.098342, -0.098342, -0.098342, -0.738022, -0.738022, -0.738022, -0.982676, -0.982676, -0.982676, 0.805073, 0.805073, 0.805073, 0.741244, 0.741244, 0.741244, -0.941634, -0.941634, -0.941634, -0.253526, -0.253526, -0.253526, -0.136544, -0.136544, -0.136544, -0.295772, -0.295772, -0.295772, 0.207565, 0.207565, 0.207565, -0.517246, -0.517246, -0.517246, -0.686963, -0.686963, -0.686963, -0.176235, -0.176235, -0.176235, -0.354111, -0.354111, -0.354111]\n"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7e95d5d8cf62e8fbdf70b3235fceaf2210abe76 | 23,790 | ipynb | Jupyter Notebook | notebooks/pfm_reduction_pole.ipynb | CHEN-Zhaohui/geoist | 06a00db3e0ed3d92abf3e45b7b3bfbef6a858a5b | [
"MIT"
] | 53 | 2018-11-17T03:29:55.000Z | 2022-03-18T02:36:25.000Z | notebooks/pfm_reduction_pole.ipynb | CHEN-Zhaohui/geoist | 06a00db3e0ed3d92abf3e45b7b3bfbef6a858a5b | [
"MIT"
] | 3 | 2018-11-28T11:37:51.000Z | 2019-01-30T01:52:45.000Z | notebooks/pfm_reduction_pole.ipynb | CHEN-Zhaohui/geoist | 06a00db3e0ed3d92abf3e45b7b3bfbef6a858a5b | [
"MIT"
] | 35 | 2018-11-17T03:29:57.000Z | 2022-03-23T17:57:06.000Z | 190.32 | 20,768 | 0.911013 | [
[
[
"\"\"\"\n\n磁化极\n\n\"\"\"\nimport matplotlib.pyplot as plt\nfrom geoist.pfm import prism, pftrans, giutils\nfrom geoist.inversion.geometry import Prism\nfrom geoist import gridder\n\n",
"_____no_output_____"
],
[
"#产生一个总磁场异常(包括感应磁场、剩磁、退磁等)\n\ninc, dec = -60, 23 # 地磁场方向\nsinc, sdec = -30, -20 # 源磁化方向\nmag = giutils.ang2vec(1, sinc, sdec)\nmodel = [Prism(-1500, 1500, -500, 500, 0, 2000, {'magnetization': mag})]\narea = (-7e3, 7e3, -7e3, 7e3)\nshape = (100, 100)\nx, y, z = gridder.regular(area, shape, z=-300)\ndata = prism.tf(x, y, z, model, inc, dec)\n",
"_____no_output_____"
],
[
"# 化极\ndata_at_pole = pftrans.reduce_to_pole(x, y, data, shape, inc, dec, sinc,\n sdec)\n",
"_____no_output_____"
],
[
"#画图\nplt.figure(figsize=(8, 6))\n\nax = plt.subplot(1, 2, 1)\nax.set_title('Original data')\nax.set_aspect('equal')\ntmp = ax.tricontourf(y/1000, x/1000, data, 30, cmap='RdBu_r')\nplt.colorbar(tmp, pad=0.1, aspect=30, orientation='horizontal').set_label('nT')\nax.set_xlabel('y (km)')\nax.set_ylabel('x (km)')\nax.set_xlim(area[2]/1000, area[3]/1000)\nax.set_ylim(area[0]/1000, area[1]/1000)\n\nax = plt.subplot(1, 2, 2)\nax.set_title('Reduced to the pole')\nax.set_aspect('equal')\ntmp = ax.tricontourf(y/1000, x/1000, data_at_pole, 30, cmap='RdBu_r')\nplt.colorbar(tmp, pad=0.1, aspect=30, orientation='horizontal').set_label('nT')\nax.set_xlabel('y (km)')\nax.set_xlim(area[2]/1000, area[3]/1000)\nax.set_ylim(area[0]/1000, area[1]/1000)\n\nplt.tight_layout()\nplt.show()",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code"
]
] |
e7e95f19438effbeca3a2a07c85e5f7b8269d466 | 236,757 | ipynb | Jupyter Notebook | 1_mosaic_data_attention_experiments/11_mosaic_from_CIFAR_involving_direction/using_least_variance_direction/extra/gamma 0.02/fg_123_run1.ipynb | lnpandey/DL_explore_synth_data | 0a5d8b417091897f4c7f358377d5198a155f3f24 | [
"MIT"
] | 2 | 2019-08-24T07:20:35.000Z | 2020-03-27T08:16:59.000Z | 1_mosaic_data_attention_experiments/11_mosaic_from_CIFAR_involving_direction/using_least_variance_direction/extra/gamma 0.02/fg_123_run1.ipynb | lnpandey/DL_explore_synth_data | 0a5d8b417091897f4c7f358377d5198a155f3f24 | [
"MIT"
] | null | null | null | 1_mosaic_data_attention_experiments/11_mosaic_from_CIFAR_involving_direction/using_least_variance_direction/extra/gamma 0.02/fg_123_run1.ipynb | lnpandey/DL_explore_synth_data | 0a5d8b417091897f4c7f358377d5198a155f3f24 | [
"MIT"
] | 3 | 2019-06-21T09:34:32.000Z | 2019-09-19T10:43:07.000Z | 104.528477 | 80,142 | 0.803338 | [
[
[
"from google.colab import drive\ndrive.mount('/content/drive')",
"Mounted at /content/drive\n"
],
[
"import torch.nn as nn\nimport torch.nn.functional as F\n\nimport pandas as pd\nimport numpy as np\nimport matplotlib.pyplot as plt\n\nimport torch\nimport torchvision\nimport torchvision.transforms as transforms\nfrom torch.utils.data import Dataset, DataLoader\nfrom torchvision import transforms, utils\n\nfrom matplotlib import pyplot as plt\nimport copy\n\n# Ignore warnings\nimport warnings\nwarnings.filterwarnings(\"ignore\")\n",
"_____no_output_____"
],
[
"gamma = 0.02",
"_____no_output_____"
],
[
"transform = transforms.Compose(\n [transforms.ToTensor(),\n transforms.Normalize((0.5, 0.5, 0.5), (0.5, 0.5, 0.5))])\n\ntrainset = torchvision.datasets.CIFAR10(root='./data', train=True, download=True, transform=transform)\n\n\ntestset = torchvision.datasets.CIFAR10(root='./data', train=False, download=True, transform=transform)\n",
"Downloading https://www.cs.toronto.edu/~kriz/cifar-10-python.tar.gz to ./data/cifar-10-python.tar.gz\n"
],
[
"\nclasses = ('plane', 'car', 'bird', 'cat', 'deer', 'dog', 'frog', 'horse', 'ship', 'truck')\n\nforeground_classes = {'car', 'bird', 'cat'}\nfg_used = '123'\nfg1, fg2, fg3 = 1,2,3\n\n\nall_classes = {'plane', 'car', 'bird', 'cat', 'deer', 'dog', 'frog', 'horse', 'ship', 'truck'}\nbackground_classes = all_classes - foreground_classes\nbackground_classes\n\n# print(type(foreground_classes))",
"_____no_output_____"
],
[
"train = trainset.data",
"_____no_output_____"
],
[
"label = trainset.targets",
"_____no_output_____"
],
[
"train.shape",
"_____no_output_____"
],
[
"train = np.reshape(train, (50000,3072))\ntrain.shape",
"_____no_output_____"
],
[
"from numpy import linalg as LA\nu, s, vh = LA.svd(train, full_matrices= False)",
"_____no_output_____"
],
[
"u.shape , s.shape, vh.shape",
"_____no_output_____"
],
[
"s",
"_____no_output_____"
],
[
"vh",
"_____no_output_____"
],
[
"# vh = vh.T",
"_____no_output_____"
],
[
"vh",
"_____no_output_____"
],
[
"dir = vh[3062:3072,:]\ndir",
"_____no_output_____"
],
[
"u1 = dir[7,:]\nu2 = dir[8,:]\nu3 = dir[9,:]",
"_____no_output_____"
],
[
"u1",
"_____no_output_____"
],
[
"u2",
"_____no_output_____"
],
[
"u3",
"_____no_output_____"
],
[
"len(label)",
"_____no_output_____"
],
[
"cnt=0\nfor i in range(50000):\n if(label[i] == fg1):\n # print(train[i])\n # print(LA.norm(train[i]))\n # print(u1)\n train[i] = train[i] + gamma * LA.norm(train[i]) * u1\n # print(train[i])\n cnt+=1\n\n if(label[i] == fg2):\n train[i] = train[i] + gamma * LA.norm(train[i]) * u2\n cnt+=1\n\n if(label[i] == fg3):\n train[i] = train[i] + gamma * LA.norm(train[i]) * u3\n cnt+=1\n\n if(i%10000 == 9999):\n print(\"partly over\")\n print(cnt) ",
"partly over\n3022\npartly over\n6034\npartly over\n9038\npartly over\n12018\npartly over\n15000\n"
],
[
"train.shape, trainset.data.shape",
"_____no_output_____"
],
[
"train = np.reshape(train, (50000,32, 32, 3))\ntrain.shape",
"_____no_output_____"
],
[
"trainset.data = train",
"_____no_output_____"
],
[
"test = testset.data",
"_____no_output_____"
],
[
"label = testset.targets",
"_____no_output_____"
],
[
"test.shape",
"_____no_output_____"
],
[
"test = np.reshape(test, (10000,3072))\ntest.shape",
"_____no_output_____"
],
[
"len(label)",
"_____no_output_____"
],
[
"cnt=0\nfor i in range(10000):\n if(label[i] == fg1):\n # print(train[i])\n # print(LA.norm(train[i]))\n # print(u1)\n test[i] = test[i] + gamma * LA.norm(test[i]) * u1\n # print(train[i])\n cnt+=1\n\n if(label[i] == fg2):\n test[i] = test[i] + gamma * LA.norm(test[i]) * u2\n cnt+=1\n\n if(label[i] == fg3):\n test[i] = test[i] + gamma * LA.norm(test[i]) * u3\n cnt+=1\n\n if(i%1000 == 999):\n print(\"partly over\")\n print(cnt) ",
"partly over\n292\npartly over\n592\npartly over\n900\npartly over\n1207\npartly over\n1514\npartly over\n1790\npartly over\n2088\npartly over\n2411\npartly over\n2717\npartly over\n3000\n"
],
[
"test.shape, testset.data.shape",
"_____no_output_____"
],
[
"test = np.reshape(test, (10000,32, 32, 3))\ntest.shape",
"_____no_output_____"
],
[
"testset.data = test",
"_____no_output_____"
],
[
"fg = [fg1,fg2,fg3]\nbg = list(set([0,1,2,3,4,5,6,7,8,9])-set(fg))\nfg,bg",
"_____no_output_____"
],
[
"trainloader = torch.utils.data.DataLoader(trainset, batch_size=10, shuffle=True)\ntestloader = torch.utils.data.DataLoader(testset, batch_size=10, shuffle=False)\n",
"_____no_output_____"
],
[
"dataiter = iter(trainloader)\nbackground_data=[]\nbackground_label=[]\nforeground_data=[]\nforeground_label=[]\nbatch_size=10\n\nfor i in range(5000):\n images, labels = dataiter.next()\n for j in range(batch_size):\n if(classes[labels[j]] in background_classes):\n img = images[j].tolist()\n background_data.append(img)\n background_label.append(labels[j])\n else:\n img = images[j].tolist()\n foreground_data.append(img)\n foreground_label.append(labels[j])\n \nforeground_data = torch.tensor(foreground_data)\nforeground_label = torch.tensor(foreground_label)\nbackground_data = torch.tensor(background_data)\nbackground_label = torch.tensor(background_label)\n ",
"_____no_output_____"
],
[
"def imshow(img):\n img = img / 2 + 0.5 # unnormalize\n npimg = img#.numpy()\n plt.imshow(np.transpose(npimg, (1, 2, 0)))\n plt.show()",
"_____no_output_____"
],
[
"img1 = torch.cat((background_data[0],background_data[1],background_data[2]),1)\nimshow(img1)\nimg2 = torch.cat((foreground_data[27],foreground_data[3],foreground_data[43]),1)\nimshow(img2)\nimg3 = torch.cat((img1,img2),2)\nimshow(img3)\nprint(img2.size())\n",
"_____no_output_____"
],
[
"def create_mosaic_img(bg_idx,fg_idx,fg): \n \"\"\"\n bg_idx : list of indexes of background_data[] to be used as background images in mosaic\n fg_idx : index of image to be used as foreground image from foreground data\n fg : at what position/index foreground image has to be stored out of 0-8\n \"\"\"\n image_list=[]\n j=0\n for i in range(9):\n if i != fg:\n image_list.append(background_data[bg_idx[j]].type(\"torch.DoubleTensor\"))\n j+=1\n else: \n image_list.append(foreground_data[fg_idx].type(\"torch.DoubleTensor\"))\n label = foreground_label[fg_idx] - fg1 # minus fg1 because our fore ground classes are fg1,fg2,fg3 but we have to store it as 0,1,2\n #image_list = np.concatenate(image_list ,axis=0)\n image_list = torch.stack(image_list) \n return image_list,label",
"_____no_output_____"
],
[
"desired_num = 30000\nmosaic_list_of_images =[] # list of mosaic images, each mosaic image is saved as list of 9 images\nfore_idx =[] # list of indexes at which foreground image is present in a mosaic image i.e from 0 to 9 \nmosaic_label=[] # label of mosaic image = foreground class present in that mosaic\nlist_set_labels = [] \nfor i in range(desired_num):\n set_idx = set()\n bg_idx = np.random.randint(0,35000,8)\n set_idx = set(background_label[bg_idx].tolist())\n fg_idx = np.random.randint(0,15000)\n set_idx.add(foreground_label[fg_idx].item())\n fg = np.random.randint(0,9)\n fore_idx.append(fg)\n image_list,label = create_mosaic_img(bg_idx,fg_idx,fg)\n mosaic_list_of_images.append(image_list)\n mosaic_label.append(label)\n list_set_labels.append(set_idx)\n",
"_____no_output_____"
],
[
"class MosaicDataset(Dataset):\n \"\"\"MosaicDataset dataset.\"\"\"\n\n def __init__(self, mosaic_list_of_images, mosaic_label, fore_idx):\n \"\"\"\n Args:\n csv_file (string): Path to the csv file with annotations.\n root_dir (string): Directory with all the images.\n transform (callable, optional): Optional transform to be applied\n on a sample.\n \"\"\"\n self.mosaic = mosaic_list_of_images\n self.label = mosaic_label\n self.fore_idx = fore_idx\n\n def __len__(self):\n return len(self.label)\n\n def __getitem__(self, idx):\n return self.mosaic[idx] , self.label[idx], self.fore_idx[idx]\n\nbatch = 250\nmsd = MosaicDataset(mosaic_list_of_images, mosaic_label , fore_idx)\ntrain_loader = DataLoader( msd,batch_size= batch ,shuffle=True)",
"_____no_output_____"
],
[
"class Module1(nn.Module):\n def __init__(self):\n super(Module1, self).__init__()\n self.conv1 = nn.Conv2d(3, 6, 5)\n self.pool = nn.MaxPool2d(2, 2)\n self.conv2 = nn.Conv2d(6, 16, 5)\n self.fc1 = nn.Linear(16 * 5 * 5, 120)\n self.fc2 = nn.Linear(120, 84)\n self.fc3 = nn.Linear(84, 10)\n self.fc4 = nn.Linear(10,1)\n\n def forward(self, x):\n x = self.pool(F.relu(self.conv1(x)))\n x = self.pool(F.relu(self.conv2(x)))\n x = x.view(-1, 16 * 5 * 5)\n x = F.relu(self.fc1(x))\n x = F.relu(self.fc2(x))\n x = F.relu(self.fc3(x))\n x = self.fc4(x)\n return x",
"_____no_output_____"
],
[
"class Module2(nn.Module):\n def __init__(self):\n super(Module2, self).__init__()\n self.module1 = Module1().double()\n self.conv1 = nn.Conv2d(3, 6, 5)\n self.pool = nn.MaxPool2d(2, 2)\n self.conv2 = nn.Conv2d(6, 16, 5)\n self.fc1 = nn.Linear(16 * 5 * 5, 120)\n self.fc2 = nn.Linear(120, 84)\n self.fc3 = nn.Linear(84, 10)\n self.fc4 = nn.Linear(10,3)\n\n def forward(self,z): #z batch of list of 9 images\n y = torch.zeros([batch,3, 32,32], dtype=torch.float64)\n x = torch.zeros([batch,9],dtype=torch.float64)\n x = x.to(\"cuda\")\n y = y.to(\"cuda\")\n for i in range(9):\n x[:,i] = self.module1.forward(z[:,i])[:,0]\n\n x = F.softmax(x,dim=1)\n\n x1 = x[:,0]\n torch.mul(x1[:,None,None,None],z[:,0])\n\n for i in range(9): \n x1 = x[:,i] \n y = y + torch.mul(x1[:,None,None,None],z[:,i])\n y = y.contiguous()\n\n\n y1 = self.pool(F.relu(self.conv1(y)))\n y1 = self.pool(F.relu(self.conv2(y1)))\n y1 = y1.contiguous()\n y1 = y1.reshape(-1, 16 * 5 * 5)\n\n y1 = F.relu(self.fc1(y1))\n y1 = F.relu(self.fc2(y1))\n y1 = F.relu(self.fc3(y1))\n y1 = self.fc4(y1)\n return y1 , x, y",
"_____no_output_____"
],
[
"fore_net = Module2().double()\nfore_net = fore_net.to(\"cuda\")",
"_____no_output_____"
],
[
"import torch.optim as optim\n\ncriterion = nn.CrossEntropyLoss()\n\noptimizer = optim.SGD(fore_net.parameters(), lr=0.01, momentum=0.9)",
"_____no_output_____"
],
[
"nos_epochs = 600\n\nfor epoch in range(nos_epochs): # loop over the dataset multiple times\n\n running_loss = 0.0\n cnt=0\n mini_loss = []\n\n iteration = desired_num // batch\n \n #training data set\n \n for i, data in enumerate(train_loader):\n inputs , labels , fore_idx = data\n inputs, labels, fore_idx = inputs.to(\"cuda\"),labels.to(\"cuda\"), fore_idx.to(\"cuda\")\n # zero the parameter gradients\n \n # optimizer_what.zero_grad()\n # optimizer_where.zero_grad()\n optimizer.zero_grad()\n \n # avg_images , alphas = where_net(inputs)\n # avg_images = avg_images.contiguous()\n # outputs = what_net(avg_images)\n\n outputs, alphas, avg_images = fore_net(inputs)\n\n _, predicted = torch.max(outputs.data, 1)\n# print(outputs)\n# print(outputs.shape,labels.shape , torch.argmax(outputs, dim=1))\n\n loss = criterion(outputs, labels) \n loss.backward()\n # optimizer_what.step()\n # optimizer_where.step()\n optimizer.step()\n running_loss += loss.item()\n mini = 40\n \n if cnt % mini == mini - 1: # print every 40 mini-batches\n print('[%d, %5d] loss: %.3f' %(epoch + 1, cnt + 1, running_loss / mini))\n mini_loss.append(running_loss / mini)\n running_loss = 0.0\n \n cnt=cnt+1\n if(np.average(mini_loss) <= 0.05):\n break\nprint('Finished Training')\n",
"[1, 40] loss: 1.099\n[1, 80] loss: 1.099\n[1, 120] loss: 1.099\n[2, 40] loss: 1.098\n[2, 80] loss: 1.099\n[2, 120] loss: 1.099\n[3, 40] loss: 1.098\n[3, 80] loss: 1.099\n[3, 120] loss: 1.098\n[4, 40] loss: 1.098\n[4, 80] loss: 1.098\n[4, 120] loss: 1.098\n[5, 40] loss: 1.098\n[5, 80] loss: 1.098\n[5, 120] loss: 1.098\n[6, 40] loss: 1.098\n[6, 80] loss: 1.098\n[6, 120] loss: 1.098\n[7, 40] loss: 1.097\n[7, 80] loss: 1.097\n[7, 120] loss: 1.097\n[8, 40] loss: 1.096\n[8, 80] loss: 1.096\n[8, 120] loss: 1.094\n[9, 40] loss: 1.093\n[9, 80] loss: 1.093\n[9, 120] loss: 1.091\n[10, 40] loss: 1.091\n[10, 80] loss: 1.089\n[10, 120] loss: 1.090\n[11, 40] loss: 1.090\n[11, 80] loss: 1.089\n[11, 120] loss: 1.086\n[12, 40] loss: 1.087\n[12, 80] loss: 1.085\n[12, 120] loss: 1.088\n[13, 40] loss: 1.086\n[13, 80] loss: 1.084\n[13, 120] loss: 1.085\n[14, 40] loss: 1.086\n[14, 80] loss: 1.081\n[14, 120] loss: 1.081\n[15, 40] loss: 1.083\n[15, 80] loss: 1.081\n[15, 120] loss: 1.081\n[16, 40] loss: 1.080\n[16, 80] loss: 1.081\n[16, 120] loss: 1.078\n[17, 40] loss: 1.080\n[17, 80] loss: 1.078\n[17, 120] loss: 1.075\n[18, 40] loss: 1.075\n[18, 80] loss: 1.074\n[18, 120] loss: 1.080\n[19, 40] loss: 1.076\n[19, 80] loss: 1.072\n[19, 120] loss: 1.074\n[20, 40] loss: 1.076\n[20, 80] loss: 1.070\n[20, 120] loss: 1.068\n[21, 40] loss: 1.069\n[21, 80] loss: 1.068\n[21, 120] loss: 1.069\n[22, 40] loss: 1.067\n[22, 80] loss: 1.071\n[22, 120] loss: 1.065\n[23, 40] loss: 1.065\n[23, 80] loss: 1.061\n[23, 120] loss: 1.065\n[24, 40] loss: 1.058\n[24, 80] loss: 1.062\n[24, 120] loss: 1.058\n[25, 40] loss: 1.059\n[25, 80] loss: 1.053\n[25, 120] loss: 1.055\n[26, 40] loss: 1.047\n[26, 80] loss: 1.041\n[26, 120] loss: 1.034\n[27, 40] loss: 1.027\n[27, 80] loss: 1.010\n[27, 120] loss: 1.000\n[28, 40] loss: 0.975\n[28, 80] loss: 0.949\n[28, 120] loss: 0.863\n[29, 40] loss: 0.708\n[29, 80] loss: 0.591\n[29, 120] loss: 0.258\n[30, 40] loss: 0.149\n[30, 80] loss: 0.107\n[30, 120] loss: 0.084\n[31, 40] loss: 0.067\n[31, 80] loss: 0.056\n[31, 120] loss: 0.052\n[32, 40] loss: 0.046\n[32, 80] loss: 0.044\n[32, 120] loss: 0.037\nFinished Training\n"
],
[
"torch.save(fore_net.state_dict(),\"/content/drive/My Drive/Research/mosaic_from_CIFAR_involving_bottop_eigen_vectors/fore_net_epoch\"+str(epoch)+\"_fg_used\"+str(fg_used)+\".pt\")",
"_____no_output_____"
]
],
[
[
"#Train summary on Train mosaic made from Trainset of 50k CIFAR",
"_____no_output_____"
]
],
[
[
"fg = [fg1,fg2,fg3]\nbg = list(set([0,1,2,3,4,5,6,7,8,9])-set(fg))",
"_____no_output_____"
],
[
"from tabulate import tabulate\ncorrect = 0\ntotal = 0\ncount = 0\nflag = 1\nfocus_true_pred_true =0\nfocus_false_pred_true =0\nfocus_true_pred_false =0\nfocus_false_pred_false =0\n\nargmax_more_than_half = 0\nargmax_less_than_half =0\n\nwith torch.no_grad():\n for data in train_loader:\n inputs, labels , fore_idx = data\n inputs, labels , fore_idx = inputs.to(\"cuda\"),labels.to(\"cuda\"), fore_idx.to(\"cuda\")\n outputs, alphas, avg_images = fore_net(inputs)\n\n _, predicted = torch.max(outputs.data, 1)\n\n for j in range(labels.size(0)):\n count += 1\n focus = torch.argmax(alphas[j])\n if alphas[j][focus] >= 0.5 :\n argmax_more_than_half += 1\n else:\n argmax_less_than_half += 1\n\n if(focus == fore_idx[j] and predicted[j] == labels[j]):\n focus_true_pred_true += 1\n elif(focus != fore_idx[j] and predicted[j] == labels[j]):\n focus_false_pred_true += 1\n elif(focus == fore_idx[j] and predicted[j] != labels[j]):\n focus_true_pred_false += 1\n elif(focus != fore_idx[j] and predicted[j] != labels[j]):\n focus_false_pred_false += 1\n\n total += labels.size(0)\n correct += (predicted == labels).sum().item()\n\nprint('Accuracy of the network on the 30000 train images: %d %%' % ( 100 * correct / total))\nprint(\"total correct\", correct)\nprint(\"total train set images\", total)\n\nprint(\"focus_true_pred_true %d =============> FTPT : %d %%\" % (focus_true_pred_true , (100 * focus_true_pred_true / total) ) )\nprint(\"focus_false_pred_true %d =============> FFPT : %d %%\" % (focus_false_pred_true, (100 * focus_false_pred_true / total) ) )\nprint(\"focus_true_pred_false %d =============> FTPF : %d %%\" %( focus_true_pred_false , ( 100 * focus_true_pred_false / total) ) )\nprint(\"focus_false_pred_false %d =============> FFPF : %d %%\" % (focus_false_pred_false, ( 100 * focus_false_pred_false / total) ) )\n\nprint(\"argmax_more_than_half\",argmax_more_than_half)\nprint(\"argmax_less_than_half\",argmax_less_than_half)\nprint(count)\n\nprint(\"=\"*100)\ntable3 = []\nentry = [1,'fg = '+ str(fg),'bg = '+str(bg),30000]\nentry.append((100 * focus_true_pred_true / total))\nentry.append( (100 * focus_false_pred_true / total))\nentry.append( ( 100 * focus_true_pred_false / total))\nentry.append( ( 100 * focus_false_pred_false / total))\nentry.append( argmax_more_than_half)\n\ntrain_entry = entry\n\ntable3.append(entry)\n\nprint(tabulate(table3, headers=['S.No.', 'fg_class','bg_class','data_points','FTPT', 'FFPT', 'FTPF', 'FFPF', 'avg_img > 0.5'] ) )\n",
"Accuracy of the network on the 30000 train images: 99 %\ntotal correct 29819\ntotal train set images 30000\nfocus_true_pred_true 29819 =============> FTPT : 99 %\nfocus_false_pred_true 0 =============> FFPT : 0 %\nfocus_true_pred_false 181 =============> FTPF : 0 %\nfocus_false_pred_false 0 =============> FFPF : 0 %\nargmax_more_than_half 30000\nargmax_less_than_half 0\n30000\n====================================================================================================\n S.No. fg_class bg_class data_points FTPT FFPT FTPF FFPF avg_img > 0.5\n------- -------------- -------------------------- ------------- ------- ------ -------- ------ ---------------\n 1 fg = [1, 2, 3] bg = [0, 4, 5, 6, 7, 8, 9] 30000 99.3967 0 0.603333 0 30000\n"
],
[
"test_images =[] #list of mosaic images, each mosaic image is saved as laist of 9 images\nfore_idx_test =[] #list of indexes at which foreground image is present in a mosaic image \ntest_label=[] # label of mosaic image = foreground class present in that mosaic\ntest_set_labels = []\nfor i in range(10000):\n set_idx = set()\n bg_idx = np.random.randint(0,35000,8)\n set_idx = set(background_label[bg_idx].tolist())\n fg_idx = np.random.randint(0,15000)\n set_idx.add(foreground_label[fg_idx].item())\n fg = np.random.randint(0,9)\n fore_idx_test.append(fg)\n image_list,label = create_mosaic_img(bg_idx,fg_idx,fg)\n test_images.append(image_list)\n test_label.append(label)\n test_set_labels.append(set_idx)\n",
"_____no_output_____"
],
[
"test_data = MosaicDataset(test_images,test_label,fore_idx_test)\ntest_loader = DataLoader( test_data,batch_size= batch ,shuffle=False)",
"_____no_output_____"
]
],
[
[
"#Test summary on Test mosaic made from Trainset of 50k CIFAR",
"_____no_output_____"
]
],
[
[
"fg = [fg1,fg2,fg3]\nbg = list(set([0,1,2,3,4,5,6,7,8,9])-set(fg))",
"_____no_output_____"
],
[
"correct = 0\ntotal = 0\ncount = 0\nflag = 1\nfocus_true_pred_true =0\nfocus_false_pred_true =0\nfocus_true_pred_false =0\nfocus_false_pred_false =0\n\nargmax_more_than_half = 0\nargmax_less_than_half =0\n\nwith torch.no_grad():\n for data in test_loader:\n inputs, labels , fore_idx = data\n inputs, labels , fore_idx = inputs.to(\"cuda\"),labels.to(\"cuda\"), fore_idx.to(\"cuda\")\n outputs, alphas, avg_images = fore_net(inputs)\n\n _, predicted = torch.max(outputs.data, 1)\n\n for j in range(labels.size(0)):\n focus = torch.argmax(alphas[j])\n if alphas[j][focus] >= 0.5 :\n argmax_more_than_half += 1\n else:\n argmax_less_than_half += 1\n\n if(focus == fore_idx[j] and predicted[j] == labels[j]):\n focus_true_pred_true += 1\n elif(focus != fore_idx[j] and predicted[j] == labels[j]):\n focus_false_pred_true += 1\n elif(focus == fore_idx[j] and predicted[j] != labels[j]):\n focus_true_pred_false += 1\n elif(focus != fore_idx[j] and predicted[j] != labels[j]):\n focus_false_pred_false += 1\n\n total += labels.size(0)\n correct += (predicted == labels).sum().item()\n\nprint('Accuracy of the network on the 10000 test images: %d %%' % (\n 100 * correct / total))\nprint(\"total correct\", correct)\nprint(\"total train set images\", total)\n\nprint(\"focus_true_pred_true %d =============> FTPT : %d %%\" % (focus_true_pred_true , (100 * focus_true_pred_true / total) ) )\nprint(\"focus_false_pred_true %d =============> FFPT : %d %%\" % (focus_false_pred_true, (100 * focus_false_pred_true / total) ) )\nprint(\"focus_true_pred_false %d =============> FTPF : %d %%\" %( focus_true_pred_false , ( 100 * focus_true_pred_false / total) ) )\nprint(\"focus_false_pred_false %d =============> FFPF : %d %%\" % (focus_false_pred_false, ( 100 * focus_false_pred_false / total) ) )\n\nprint(\"argmax_more_than_half\",argmax_more_than_half)\nprint(\"argmax_less_than_half\",argmax_less_than_half)\n\nprint(\"=\"*100)\n# table4 = []\nentry = [2,'fg = '+ str(fg),'bg = '+str(bg),10000]\nentry.append((100 * focus_true_pred_true / total))\nentry.append( (100 * focus_false_pred_true / total))\nentry.append( ( 100 * focus_true_pred_false / total))\nentry.append( ( 100 * focus_false_pred_false / total))\nentry.append( argmax_more_than_half)\n\ntest_entry = entry \n\ntable3.append(entry)\n\nprint(tabulate(table3, headers=['S.No.', 'fg_class','bg_class','data_points','FTPT', 'FFPT', 'FTPF', 'FFPF', 'avg_img > 0.5'] ) )\n",
"Accuracy of the network on the 10000 test images: 99 %\ntotal correct 9947\ntotal train set images 10000\nfocus_true_pred_true 9947 =============> FTPT : 99 %\nfocus_false_pred_true 0 =============> FFPT : 0 %\nfocus_true_pred_false 53 =============> FTPF : 0 %\nfocus_false_pred_false 0 =============> FFPF : 0 %\nargmax_more_than_half 10000\nargmax_less_than_half 0\n====================================================================================================\n S.No. fg_class bg_class data_points FTPT FFPT FTPF FFPF avg_img > 0.5\n------- -------------- -------------------------- ------------- ------- ------ -------- ------ ---------------\n 1 fg = [1, 2, 3] bg = [0, 4, 5, 6, 7, 8, 9] 30000 99.3967 0 0.603333 0 30000\n 2 fg = [1, 2, 3] bg = [0, 4, 5, 6, 7, 8, 9] 10000 99.47 0 0.53 0 10000\n"
],
[
"dataiter = iter(testloader)\nbackground_data=[]\nbackground_label=[]\nforeground_data=[]\nforeground_label=[]\nbatch_size=10\n\nfor i in range(1000):\n images, labels = dataiter.next()\n for j in range(batch_size):\n if(classes[labels[j]] in background_classes):\n img = images[j].tolist()\n background_data.append(img)\n background_label.append(labels[j])\n else:\n img = images[j].tolist()\n foreground_data.append(img)\n foreground_label.append(labels[j])\n \nforeground_data = torch.tensor(foreground_data)\nforeground_label = torch.tensor(foreground_label)\nbackground_data = torch.tensor(background_data)\nbackground_label = torch.tensor(background_label)",
"_____no_output_____"
],
[
"test_images =[] #list of mosaic images, each mosaic image is saved as laist of 9 images\nfore_idx_test =[] #list of indexes at which foreground image is present in a mosaic image \ntest_label=[] # label of mosaic image = foreground class present in that mosaic\ntest_set_labels = []\nfor i in range(10000):\n set_idx = set()\n bg_idx = np.random.randint(0,7000,8)\n set_idx = set(background_label[bg_idx].tolist())\n fg_idx = np.random.randint(0,3000)\n set_idx.add(foreground_label[fg_idx].item())\n fg = np.random.randint(0,9)\n fore_idx_test.append(fg)\n image_list,label = create_mosaic_img(bg_idx,fg_idx,fg)\n test_images.append(image_list)\n test_label.append(label)\n test_set_labels.append(set_idx)\n",
"_____no_output_____"
],
[
"test_data = MosaicDataset(test_images,test_label,fore_idx_test)\nunseen_test_loader = DataLoader( test_data,batch_size= batch ,shuffle=False)",
"_____no_output_____"
]
],
[
[
"# Test summary on Test mosaic made from Testset of 10k CIFAR",
"_____no_output_____"
]
],
[
[
"fg = [fg1,fg2,fg3]\nbg = list(set([0,1,2,3,4,5,6,7,8,9])-set(fg))",
"_____no_output_____"
],
[
"correct = 0\ntotal = 0\ncount = 0\nflag = 1\nfocus_true_pred_true =0\nfocus_false_pred_true =0\nfocus_true_pred_false =0\nfocus_false_pred_false =0\n\nargmax_more_than_half = 0\nargmax_less_than_half =0\n\nwith torch.no_grad():\n for data in unseen_test_loader:\n inputs, labels , fore_idx = data\n inputs, labels , fore_idx = inputs.to(\"cuda\"),labels.to(\"cuda\"), fore_idx.to(\"cuda\")\n outputs, alphas, avg_images = fore_net(inputs)\n\n _, predicted = torch.max(outputs.data, 1)\n\n for j in range(labels.size(0)):\n focus = torch.argmax(alphas[j])\n if alphas[j][focus] >= 0.5 :\n argmax_more_than_half += 1\n else:\n argmax_less_than_half += 1\n\n if(focus == fore_idx[j] and predicted[j] == labels[j]):\n focus_true_pred_true += 1\n elif(focus != fore_idx[j] and predicted[j] == labels[j]):\n focus_false_pred_true += 1\n elif(focus == fore_idx[j] and predicted[j] != labels[j]):\n focus_true_pred_false += 1\n elif(focus != fore_idx[j] and predicted[j] != labels[j]):\n focus_false_pred_false += 1\n\n total += labels.size(0)\n correct += (predicted == labels).sum().item()\n\nprint('Accuracy of the network on the 10000 test images: %d %%' % (\n 100 * correct / total))\nprint(\"total correct\", correct)\nprint(\"total train set images\", total)\n\nprint(\"focus_true_pred_true %d =============> FTPT : %d %%\" % (focus_true_pred_true , (100 * focus_true_pred_true / total) ) )\nprint(\"focus_false_pred_true %d =============> FFPT : %d %%\" % (focus_false_pred_true, (100 * focus_false_pred_true / total) ) )\nprint(\"focus_true_pred_false %d =============> FTPF : %d %%\" %( focus_true_pred_false , ( 100 * focus_true_pred_false / total) ) )\nprint(\"focus_false_pred_false %d =============> FFPF : %d %%\" % (focus_false_pred_false, ( 100 * focus_false_pred_false / total) ) )\n\nprint(\"argmax_more_than_half\",argmax_more_than_half)\nprint(\"argmax_less_than_half\",argmax_less_than_half)\n\nprint(\"=\"*100)\n# table4 = []\nentry = [3,'fg = '+ str(fg),'bg = '+str(bg),10000]\nentry.append((100 * focus_true_pred_true / total))\nentry.append( (100 * focus_false_pred_true / total))\nentry.append( ( 100 * focus_true_pred_false / total))\nentry.append( ( 100 * focus_false_pred_false / total))\nentry.append( argmax_more_than_half)\n\ntest_entry = entry \n\ntable3.append(entry)\n\nprint(tabulate(table3, headers=['S.No.', 'fg_class','bg_class','data_points','FTPT', 'FFPT', 'FTPF', 'FFPF', 'avg_img > 0.5'] ) )\n",
"Accuracy of the network on the 10000 test images: 98 %\ntotal correct 9860\ntotal train set images 10000\nfocus_true_pred_true 9860 =============> FTPT : 98 %\nfocus_false_pred_true 0 =============> FFPT : 0 %\nfocus_true_pred_false 140 =============> FTPF : 1 %\nfocus_false_pred_false 0 =============> FFPF : 0 %\nargmax_more_than_half 10000\nargmax_less_than_half 0\n====================================================================================================\n S.No. fg_class bg_class data_points FTPT FFPT FTPF FFPF avg_img > 0.5\n------- -------------- -------------------------- ------------- ------- ------ -------- ------ ---------------\n 1 fg = [1, 2, 3] bg = [0, 4, 5, 6, 7, 8, 9] 30000 99.3967 0 0.603333 0 30000\n 2 fg = [1, 2, 3] bg = [0, 4, 5, 6, 7, 8, 9] 10000 99.47 0 0.53 0 10000\n 3 fg = [1, 2, 3] bg = [0, 4, 5, 6, 7, 8, 9] 10000 98.6 0 1.4 0 10000\n"
],
[
"",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
]
] |
e7e96e75f701487819fc5931fd50bfd12a2bf303 | 186,803 | ipynb | Jupyter Notebook | code/CH05/CH05_SEC06_1_LDA_Classify.ipynb | ksang/data-book-python | 08b5d3ae76980628887bc1ab2e17d83ddf07b00c | [
"MIT"
] | 1 | 2022-01-22T03:45:05.000Z | 2022-01-22T03:45:05.000Z | code/CH05/CH05_SEC06_1_LDA_Classify.ipynb | ksang/data-book-python | 08b5d3ae76980628887bc1ab2e17d83ddf07b00c | [
"MIT"
] | null | null | null | code/CH05/CH05_SEC06_1_LDA_Classify.ipynb | ksang/data-book-python | 08b5d3ae76980628887bc1ab2e17d83ddf07b00c | [
"MIT"
] | null | null | null | 600.652733 | 71,656 | 0.94529 | [
[
[
"import numpy as np\nimport matplotlib.pyplot as plt\nfrom matplotlib import rcParams\nfrom scipy import io\nimport os\nfrom mpl_toolkits import mplot3d\nfrom sklearn.discriminant_analysis import LinearDiscriminantAnalysis\nfrom sklearn.discriminant_analysis import QuadraticDiscriminantAnalysis\n\nrcParams.update({'font.size': 18})\nplt.rcParams['figure.figsize'] = [12, 12]",
"_____no_output_____"
],
[
"dogdata_w_mat = io.loadmat(os.path.join('..','DATA','dogData_w.mat'))\ncatdata_w_mat = io.loadmat(os.path.join('..','DATA','catData_w.mat'))\n\ndog_wave = dogdata_w_mat['dog_wave']\ncat_wave = catdata_w_mat['cat_wave']\n\nCD = np.concatenate((dog_wave,cat_wave),axis=1)\n\nu,s,vT = np.linalg.svd(CD-np.mean(CD),full_matrices=0)\nv = vT.T",
"_____no_output_____"
],
[
"xtrain = np.concatenate((v[:60,np.array([1,3])],v[80:140,np.array([1,3])]))\nlabel = np.repeat(np.array([1,-1]),60)\ntest = np.concatenate((v[60:80,np.array([1,3])],v[140:160,np.array([1,3])]))\n\nlda = LinearDiscriminantAnalysis()\ntest_class = lda.fit(xtrain, label).predict(test)\n\ntruth = np.repeat(np.array([1,-1]),20)\nE = 100*(1-np.sum(0.5*np.abs(test_class - truth))/40)\n\nfig,axs = plt.subplots(2)\naxs[0].bar(range(40),test_class)\n\naxs[1].plot(v[:80,1],v[:80,3],'ro',MarkerFaceColor=(0,1,0.2),MarkerEdgeColor='k',ms=12)\naxs[1].plot(v[80:,1],v[80:,3],'bo',MarkerFaceColor=(0.9,0,1),MarkerEdgeColor='k',ms=12)\n\nplt.show()",
"_____no_output_____"
],
[
"plt.rcParams['figure.figsize'] = [8,4]\nfig,axs = plt.subplots(1,2)\nfor j in range(2):\n U3 = np.flipud(np.reshape(u[:,2*j+1],(32,32)))\n axs[j].pcolor(np.rot90(U3),cmap='hot')\n axs[j].axis('off')",
"_____no_output_____"
],
[
"dogdata_mat = io.loadmat(os.path.join('..','DATA','dogData.mat'))\ncatdata_mat = io.loadmat(os.path.join('..','DATA','catData.mat'))\n\ndog = dogdata_mat['dog']\ncat = catdata_mat['cat']\n\nCD = np.concatenate((dog,cat),axis=1)\n\nu,s,vT = np.linalg.svd(CD-np.mean(CD),full_matrices=0)\nv = vT.T",
"_____no_output_____"
],
[
"plt.rcParams['figure.figsize'] = [12, 12]\nxtrain = np.concatenate((v[:60,np.array([1,3])],v[80:140,np.array([1,3])]))\nlabel = np.repeat(np.array([1,-1]),60)\ntest = np.concatenate((v[60:80,np.array([1,3])],v[140:160,np.array([1,3])]))\n\nlda = LinearDiscriminantAnalysis()\ntest_class = lda.fit(xtrain, label).predict(test)\n\nfig,axs = plt.subplots(2)\naxs[0].bar(range(40),test_class)\n\naxs[1].plot(v[:80,1],v[:80,3],'ro',MarkerFaceColor=(0,1,0.2),MarkerEdgeColor='k',ms=12)\naxs[1].plot(v[80:,1],v[80:,3],'bo',MarkerFaceColor=(0.9,0,1),MarkerEdgeColor='k',ms=12)\n\nplt.show()",
"_____no_output_____"
],
[
"## Cross-validate\nE = np.zeros(100)\n\nfor jj in range(100):\n r1 = np.random.permutation(80)\n r2 = np.random.permutation(80)\n ind1 = r1[:60]\n ind2 = r2[:60]+60\n ind1t = r1[60:80]\n ind2t = r2[60:80]+60\n \n xtrain = np.concatenate((v[ind1[:, np.newaxis],np.array([1,3])], v[ind2[:, np.newaxis],np.array([1,3])]))\n test = np.concatenate((v[ind1t[:, np.newaxis],np.array([1,3])], v[ind2t[:, np.newaxis],np.array([1,3])]))\n \n label = np.repeat(np.array([1,-1]),60)\n\n lda = LinearDiscriminantAnalysis()\n test_class = lda.fit(xtrain, label).predict(test)\n\n truth = np.repeat(np.array([1,-1]),20)\n E[jj] = 100*np.sum(np.abs(test_class-truth))/40\n \nplt.bar(range(100),E,color=(0.5,0.5,0.5))\nplt.plot(range(100),np.mean(E)*np.ones(100),'r:',LineWidth=3)\nplt.show()",
"_____no_output_____"
],
[
"dogdata_w_mat = io.loadmat(os.path.join('..','DATA','dogData_w.mat'))\ncatdata_w_mat = io.loadmat(os.path.join('..','DATA','catData_w.mat'))\n\ndog_wave = dogdata_w_mat['dog_wave']\ncat_wave = catdata_w_mat['cat_wave']\n\nCD = np.concatenate((dog_wave,cat_wave),axis=1)\n\nu,s,vT = np.linalg.svd(CD-np.mean(CD),full_matrices=0)\nv = vT.T",
"_____no_output_____"
],
[
"plt.rcParams['figure.figsize'] = [12, 6]\nfig,axs = plt.subplots(1,2)\nfor j in range(2):\n axs[j].plot(v[:80,1],v[:80,3],'ro',MarkerFaceColor=(0,1,0.2),MarkerEdgeColor='k',ms=12)\n axs[j].plot(v[80:,1],v[80:,3],'bo',MarkerFaceColor=(0.9,0,1),MarkerEdgeColor='k',ms=12)\n\n# Linear Discriminant\nxtrain = np.concatenate((v[:60,np.array([1,3])],v[80:140,np.array([1,3])]))\ntest = np.concatenate((v[60:80,np.array([1,3])],v[140:160,np.array([1,3])]))\nlabel = np.repeat(np.array([1,-1]),60)\n\n\n\nlda = LinearDiscriminantAnalysis().fit(xtrain, label)\ntest_class = lda.predict(test)\nK = -lda.intercept_[0]\nL = -lda.coef_[0]\n\n\nx = np.arange(-0.15,0.25,0.005)\naxs[0].plot(x,-(L[0]*x+K)/L[1],'k',LineWidth=2)\n\n\n# Quadratic Discriminant\nqda = QuadraticDiscriminantAnalysis().fit(xtrain, label)\ny = np.arange(-0.3,0.25,0.005)\nX,Y = np.meshgrid(x,y)\n\nZ = np.zeros_like(X)\n\nfor jj in range(len(x)):\n for kk in range(len(y)):\n Z[kk,jj] = qda.predict(np.array([[x[jj],y[kk]]]))\n\naxs[1].contour(X,Y,Z,0,colors='k',linewidths=2) \n\nplt.show()\n",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e974b6f5506cca8d9c16183decd3b917a89695 | 27,846 | ipynb | Jupyter Notebook | notebooks/example_container.ipynb | Mike7477/cta-lstchain | 977de95d7dfe046cd402dbf1df628384724a4316 | [
"BSD-3-Clause"
] | null | null | null | notebooks/example_container.ipynb | Mike7477/cta-lstchain | 977de95d7dfe046cd402dbf1df628384724a4316 | [
"BSD-3-Clause"
] | null | null | null | notebooks/example_container.ipynb | Mike7477/cta-lstchain | 977de95d7dfe046cd402dbf1df628384724a4316 | [
"BSD-3-Clause"
] | null | null | null | 35.654289 | 185 | 0.419809 | [
[
[
"# Example to use the custom Container for mono reconstruction",
"_____no_output_____"
]
],
[
[
"from lstchain.io.containers import DL1ParametersContainer\nfrom lstchain.reco.utils import guess_type\n\nfrom ctapipe.utils import get_dataset_path\nfrom ctapipe.io import HDF5TableWriter, HDF5TableReader\nfrom ctapipe.calib import CameraCalibrator\nfrom ctapipe.image import tailcuts_clean\nfrom ctapipe.io import event_source\nfrom ctapipe.image import hillas_parameters",
"_____no_output_____"
]
],
[
[
"## Options",
"_____no_output_____"
]
],
[
[
"infile = get_dataset_path('gamma_test_large.simtel.gz') \n\ndl1_parameters_filename = 'dl1.h5'\n\nallowed_tels = {1} # select LST1 only\nmax_events = 300 # limit the number of events to analyse in files - None if no limit\n\ncal = CameraCalibrator(r1_product='HESSIOR1Calibrator', extractor_product='NeighbourPeakIntegrator')\n\ncleaning_method = tailcuts_clean\ncleaning_parameters = {'boundary_thresh': 0,\n 'picture_thresh': 6,\n 'min_number_picture_neighbors': 2}",
"_____no_output_____"
]
],
[
[
"## R0 to DL1",
"_____no_output_____"
]
],
[
[
"dl1_container = DL1ParametersContainer() ",
"_____no_output_____"
],
[
"with HDF5TableWriter(filename=dl1_parameters_filename, group_name='events', overwrite=True) as writer:\n\n source = event_source(infile)\n source.allowed_tels = allowed_tels\n source.max_events = max_events\n\n for i, event in enumerate(source):\n if i%100==0:\n print(i)\n cal.calibrate(event)\n\n for telescope_id, dl1 in event.dl1.tel.items():\n tel = event.inst.subarray.tels[telescope_id]\n camera = tel.camera\n signal_pixels = cleaning_method(camera, dl1.image[0], \n **cleaning_parameters)\n\n image = dl1.image[0]\n image[~signal_pixels] = 0\n \n peakpos = dl1.peakpos[0]\n\n if image.sum() > 0:\n try:\n hillas = hillas_parameters(\n camera,\n image\n )\n \n except:\n break\n\n ## Fill container ##\n dl1_container.fill_mc(event)\n dl1_container.fill_hillas(hillas)\n dl1_container.fill_event_info(event)\n dl1_container.set_mc_core_distance(event, telescope_id)\n dl1_container.mc_type = guess_type(infile)\n dl1_container.set_timing_features(camera, image, peakpos, hillas)\n dl1_container.set_source_camera_position(event, telescope_id)\n dl1_container.set_disp([dl1_container.src_x, dl1_container.src_y], hillas)\n\n\n ## Save parameters for later training ##\n writer.write(camera.cam_id, [dl1_container])\n \n ",
"WARNING:ctapipe.io.hessioeventsource.HESSIOEventSource:Only one pyhessio event_source allowed at a time. Previous hessio file will be closed.\n"
],
[
"# The file has been created\n!ls -lsh dl1.h5",
"152 -rw-r--r-- 1 thomasvuillaume staff 75K Nov 19 14:31 dl1.h5\r\n"
]
],
[
[
"## Transparent data reading into the container",
"_____no_output_____"
]
],
[
[
"from ctapipe.io import HDF5TableReader\nwith HDF5TableReader(dl1_parameters_filename, mode='r+') as table:\n for c in table.read('/events/LSTCam', DL1ParametersContainer()):\n print(c.disp)",
"WARNING:ctapipe.io.hdf5tableio.HDF5TableReader:Table '/events/LSTCam' is missing column 'mc_type' that is in container DL1ParametersContainer. It will be skipped.\nWARNING:ctapipe.io.hdf5tableio.HDF5TableReader:Table '/events/LSTCam' is missing column 'impact' that is in container DL1ParametersContainer. It will be skipped.\nWARNING:ctapipe.io.hdf5tableio.HDF5TableReader:Table '/events/LSTCam' is missing column 'hadroness' that is in container DL1ParametersContainer. It will be skipped.\nWARNING:ctapipe.io.hdf5tableio.HDF5TableReader:Table '/events/LSTCam' is missing column 'wl' that is in container DL1ParametersContainer. It will be skipped.\nWARNING:ctapipe.io.hdf5tableio.HDF5TableReader:Table '/events/LSTCam' is missing column 'mc_shower_primary_id' that is in container DL1ParametersContainer. It will be skipped.\n"
]
],
[
[
"## The hdf5 file is also very easy to read with pandas",
"_____no_output_____"
]
],
[
[
"import pandas as pd\npd.read_hdf(dl1_parameters_filename, key='events/LSTCam')",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7e98024fd0eec7410729ab2ae0a4efc2cd1f218 | 52,729 | ipynb | Jupyter Notebook | Data Cleaning and Exploration.ipynb | DAatDU/CSM-consulting | 9cec7f0d25432de2eb42be85b86ec3df70e7faaf | [
"CC0-1.0"
] | null | null | null | Data Cleaning and Exploration.ipynb | DAatDU/CSM-consulting | 9cec7f0d25432de2eb42be85b86ec3df70e7faaf | [
"CC0-1.0"
] | null | null | null | Data Cleaning and Exploration.ipynb | DAatDU/CSM-consulting | 9cec7f0d25432de2eb42be85b86ec3df70e7faaf | [
"CC0-1.0"
] | null | null | null | 39.116469 | 612 | 0.435965 | [
[
[
"# Import pandas and read in the datasets\nimport pandas as pd\nimport os \nimport seaborn as sns\nimport matplotlib.pyplot as plt\nimport numpy as np\n\npath = 'C:/Users/petermorley/Desktop/DA301/Data/Cleaning_Stage'\nweather2010 = pd.read_csv('2010_weather_dataset.csv')\nweather2011 = pd.read_csv('2011_weather_dataset.csv')\nweather2012 = pd.read_csv('2012_weather_dataset.csv')\nweather2013 = pd.read_csv('2013_weather_dataset.csv')\nweather2014 = pd.read_csv('2014_weather_dataset.csv')\nweather2015 = pd.read_csv('2015_weather_dataset.csv')\nweather2016 = pd.read_csv('2016_weather_dataset.csv')\nweather2017 = pd.read_csv('2017_weather_dataset.csv')\nweather2018 = pd.read_csv('2018_weather_dataset.csv')\nweather2019 = pd.read_csv('2019_weather_dataset.csv')\nweather2020 = pd.read_csv('2020_weather_dataset.csv')",
"_____no_output_____"
],
[
"# In each year, select the counties that we will be analyzing\nriverside2011DF = weather2011[weather2011['Name'] == 'Riverside, CA, United States']\nbernadino2011DF = weather2011[weather2011['Name'] == 'San Bernardino, CA, United States']\nriverside2012DF = weather2012[weather2012['Name'] == 'Riverside, CA, United States']\nbernadino2012DF = weather2012[weather2012['Name'] == 'San Bernardino, CA, United States']\nriverside2013DF = weather2013[weather2013['Name'] == 'Riverside, CA, United States']\nbernadino2013DF = weather2013[weather2013['Name'] == 'San Bernardino, CA, United States']\nriverside2014DF = weather2014[weather2014['Name'] == 'Riverside, CA, United States']\nbernadino2014DF = weather2014[weather2014['Name'] == 'San Bernardino, CA, United States']\nriverside2015DF = weather2015[weather2015['Name'] == 'Riverside, CA, United States']\nbernadino2015DF = weather2015[weather2015['Name'] == 'San Bernardino, CA, United States']\nriverside2016DF = weather2016[weather2016['Name'] == 'Riverside, CA, United States']\nbernadino2016DF = weather2016[weather2016['Name'] == 'San Bernardino, CA, United States']\nriverside2017DF = weather2017[weather2017['Name'] == 'Riverside, CA, United States']\nbernadino2017DF = weather2017[weather2017['Name'] == 'San Bernardino, CA, United States']\nriverside2018DF = weather2018[weather2018['Name'] == 'Riverside, CA, United States']\nbernadino2018DF = weather2018[weather2018['Name'] == 'San Bernardino, CA, United States']\nriverside2019DF = weather2019[weather2019['Name'] == 'Riverside, CA, United States']\nbernadino2019DF = weather2019[weather2019['Name'] == 'San Bernardino, CA, United States']\nriverside2020DF = weather2020[weather2020['Name'] == 'Riverside, CA, United States']\nbernadino2020DF = weather2020[weather2020['Name'] == 'San Bernardino, CA, United States']\n#Combine all years and counties that we will be using into one dataset \ncountiesTotal = pd.concat([riverside2011DF, bernadino2011DF, riverside2012DF, bernadino2012DF, \n riverside2013DF, bernadino2013DF, riverside2014DF, bernadino2014DF, \n riverside2015DF, bernadino2015DF, riverside2016DF, bernadino2016DF, \n riverside2017DF, bernadino2017DF, riverside2018DF, bernadino2018DF, \n riverside2019DF, bernadino2019DF, riverside2020DF, bernadino2020DF])\nriversideTotal = pd.concat([riverside2011DF, riverside2012DF, riverside2013DF, riverside2014DF, \n riverside2015DF, riverside2016DF, riverside2017DF, riverside2018DF, \n riverside2018DF, riverside2020DF])\n \nbernardinoTotal = pd.concat([bernadino2011DF, bernadino2012DF, bernadino2013DF, bernadino2014DF, \n bernadino2015DF, bernadino2016DF, bernadino2017DF, bernadino2018DF, \n bernadino2019DF, bernadino2020DF])",
"_____no_output_____"
],
[
"# Cleaning the data further to split up the date column into individual columns \n\nDay = lambda x: int(x.split(\"/\")[1]) # int because the values for the date columns are still strings \nMonth = lambda x: int(x.split(\"/\")[0])\nYear = lambda x: int(x.split(\"/\")[2]) \n\nriversideTcopy = riversideTotal.copy() \nriversideTcopy[\"Year\"] = riversideTcopy[riversideTcopy[\"Date time\"].notna()][\"Date time\"].apply(Year) # making the new column and appling the lambda function above to the following 5 lines after this \nriversideTcopy[\"Month\"] = riversideTcopy[riversideTcopy[\"Date time\"].notna()][\"Date time\"].apply(Month)\nriversideTcopy[\"Day\"] = riversideTcopy[riversideTcopy[\"Date time\"].notna()][\"Date time\"].apply(Day)\nriversideTcopy.drop(columns=[\"Date time\"], inplace=True)\nriversideTcopy.set_index([\"Year\",\"Month\",\"Day\"])\n\nbernardinoTcopy = bernardinoTotal.copy() \nbernardinoTcopy[\"Year\"] = bernardinoTcopy[bernardinoTcopy[\"Date time\"].notna()][\"Date time\"].apply(Year) # making the new column and appling the lambda function above to the following 5 lines after this \nbernardinoTcopy[\"Month\"] = bernardinoTcopy[bernardinoTcopy[\"Date time\"].notna()][\"Date time\"].apply(Month)\nbernardinoTcopy[\"Day\"] = bernardinoTcopy[bernardinoTcopy[\"Date time\"].notna()][\"Date time\"].apply(Day)\nbernardinoTcopy.drop(columns=[\"Date time\"], inplace=True)\nbernardinoTcopy.set_index([\"Year\",\"Month\",\"Day\"])",
"_____no_output_____"
],
[
"# Computes the heat index based on relative humidity and temperature\nheatIndex = -42.379 + 2.04901523*countiesTotal[\"Temperature\"] + 10.14333127*countiesTotal[\"Relative Humidity\"] - 0.22475541*countiesTotal[\"Temperature\"]*countiesTotal[\"Relative Humidity\"] - 0.683783*(10**(-3))*(countiesTotal[\"Temperature\"])**2 - 5.481717*(10**(-2))*(countiesTotal[\"Relative Humidity\"])**2 + 1.22874*(10**(-3))*(countiesTotal[\"Temperature\"])**2 *countiesTotal[\"Relative Humidity\"] + 8.5282*(10**(-4))*(countiesTotal[\"Relative Humidity\"])**2 *countiesTotal[\"Temperature\"] - 1.99*(10**(-6))*(countiesTotal[\"Relative Humidity\"])**2 *(countiesTotal[\"Temperature\"])**2\ncountiesTotal[\"Heat Index\"] = heatIndex",
"_____no_output_____"
],
[
"# Creates the weather dataset containing the columns that we're interested in\nweather = countiesTotal[['Name', 'Date time', 'Maximum Temperature', 'Minimum Temperature', 'Temperature', \n 'Heat Index', 'Wind Chill','Precipitation', 'Wind Speed', 'Wind Direction', 'Wind Gust', \n 'Visibility', 'Cloud Cover', 'Relative Humidity', 'Conditions']]",
"_____no_output_____"
],
[
"weather.describe() # Outputs summary statistics. We're missing some values for wind chill so we will calculate those\n # using the wind chill formula. ",
"_____no_output_____"
],
[
"# We are not missing any values for the data that we need to calculate wind chill\nweather['Temperature'].isnull().sum()\nweather['Wind Speed'].isnull().sum()",
"_____no_output_____"
],
[
"# Computing the value for wind chill and assigning it as a column to the weather dataset\nwindChill = 35.78 + 0.6215*weather['Temperature'] - 35.75*(weather['Wind Speed']**0.16) + 0.4275*weather['Temperature']*(weather['Wind Speed']**0.16)\nweather[\"Wind Chill\"] = windChill",
"/opt/anaconda3/lib/python3.7/site-packages/ipykernel_launcher.py:3: SettingWithCopyWarning: \nA value is trying to be set on a copy of a slice from a DataFrame.\nTry using .loc[row_indexer,col_indexer] = value instead\n\nSee the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy\n This is separate from the ipykernel package so we can avoid doing imports until\n"
],
[
"# Created a value for wind chill for every row in the column\nweather['Wind Chill'].isnull().sum()",
"_____no_output_____"
],
[
"# We're still missing some values for wind gust. Since we don't have a formula to calculate this, we will remove\n# it from the dataset\nweather.describe()",
"_____no_output_____"
],
[
"weather['Wind Gust'].isnull().sum()",
"_____no_output_____"
],
[
"# Drop wind gust as a column in our data frame\nweather.drop([\"Wind Gust\"], axis =1, inplace=True) ",
"/opt/anaconda3/lib/python3.7/site-packages/pandas/core/frame.py:3997: SettingWithCopyWarning: \nA value is trying to be set on a copy of a slice from a DataFrame\n\nSee the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy\n errors=errors,\n"
],
[
"# Now we are no longer missing any value in the data frame \nweather.describe()",
"_____no_output_____"
],
[
"# The conditions variable has nine unique values. These can be generalized into four main categories:\n# clear, rain, cloudy, snow. We will assign these values to each corresponding value in the data frame column.\nprint(pd.unique(weather['Conditions']))",
"['Clear' 'Rain, Overcast' 'Partially cloudy' 'Overcast' 'Rain'\n 'Rain, Partially cloudy' 'Snow' 'Snow, Partially cloudy' 'Snow, Overcast']\n"
],
[
"#weather.loc[weather.Conditions == \"Clear\", 'Conditions'] = 0\n#weather.loc[weather.Conditions == \"Rain, Overcast\" or \"Rain\" or \"Rain, Partially cloudy\", 'Conditions'] = 1\n#weather.loc[weather.Conditions == \"Partially cloudy\" or \"Overcast\", 'Conditions'] = 2\n#weather.loc[weather.Conditions == \"Snow\" or \"Snow, Partially cloudy\" or \"Snow, Overcast\", 'Conditions'] = 3\n\n# Not sure how to make the conditions variable into the 4 groups",
"_____no_output_____"
],
[
"weather.describe()",
"_____no_output_____"
],
[
"weather.head()",
"_____no_output_____"
],
[
"sanBernadinoFires = pd.read_csv('hist_incident_data-san bernardino.csv')\nriversideFires = pd.read_csv('hist_incident_data-riverside.csv')",
"_____no_output_____"
],
[
"riversideFires.head()",
"_____no_output_____"
],
[
"riversideFires = riversideFires[['incident_date_created', 'incident_county', 'incident_acres_burned',\n 'incident_longitude', 'incident_latitude']]\nsanBernadinoFires = sanBernadinoFires[['incident_date_created', 'incident_county', 'incident_acres_burned',\n 'incident_longitude', 'incident_latitude']]",
"_____no_output_____"
],
[
"# From here we should decide how we want to join the data. I think leaving them separate is good for now since\n# we have to analyze weather and fires individually first. ",
"_____no_output_____"
],
[
"RcorrMatrix = riversideTotal.corr()\nRcorrMatrix",
"_____no_output_____"
],
[
"SBcorrMatrix = bernardinoTotal.corr()\nSBcorrMatrix",
"_____no_output_____"
],
[
"# trying to see a line graph for temperature over the past decade using all the temperature variables for both San Bernardino\n# and Riverside \nimport seaborn as sns\n\nRiversideTempPlot = sns.lineplot(x='Year', y='Temperature', data=riversideTcopy, sort = False)",
"_____no_output_____"
],
[
"RiversideMaxTempPlot = sns.lineplot(x='Year', y='Maximum Temperature', data=riversideTcopy, sort = False)",
"_____no_output_____"
],
[
"RiversideMinTempPlot = sns.lineplot(x='Year', y='Minimum Temperature', data=riversideTcopy, sort = False)",
"_____no_output_____"
],
[
"bernardinoTempPlot = sns.lineplot(x=\"Year\", y='Temperature', data=bernardinoTcopy, sort=False)",
"_____no_output_____"
],
[
"bernardinoMaxTempPlot = sns.lineplot(x=\"Year\", y='Maximum Temperature', data=bernardinoTcopy, sort=False)",
"_____no_output_____"
],
[
"bernardinoMinTempPlot = sns.lineplot(x=\"Year\", y='Minimum Temperature', data=bernardinoTcopy, sort=False)",
"_____no_output_____"
],
[
"# tried to use the original Date time column but it does not give a clear x axis\nbernardinoTemps = np.array(bernardinoTotal[\"Temperature\"])\nbernardinoYears = np.array(bernardinoTotal[\"Date time\"])\nplt.plot(bernardinoYears, bernardinoTemps)\nplt.show()",
"_____no_output_____"
],
[
"# looking at July 2013 in riverside for linegraph \nRiverside2013 = riversideTcopy[riversideTcopy[\"Year\"]==2013]\nJuly2013Riverside = Riverside2013[Riverside2013[\"Month\"]==7]\n\nriversideJ2013Days = July2013Riverside[\"Day\"]\nriversideJ2013Temps = July2013Riverside[\"Temperature\"]\n\nplt.plot(riversideJ2013Days, riversideJ2013Temps)\nplt.xlabel(\"Days of the July\")\nplt.ylabel(\"Temperature\")\nplt.title(\"Temperature Change in Riverside, CA in July 2013\")\nplt.show()",
"_____no_output_____"
],
[
"import dash\nimport dash_html_components as html\nimport plotly.graph_objects as go\nimport dash_core_components as dcc\nimport plotly.express as px\nfrom dash.dependencies import Input, Output\n\n\napp = dash.Dash()\n\ndf = riversideTcopy\n\napp.layout = html.Div(id = 'parent', children = [\n html.H1(id = 'H1', children = 'Styling using html components', style = {'textAlign':'center',\\\n 'marginTop':40,'marginBottom':40}),\n\n dcc.Dropdown( id = 'dropdown',\n options = [\n {'label':'Temperature', 'value':'Temperature' },\n {'label': 'Maximum Temperature', 'value':'Maximum Temperature'},\n {'label': 'Minimum Temperature', 'value':'Minimum Temperature'},\n ],\n value = 'Temperature'),\n dcc.Graph(id=\"box-plot\")\n ])\n\n \[email protected](\n Output(\"box-plot\", \"figure\"), \n [Input(\"x-axis\", \"value\"), \n Input(\"y-axis\", \"value\")])\ndef generate_chart(x, y):\n fig = px.box(df, x=x, y=y)\n return fig\n\n\n\nif __name__ == '__main__': \n app.run_server()",
"_____no_output_____"
],
[
"import dash\nimport dash_html_components as html\nimport plotly.graph_objects as go\nimport dash_core_components as dcc\nimport plotly.express as px\nfrom dash.dependencies import Input, Output\n\n\napp = dash.Dash()\n\ndf = px.data.stocks()\n\n\napp.layout = html.Div(id = 'parent', children = [\n html.H1(id = 'H1', children = 'Styling using html components', style = {'textAlign':'center',\\\n 'marginTop':40,'marginBottom':40}),\n\n dcc.Dropdown( id = 'dropdown',\n options = [\n {'label':'Google', 'value':'GOOG' },\n {'label': 'Apple', 'value':'AAPL'},\n {'label': 'Amazon', 'value':'AMZN'},\n ],\n value = 'GOOG'),\n dcc.Graph(id = 'bar_plot')\n ])\n \n \[email protected](Output(component_id='bar_plot', component_property= 'figure'),\n [Input(component_id='dropdown', component_property= 'value')])\ndef graph_update(dropdown_value):\n print(dropdown_value)\n fig = go.Figure([go.Scatter(x = df['date'], y = df['{}'.format(dropdown_value)],\\\n line = dict(color = 'firebrick', width = 4))\n ])\n \n fig.update_layout(title = 'Stock prices over time',\n xaxis_title = 'Dates',\n yaxis_title = 'Prices'\n )\n return fig \n\n\n\nif __name__ == '__main__': \n app.run_server()",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e981f3cfb0a241ef5a4835db5233b1e232ff43 | 53,212 | ipynb | Jupyter Notebook | DataProcessing_Trang.ipynb | trangtran72/CS585_FinalProject | 8d91a05d1720f6dffe5e90cb82eabc377a9f3aff | [
"MIT"
] | null | null | null | DataProcessing_Trang.ipynb | trangtran72/CS585_FinalProject | 8d91a05d1720f6dffe5e90cb82eabc377a9f3aff | [
"MIT"
] | null | null | null | DataProcessing_Trang.ipynb | trangtran72/CS585_FinalProject | 8d91a05d1720f6dffe5e90cb82eabc377a9f3aff | [
"MIT"
] | null | null | null | 44.306411 | 117 | 0.38232 | [
[
[
"import pandas as pd\nimport numpy as np",
"_____no_output_____"
],
[
"def readReview (filename):\n \"\"\"\n A function to read a movie review file, and return a Pandas data frame\n \n Parameter:\n filename: The txt file contains movie reviews\n Return: A Pandas dataframe\n \"\"\"\n \n dict ={}\n keys = []\n values = []\n \n f = open(filename, encoding = 'utf-8', errors = 'ignore')\n lines = f.readlines()\n for line in lines:\n line = line.strip()\n row = line.split('\\t')\n key = [words for segments in row for words in segments.split(':')][0]\n value =[words for segments in row for words in segments.split(':')][-1]\n keys.append(key)\n values.append(value)\n f.close()\n \n dict = dict.fromkeys(keys, 0)\n\n index_list = {k:[] for k in keys}\n index_list['product/productId'].append(list(range(0, len(keys), 9)))\n index_list['review/userId'].append(range(1, len(keys), 9))\n index_list['review/score'].append(range(4, len(keys), 9))\n index_list['review/summary'].append(range(6, len(keys), 9))\n index_list['review/text'].append(range(7, len(keys), 9))\n\n\n dict['product/productId'] = [values[i] for i in [j for i in index_list['product/productId'] for j in i]]\n dict['review/userId'] = [values[i] for i in [j for i in index_list['review/userId'] for j in i]]\n dict['review/score'] = [values[i] for i in [j for i in index_list['review/score'] for j in i]]\n dict['review/summary'] = [values[i] for i in [j for i in index_list['review/summary'] for j in i]]\n dict['review/text'] = [values[i] for i in [j for i in index_list['review/text'] for j in i]]\n\n df= pd.DataFrame(dict)\n df = df.dropna(how='all', axis=1)\n \n return df ",
"_____no_output_____"
],
[
"movies = readReview ('outaa.txt')",
"_____no_output_____"
],
[
"df = df.dropna(how='all', axis='columns')\ndf",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code"
]
] |
e7e983c387b1bced45ec577770ccd5d3df1b6738 | 6,994 | ipynb | Jupyter Notebook | notebooks/2021-09-08-cloudknot-save-all-tfrecs.ipynb | richford/hbn-pod2-qc | cf565595e0fae2f630c76904a04803b0caf5fedc | [
"BSD-3-Clause"
] | 2 | 2022-02-12T01:03:08.000Z | 2022-03-17T18:41:28.000Z | notebooks/2021-09-08-cloudknot-save-all-tfrecs.ipynb | richford/hbn-pod2-qc | cf565595e0fae2f630c76904a04803b0caf5fedc | [
"BSD-3-Clause"
] | 1 | 2022-03-17T16:13:19.000Z | 2022-03-17T19:13:16.000Z | notebooks/2021-09-08-cloudknot-save-all-tfrecs.ipynb | richford/hbn-pod2-qc | cf565595e0fae2f630c76904a04803b0caf5fedc | [
"BSD-3-Clause"
] | null | null | null | 28.315789 | 206 | 0.522162 | [
[
[
"import cloudknot as ck\nimport itertools\nimport numpy as np\nimport pandas as pd",
"_____no_output_____"
],
[
"input_dirs = [\"b0-tensorfa-dwiqc\"]",
"_____no_output_____"
],
[
"def create_tfrecs(s3_input_dir):\n import nobrainer\n import numpy as np\n import os\n import os.path as op\n import pandas as pd\n import re\n \n from glob import glob\n from s3fs import S3FileSystem\n\n # Download the QC scores from S3 FCP-INDI\n df_qc = pd.read_csv(\n \"s3://fcp-indi/data/Projects/HBN/BIDS_curated/derivatives/qsiprep/participants.tsv\",\n sep=\"\\t\",\n index_col=\"subject_id\"\n )\n\n # Download nifti files from S3 to local\n local_nifti_dir = \"niftis\"\n local_tfrec_dir = \"tfrecs\"\n os.makedirs(local_nifti_dir, exist_ok=True)\n os.makedirs(local_tfrec_dir, exist_ok=True)\n \n fs = S3FileSystem()\n fs.get(f\"hbn-pod2-deep-learning/{s3_input_dir}\", local_nifti_dir, recursive=True)\n\n nifti_files = [op.abspath(filename) for filename in glob(f\"{local_nifti_dir}/*.nii.gz\")]\n nifti_files = [fn for fn in nifti_files if \"irregularsize\" not in fn]\n sub_id_pattern = re.compile(\"sub-[a-zA-Z0-9]*\")\n subjects = [sub_id_pattern.search(s).group(0) for s in nifti_files]\n \n df_nifti = pd.DataFrame(data=nifti_files, index=subjects, columns=[\"features\"])\n df_nifti = df_nifti.merge(df_qc, left_index=True, right_index=True, how=\"left\")\n df_nifti.drop(\"scan_site_id\", axis=\"columns\", inplace=True)\n df_nifti.rename(columns={\"fibr + qsiprep rating\": \"labels\"}, inplace=True)\n\n filepaths = list(df_nifti.itertuples(index=False, name=None))\n \n n_channels = {\n \"b0-colorfa-rgb\": 3,\n \"combined\": 4,\n \"b0-tensorfa-dwiqc\": 5,\n }\n \n # Verify that all volumes have the same shape\n invalid = nobrainer.io.verify_features_labels(\n filepaths, volume_shape=(128, 128, 128, n_channels[s3_input_dir]),\n check_labels_int=False,\n check_labels_gte_zero=False,\n )\n print(\"Invalid:\", invalid)\n assert not invalid \n \n os.makedirs(local_tfrec_dir, exist_ok=True)\n\n nobrainer.tfrecord.write(\n features_labels=filepaths,\n filename_template=local_tfrec_dir + \"/data-all_shard-{shard:03d}.tfrec\",\n examples_per_shard=20\n )\n \n output_s3_dirs = {\n \"b0-colorfa-rgb\": \"tfrecs/b0-colorfa-rgb-nosplit\",\n \"combined\": \"tfrecs/b0-colorfa-4channel-nosplit\",\n \"b0-tensorfa-dwiqc\": \"tfrecs/b0-tensorfa-dwiqc-nosplit\"\n }\n \n df_nifti.to_csv(op.join(local_tfrec_dir, \"filepaths.csv\"))\n\n fs = S3FileSystem()\n fs.put(\n local_tfrec_dir,\n f\"hbn-pod2-deep-learning/{output_s3_dirs[s3_input_dir]}\",\n recursive=True\n )",
"_____no_output_____"
],
[
"di = ck.DockerImage(\n func=create_tfrecs,\n base_image=\"python:3.8\",\n github_installs=\"https://github.com/richford/nobrainer.git@enh/four-d\",\n overwrite=True\n)",
"WARNING:cloudknot.dockerimage:Warning, some imports not found by pipreqs. You will need to edit the Dockerfile by hand, e.g by installing from github. You need to install the following packages []\n"
],
[
"di.build(tags=[\"hbn-pod2-tfrecs-20210908\"])",
"_____no_output_____"
],
[
"repo = ck.aws.DockerRepo(name=ck.get_ecr_repo())",
"_____no_output_____"
],
[
"# The very first time you run this, this command could take a few minutes\ndi.push(repo=repo)",
"_____no_output_____"
],
[
"# Specify bid_percentage to use Spot instances\n# And make sure the volume size is large enough. 55-60 GB seems about right for HBN preprocessing. YMMV.\nknot = ck.Knot(\n name=\"hbn-pod2-tfrecs-20210917-0\",\n docker_image=di,\n pars_policies=(\"AmazonS3FullAccess\",),\n bid_percentage=100,\n memory=8000,\n job_def_vcpus=8,\n volume_size=100,\n max_vcpus=64,\n retries=1,\n aws_resource_tags={\"Project\": \"HBN-FCP-INDI\"},\n)",
"_____no_output_____"
],
[
"results = knot.map(input_dirs)",
"_____no_output_____"
],
[
"knot.view_jobs()",
"Job ID Name Status \n---------------------------------------------------------\nd95f98ad-22a8-4d7e-8a7d-62003b860776 hbn-pod2-tfrecs-20210917-0-0 SUBMITTED\n"
],
[
"knot.clobber(clobber_pars=True)",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e99206eec85cd9fe31e63e5c9d0cd8d6167a2a | 81,809 | ipynb | Jupyter Notebook | Part 8 - Transfer Learning.ipynb | Hamez/DL_PyTorch | 4c2367ec8f46aac7951ca3a2963fcf3d611cef6b | [
"MIT"
] | null | null | null | Part 8 - Transfer Learning.ipynb | Hamez/DL_PyTorch | 4c2367ec8f46aac7951ca3a2963fcf3d611cef6b | [
"MIT"
] | null | null | null | Part 8 - Transfer Learning.ipynb | Hamez/DL_PyTorch | 4c2367ec8f46aac7951ca3a2963fcf3d611cef6b | [
"MIT"
] | 2 | 2018-11-11T04:37:06.000Z | 2019-11-26T12:02:20.000Z | 63.319659 | 662 | 0.53642 | [
[
[
"# Transfer Learning\n\nIn this notebook, you'll learn how to use pre-trained networks to solved challenging problems in computer vision. Specifically, you'll use networks trained on [ImageNet](http://www.image-net.org/) [available from torchvision](http://pytorch.org/docs/0.3.0/torchvision/models.html). \n\nImageNet is a massive dataset with over 1 million labeled images in 1000 categories. It's used to train deep neural networks using an architecture called convolutional layers. I'm not going to get into the details of convolutional networks here, but if you want to learn more about them, please [watch this](https://www.youtube.com/watch?v=2-Ol7ZB0MmU).\n\nOnce trained, these models work astonishingly well as feature detectors for images they weren't trained on. Using a pre-trained network on images not in the training set is called transfer learning. Here we'll use transfer learning to train a network that can classify our cat and dog photos with near perfect accuracy.\n\nWith `torchvision.models` you can download these pre-trained networks and use them in your applications. We'll include `models` in our imports now.",
"_____no_output_____"
]
],
[
[
"%matplotlib inline\n%config InlineBackend.figure_format = 'retina'\n\nimport matplotlib.pyplot as plt\n\nimport torch\nfrom torch import nn\nfrom torch import optim\nimport torch.nn.functional as F\nfrom torchvision import datasets, transforms, models",
"_____no_output_____"
]
],
[
[
"Most of the pretrained models require the input to be 224x224 images. Also, we'll need to match the normalization used when the models were trained. Each color channel was normalized separately, the means are `[0.485, 0.456, 0.406]` and the standard deviations are `[0.229, 0.224, 0.225]`.",
"_____no_output_____"
]
],
[
[
"data_dir = 'Cat_Dog_data'\n\n# TODO: Define transforms for the training data and testing data\ntrain_transforms = transforms.Compose([transforms.RandomRotation(30),\n transforms.RandomResizedCrop(224),\n transforms.RandomHorizontalFlip(),\n transforms.ToTensor(),\n transforms.Normalize([0.485, 0.456, 0.406], \n [0.229, 0.224, 0.225])])\ntest_transforms = transforms.Compose([transforms.Resize(256),\n transforms.CenterCrop(224),\n transforms.ToTensor(),\n transforms.Normalize([0.485, 0.456, 0.406], \n [0.229, 0.224, 0.225])])\n\n# Pass transforms in here, then run the next cell to see how the transforms look\ntrain_data = datasets.ImageFolder(data_dir + '/train', transform=train_transforms)\ntest_data = datasets.ImageFolder(data_dir + '/test', transform=test_transforms)\n\ntrainloader = torch.utils.data.DataLoader(train_data, batch_size=64, shuffle=True)\ntestloader = torch.utils.data.DataLoader(test_data, batch_size=32)",
"_____no_output_____"
]
],
[
[
"We can load in a model such as [DenseNet](http://pytorch.org/docs/0.3.0/torchvision/models.html#id5). Let's print out the model architecture so we can see what's going on.",
"_____no_output_____"
]
],
[
[
"model = models.densenet121(pretrained=True)\nmodel",
"C:\\Users\\jdmcd\\Anaconda3\\lib\\site-packages\\torchvision\\models\\densenet.py:212: UserWarning: nn.init.kaiming_normal is now deprecated in favor of nn.init.kaiming_normal_.\n nn.init.kaiming_normal(m.weight.data)\n"
]
],
[
[
"This model is built out of two main parts, the features and the classifier. The features part is a stack of convolutional layers and overall works as a feature detector that can be fed into a classifier. The classifier part is a single fully-connected layer `(classifier): Linear(in_features=1024, out_features=1000)`. This layer was trained on the ImageNet dataset, so it won't work for our specific problem. That means we need to replace the classifier, but the features will work perfectly on their own. In general, I think about pre-trained networks as amazingly good feature detectors that can be used as the input for simple feed-forward classifiers.",
"_____no_output_____"
]
],
[
[
"# Freeze parameters so we don't backprop through them\nfor param in model.parameters():\n param.requires_grad = False\n\nfrom collections import OrderedDict\nclassifier = nn.Sequential(OrderedDict([\n ('fc1', nn.Linear(1024, 500)),\n ('relu', nn.ReLU()),\n ('fc2', nn.Linear(500, 2)),\n ('output', nn.LogSoftmax(dim=1))\n ]))\n \nmodel.classifier = classifier\nmodel",
"_____no_output_____"
]
],
[
[
"With our model built, we need to train the classifier. However, now we're using a **really deep** neural network. If you try to train this on a CPU like normal, it will take a long, long time. Instead, we're going to use the GPU to do the calculations. The linear algebra computations are done in parallel on the GPU leading to 100x increased training speeds. It's also possible to train on multiple GPUs, further decreasing training time.\n\nPyTorch, along with pretty much every other deep learning framework, uses [CUDA](https://developer.nvidia.com/cuda-zone) to efficiently compute the forward and backwards passes on the GPU. In PyTorch, you move your model parameters and other tensors to the GPU memory using `model.to('cuda')`. You can move them back from the GPU with `model.to('cpu')` which you'll commonly do when you need to operate on the network output outside of PyTorch. As a demonstration of the increased speed, I'll compare how long it takes to perform a forward and backward pass with and without a GPU.",
"_____no_output_____"
]
],
[
[
"import time",
"_____no_output_____"
],
[
"#for device in ['cpu', 'cuda']:\ndevice = 'cpu'\ncriterion = nn.NLLLoss()\n# Only train the classifier parameters, feature parameters are frozen\noptimizer = optim.Adam(model.classifier.parameters(), lr=0.001)\n\nmodel.to(device)\n\nfor ii, (inputs, labels) in enumerate(trainloader):\n\n # Move input and label tensors to the GPU\n inputs, labels = inputs.to(device), labels.to(device)\n\n start = time.time()\n\n outputs = model.forward(inputs)\n loss = criterion(outputs, labels)\n loss.backward()\n optimizer.step()\n\n if ii==3:\n break\n\nprint(f\"Device = {device}; Time per batch: {(time.time() - start)/3:.3f} seconds\")",
"Device = cpu; Time per batch: 6.865 seconds\n"
]
],
[
[
"You can write device agnostic code which will automatically use CUDA if it's enabled like so:\n```python\n# at beginning of the script\ndevice = torch.device(\"cuda:0\" if torch.cuda.is_available() else \"cpu\")\n\n...\n\n# then whenever you get a new Tensor or Module\n# this won't copy if they are already on the desired device\ninput = data.to(device)\nmodel = MyModule(...).to(device)\n```\n\nFrom here, I'll let you finish training the model. The process is the same as before except now your model is much more powerful. You should get better than 95% accuracy easily.\n\n>**Exercise:** Train a pretrained models to classify the cat and dog images. Continue with the DenseNet model, or try ResNet, it's also a good model to try out first. Make sure you are only training the classifier and the parameters for the features part are frozen.",
"_____no_output_____"
]
],
[
[
"# TODO: Train a model with a pre-trained network\ndevice = 'cpu'\ncriterion = nn.NLLLoss()\n# Only train the classifier parameters, feature parameters are frozen\noptimizer = optim.Adam(model.classifier.parameters(), lr=0.001)\nmodel.classifier.fc1.weight\nmodel.to(device)\n\nfor ii, (inputs, labels) in enumerate(trainloader):\n\n # Move input and label tensors to the GPU\n inputs, labels = inputs.to(device), labels.to(device)\n\n start = time.time()\n\n outputs = model.forward(inputs)\n loss = criterion(outputs, labels)\n loss.backward()\n optimizer.step()\n \nmodel.classifier.fc1.weight",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7e9ae57e51c44eb201d9d1ed53eda25793e1bd5 | 52,913 | ipynb | Jupyter Notebook | PyCitySchools/.ipynb_checkpoints/PyCitySchools_starter-checkpoint.ipynb | seidyp/pandas-analysis-education | 576478d9db6b70d532d02e9daf6fdbae7a0d9b41 | [
"Apache-2.0"
] | null | null | null | PyCitySchools/.ipynb_checkpoints/PyCitySchools_starter-checkpoint.ipynb | seidyp/pandas-analysis-education | 576478d9db6b70d532d02e9daf6fdbae7a0d9b41 | [
"Apache-2.0"
] | null | null | null | PyCitySchools/.ipynb_checkpoints/PyCitySchools_starter-checkpoint.ipynb | seidyp/pandas-analysis-education | 576478d9db6b70d532d02e9daf6fdbae7a0d9b41 | [
"Apache-2.0"
] | null | null | null | 34.926073 | 202 | 0.393797 | [
[
[
"### Note\n* Instructions have been included for each segment. You do not have to follow them exactly, but they are included to help you think through the steps.",
"_____no_output_____"
]
],
[
[
"# Dependencies and Setup\nimport pandas as pd\n\n# File to Load (Remember to Change These)\nschool_data_to_load = \"Resources/schools_complete.csv\"\nstudent_data_to_load = \"Resources/students_complete.csv\"\n\n# Read School and Student Data File and store into Pandas DataFrames\nschool_data = pd.read_csv(school_data_to_load)\nstudent_data = pd.read_csv(student_data_to_load)\n\n# Combine the data into a single dataset. \nschool_data_complete = pd.merge(student_data, school_data, how=\"left\", on=[\"school_name\", \"school_name\"])",
"_____no_output_____"
]
],
[
[
"## District Summary\n\n* Calculate the total number of schools\n\n* Calculate the total number of students\n\n* Calculate the total budget\n\n* Calculate the average math score \n\n* Calculate the average reading score\n\n* Calculate the percentage of students with a passing math score (70 or greater)\n\n* Calculate the percentage of students with a passing reading score (70 or greater)\n\n* Calculate the percentage of students who passed math **and** reading (% Overall Passing)\n\n* Create a dataframe to hold the above results\n\n* Optional: give the displayed data cleaner formatting",
"_____no_output_____"
],
[
"## School Summary",
"_____no_output_____"
],
[
"* Create an overview table that summarizes key metrics about each school, including:\n * School Name\n * School Type\n * Total Students\n * Total School Budget\n * Per Student Budget\n * Average Math Score\n * Average Reading Score\n * % Passing Math\n * % Passing Reading\n * % Overall Passing (The percentage of students that passed math **and** reading.)\n \n* Create a dataframe to hold the above results",
"_____no_output_____"
],
[
"## Top Performing Schools (By % Overall Passing)",
"_____no_output_____"
],
[
"* Sort and display the top five performing schools by % overall passing.",
"_____no_output_____"
],
[
"## Bottom Performing Schools (By % Overall Passing)",
"_____no_output_____"
],
[
"* Sort and display the five worst-performing schools by % overall passing.",
"_____no_output_____"
],
[
"## Math Scores by Grade",
"_____no_output_____"
],
[
"* Create a table that lists the average Reading Score for students of each grade level (9th, 10th, 11th, 12th) at each school.\n\n * Create a pandas series for each grade. Hint: use a conditional statement.\n \n * Group each series by school\n \n * Combine the series into a dataframe\n \n * Optional: give the displayed data cleaner formatting",
"_____no_output_____"
],
[
"## Reading Score by Grade ",
"_____no_output_____"
],
[
"* Perform the same operations as above for reading scores",
"_____no_output_____"
],
[
"## Scores by School Spending",
"_____no_output_____"
],
[
"* Create a table that breaks down school performances based on average Spending Ranges (Per Student). Use 4 reasonable bins to group school spending. Include in the table each of the following:\n * Average Math Score\n * Average Reading Score\n * % Passing Math\n * % Passing Reading\n * Overall Passing Rate (Average of the above two)",
"_____no_output_____"
],
[
"## Scores by School Size",
"_____no_output_____"
],
[
"* Perform the same operations as above, based on school size.",
"_____no_output_____"
],
[
"## Scores by School Type",
"_____no_output_____"
],
[
"* Perform the same operations as above, based on school type",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
]
] |
e7e9b248c1dbbb5b6a3684b59ccbdc8b212ac968 | 599,027 | ipynb | Jupyter Notebook | notebooks/viz_2.ipynb | jf-silverman/energy_forecasting | 4550b622641676c899719069bbac06600a23cc76 | [
"CC0-1.0"
] | null | null | null | notebooks/viz_2.ipynb | jf-silverman/energy_forecasting | 4550b622641676c899719069bbac06600a23cc76 | [
"CC0-1.0"
] | null | null | null | notebooks/viz_2.ipynb | jf-silverman/energy_forecasting | 4550b622641676c899719069bbac06600a23cc76 | [
"CC0-1.0"
] | 1 | 2022-03-30T03:25:53.000Z | 2022-03-30T03:25:53.000Z | 612.502045 | 100,664 | 0.941453 | [
[
[
"# Visualizations 2 #\n## A notebook of exploratory data analysis for energy forecasting ##",
"_____no_output_____"
],
[
"**Run Imports**",
"_____no_output_____"
]
],
[
[
"import pandas as pd\nimport numpy as np\nimport matplotlib.pyplot as plt\nimport seaborn as sns\nfrom datetime import datetime as dt",
"_____no_output_____"
]
],
[
[
"**Seaborn plot formatting**",
"_____no_output_____"
]
],
[
[
"sns.set_context('paper', font_scale= 1.5) # size of text/graph elements\nsns.despine() # takes away axes on charts with no grid\nsns.set_style('darkgrid') # background of chart color and if grid is present",
"_____no_output_____"
]
],
[
[
"**Reading in the data**",
"_____no_output_____"
]
],
[
[
"nrg = pd.read_csv('../data/all_erco_energy_cst.csv')",
"_____no_output_____"
]
],
[
[
"**Converting the date column to datetime type for indexing**",
"_____no_output_____"
]
],
[
[
"nrg['datetime'] = pd.to_datetime(nrg['datetime'].str[:-3],format='%Y%m%dT%H')",
"_____no_output_____"
]
],
[
[
"**Setting the date column as index**",
"_____no_output_____"
]
],
[
[
"nrg.info()",
"<class 'pandas.core.frame.DataFrame'>\nRangeIndex: 57865 entries, 0 to 57864\nData columns (total 13 columns):\n # Column Non-Null Count Dtype \n--- ------ -------------- ----- \n 0 datetime 57865 non-null datetime64[ns]\n 1 demand 57660 non-null float64 \n 2 plant 57852 non-null object \n 3 net_generation 57649 non-null float64 \n 4 coal 31513 non-null float64 \n 5 hydro 31513 non-null float64 \n 6 natural_gas 31513 non-null float64 \n 7 nuclear 31513 non-null float64 \n 8 other 31513 non-null float64 \n 9 solar 31513 non-null float64 \n 10 wind 31513 non-null float64 \n 11 total_interchange 57631 non-null float64 \n 12 forecast 57817 non-null float64 \ndtypes: datetime64[ns](1), float64(11), object(1)\nmemory usage: 5.7+ MB\n"
]
],
[
[
"**Dropping records that in the data for forcasting that don't have energy type info**",
"_____no_output_____"
]
],
[
[
"nrg.dropna(inplace=True) # removes 26,000 records from before July 2018",
"_____no_output_____"
]
],
[
[
"**Making an hour column for plotting (0 to 23 hrs)**",
"_____no_output_____"
]
],
[
[
"nrg['hour_num'] = pd.to_numeric(nrg['datetime'].dt.strftime('%H'),downcast='integer')",
"_____no_output_____"
],
[
"# making a 'day_night' column, where 6am to 6pm is the day value; rest is night.\ndef calc_col_vals(row):\n if row['hour_num']<6 or row['hour_num']>17:\n return 'Night'\n elif row['hour_num']>5 and row['hour_num']<18:\n return 'Day'\n else:\n return 'WTF'",
"_____no_output_____"
],
[
"# applying day/night values to new column 'day_night'\nnrg['day_night'] = nrg.apply(lambda row: calc_col_vals(row), axis=1)",
"_____no_output_____"
],
[
"# checking the value counts\nnrg.apply(lambda row: calc_col_vals(row), axis=1).value_counts()",
"_____no_output_____"
],
[
"# day of the week\nnrg['weekday'] = nrg['datetime'].dt.strftime('%a')",
"_____no_output_____"
],
[
"# numbered day of week for chart order\nnrg['weekday_num'] = pd.to_numeric(nrg['datetime'].dt.strftime('%w'))",
"_____no_output_____"
],
[
"# an am/pm column, but not sure this is useful\n#msnrg['am_pm'] = msnrg['datetime'].dt.strftime('%p')",
"_____no_output_____"
],
[
"nrg['month'] = nrg['datetime'].dt.strftime('%b')",
"_____no_output_____"
],
[
"nrg['month_num'] = nrg['datetime'].dt.strftime('%m')",
"_____no_output_____"
],
[
"nrg['month_num'] = pd.to_numeric(nrg['month_num'])",
"_____no_output_____"
],
[
"nrg.isnull().sum()",
"_____no_output_____"
],
[
"nrg.info()",
"<class 'pandas.core.frame.DataFrame'>\nInt64Index: 31460 entries, 11 to 31523\nData columns (total 19 columns):\n # Column Non-Null Count Dtype \n--- ------ -------------- ----- \n 0 datetime 31460 non-null datetime64[ns]\n 1 demand 31460 non-null float64 \n 2 plant 31460 non-null object \n 3 net_generation 31460 non-null float64 \n 4 coal 31460 non-null float64 \n 5 hydro 31460 non-null float64 \n 6 natural_gas 31460 non-null float64 \n 7 nuclear 31460 non-null float64 \n 8 other 31460 non-null float64 \n 9 solar 31460 non-null float64 \n 10 wind 31460 non-null float64 \n 11 total_interchange 31460 non-null float64 \n 12 forecast 31460 non-null float64 \n 13 hour_num 31460 non-null int8 \n 14 day_night 31460 non-null object \n 15 weekday 31460 non-null object \n 16 weekday_num 31460 non-null int64 \n 17 month 31460 non-null object \n 18 month_num 31460 non-null int64 \ndtypes: datetime64[ns](1), float64(11), int64(2), int8(1), object(4)\nmemory usage: 4.6+ MB\n"
],
[
"# Melting Energy Sources into a single column for seaborn viz\nmelt_nrg = pd.melt(nrg, \n id_vars= ['datetime', 'month','month_num','weekday','weekday_num',\n 'day_night', 'hour_num'],\n value_vars=['net_generation','coal',\n 'hydro','natural_gas','nuclear', 'other',\n 'solar','wind'], \n var_name='energy_type',\n value_name='energy_mwh')",
"_____no_output_____"
],
[
"melt_nrg.info()",
"<class 'pandas.core.frame.DataFrame'>\nRangeIndex: 251680 entries, 0 to 251679\nData columns (total 9 columns):\n # Column Non-Null Count Dtype \n--- ------ -------------- ----- \n 0 datetime 251680 non-null datetime64[ns]\n 1 month 251680 non-null object \n 2 month_num 251680 non-null int64 \n 3 weekday 251680 non-null object \n 4 weekday_num 251680 non-null int64 \n 5 day_night 251680 non-null object \n 6 hour_num 251680 non-null int8 \n 7 energy_type 251680 non-null object \n 8 energy_mwh 251680 non-null float64 \ndtypes: datetime64[ns](1), float64(1), int64(2), int8(1), object(4)\nmemory usage: 15.6+ MB\n"
],
[
"melt_nrg.set_index('datetime')",
"_____no_output_____"
],
[
"#['net_generation','coal', 'hydro','natural_gas','nuclear', 'other', 'solar','wind']\n#colors={'demand':'#007EB5', 'net_generation':'#EC5064', 'coal':'#F9A834','hydro':'#B4CF68', 'natural gas':'#EF8D00', 'oil':'#FFD083', 'nuclear':'#007043', 'other':'#00F995', 'solar':'#00D982', 'wind':'#00965A', 'total_interchange': '#2A2A2A', 'forecast': '#84#1F9'}\ncolors=['#EC5064','#2A2A2A','#007EB5', '#EF8D00','#FFAAB0','#007043','#FFD083', '#00F995']\ncustomPallete = sns.set_palette(sns.color_palette(colors))\n\nfigtitle = \"Mean Monthly Energy Generation 2018-2021\" \nplt.figure(figsize=(14,6))\nsns.lineplot(x='month_num', y='energy_mwh', hue='energy_type', style='energy_type',\n data=melt_nrg, legend='brief'); \nplt.xticks(ticks=[1,2,3,4,5,6,7,8,9,10,11,12], \n labels=['Jan','Feb','Mar','Apr','May','Jun',\n 'Jul','Aug','Sep','Oct','Nov','Dec']); #use ticks to get months\nplt.legend(labels=['Net Generation', 'Coal', 'Hydro', 'Natural Gas', 'Nuclear', 'Other', 'Solar', 'Wind'])\nplt.ylabel(\"Mega Watt Hours\")\nplt.title(figtitle)\nplt.savefig('../output/' + figtitle + '.png', dpi=300) ",
"_____no_output_____"
],
[
"#nrg = nrg.set_index('datetime');",
"_____no_output_____"
],
[
"plt.figure(figsize=(14,6));\nfigtitle = 'Mean Monthly Hyrdo Generation 2018-2021'\n# Draw a nested boxplot to show bills by day and time\nsns.barplot(x=\"month\", y=\"hydro\",\n data=nrg.sort_values(by='month_num'), palette='Blues_r')\nsns.despine(offset=10, trim=True)\n\nplt.ylabel(\"Mega Watt Hours\")\nplt.xlabel('')\nplt.title(figtitle)\nplt.savefig('../output/' + figtitle + '.png', dpi=300)",
"_____no_output_____"
],
[
"plt.figure(figsize=(14,6));\nfigtitle = \"Mean Monthly Solar Generation 2018-2021\"\n# Draw a nested boxplot to show bills by day and time\nsns.barplot(x=\"month\", y=\"solar\",\n data=nrg.sort_values(by='month_num'), palette='Spectral')\nsns.despine(offset=10, trim=True)\n\nplt.ylabel(\"Mega Watt Hours\")\nplt.xlabel('')\nplt.title(figtitle)\nplt.savefig('../output/' + figtitle + '.png', dpi=300)",
"_____no_output_____"
],
[
"df = nrg[(nrg['datetime'] > '2019-05-01 01:00:00') & (nrg['datetime'] < '2019-05-31 01:00:00')].set_index('datetime')\n\nfigtitle = \"Daily Solar Generation for May 2019\"\nplt.figure(figsize=(16,4))\nsns.lineplot(x='datetime', y='solar', data= df)\n\nplt.ylim(bottom=0, top=2000)\nplt.title(figtitle)\nplt.xlabel('')\nplt.ylabel(\"Mega Watt Hours\")\nplt.savefig('../output/' + figtitle + '.png', dpi=300)",
"_____no_output_____"
],
[
"df = nrg[(nrg['datetime'] > '2019-07-31 01:00:00') & (nrg['datetime'] < '2019-08-31 01:00:00')]\n\nfigtitle = \"Daily Solar Generation for August 2019\"\nplt.figure(figsize=(16,4))\nsns.lineplot(x='datetime', y='solar', data= df)\nplt.ylim(bottom=0, top=2000)\nplt.title(figtitle)\nplt.xlabel('')\nplt.ylabel(\"Mega Watt Hours\")\nplt.savefig('../output/' + figtitle + '.png', dpi=300)",
"_____no_output_____"
],
[
"df = nrg[(nrg['datetime'] > '2019-01-01 01:00:00') & (nrg['datetime'] < '2019-01-31 01:00:00')]\n\nfigtitle = \"Daily Solar Generation for January 2019\"\nplt.figure(figsize=(16,4))\nsns.lineplot(x='datetime', y='solar', data= df)\nplt.ylim(bottom=-100, top=2000)\nplt.title(figtitle)\nplt.xlabel('')\nplt.ylabel(\"Mega Watt Hours\")\nplt.savefig('../output/' + figtitle + '.png', dpi=300)",
"_____no_output_____"
],
[
"#plt.figure(figsize=(9,6));\nfigtitle = \"Mean Weekly Energy Demand 2018-2021\"\nsns.relplot(x=\"weekday\", y='demand',kind=\"line\", color='#007EB5',\n data= nrg.sort_values(by='weekday_num'), height= 4, \n aspect=3).set(title= figtitle, xlabel='', ylabel='Megawatt Hours');\nplt.savefig('../output/' + figtitle + '.png', dpi=300) ",
"_____no_output_____"
],
[
"figtitle = \"Mean Daily Energy Demand\"\n#plt.figure(figsize=(9,6));\nsns.relplot(x=\"hour_num\", y='demand',kind=\"line\",\n data=nrg, height=4, aspect=3).set(xlabel='',ylabel='Megawatt Hours', title=figtitle);\nplt.savefig('../output/' + figtitle + '.png', dpi=300) ",
"_____no_output_____"
],
[
"plt.figure(figsize=(9,6));\nfigtitle = \"Variation in Weekly Demand\"\nsns.violinplot(data=nrg, order=['Sun','Mon','Tue','Wed','Thu','Fri','Sat'], \n x=\"weekday\", y=\"demand\", hue=\"day_night\", hue_order=['Day','Night'],\n split=True, inner=\"quart\", linewidth=1,\n palette='viridis_r').set(title=figtitle, xlabel='', ylabel='Megawatt Hours')\n\nsns.despine(left=True)\nplt.savefig('../output/' + figtitle + '.png', dpi=300) ",
"_____no_output_____"
],
[
"plt.figure(figsize=(6,6));\nfigtitle = \"Variation in Daily Demand\"\nsns.violinplot(data=nrg, \n x=\"day_night\", y=\"demand\", order=['Day','Night'],\n split=False, inner=\"quart\", linewidth=1,\n palette='viridis_r').set(title=figtitle,ylabel='Megawatt Hours') \nsns.despine(left=True)\nplt.savefig('../output/' + figtitle + '.png', dpi=300) #6am to 6pm and 6pm to 6pm",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e9b7ec95f696e3b4882ede24ddf6082a18303f | 65,251 | ipynb | Jupyter Notebook | Coursework/Part 2 Q-Learning for FrozenLake-v0.ipynb | vieliashevskyi/lembs-datascience-school | 0335ffad7fcbfe2cb9bd2ae3a4af5a971062c4bb | [
"MIT"
] | null | null | null | Coursework/Part 2 Q-Learning for FrozenLake-v0.ipynb | vieliashevskyi/lembs-datascience-school | 0335ffad7fcbfe2cb9bd2ae3a4af5a971062c4bb | [
"MIT"
] | 2 | 2020-06-26T12:04:34.000Z | 2021-02-09T16:37:40.000Z | Coursework/Part 2 Q-Learning for FrozenLake-v0.ipynb | vieliashevskyi/lembs-datascience-school | 0335ffad7fcbfe2cb9bd2ae3a4af5a971062c4bb | [
"MIT"
] | null | null | null | 49.320484 | 551 | 0.628741 | [
[
[
"## Table of Contents\n- [Part 2: Q-Learning for FrozenLake-v0, OpenAI Gym environment](#frozen-lake)\n - [Q-Learning Approach](#q-learn)\n - [OpenAI Gym Stochastic FrozenLake approach](#stochastic-field)\n - [Personal Deterministic FrozenLake approach](#deterministic-field)\n - [Play Against Environment](#pve)\n - [DNN Approach](#dnn)",
"_____no_output_____"
],
[
"## Part 2: Q-Learning for FrozenLake-v0, OpenAI Gym environment <a class=\"anchor\" id=\"frozen-lake\"></a>",
"_____no_output_____"
],
[
"The idea behind this is, just parsing data is not that exciting... aaannnd, I don't have time to make **Self taught Quantum Checkers** the _**thing**_. So I've used existing <a href=\"https://gym.openai.com/\">environment</a> and a <a href=\"https://gym.openai.com/envs/FrozenLake8x8-v0/\">Frozen Lake 8x8</a> in particular.\n\n**Basic Idea**:\nThe agent controls the movement of a character in a grid world. Some tiles of the grid are walkable, and others lead to the agent falling into the water. Additionally, the movement direction of the agent is uncertain and only partially depends on the chosen direction. The agent is rewarded for finding a walkable path to a goal tile.\n\n_Winter is here. You and your friends were tossing around a frisbee at the park when you made a wild throw that left the frisbee out in the middle of the lake. The water is mostly frozen, but there are a few holes where the ice has melted. If you step into one of those holes, you'll fall into the freezing water. At this time, there's an international frisbee shortage, so it's absolutely imperative that you navigate across the lake and retrieve the disc. However, the ice is slippery, so you won't always move in the direction you intend._\n\n**Grid is Described**:\n- S: starting point, safe\n- F: frozen surface, safe\n- H: hole, fall to your doom\n- G: goal, where the frisbee is located\n\n**Example**: <br>\nSFFF <br>\nFHFH <br>\nFFFH <br> \nHFFG <br>\n\n**Glossary**:\n- **environment** — It is like an object or interface through which we or our game bot(agent) can interact with the game and get details of current state and etc. There are several different games or environments available. You can find them here. \n- **step** - It’s a function through which we can do an action like what actually we want to do to at the current state/stage of the game.\n- **action** - It’s a value or object which we basically want to do at the current state/stage of the game. Like moving right or left or jump or etc.\n- **observation (object)** - An environment-specific object representing your observation of the environment. For example, pixel data from a camera, joint angles and joint velocities of a robot, or the board state in a board game.\n- **reward (float)** - Amount of reward achieved by the previous action. The scale varies between environments, but the goal is always to increase your total reward.\n- **done (boolean)** - whether it’s time to reset the environment again. Most (but not all) tasks are divided up into well-defined episodes, and done being True indicates the episode has terminated. (For example, perhaps the pole tipped too far, or you lost your last life.)\n- **info (dictionary)** - diagnostic information useful for debugging. It can sometimes be useful for learning (for example, it might contain the raw probabilities behind the environment’s last state change). However, official evaluations of your agent are not allowed to use this for learning.",
"_____no_output_____"
]
],
[
[
"'''\n If following import fails, just install gym from anaconda console, using:\n pip install gym\n'''\nimport gym\nimport numpy as np\nimport time, pickle, os",
"_____no_output_____"
],
[
"# Global constants\nepsilon = 0.9\ntotal_epoches = 10000\nmax_steps = 1000\n\nlr_rate = 0.81\ngamma = 0.96",
"_____no_output_____"
]
],
[
[
"### Q-Learning Approach <a class=\"anchor\" id=\"q-learn\"></a>",
"_____no_output_____"
],
[
"Before moving to DNN implementation, I've decided to use <a href=\"https://en.wikipedia.org/wiki/Q-learning\">Q-learning</a> algorithm.\nThe goal of Q-learning is to learn a policy, which tells an agent what action to take under what circumstances.",
"_____no_output_____"
],
[
"#### OpenAI Gym Stochastic FrozenLake Approach <a class=\"anchor\" id=\"stochastic-field\"></a>",
"_____no_output_____"
],
[
"In this context, **stochastic** means that upon action selection there still is 1/3 of a chance to end up on different tile, due to environment. You see - ice is slippery.",
"_____no_output_____"
]
],
[
[
"# Load OpenAI gym environment\nenv_stochastic = gym.make('FrozenLake-v0')\n\n# Define our Q-Learn matrix\nQ_stochastic = np.zeros((env_stochastic.observation_space.n, env_stochastic.action_space.n))\nfile_Q_stochastic = \"frozenLake_stochastic_qTable.pkl\"\n\n# Preview board\nenv_stochastic.render()",
"\n\u001b[41mS\u001b[0mFFF\nFHFH\nFFFH\nHFFG\n"
],
[
"# Defines all possible actions our agent can take. In our case it's a space of 4 actions: Left, Right, Up, Down and they are coded as [0,1,2,3].\ndisplay(env_stochastic.action_space)\n\n# Defines observation space for our agent. In our case it's a descrete 4x4 board.\ndisplay(env_stochastic.observation_space)\n\n# Defines reward range. In our case o if failed and 1 if succeded\ndisplay(env_stochastic.reward_range)",
"_____no_output_____"
],
[
"# Used to dump Q-Matrices to files\ndef SaveQTableToFile(Q, name):\n with open(name, 'wb') as f:\n pickle.dump(Q, f)",
"_____no_output_____"
],
[
"# \"Epsilon Greedy\" action selection\ndef choose_action(state, environment, Q):\n action=0\n if np.random.uniform(0, 1) < epsilon:\n action = environment.action_space.sample()\n else:\n action = np.argmax(Q[state, :])\n return action\n\n# Learning function\ndef learn(state, state2, reward, action, Q):\n predict = Q[state, action]\n target = reward + gamma * np.max(Q[state2, :])\n Q[state, action] = Q[state, action] + lr_rate * (target - predict)\n\n# Train function\ndef initQLearn(epoches, environment, Q, showOutput=False):\n for episode in range(epoches):\n state = environment.reset()\n t = 0\n while t < max_steps:\n if showOutput == True:\n environment.render()\n\n action = choose_action(state, environment, Q) \n state2, reward, done, info = environment.step(action)\n if showOutput == True:\n print(\"Reward:\", reward)\n print(\"Info:\", info)\n \n learn(state, state2, reward, action, Q)\n state = state2\n t += 1\n \n if done == True and reward == 1:\n print('Episode', episode, 'was successful. Agent has reached the Exit.')\n break\n\n if showOutput == True:\n time.sleep(0.1)\n \n print('Our Q is equal:\\n', Q)",
"_____no_output_____"
],
[
"# Train Q-Learn model on stochastic environment\ninitQLearn(total_epoches, env_stochastic, Q_stochastic, False)",
"Episode 72 was successful. Agent has reached the Exit.\nEpisode 105 was successful. Agent has reached the Exit.\nEpisode 118 was successful. Agent has reached the Exit.\nEpisode 148 was successful. Agent has reached the Exit.\nEpisode 191 was successful. Agent has reached the Exit.\nEpisode 224 was successful. Agent has reached the Exit.\nEpisode 333 was successful. Agent has reached the Exit.\nEpisode 355 was successful. Agent has reached the Exit.\nEpisode 371 was successful. Agent has reached the Exit.\nEpisode 479 was successful. Agent has reached the Exit.\nEpisode 673 was successful. Agent has reached the Exit.\nEpisode 752 was successful. Agent has reached the Exit.\nEpisode 867 was successful. Agent has reached the Exit.\nEpisode 869 was successful. Agent has reached the Exit.\nEpisode 898 was successful. Agent has reached the Exit.\nEpisode 1058 was successful. Agent has reached the Exit.\nEpisode 1115 was successful. Agent has reached the Exit.\nEpisode 1145 was successful. Agent has reached the Exit.\nEpisode 1161 was successful. Agent has reached the Exit.\nEpisode 1168 was successful. Agent has reached the Exit.\nEpisode 1273 was successful. Agent has reached the Exit.\nEpisode 1318 was successful. Agent has reached the Exit.\nEpisode 1373 was successful. Agent has reached the Exit.\nEpisode 1387 was successful. Agent has reached the Exit.\nEpisode 1431 was successful. Agent has reached the Exit.\nEpisode 1432 was successful. Agent has reached the Exit.\nEpisode 1503 was successful. Agent has reached the Exit.\nEpisode 1538 was successful. Agent has reached the Exit.\nEpisode 1561 was successful. Agent has reached the Exit.\nEpisode 1578 was successful. Agent has reached the Exit.\nEpisode 1587 was successful. Agent has reached the Exit.\nEpisode 1624 was successful. Agent has reached the Exit.\nEpisode 1641 was successful. Agent has reached the Exit.\nEpisode 1709 was successful. Agent has reached the Exit.\nEpisode 1796 was successful. Agent has reached the Exit.\nEpisode 1807 was successful. Agent has reached the Exit.\nEpisode 1876 was successful. Agent has reached the Exit.\nEpisode 1918 was successful. Agent has reached the Exit.\nEpisode 1956 was successful. Agent has reached the Exit.\nEpisode 1980 was successful. Agent has reached the Exit.\nEpisode 2010 was successful. Agent has reached the Exit.\nEpisode 2451 was successful. Agent has reached the Exit.\nEpisode 2456 was successful. Agent has reached the Exit.\nEpisode 2612 was successful. Agent has reached the Exit.\nEpisode 2619 was successful. Agent has reached the Exit.\nEpisode 2632 was successful. Agent has reached the Exit.\nEpisode 2677 was successful. Agent has reached the Exit.\nEpisode 2685 was successful. Agent has reached the Exit.\nEpisode 2713 was successful. Agent has reached the Exit.\nEpisode 2794 was successful. Agent has reached the Exit.\nEpisode 2815 was successful. Agent has reached the Exit.\nEpisode 2875 was successful. Agent has reached the Exit.\nEpisode 2903 was successful. Agent has reached the Exit.\nEpisode 2912 was successful. Agent has reached the Exit.\nEpisode 2971 was successful. Agent has reached the Exit.\nEpisode 3033 was successful. Agent has reached the Exit.\nEpisode 3246 was successful. Agent has reached the Exit.\nEpisode 3249 was successful. Agent has reached the Exit.\nEpisode 3628 was successful. Agent has reached the Exit.\nEpisode 3667 was successful. Agent has reached the Exit.\nEpisode 3830 was successful. Agent has reached the Exit.\nEpisode 3858 was successful. Agent has reached the Exit.\nEpisode 3867 was successful. Agent has reached the Exit.\nEpisode 3930 was successful. Agent has reached the Exit.\nEpisode 3958 was successful. Agent has reached the Exit.\nEpisode 3970 was successful. Agent has reached the Exit.\nEpisode 3995 was successful. Agent has reached the Exit.\nEpisode 4066 was successful. Agent has reached the Exit.\nEpisode 4075 was successful. Agent has reached the Exit.\nEpisode 4125 was successful. Agent has reached the Exit.\nEpisode 4223 was successful. Agent has reached the Exit.\nEpisode 4256 was successful. Agent has reached the Exit.\nEpisode 4416 was successful. Agent has reached the Exit.\nEpisode 4450 was successful. Agent has reached the Exit.\nEpisode 4451 was successful. Agent has reached the Exit.\nEpisode 4495 was successful. Agent has reached the Exit.\nEpisode 4519 was successful. Agent has reached the Exit.\nEpisode 4521 was successful. Agent has reached the Exit.\nEpisode 4561 was successful. Agent has reached the Exit.\nEpisode 4569 was successful. Agent has reached the Exit.\nEpisode 4583 was successful. Agent has reached the Exit.\nEpisode 4608 was successful. Agent has reached the Exit.\nEpisode 4639 was successful. Agent has reached the Exit.\nEpisode 4691 was successful. Agent has reached the Exit.\nEpisode 4723 was successful. Agent has reached the Exit.\nEpisode 4733 was successful. Agent has reached the Exit.\nEpisode 4883 was successful. Agent has reached the Exit.\nEpisode 4940 was successful. Agent has reached the Exit.\nEpisode 5094 was successful. Agent has reached the Exit.\nEpisode 5121 was successful. Agent has reached the Exit.\nEpisode 5210 was successful. Agent has reached the Exit.\nEpisode 5211 was successful. Agent has reached the Exit.\nEpisode 5249 was successful. Agent has reached the Exit.\nEpisode 5268 was successful. Agent has reached the Exit.\nEpisode 5346 was successful. Agent has reached the Exit.\nEpisode 5355 was successful. Agent has reached the Exit.\nEpisode 5423 was successful. Agent has reached the Exit.\nEpisode 5492 was successful. Agent has reached the Exit.\nEpisode 5628 was successful. Agent has reached the Exit.\nEpisode 5713 was successful. Agent has reached the Exit.\nEpisode 5738 was successful. Agent has reached the Exit.\nEpisode 5766 was successful. Agent has reached the Exit.\nEpisode 5824 was successful. Agent has reached the Exit.\nEpisode 5930 was successful. Agent has reached the Exit.\nEpisode 5940 was successful. Agent has reached the Exit.\nEpisode 5950 was successful. Agent has reached the Exit.\nEpisode 6002 was successful. Agent has reached the Exit.\nEpisode 6087 was successful. Agent has reached the Exit.\nEpisode 6090 was successful. Agent has reached the Exit.\nEpisode 6152 was successful. Agent has reached the Exit.\nEpisode 6155 was successful. Agent has reached the Exit.\nEpisode 6338 was successful. Agent has reached the Exit.\nEpisode 6383 was successful. Agent has reached the Exit.\nEpisode 6511 was successful. Agent has reached the Exit.\nEpisode 6542 was successful. Agent has reached the Exit.\nEpisode 6593 was successful. Agent has reached the Exit.\nEpisode 6594 was successful. Agent has reached the Exit.\nEpisode 6597 was successful. Agent has reached the Exit.\nEpisode 6598 was successful. Agent has reached the Exit.\nEpisode 6616 was successful. Agent has reached the Exit.\nEpisode 6631 was successful. Agent has reached the Exit.\nEpisode 6643 was successful. Agent has reached the Exit.\nEpisode 6731 was successful. Agent has reached the Exit.\nEpisode 6779 was successful. Agent has reached the Exit.\nEpisode 6943 was successful. Agent has reached the Exit.\nEpisode 6998 was successful. Agent has reached the Exit.\nEpisode 7039 was successful. Agent has reached the Exit.\nEpisode 7041 was successful. Agent has reached the Exit.\nEpisode 7045 was successful. Agent has reached the Exit.\nEpisode 7203 was successful. Agent has reached the Exit.\nEpisode 7212 was successful. Agent has reached the Exit.\nEpisode 7220 was successful. Agent has reached the Exit.\nEpisode 7296 was successful. Agent has reached the Exit.\nEpisode 7364 was successful. Agent has reached the Exit.\nEpisode 7372 was successful. Agent has reached the Exit.\nEpisode 7449 was successful. Agent has reached the Exit.\nEpisode 7533 was successful. Agent has reached the Exit.\nEpisode 7603 was successful. Agent has reached the Exit.\nEpisode 7677 was successful. Agent has reached the Exit.\nEpisode 7684 was successful. Agent has reached the Exit.\nEpisode 7688 was successful. Agent has reached the Exit.\nEpisode 7849 was successful. Agent has reached the Exit.\nEpisode 7850 was successful. Agent has reached the Exit.\nEpisode 7911 was successful. Agent has reached the Exit.\nEpisode 7945 was successful. Agent has reached the Exit.\nEpisode 7975 was successful. Agent has reached the Exit.\nEpisode 7985 was successful. Agent has reached the Exit.\nEpisode 8032 was successful. Agent has reached the Exit.\nEpisode 8076 was successful. Agent has reached the Exit.\nEpisode 8107 was successful. Agent has reached the Exit.\nEpisode 8117 was successful. Agent has reached the Exit.\nEpisode 8154 was successful. Agent has reached the Exit.\nEpisode 8287 was successful. Agent has reached the Exit.\nEpisode 8388 was successful. Agent has reached the Exit.\nEpisode 8476 was successful. Agent has reached the Exit.\nEpisode 8549 was successful. Agent has reached the Exit.\nEpisode 8572 was successful. Agent has reached the Exit.\nEpisode 8641 was successful. Agent has reached the Exit.\nEpisode 8653 was successful. Agent has reached the Exit.\nEpisode 8675 was successful. Agent has reached the Exit.\nEpisode 8720 was successful. Agent has reached the Exit.\nEpisode 8731 was successful. Agent has reached the Exit.\nEpisode 8805 was successful. Agent has reached the Exit.\nEpisode 9005 was successful. Agent has reached the Exit.\nEpisode 9031 was successful. Agent has reached the Exit.\nEpisode 9055 was successful. Agent has reached the Exit.\nEpisode 9143 was successful. Agent has reached the Exit.\nEpisode 9166 was successful. Agent has reached the Exit.\nEpisode 9230 was successful. Agent has reached the Exit.\nEpisode 9328 was successful. Agent has reached the Exit.\nEpisode 9337 was successful. Agent has reached the Exit.\nEpisode 9437 was successful. Agent has reached the Exit.\nEpisode 9503 was successful. Agent has reached the Exit.\nEpisode 9566 was successful. Agent has reached the Exit.\nEpisode 9568 was successful. Agent has reached the Exit.\nEpisode 9656 was successful. Agent has reached the Exit.\nEpisode 9746 was successful. Agent has reached the Exit.\nEpisode 9788 was successful. Agent has reached the Exit.\nEpisode 9792 was successful. Agent has reached the Exit.\nEpisode 9893 was successful. Agent has reached the Exit.\nEpisode 9950 was successful. Agent has reached the Exit.\nEpisode 9978 was successful. Agent has reached the Exit.\nOur Q is equal:\n [[0.63155759 0.7260818 0.63098495 0.64096586]\n [0.08923413 0.59320009 0.4857933 0.5864178 ]\n [0.58253307 0.54801955 0.74002867 0.5670273 ]\n [0.09847175 0.43629738 0.52282304 0.5225726 ]\n [0.69112814 0.72705092 0.76905186 0.12600166]\n [0. 0. 0. 0. ]\n [0.59500203 0.00506861 0.15284422 0.46626663]\n [0. 0. 0. 0. ]\n [0.73769729 0.12177414 0.02240348 0.75699989]\n [0.02665465 0.75511081 0.84217728 0.79496329]\n [0.88576406 0.8295706 0.79526765 0.79497617]\n [0. 0. 0. 0. ]\n [0. 0. 0. 0. ]\n [0.15828885 0.02537745 0.92143326 0.16265478]\n [0.83612259 0.99437935 0.90915363 0.94866158]\n [0. 0. 0. 0. ]]\n"
],
[
"# Save our Q-Matrix to file\nSaveQTableToFile(Q_stochastic, file_Q_stochastic)",
"_____no_output_____"
]
],
[
[
"Below function does not depend on any additional learning, so it can be freely changed.",
"_____no_output_____"
]
],
[
[
"# This function will select Move (Action) based on State and all previous Experience saved in model.\ndef choose_QModel_action(state, verbose, Q):\n action = np.argmax(Q[state, :])\n if verbose == True:\n print (action)\n return action\n\ndef initPlayByQModel(episodes_count, environment, file_QTable, showOutput=True, verbose=False):\n with open(file_QTable, 'rb') as f:\n _Q = pickle.load(f)\n\n for episode in range(episodes_count):\n state = environment.reset()\n \n print(\"*** Starting Episode: \", episode)\n t = 0\n \n while t < max_steps:\n if showOutput == True:\n environment.render()\n\n action = choose_QModel_action(state, verbose, _Q)\n state2, reward, done, info = environment.step(action)\n \n if verbose == True:\n print(\"Reward:\", reward)\n print(\"Info:\", info)\n print(\"State2:\", state2)\n \n state = state2\n t += 1\n\n if done == True and reward == 1:\n print('Success: Agent passed the Lake!')\n break\n \n if done == True and reward == 0:\n print('Agent died in vain!')\n break\n \n os.system('clear')",
"_____no_output_____"
],
[
"# Due to environment running it few times, may and will produce different outcomes.\ninitPlayByQModel(10, env_stochastic, file_Q_stochastic, False, False)",
"*** Starting Episode: 0\nSuccess: Agent passed the Lake!\n*** Starting Episode: 1\nSuccess: Agent passed the Lake!\n*** Starting Episode: 2\nAgent died in vain!\n*** Starting Episode: 3\nAgent died in vain!\n*** Starting Episode: 4\nSuccess: Agent passed the Lake!\n*** Starting Episode: 5\nAgent died in vain!\n*** Starting Episode: 6\nAgent died in vain!\n*** Starting Episode: 7\nAgent died in vain!\n*** Starting Episode: 8\nAgent died in vain!\n*** Starting Episode: 9\nAgent died in vain!\n"
]
],
[
[
"#### Personal Deterministic FrozenLake Approach <a class=\"anchor\" id=\"deterministic-field\"></a>",
"_____no_output_____"
],
[
"So, as you can see stochastic environment ain't that good for Q-Learning (Let's run few more times here, just to show how bad it is). Let's make environment **deterministic**!\nFor any references about arguments or environment we can look directly in OpenAI FrozenLake <a href=\"https://github.com/openai/gym/blob/master/gym/envs/toy_text/frozen_lake.py\">implementation</a>.",
"_____no_output_____"
]
],
[
[
"# First, we need to register our new environment we going to work with\nfrom gym.envs.registration import register\n\nregister(id='Deterministic-FrozenLake4x4-v0',\n entry_point='gym.envs.toy_text.frozen_lake:FrozenLakeEnv',\n kwargs={'map_name': '4x4', 'is_slippery': False}\n)",
"_____no_output_____"
],
[
"# Create new environment instance to work with\nenv_deterministic = gym.make('Deterministic-FrozenLake4x4-v0')\n\n# Define our Q-Learn matrix\nQ_deterministic = np.zeros((env_deterministic.observation_space.n, env_deterministic.action_space.n))\nfile_Q_deterministic = \"frozenLake_deterministic_qTable.pkl\"\n\n# Preview board\nenv_deterministic.render()",
"\n\u001b[41mS\u001b[0mFFF\nFHFH\nFFFH\nHFFG\n"
],
[
"# Same learning function but for different environment\ninitQLearn(total_epoches, env_deterministic, Q_deterministic, False)",
"Episode 19 was successful. Agent has reached the Exit.\nEpisode 39 was successful. Agent has reached the Exit.\nEpisode 52 was successful. Agent has reached the Exit.\nEpisode 66 was successful. Agent has reached the Exit.\nEpisode 98 was successful. Agent has reached the Exit.\nEpisode 124 was successful. Agent has reached the Exit.\nEpisode 199 was successful. Agent has reached the Exit.\nEpisode 263 was successful. Agent has reached the Exit.\nEpisode 295 was successful. Agent has reached the Exit.\nEpisode 331 was successful. Agent has reached the Exit.\nEpisode 394 was successful. Agent has reached the Exit.\nEpisode 406 was successful. Agent has reached the Exit.\nEpisode 412 was successful. Agent has reached the Exit.\nEpisode 434 was successful. Agent has reached the Exit.\nEpisode 498 was successful. Agent has reached the Exit.\nEpisode 500 was successful. Agent has reached the Exit.\nEpisode 513 was successful. Agent has reached the Exit.\nEpisode 519 was successful. Agent has reached the Exit.\nEpisode 543 was successful. Agent has reached the Exit.\nEpisode 577 was successful. Agent has reached the Exit.\nEpisode 583 was successful. Agent has reached the Exit.\nEpisode 605 was successful. Agent has reached the Exit.\nEpisode 615 was successful. Agent has reached the Exit.\nEpisode 624 was successful. Agent has reached the Exit.\nEpisode 647 was successful. Agent has reached the Exit.\nEpisode 671 was successful. Agent has reached the Exit.\nEpisode 672 was successful. Agent has reached the Exit.\nEpisode 687 was successful. Agent has reached the Exit.\nEpisode 697 was successful. Agent has reached the Exit.\nEpisode 724 was successful. Agent has reached the Exit.\nEpisode 752 was successful. Agent has reached the Exit.\nEpisode 761 was successful. Agent has reached the Exit.\nEpisode 765 was successful. Agent has reached the Exit.\nEpisode 794 was successful. Agent has reached the Exit.\nEpisode 830 was successful. Agent has reached the Exit.\nEpisode 866 was successful. Agent has reached the Exit.\nEpisode 898 was successful. Agent has reached the Exit.\nEpisode 912 was successful. Agent has reached the Exit.\nEpisode 930 was successful. Agent has reached the Exit.\nEpisode 1078 was successful. Agent has reached the Exit.\nEpisode 1107 was successful. Agent has reached the Exit.\nEpisode 1110 was successful. Agent has reached the Exit.\nEpisode 1112 was successful. Agent has reached the Exit.\nEpisode 1206 was successful. Agent has reached the Exit.\nEpisode 1212 was successful. Agent has reached the Exit.\nEpisode 1216 was successful. Agent has reached the Exit.\nEpisode 1248 was successful. Agent has reached the Exit.\nEpisode 1346 was successful. Agent has reached the Exit.\nEpisode 1393 was successful. Agent has reached the Exit.\nEpisode 1412 was successful. Agent has reached the Exit.\nEpisode 1430 was successful. Agent has reached the Exit.\nEpisode 1452 was successful. Agent has reached the Exit.\nEpisode 1470 was successful. Agent has reached the Exit.\nEpisode 1489 was successful. Agent has reached the Exit.\nEpisode 1506 was successful. Agent has reached the Exit.\nEpisode 1512 was successful. Agent has reached the Exit.\nEpisode 1545 was successful. Agent has reached the Exit.\nEpisode 1562 was successful. Agent has reached the Exit.\nEpisode 1576 was successful. Agent has reached the Exit.\nEpisode 1610 was successful. Agent has reached the Exit.\nEpisode 1632 was successful. Agent has reached the Exit.\nEpisode 1658 was successful. Agent has reached the Exit.\nEpisode 1709 was successful. Agent has reached the Exit.\nEpisode 1719 was successful. Agent has reached the Exit.\nEpisode 1724 was successful. Agent has reached the Exit.\nEpisode 1734 was successful. Agent has reached the Exit.\nEpisode 1752 was successful. Agent has reached the Exit.\nEpisode 1766 was successful. Agent has reached the Exit.\nEpisode 1804 was successful. Agent has reached the Exit.\nEpisode 1860 was successful. Agent has reached the Exit.\nEpisode 1892 was successful. Agent has reached the Exit.\nEpisode 1896 was successful. Agent has reached the Exit.\nEpisode 1957 was successful. Agent has reached the Exit.\nEpisode 1980 was successful. Agent has reached the Exit.\nEpisode 1985 was successful. Agent has reached the Exit.\nEpisode 1990 was successful. Agent has reached the Exit.\nEpisode 1993 was successful. Agent has reached the Exit.\nEpisode 2065 was successful. Agent has reached the Exit.\nEpisode 2132 was successful. Agent has reached the Exit.\nEpisode 2151 was successful. Agent has reached the Exit.\nEpisode 2163 was successful. Agent has reached the Exit.\nEpisode 2169 was successful. Agent has reached the Exit.\nEpisode 2184 was successful. Agent has reached the Exit.\nEpisode 2187 was successful. Agent has reached the Exit.\nEpisode 2192 was successful. Agent has reached the Exit.\nEpisode 2255 was successful. Agent has reached the Exit.\nEpisode 2274 was successful. Agent has reached the Exit.\nEpisode 2287 was successful. Agent has reached the Exit.\nEpisode 2289 was successful. Agent has reached the Exit.\nEpisode 2328 was successful. Agent has reached the Exit.\nEpisode 2350 was successful. Agent has reached the Exit.\nEpisode 2363 was successful. Agent has reached the Exit.\nEpisode 2375 was successful. Agent has reached the Exit.\nEpisode 2442 was successful. Agent has reached the Exit.\nEpisode 2443 was successful. Agent has reached the Exit.\nEpisode 2454 was successful. Agent has reached the Exit.\nEpisode 2506 was successful. Agent has reached the Exit.\nEpisode 2536 was successful. Agent has reached the Exit.\nEpisode 2551 was successful. Agent has reached the Exit.\nEpisode 2568 was successful. Agent has reached the Exit.\nEpisode 2570 was successful. Agent has reached the Exit.\nEpisode 2573 was successful. Agent has reached the Exit.\nEpisode 2593 was successful. Agent has reached the Exit.\nEpisode 2612 was successful. Agent has reached the Exit.\nEpisode 2631 was successful. Agent has reached the Exit.\nEpisode 2639 was successful. Agent has reached the Exit.\nEpisode 2666 was successful. Agent has reached the Exit.\nEpisode 2670 was successful. Agent has reached the Exit.\nEpisode 2702 was successful. Agent has reached the Exit.\nEpisode 2715 was successful. Agent has reached the Exit.\nEpisode 2726 was successful. Agent has reached the Exit.\nEpisode 2738 was successful. Agent has reached the Exit.\nEpisode 2768 was successful. Agent has reached the Exit.\nEpisode 2784 was successful. Agent has reached the Exit.\nEpisode 2808 was successful. Agent has reached the Exit.\nEpisode 2844 was successful. Agent has reached the Exit.\nEpisode 2886 was successful. Agent has reached the Exit.\nEpisode 2905 was successful. Agent has reached the Exit.\nEpisode 2925 was successful. Agent has reached the Exit.\nEpisode 2951 was successful. Agent has reached the Exit.\nEpisode 2971 was successful. Agent has reached the Exit.\nEpisode 2990 was successful. Agent has reached the Exit.\nEpisode 2996 was successful. Agent has reached the Exit.\nEpisode 2999 was successful. Agent has reached the Exit.\nEpisode 3065 was successful. Agent has reached the Exit.\nEpisode 3084 was successful. Agent has reached the Exit.\nEpisode 3101 was successful. Agent has reached the Exit.\nEpisode 3135 was successful. Agent has reached the Exit.\nEpisode 3180 was successful. Agent has reached the Exit.\nEpisode 3183 was successful. Agent has reached the Exit.\nEpisode 3200 was successful. Agent has reached the Exit.\nEpisode 3204 was successful. Agent has reached the Exit.\nEpisode 3205 was successful. Agent has reached the Exit.\nEpisode 3248 was successful. Agent has reached the Exit.\nEpisode 3303 was successful. Agent has reached the Exit.\nEpisode 3332 was successful. Agent has reached the Exit.\nEpisode 3339 was successful. Agent has reached the Exit.\nEpisode 3368 was successful. Agent has reached the Exit.\nEpisode 3390 was successful. Agent has reached the Exit.\nEpisode 3425 was successful. Agent has reached the Exit.\nEpisode 3471 was successful. Agent has reached the Exit.\nEpisode 3480 was successful. Agent has reached the Exit.\nEpisode 3492 was successful. Agent has reached the Exit.\nEpisode 3504 was successful. Agent has reached the Exit.\nEpisode 3507 was successful. Agent has reached the Exit.\nEpisode 3520 was successful. Agent has reached the Exit.\nEpisode 3534 was successful. Agent has reached the Exit.\nEpisode 3543 was successful. Agent has reached the Exit.\nEpisode 3551 was successful. Agent has reached the Exit.\nEpisode 3643 was successful. Agent has reached the Exit.\nEpisode 3651 was successful. Agent has reached the Exit.\nEpisode 3658 was successful. Agent has reached the Exit.\nEpisode 3669 was successful. Agent has reached the Exit.\nEpisode 3670 was successful. Agent has reached the Exit.\nEpisode 3684 was successful. Agent has reached the Exit.\nEpisode 3688 was successful. Agent has reached the Exit.\nEpisode 3692 was successful. Agent has reached the Exit.\nEpisode 3705 was successful. Agent has reached the Exit.\nEpisode 3707 was successful. Agent has reached the Exit.\nEpisode 3743 was successful. Agent has reached the Exit.\nEpisode 3806 was successful. Agent has reached the Exit.\nEpisode 3838 was successful. Agent has reached the Exit.\nEpisode 3884 was successful. Agent has reached the Exit.\nEpisode 3889 was successful. Agent has reached the Exit.\nEpisode 3893 was successful. Agent has reached the Exit.\nEpisode 3986 was successful. Agent has reached the Exit.\nEpisode 4056 was successful. Agent has reached the Exit.\nEpisode 4103 was successful. Agent has reached the Exit.\nEpisode 4163 was successful. Agent has reached the Exit.\nEpisode 4166 was successful. Agent has reached the Exit.\nEpisode 4181 was successful. Agent has reached the Exit.\nEpisode 4197 was successful. Agent has reached the Exit.\nEpisode 4208 was successful. Agent has reached the Exit.\nEpisode 4248 was successful. Agent has reached the Exit.\nEpisode 4262 was successful. Agent has reached the Exit.\nEpisode 4270 was successful. Agent has reached the Exit.\nEpisode 4280 was successful. Agent has reached the Exit.\nEpisode 4291 was successful. Agent has reached the Exit.\nEpisode 4297 was successful. Agent has reached the Exit.\nEpisode 4355 was successful. Agent has reached the Exit.\nEpisode 4415 was successful. Agent has reached the Exit.\nEpisode 4431 was successful. Agent has reached the Exit.\nEpisode 4441 was successful. Agent has reached the Exit.\nEpisode 4456 was successful. Agent has reached the Exit.\nEpisode 4477 was successful. Agent has reached the Exit.\nEpisode 4489 was successful. Agent has reached the Exit.\nEpisode 4528 was successful. Agent has reached the Exit.\nEpisode 4563 was successful. Agent has reached the Exit.\nEpisode 4566 was successful. Agent has reached the Exit.\nEpisode 4570 was successful. Agent has reached the Exit.\nEpisode 4573 was successful. Agent has reached the Exit.\nEpisode 4596 was successful. Agent has reached the Exit.\nEpisode 4601 was successful. Agent has reached the Exit.\nEpisode 4605 was successful. Agent has reached the Exit.\nEpisode 4625 was successful. Agent has reached the Exit.\nEpisode 4703 was successful. Agent has reached the Exit.\nEpisode 4710 was successful. Agent has reached the Exit.\nEpisode 4717 was successful. Agent has reached the Exit.\nEpisode 4796 was successful. Agent has reached the Exit.\nEpisode 4798 was successful. Agent has reached the Exit.\nEpisode 4803 was successful. Agent has reached the Exit.\nEpisode 4827 was successful. Agent has reached the Exit.\nEpisode 4829 was successful. Agent has reached the Exit.\nEpisode 4839 was successful. Agent has reached the Exit.\nEpisode 4890 was successful. Agent has reached the Exit.\nEpisode 4893 was successful. Agent has reached the Exit.\nEpisode 4931 was successful. Agent has reached the Exit.\nEpisode 4977 was successful. Agent has reached the Exit.\nEpisode 4991 was successful. Agent has reached the Exit.\nEpisode 5090 was successful. Agent has reached the Exit.\nEpisode 5108 was successful. Agent has reached the Exit.\nEpisode 5117 was successful. Agent has reached the Exit.\nEpisode 5222 was successful. Agent has reached the Exit.\nEpisode 5228 was successful. Agent has reached the Exit.\nEpisode 5240 was successful. Agent has reached the Exit.\nEpisode 5251 was successful. Agent has reached the Exit.\nEpisode 5254 was successful. Agent has reached the Exit.\nEpisode 5270 was successful. Agent has reached the Exit.\nEpisode 5271 was successful. Agent has reached the Exit.\nEpisode 5312 was successful. Agent has reached the Exit.\nEpisode 5313 was successful. Agent has reached the Exit.\nEpisode 5320 was successful. Agent has reached the Exit.\nEpisode 5344 was successful. Agent has reached the Exit.\nEpisode 5355 was successful. Agent has reached the Exit.\nEpisode 5370 was successful. Agent has reached the Exit.\nEpisode 5371 was successful. Agent has reached the Exit.\nEpisode 5378 was successful. Agent has reached the Exit.\nEpisode 5388 was successful. Agent has reached the Exit.\nEpisode 5519 was successful. Agent has reached the Exit.\nEpisode 5625 was successful. Agent has reached the Exit.\nEpisode 5684 was successful. Agent has reached the Exit.\nEpisode 5715 was successful. Agent has reached the Exit.\nEpisode 5753 was successful. Agent has reached the Exit.\nEpisode 5768 was successful. Agent has reached the Exit.\nEpisode 5787 was successful. Agent has reached the Exit.\nEpisode 5815 was successful. Agent has reached the Exit.\nEpisode 5840 was successful. Agent has reached the Exit.\nEpisode 5845 was successful. Agent has reached the Exit.\nEpisode 5853 was successful. Agent has reached the Exit.\nEpisode 5872 was successful. Agent has reached the Exit.\nEpisode 5882 was successful. Agent has reached the Exit.\nEpisode 5928 was successful. Agent has reached the Exit.\nEpisode 5932 was successful. Agent has reached the Exit.\nEpisode 5950 was successful. Agent has reached the Exit.\nEpisode 5983 was successful. Agent has reached the Exit.\nEpisode 6000 was successful. Agent has reached the Exit.\nEpisode 6022 was successful. Agent has reached the Exit.\nEpisode 6025 was successful. Agent has reached the Exit.\nEpisode 6032 was successful. Agent has reached the Exit.\nEpisode 6048 was successful. Agent has reached the Exit.\nEpisode 6075 was successful. Agent has reached the Exit.\nEpisode 6084 was successful. Agent has reached the Exit.\nEpisode 6100 was successful. Agent has reached the Exit.\nEpisode 6109 was successful. Agent has reached the Exit.\nEpisode 6130 was successful. Agent has reached the Exit.\nEpisode 6132 was successful. Agent has reached the Exit.\nEpisode 6133 was successful. Agent has reached the Exit.\nEpisode 6134 was successful. Agent has reached the Exit.\nEpisode 6152 was successful. Agent has reached the Exit.\nEpisode 6154 was successful. Agent has reached the Exit.\nEpisode 6157 was successful. Agent has reached the Exit.\nEpisode 6180 was successful. Agent has reached the Exit.\nEpisode 6219 was successful. Agent has reached the Exit.\nEpisode 6242 was successful. Agent has reached the Exit.\nEpisode 6302 was successful. Agent has reached the Exit.\nEpisode 6315 was successful. Agent has reached the Exit.\nEpisode 6331 was successful. Agent has reached the Exit.\nEpisode 6333 was successful. Agent has reached the Exit.\nEpisode 6343 was successful. Agent has reached the Exit.\nEpisode 6386 was successful. Agent has reached the Exit.\nEpisode 6414 was successful. Agent has reached the Exit.\nEpisode 6416 was successful. Agent has reached the Exit.\nEpisode 6424 was successful. Agent has reached the Exit.\nEpisode 6426 was successful. Agent has reached the Exit.\nEpisode 6472 was successful. Agent has reached the Exit.\nEpisode 6483 was successful. Agent has reached the Exit.\nEpisode 6495 was successful. Agent has reached the Exit.\nEpisode 6500 was successful. Agent has reached the Exit.\nEpisode 6530 was successful. Agent has reached the Exit.\nEpisode 6536 was successful. Agent has reached the Exit.\nEpisode 6572 was successful. Agent has reached the Exit.\nEpisode 6588 was successful. Agent has reached the Exit.\nEpisode 6599 was successful. Agent has reached the Exit.\nEpisode 6606 was successful. Agent has reached the Exit.\nEpisode 6638 was successful. Agent has reached the Exit.\nEpisode 6667 was successful. Agent has reached the Exit.\nEpisode 6726 was successful. Agent has reached the Exit.\nEpisode 6786 was successful. Agent has reached the Exit.\nEpisode 6788 was successful. Agent has reached the Exit.\nEpisode 6792 was successful. Agent has reached the Exit.\nEpisode 6802 was successful. Agent has reached the Exit.\nEpisode 6803 was successful. Agent has reached the Exit.\nEpisode 6813 was successful. Agent has reached the Exit.\nEpisode 6821 was successful. Agent has reached the Exit.\nEpisode 6826 was successful. Agent has reached the Exit.\nEpisode 6844 was successful. Agent has reached the Exit.\nEpisode 6853 was successful. Agent has reached the Exit.\nEpisode 6873 was successful. Agent has reached the Exit.\nEpisode 6900 was successful. Agent has reached the Exit.\nEpisode 6921 was successful. Agent has reached the Exit.\nEpisode 6923 was successful. Agent has reached the Exit.\nEpisode 6948 was successful. Agent has reached the Exit.\nEpisode 6951 was successful. Agent has reached the Exit.\nEpisode 6952 was successful. Agent has reached the Exit.\nEpisode 6988 was successful. Agent has reached the Exit.\nEpisode 7017 was successful. Agent has reached the Exit.\nEpisode 7035 was successful. Agent has reached the Exit.\nEpisode 7048 was successful. Agent has reached the Exit.\nEpisode 7053 was successful. Agent has reached the Exit.\nEpisode 7102 was successful. Agent has reached the Exit.\nEpisode 7130 was successful. Agent has reached the Exit.\nEpisode 7164 was successful. Agent has reached the Exit.\nEpisode 7169 was successful. Agent has reached the Exit.\nEpisode 7171 was successful. Agent has reached the Exit.\nEpisode 7217 was successful. Agent has reached the Exit.\nEpisode 7254 was successful. Agent has reached the Exit.\nEpisode 7305 was successful. Agent has reached the Exit.\nEpisode 7314 was successful. Agent has reached the Exit.\nEpisode 7383 was successful. Agent has reached the Exit.\nEpisode 7384 was successful. Agent has reached the Exit.\nEpisode 7385 was successful. Agent has reached the Exit.\nEpisode 7388 was successful. Agent has reached the Exit.\nEpisode 7403 was successful. Agent has reached the Exit.\nEpisode 7408 was successful. Agent has reached the Exit.\nEpisode 7417 was successful. Agent has reached the Exit.\nEpisode 7423 was successful. Agent has reached the Exit.\nEpisode 7424 was successful. Agent has reached the Exit.\nEpisode 7449 was successful. Agent has reached the Exit.\nEpisode 7450 was successful. Agent has reached the Exit.\nEpisode 7470 was successful. Agent has reached the Exit.\nEpisode 7515 was successful. Agent has reached the Exit.\nEpisode 7543 was successful. Agent has reached the Exit.\nEpisode 7562 was successful. Agent has reached the Exit.\nEpisode 7599 was successful. Agent has reached the Exit.\nEpisode 7602 was successful. Agent has reached the Exit.\nEpisode 7604 was successful. Agent has reached the Exit.\nEpisode 7628 was successful. Agent has reached the Exit.\nEpisode 7635 was successful. Agent has reached the Exit.\nEpisode 7709 was successful. Agent has reached the Exit.\nEpisode 7719 was successful. Agent has reached the Exit.\nEpisode 7762 was successful. Agent has reached the Exit.\nEpisode 7767 was successful. Agent has reached the Exit.\nEpisode 7773 was successful. Agent has reached the Exit.\nEpisode 7781 was successful. Agent has reached the Exit.\nEpisode 7804 was successful. Agent has reached the Exit.\nEpisode 7831 was successful. Agent has reached the Exit.\nEpisode 7838 was successful. Agent has reached the Exit.\nEpisode 7886 was successful. Agent has reached the Exit.\nEpisode 7915 was successful. Agent has reached the Exit.\nEpisode 7918 was successful. Agent has reached the Exit.\nEpisode 7928 was successful. Agent has reached the Exit.\nEpisode 7942 was successful. Agent has reached the Exit.\nEpisode 7944 was successful. Agent has reached the Exit.\nEpisode 7974 was successful. Agent has reached the Exit.\nEpisode 8024 was successful. Agent has reached the Exit.\nEpisode 8072 was successful. Agent has reached the Exit.\nEpisode 8135 was successful. Agent has reached the Exit.\nEpisode 8140 was successful. Agent has reached the Exit.\nEpisode 8148 was successful. Agent has reached the Exit.\nEpisode 8177 was successful. Agent has reached the Exit.\nEpisode 8190 was successful. Agent has reached the Exit.\nEpisode 8196 was successful. Agent has reached the Exit.\nEpisode 8223 was successful. Agent has reached the Exit.\nEpisode 8236 was successful. Agent has reached the Exit.\nEpisode 8316 was successful. Agent has reached the Exit.\nEpisode 8329 was successful. Agent has reached the Exit.\nEpisode 8332 was successful. Agent has reached the Exit.\nEpisode 8369 was successful. Agent has reached the Exit.\nEpisode 8373 was successful. Agent has reached the Exit.\nEpisode 8406 was successful. Agent has reached the Exit.\nEpisode 8446 was successful. Agent has reached the Exit.\nEpisode 8448 was successful. Agent has reached the Exit.\nEpisode 8477 was successful. Agent has reached the Exit.\nEpisode 8526 was successful. Agent has reached the Exit.\nEpisode 8556 was successful. Agent has reached the Exit.\nEpisode 8587 was successful. Agent has reached the Exit.\nEpisode 8615 was successful. Agent has reached the Exit.\nEpisode 8677 was successful. Agent has reached the Exit.\nEpisode 8700 was successful. Agent has reached the Exit.\nEpisode 8701 was successful. Agent has reached the Exit.\nEpisode 8711 was successful. Agent has reached the Exit.\nEpisode 8737 was successful. Agent has reached the Exit.\nEpisode 8783 was successful. Agent has reached the Exit.\nEpisode 8801 was successful. Agent has reached the Exit.\nEpisode 8819 was successful. Agent has reached the Exit.\nEpisode 8829 was successful. Agent has reached the Exit.\nEpisode 8847 was successful. Agent has reached the Exit.\nEpisode 8863 was successful. Agent has reached the Exit.\nEpisode 8907 was successful. Agent has reached the Exit.\nEpisode 8917 was successful. Agent has reached the Exit.\nEpisode 8921 was successful. Agent has reached the Exit.\nEpisode 8954 was successful. Agent has reached the Exit.\nEpisode 8966 was successful. Agent has reached the Exit.\nEpisode 8971 was successful. Agent has reached the Exit.\nEpisode 9011 was successful. Agent has reached the Exit.\nEpisode 9077 was successful. Agent has reached the Exit.\nEpisode 9083 was successful. Agent has reached the Exit.\nEpisode 9085 was successful. Agent has reached the Exit.\nEpisode 9155 was successful. Agent has reached the Exit.\nEpisode 9207 was successful. Agent has reached the Exit.\nEpisode 9247 was successful. Agent has reached the Exit.\nEpisode 9258 was successful. Agent has reached the Exit.\nEpisode 9306 was successful. Agent has reached the Exit.\nEpisode 9323 was successful. Agent has reached the Exit.\nEpisode 9340 was successful. Agent has reached the Exit.\nEpisode 9355 was successful. Agent has reached the Exit.\nEpisode 9357 was successful. Agent has reached the Exit.\nEpisode 9421 was successful. Agent has reached the Exit.\nEpisode 9448 was successful. Agent has reached the Exit.\nEpisode 9484 was successful. Agent has reached the Exit.\nEpisode 9501 was successful. Agent has reached the Exit.\nEpisode 9512 was successful. Agent has reached the Exit.\nEpisode 9515 was successful. Agent has reached the Exit.\nEpisode 9525 was successful. Agent has reached the Exit.\nEpisode 9550 was successful. Agent has reached the Exit.\nEpisode 9564 was successful. Agent has reached the Exit.\nEpisode 9595 was successful. Agent has reached the Exit.\nEpisode 9609 was successful. Agent has reached the Exit.\nEpisode 9625 was successful. Agent has reached the Exit.\nEpisode 9632 was successful. Agent has reached the Exit.\nEpisode 9689 was successful. Agent has reached the Exit.\nEpisode 9692 was successful. Agent has reached the Exit.\nEpisode 9698 was successful. Agent has reached the Exit.\nEpisode 9706 was successful. Agent has reached the Exit.\nEpisode 9724 was successful. Agent has reached the Exit.\nEpisode 9743 was successful. Agent has reached the Exit.\nEpisode 9755 was successful. Agent has reached the Exit.\nEpisode 9771 was successful. Agent has reached the Exit.\nEpisode 9778 was successful. Agent has reached the Exit.\nEpisode 9794 was successful. Agent has reached the Exit.\nEpisode 9844 was successful. Agent has reached the Exit.\nEpisode 9883 was successful. Agent has reached the Exit.\nEpisode 9906 was successful. Agent has reached the Exit.\nEpisode 9916 was successful. Agent has reached the Exit.\nEpisode 9953 was successful. Agent has reached the Exit.\nOur Q is equal:\n [[0.78275779 0.8153727 0.8153727 0.78275779]\n [0.78275779 0. 0.84934656 0.8153727 ]\n [0.8153727 0.884736 0.8153727 0.84934656]\n [0.84934656 0. 0.8153727 0.8153727 ]\n [0.8153727 0.84934656 0. 0.78275779]\n [0. 0. 0. 0. ]\n [0. 0.9216 0. 0.84934656]\n [0. 0. 0. 0. ]\n [0.84934656 0. 0.884736 0.8153727 ]\n [0.84934656 0.9216 0.9216 0. ]\n [0.884736 0.96 0. 0.884736 ]\n [0. 0. 0. 0. ]\n [0. 0. 0. 0. ]\n [0. 0.9216 0.96 0.884736 ]\n [0.9216 0.96 1. 0.9216 ]\n [0. 0. 0. 0. ]]\n"
],
[
"# Save our Q-Matrix to file\nSaveQTableToFile(Q_deterministic, file_Q_deterministic)",
"_____no_output_____"
],
[
"initPlayByQModel(10, env_deterministic, file_Q_deterministic, False, False)",
"*** Starting Episode: 0\nSuccess: Agent passed the Lake!\n*** Starting Episode: 1\nSuccess: Agent passed the Lake!\n*** Starting Episode: 2\nSuccess: Agent passed the Lake!\n*** Starting Episode: 3\nSuccess: Agent passed the Lake!\n*** Starting Episode: 4\nSuccess: Agent passed the Lake!\n*** Starting Episode: 5\nSuccess: Agent passed the Lake!\n*** Starting Episode: 6\nSuccess: Agent passed the Lake!\n*** Starting Episode: 7\nSuccess: Agent passed the Lake!\n*** Starting Episode: 8\nSuccess: Agent passed the Lake!\n*** Starting Episode: 9\nSuccess: Agent passed the Lake!\n"
]
],
[
[
"Wow. Environment definitely takes huge place in results.\nLet's check differences in learned Q-Matrices:",
"_____no_output_____"
]
],
[
[
"print(\"Stochastic:\\n\\n\", Q_stochastic, \"\\n\\n\\nDeterministic:\\n\\n\", Q_deterministic)",
"Stochastic:\n\n [[0.63155759 0.7260818 0.63098495 0.64096586]\n [0.08923413 0.59320009 0.4857933 0.5864178 ]\n [0.58253307 0.54801955 0.74002867 0.5670273 ]\n [0.09847175 0.43629738 0.52282304 0.5225726 ]\n [0.69112814 0.72705092 0.76905186 0.12600166]\n [0. 0. 0. 0. ]\n [0.59500203 0.00506861 0.15284422 0.46626663]\n [0. 0. 0. 0. ]\n [0.73769729 0.12177414 0.02240348 0.75699989]\n [0.02665465 0.75511081 0.84217728 0.79496329]\n [0.88576406 0.8295706 0.79526765 0.79497617]\n [0. 0. 0. 0. ]\n [0. 0. 0. 0. ]\n [0.15828885 0.02537745 0.92143326 0.16265478]\n [0.83612259 0.99437935 0.90915363 0.94866158]\n [0. 0. 0. 0. ]] \n\n\nDeterministic:\n\n [[0.78275779 0.8153727 0.8153727 0.78275779]\n [0.78275779 0. 0.84934656 0.8153727 ]\n [0.8153727 0.884736 0.8153727 0.84934656]\n [0.84934656 0. 0.8153727 0.8153727 ]\n [0.8153727 0.84934656 0. 0.78275779]\n [0. 0. 0. 0. ]\n [0. 0.9216 0. 0.84934656]\n [0. 0. 0. 0. ]\n [0.84934656 0. 0.884736 0.8153727 ]\n [0.84934656 0.9216 0.9216 0. ]\n [0.884736 0.96 0. 0.884736 ]\n [0. 0. 0. 0. ]\n [0. 0. 0. 0. ]\n [0. 0.9216 0.96 0.884736 ]\n [0.9216 0.96 1. 0.9216 ]\n [0. 0. 0. 0. ]]\n"
]
],
[
[
"#### Play against environment <a class=\"anchor\" id=\"pve\"></a>",
"_____no_output_____"
],
[
"Let's adapt environment to be human agent friendly",
"_____no_output_____"
]
],
[
[
"def initPlayVsEnv(environment, showOutput=True):\n state = environment.reset()\n \n while True:\n if showOutput == True:\n environment.render()\n\n action = input('Your action? 0 -> Left, 1 -> Down, 2 -> Right, 3 -> Up')\n action = int(action)\n\n if action >= 4:\n print ('No such input! Try once more (0 to 3)')\n break\n\n state2, reward, done, info = environment.step(action)\n state = state2\n \n if done == True and reward == 1:\n print('Success: You have passed the lake!')\n break\n \n if done == True and reward == 0:\n print('Bottom of the Lake is Dark and Full of Terrors!')\n break",
"_____no_output_____"
],
[
"initPlayVsEnv(env_stochastic, True)",
"\n\u001b[41mS\u001b[0mFFF\nFHFH\nFFFH\nHFFG\n"
]
],
[
[
"For some reason it halts :(",
"_____no_output_____"
],
[
"### DNN Approach <a class=\"anchor\" id=\"dnn\"></a>",
"_____no_output_____"
],
[
"I had no time :'(",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown",
"markdown"
]
] |
e7e9b8ea469e1d19e4d30387b22958d67ee0926d | 522,135 | ipynb | Jupyter Notebook | pensamento_computacional/PC_exercicio3_aula3.ipynb | biamuniz/MJDA_Insper | 9fd083833ecab54ec947ffbdd70e645863337914 | [
"MIT"
] | 2 | 2021-12-19T21:54:34.000Z | 2022-01-15T00:23:48.000Z | pensamento_computacional/PC_exercicio3_aula3.ipynb | biamuniz/mjda_insper | 906bde0e34f4d2e4ec6f61be35cd56b0d79ba4ee | [
"MIT"
] | null | null | null | pensamento_computacional/PC_exercicio3_aula3.ipynb | biamuniz/mjda_insper | 906bde0e34f4d2e4ec6f61be35cd56b0d79ba4ee | [
"MIT"
] | null | null | null | 103.209132 | 145 | 0.712396 | [
[
[
"\n#Pensamento Computacional\n####Aluna: Bianca Muniz\n####Exercício : calculando a densidade demográfica\n",
"_____no_output_____"
]
],
[
[
"import csv\narquivo = open(\"brasil.csv\")\nleitor = csv.DictReader(arquivo)\nfor registro in leitor:\n registro[\"densidade\"] = int(registro[\"habitantes\"])/float(registro[\"area\"])\n print(f\"O município {registro['municipio']}/{registro['estado']} possui densidade demográfica de {registro['densidade']} hab/km².\")",
"\u001b[1;30;43mA saída de streaming foi truncada nas últimas 5000 linhas.\u001b[0m\nO município Sapeaçu/BA possui densidade demográfica de 141.4981656855217 hab/km².\nO município Sátiro Dias/BA possui densidade demográfica de 18.77530815306173 hab/km².\nO município Saubara/BA possui densidade demográfica de 68.50764525993884 hab/km².\nO município Saúde/BA possui densidade demográfica de 23.48753742737602 hab/km².\nO município Seabra/BA possui densidade demográfica de 16.60436421707471 hab/km².\nO município Sebastião Laranjeiras/BA possui densidade demográfica de 5.322255351250379 hab/km².\nO município Senhor do Bonfim/BA possui densidade demográfica de 89.93341309260535 hab/km².\nO município Sento Sé/BA possui densidade demográfica de 2.947149749856482 hab/km².\nO município Serra do Ramalho/BA possui densidade demográfica de 12.200229057970176 hab/km².\nO município Serra Dourada/BA possui densidade demográfica de 13.449871159858311 hab/km².\nO município Serra Preta/BA possui densidade demográfica de 28.706965647076366 hab/km².\nO município Serrinha/BA possui densidade demográfica de 122.9706999022796 hab/km².\nO município Serrolândia/BA possui densidade demográfica de 41.72384654385668 hab/km².\nO município Simões Filho/BA possui densidade demográfica de 586.6563959844946 hab/km².\nO município Sítio do Mato/BA possui densidade demográfica de 6.880917303365654 hab/km².\nO município Sítio do Quinto/BA possui densidade demográfica de 17.984203836211208 hab/km².\nO município Sobradinho/BA possui densidade demográfica de 17.757401607852 hab/km².\nO município Souto Soares/BA possui densidade demográfica de 16.002858552002497 hab/km².\nO município Tabocas do Brejo Velho/BA possui densidade demográfica de 8.308982801982932 hab/km².\nO município Tanhaçu/BA possui densidade demográfica de 16.212209584912998 hab/km².\nO município Tanque Novo/BA possui densidade demográfica de 22.31013971503666 hab/km².\nO município Tanquinho/BA possui densidade demográfica de 36.424835114851035 hab/km².\nO município Taperoá/BA possui densidade demográfica de 45.638890917500426 hab/km².\nO município Tapiramutá/BA possui densidade demográfica de 24.877989998192444 hab/km².\nO município Teixeira de Freitas/BA possui densidade demográfica de 118.86701666050884 hab/km².\nO município Teodoro Sampaio/BA possui densidade demográfica de 34.097780081195474 hab/km².\nO município Teofilândia/BA possui densidade demográfica de 64.02217321332776 hab/km².\nO município Teolândia/BA possui densidade demográfica de 46.679042255293716 hab/km².\nO município Terra Nova/BA possui densidade demográfica de 64.35932237470466 hab/km².\nO município Tremedal/BA possui densidade demográfica de 10.139568670882307 hab/km².\nO município Tucano/BA possui densidade demográfica de 18.72639908543665 hab/km².\nO município Uauá/BA possui densidade demográfica de 8.003979915920983 hab/km².\nO município Ubaíra/BA possui densidade demográfica de 27.1941178090491 hab/km².\nO município Ubaitaba/BA possui densidade demográfica de 115.71500475364913 hab/km².\nO município Ubatã/BA possui densidade demográfica de 93.21503131524008 hab/km².\nO município Uibaí/BA possui densidade demográfica de 24.728216483057768 hab/km².\nO município Umburanas/BA possui densidade demográfica de 10.17708121310808 hab/km².\nO município Una/BA possui densidade demográfica de 20.476627259138468 hab/km².\nO município Urandi/BA possui densidade demográfica de 16.984888338748775 hab/km².\nO município Uruçuca/BA possui densidade demográfica de 50.60717383539976 hab/km².\nO município Utinga/BA possui densidade demográfica de 28.474061075160993 hab/km².\nO município Valença/BA possui densidade demográfica de 74.35205138310094 hab/km².\nO município Valente/BA possui densidade demográfica de 63.901753655617426 hab/km².\nO município Várzea da Roça/BA possui densidade demográfica de 26.82518679950187 hab/km².\nO município Várzea do Poço/BA possui densidade demográfica de 42.26733687960568 hab/km².\nO município Várzea Nova/BA possui densidade demográfica de 10.95873186188628 hab/km².\nO município Varzedo/BA possui densidade demográfica de 40.16313932980599 hab/km².\nO município Vera Cruz/BA possui densidade demográfica de 125.33613585560337 hab/km².\nO município Vereda/BA possui densidade demográfica de 7.7773838253291085 hab/km².\nO município Vitória da Conquista/BA possui densidade demográfica de 91.41377882504342 hab/km².\nO município Wagner/BA possui densidade demográfica de 21.33729216152019 hab/km².\nO município Wanderley/BA possui densidade demográfica de 4.218603755351392 hab/km².\nO município Wenceslau Guimarães/BA possui densidade demográfica de 32.91989970772814 hab/km².\nO município Xique-Xique/BA possui densidade demográfica de 8.275766811514394 hab/km².\nO município Abaiara/CE possui densidade demográfica de 58.69261309623665 hab/km².\nO município Acarape/CE possui densidade demográfica de 98.52261048304213 hab/km².\nO município Acaraú/CE possui densidade demográfica de 68.30492783896696 hab/km².\nO município Acopiara/CE possui densidade demográfica de 22.583706711987112 hab/km².\nO município Aiuaba/CE possui densidade demográfica de 6.655794809441263 hab/km².\nO município Alcântaras/CE possui densidade demográfica de 77.70723613014933 hab/km².\nO município Altaneira/CE possui densidade demográfica de 93.53342428376536 hab/km².\nO município Alto Santo/CE possui densidade demográfica de 12.224538749523617 hab/km².\nO município Amontada/CE possui densidade demográfica de 33.27452842990908 hab/km².\nO município Antonina do Norte/CE possui densidade demográfica de 26.85121107266436 hab/km².\nO município Apuiarés/CE possui densidade demográfica de 25.542959864993765 hab/km².\nO município Aquiraz/CE possui densidade demográfica de 150.5025177694428 hab/km².\nO município Aracati/CE possui densidade demográfica de 56.315652329690735 hab/km².\nO município Aracoiaba/CE possui densidade demográfica de 38.67042339323789 hab/km².\nO município Ararendá/CE possui densidade demográfica de 30.485572312788772 hab/km².\nO município Araripe/CE possui densidade demográfica de 18.8057421835935 hab/km².\nO município Aratuba/CE possui densidade demográfica de 100.43557801202195 hab/km².\nO município Arneiroz/CE possui densidade demográfica de 7.173937507033273 hab/km².\nO município Assaré/CE possui densidade demográfica de 20.106061827595784 hab/km².\nO município Aurora/CE possui densidade demográfica de 27.73187031518107 hab/km².\nO município Baixio/CE possui densidade demográfica de 41.15276924127569 hab/km².\nO município Banabuiú/CE possui densidade demográfica de 16.027510112650766 hab/km².\nO município Barbalha/CE possui densidade demográfica de 97.14140225808151 hab/km².\nO município Barreira/CE possui densidade demográfica de 79.62654082421382 hab/km².\nO município Barro/CE possui densidade demográfica de 30.220961103541278 hab/km².\nO município Barroquinha/CE possui densidade demográfica de 37.75592707545447 hab/km².\nO município Baturité/CE possui densidade demográfica de 107.98172272992417 hab/km².\nO município Beberibe/CE possui densidade demográfica de 30.365973064678023 hab/km².\nO município Bela Cruz/CE possui densidade demográfica de 36.627838010960595 hab/km².\nO município Boa Viagem/CE possui densidade demográfica de 18.506193642087155 hab/km².\nO município Brejo Santo/CE possui densidade demográfica de 68.12022368599551 hab/km².\nO município Camocim/CE possui densidade demográfica de 53.484236917441635 hab/km².\nO município Campos Sales/CE possui densidade demográfica de 24.47980642241658 hab/km².\nO município Canindé/CE possui densidade demográfica de 23.139183714051352 hab/km².\nO município Capistrano/CE possui densidade demográfica de 76.66591777128735 hab/km².\nO município Caridade/CE possui densidade demográfica de 23.650045480856694 hab/km².\nO município Cariré/CE possui densidade demográfica de 24.240302293626467 hab/km².\nO município Caririaçu/CE possui densidade demográfica de 42.32631984091347 hab/km².\nO município Cariús/CE possui densidade demográfica de 17.486343944245622 hab/km².\nO município Carnaubal/CE possui densidade demográfica de 45.90334694772621 hab/km².\nO município Cascavel/CE possui densidade demográfica de 78.99155649504974 hab/km².\nO município Catarina/CE possui densidade demográfica de 38.50182804091525 hab/km².\nO município Catunda/CE possui densidade demográfica de 12.586156745203677 hab/km².\nO município Caucaia/CE possui densidade demográfica de 264.9070825634305 hab/km².\nO município Cedro/CE possui densidade demográfica de 33.79305593827501 hab/km².\nO município Chaval/CE possui densidade demográfica de 52.95302858582043 hab/km².\nO município Choró/CE possui densidade demográfica de 15.755666425585643 hab/km².\nO município Chorozinho/CE possui densidade demográfica de 67.93936999389389 hab/km².\nO município Coreaú/CE possui densidade demográfica de 28.2985305491106 hab/km².\nO município Crateús/CE possui densidade demográfica de 24.3914858264604 hab/km².\nO município Crato/CE possui densidade demográfica de 103.2138516069258 hab/km².\nO município Croatá/CE possui densidade demográfica de 24.48994232259175 hab/km².\nO município Cruz/CE possui densidade demográfica de 68.12850431883619 hab/km².\nO município Deputado Irapuan Pinheiro/CE possui densidade demográfica de 19.333375847628766 hab/km².\nO município Ererê/CE possui densidade demográfica de 17.872540565963785 hab/km².\nO município Eusébio/CE possui densidade demográfica de 582.622452854069 hab/km².\nO município Farias Brito/CE possui densidade demográfica de 37.74075691989992 hab/km².\nO município Forquilha/CE possui densidade demográfica de 42.14008007891835 hab/km².\nO município Fortaleza/CE possui densidade demográfica de 7786.444606738005 hab/km².\nO município Fortim/CE possui densidade demográfica de 53.15134340137031 hab/km².\nO município Frecheirinha/CE possui densidade demográfica de 71.67843743103067 hab/km².\nO município General Sampaio/CE possui densidade demográfica de 30.212331762305038 hab/km².\nO município Graça/CE possui densidade demográfica de 53.38986057402349 hab/km².\nO município Granja/CE possui densidade demográfica de 19.518244711221186 hab/km².\nO município Granjeiro/CE possui densidade demográfica de 46.22990112853291 hab/km².\nO município Groaíras/CE possui densidade demográfica de 65.58512343699904 hab/km².\nO município Guaiúba/CE possui densidade demográfica de 90.18455433683974 hab/km².\nO município Guaraciaba do Norte/CE possui densidade demográfica de 61.77836653256141 hab/km².\nO município Guaramiranga/CE possui densidade demográfica de 70.05383580080753 hab/km².\nO município Hidrolândia/CE possui densidade demográfica de 19.987588560790194 hab/km².\nO município Horizonte/CE possui densidade demográfica de 344.9618702337792 hab/km².\nO município Ibaretama/CE possui densidade demográfica de 14.729954631466157 hab/km².\nO município Ibiapina/CE possui densidade demográfica de 57.37697016436111 hab/km².\nO município Ibicuitinga/CE possui densidade demográfica de 26.717737183264585 hab/km².\nO município Icapuí/CE possui densidade demográfica de 43.433699374188215 hab/km².\nO município Icó/CE possui densidade demográfica de 34.965811965811966 hab/km².\nO município Iguatu/CE possui densidade demográfica de 93.75637624974495 hab/km².\nO município Independência/CE possui densidade demográfica de 7.9451824971727545 hab/km².\nO município Ipaporanga/CE possui densidade demográfica de 16.154897883612954 hab/km².\nO município Ipaumirim/CE possui densidade demográfica de 43.855676879815945 hab/km².\nO município Ipu/CE possui densidade demográfica de 64.03101760630521 hab/km².\nO município Ipueiras/CE possui densidade demográfica de 25.627280172734718 hab/km².\nO município Iracema/CE possui densidade demográfica de 16.70867579908676 hab/km².\nO município Irauçuba/CE possui densidade demográfica de 15.277331052181351 hab/km².\nO município Itaiçaba/CE possui densidade demográfica de 34.49153740983452 hab/km².\nO município Itaitinga/CE possui densidade demográfica de 236.5095087163233 hab/km².\nO município Itapagé/CE possui densidade demográfica de 110.00887351823621 hab/km².\nO município Itapipoca/CE possui densidade demográfica de 71.90427219110869 hab/km².\nO município Itapiúna/CE possui densidade demográfica de 31.639205028027856 hab/km².\nO município Itarema/CE possui densidade demográfica de 51.995393111869674 hab/km².\nO município Itatira/CE possui densidade demográfica de 24.116716021648113 hab/km².\nO município Jaguaretama/CE possui densidade demográfica de 10.152893031715356 hab/km².\nO município Jaguaribara/CE possui densidade demográfica de 15.550139067500075 hab/km².\nO município Jaguaribe/CE possui densidade demográfica de 18.33376846883808 hab/km².\nO município Jaguaruana/CE possui densidade demográfica de 37.15708423624879 hab/km².\nO município Jardim/CE possui densidade demográfica de 48.31106766590638 hab/km².\nO município Jati/CE possui densidade demográfica de 21.214722906915558 hab/km².\nO município Jijoca de Jericoacoara/CE possui densidade demográfica de 83.02163191562089 hab/km².\nO município Juazeiro do Norte/CE possui densidade demográfica de 1004.4568580958887 hab/km².\nO município Jucás/CE possui densidade demográfica de 25.402533104279815 hab/km².\nO município Lavras da Mangabeira/CE possui densidade demográfica de 32.79639651043809 hab/km².\nO município Limoeiro do Norte/CE possui densidade demográfica de 74.91179250935332 hab/km².\nO município Madalena/CE possui densidade demográfica de 17.481057677439306 hab/km².\nO município Maracanaú/CE possui densidade demográfica de 1960.2156586966712 hab/km².\nO município Maranguape/CE possui densidade demográfica de 192.1928681435849 hab/km².\nO município Marco/CE possui densidade demográfica de 43.02609119726896 hab/km².\nO município Martinópole/CE possui densidade demográfica de 34.16510569975917 hab/km².\nO município Massapê/CE possui densidade demográfica de 62.11126407568216 hab/km².\nO município Mauriti/CE possui densidade demográfica de 42.15380804009566 hab/km².\nO município Meruoca/CE possui densidade demográfica de 91.37804471137805 hab/km².\nO município Milagres/CE possui densidade demográfica de 46.69217070114108 hab/km².\nO município Milhã/CE possui densidade demográfica de 26.050085599394833 hab/km².\nO município Miraíma/CE possui densidade demográfica de 18.286759243385337 hab/km².\nO município Missão Velha/CE possui densidade demográfica de 53.080377884466465 hab/km².\nO município Mombaça/CE possui densidade demográfica de 20.141732877875704 hab/km².\nO município Monsenhor Tabosa/CE possui densidade demográfica de 18.851423025707 hab/km².\nO município Morada Nova/CE possui densidade demográfica de 22.331564270936404 hab/km².\nO município Moraújo/CE possui densidade demográfica de 19.416307773741067 hab/km².\nO município Morrinhos/CE possui densidade demográfica de 49.812301472711525 hab/km².\nO município Mucambo/CE possui densidade demográfica de 73.98740818467996 hab/km².\nO município Mulungu/CE possui densidade demográfica de 85.34591662331873 hab/km².\nO município Nova Olinda/CE possui densidade demográfica de 50.12658227848102 hab/km².\nO município Nova Russas/CE possui densidade demográfica de 41.68854423307349 hab/km².\nO município Novo Oriente/CE possui densidade demográfica de 28.916462149380127 hab/km².\nO município Ocara/CE possui densidade demográfica de 31.364889405678003 hab/km².\nO município Orós/CE possui densidade demográfica de 37.11628229822826 hab/km².\nO município Pacajus/CE possui densidade demográfica de 242.99748506758883 hab/km².\nO município Pacatuba/CE possui densidade demográfica de 547.7611940298507 hab/km².\nO município Pacoti/CE possui densidade demográfica de 103.61542581681843 hab/km².\nO município Pacujá/CE possui densidade demográfica de 78.62866150006568 hab/km².\nO município Palhano/CE possui densidade demográfica de 20.13261274353967 hab/km².\nO município Palmácia/CE possui densidade demográfica de 101.90136660724896 hab/km².\nO município Paracuru/CE possui densidade demográfica de 105.35149355622897 hab/km².\nO município Paraipaba/CE possui densidade demográfica de 99.83051973946563 hab/km².\nO município Parambu/CE possui densidade demográfica de 13.591689312970471 hab/km².\nO município Paramoti/CE possui densidade demográfica de 23.431898713193394 hab/km².\nO município Pedra Branca/CE possui densidade demográfica de 32.14173361262651 hab/km².\nO município Penaforte/CE possui densidade demográfica de 57.95814838300571 hab/km².\nO município Pentecoste/CE possui densidade demográfica de 25.68362705051839 hab/km².\nO município Pereiro/CE possui densidade demográfica de 36.34748910059745 hab/km².\nO município Pindoretama/CE possui densidade demográfica de 256.07182017543863 hab/km².\nO município Piquet Carneiro/CE possui densidade demográfica de 26.309791113832755 hab/km².\nO município Pires Ferreira/CE possui densidade demográfica de 42.02385849444673 hab/km².\nO município Poranga/CE possui densidade demográfica de 9.16624658203871 hab/km².\nO município Porteiras/CE possui densidade demográfica de 69.22051659159848 hab/km².\nO município Potengi/CE possui densidade demográfica de 30.33684645587931 hab/km².\nO município Potiretama/CE possui densidade demográfica de 14.929083199298145 hab/km².\nO município Quiterianópolis/CE possui densidade demográfica de 19.136591129597786 hab/km².\nO município Quixadá/CE possui densidade demográfica de 39.90632875043939 hab/km².\nO município Quixelô/CE possui densidade demográfica de 26.806776753163202 hab/km².\nO município Quixeramobim/CE possui densidade demográfica de 21.946007332940532 hab/km².\nO município Quixeré/CE possui densidade demográfica de 31.686853187946852 hab/km².\nO município Redenção/CE possui densidade demográfica de 117.23847143935022 hab/km².\nO município Reriutaba/CE possui densidade demográfica de 50.75393926745278 hab/km².\nO município Russas/CE possui densidade demográfica de 43.91432578087171 hab/km².\nO município Saboeiro/CE possui densidade demográfica de 11.385780784687888 hab/km².\nO município Salitre/CE possui densidade demográfica de 19.2115470684768 hab/km².\nO município Santa Quitéria/CE possui densidade demográfica de 10.037131966351211 hab/km².\nO município Santana do Acaraú/CE possui densidade demográfica de 30.8935037603293 hab/km².\nO município Santana do Cariri/CE possui densidade demográfica de 20.068726915704335 hab/km².\nO município São Benedito/CE possui densidade demográfica de 130.60753880266074 hab/km².\nO município São Gonçalo do Amarante/CE possui densidade demográfica de 52.5975193241057 hab/km².\nO município São João do Jaguaribe/CE possui densidade demográfica de 28.16800969835271 hab/km².\nO município São Luís do Curu/CE possui densidade demográfica de 100.73517399117792 hab/km².\nO município Senador Pompeu/CE possui densidade demográfica de 26.412740861964018 hab/km².\nO município Senador Sá/CE possui densidade demográfica de 16.163427061709754 hab/km².\nO município Sobral/CE possui densidade demográfica de 88.66786000282632 hab/km².\nO município Solonópole/CE possui densidade demográfica de 11.499378324013618 hab/km².\nO município Tabuleiro do Norte/CE possui densidade demográfica de 33.886033208405365 hab/km².\nO município Tamboril/CE possui densidade demográfica de 12.976530992041035 hab/km².\nO município Tarrafas/CE possui densidade demográfica de 19.60870617751271 hab/km².\nO município Tauá/CE possui densidade demográfica de 13.866048141437872 hab/km².\nO município Tejuçuoca/CE possui densidade demográfica de 22.417169577554855 hab/km².\nO município Tianguá/CE possui densidade demográfica de 75.79795134724775 hab/km².\nO município Trairi/CE possui densidade demográfica de 55.548113900531476 hab/km².\nO município Tururu/CE possui densidade demográfica de 71.22800079098279 hab/km².\nO município Ubajara/CE possui densidade demográfica de 75.49818302733773 hab/km².\nO município Umari/CE possui densidade demográfica de 28.587125374152237 hab/km².\nO município Umirim/CE possui densidade demográfica de 59.346000883782594 hab/km².\nO município Uruburetama/CE possui densidade demográfica de 203.6159472545586 hab/km².\nO município Uruoca/CE possui densidade demográfica de 18.490132759239327 hab/km².\nO município Varjota/CE possui densidade demográfica de 98.06577480490523 hab/km².\nO município Várzea Alegre/CE possui densidade demográfica de 45.98963755369685 hab/km².\nO município Viçosa do Ceará/CE possui densidade demográfica de 41.89824874392931 hab/km².\nO município Brasília/DF possui densidade demográfica de 444.6643598615917 hab/km².\nO município Afonso Cláudio/ES possui densidade demográfica de 32.678522629332996 hab/km².\nO município Água Doce do Norte/ES possui densidade demográfica de 24.847486965148924 hab/km².\nO município Águia Branca/ES possui densidade demográfica de 20.946198701727365 hab/km².\nO município Alegre/ES possui densidade demográfica de 39.854922279792746 hab/km².\nO município Alfredo Chaves/ES possui densidade demográfica de 22.66194644278082 hab/km².\nO município Alto Rio Novo/ES possui densidade demográfica de 32.14426920880376 hab/km².\nO município Anchieta/ES possui densidade demográfica de 58.407252645211734 hab/km².\nO município Apiacá/ES possui densidade demográfica de 38.723645548739626 hab/km².\nO município Aracruz/ES possui densidade demográfica de 57.4715388343037 hab/km².\nO município Atilio Vivacqua/ES possui densidade demográfica de 44.08144998881182 hab/km².\nO município Baixo Guandu/ES possui densidade demográfica de 31.71077453193322 hab/km².\nO município Barra de São Francisco/ES possui densidade demográfica de 43.16096835846252 hab/km².\nO município Boa Esperança/ES possui densidade demográfica de 33.1365227537923 hab/km².\nO município Bom Jesus do Norte/ES possui densidade demográfica de 106.37629097440504 hab/km².\nO município Brejetuba/ES possui densidade demográfica de 34.61951942354069 hab/km².\nO município Cachoeiro de Itapemirim/ES possui densidade demográfica de 216.23015782641374 hab/km².\nO município Cariacica/ES possui densidade demográfica de 1246.1159151004074 hab/km².\nO município Castelo/ES possui densidade demográfica de 52.325091106225344 hab/km².\nO município Colatina/ES possui densidade demográfica de 78.90175042348956 hab/km².\nO município Conceição da Barra/ES possui densidade demográfica de 24.0094184368433 hab/km².\nO município Conceição do Castelo/ES possui densidade demográfica de 31.636107575224113 hab/km².\nO município Divino de São Lourenço/ES possui densidade demográfica de 25.971934667586844 hab/km².\nO município Domingos Martins/ES possui densidade demográfica de 25.926649570562137 hab/km².\nO município Dores do Rio Preto/ES possui densidade demográfica de 40.15693659761456 hab/km².\nO município Ecoporanga/ES possui densidade demográfica de 10.156779864967161 hab/km².\nO município Fundão/ES possui densidade demográfica de 58.96716541978387 hab/km².\nO município Governador Lindenberg/ES possui densidade demográfica de 30.193344074670815 hab/km².\nO município Guaçuí/ES possui densidade demográfica de 59.467480889951744 hab/km².\nO município Guarapari/ES possui densidade demográfica de 177.10306312974146 hab/km².\nO município Ibatiba/ES possui densidade demográfica de 92.98245614035088 hab/km².\nO município Ibiraçu/ES possui densidade demográfica de 55.542857142857144 hab/km².\nO município Ibitirama/ES possui densidade demográfica de 27.15312092642556 hab/km².\nO município Iconha/ES possui densidade demográfica de 61.52901292192797 hab/km².\nO município Irupi/ES possui densidade demográfica de 63.522080736927656 hab/km².\nO município Itaguaçu/ES possui densidade demográfica de 26.59266227657573 hab/km².\nO município Itapemirim/ES possui densidade demográfica de 55.151547510990085 hab/km².\nO município Itarana/ES possui densidade demográfica de 36.42053822466194 hab/km².\nO município Iúna/ES possui densidade demográfica de 59.269541077470286 hab/km².\nO município Jaguaré/ES possui densidade demográfica de 37.40507768093975 hab/km².\nO município Jerônimo Monteiro/ES possui densidade demográfica de 67.16261266823065 hab/km².\nO município João Neiva/ES possui densidade demográfica de 55.5227759631932 hab/km².\nO município Laranja da Terra/ES possui densidade demográfica de 23.618474158430963 hab/km².\nO município Linhares/ES possui densidade demográfica de 40.32544361812028 hab/km².\nO município Mantenópolis/ES possui densidade demográfica de 42.349573766411545 hab/km².\nO município Marataízes/ES possui densidade demográfica de 256.53742110009017 hab/km².\nO município Marechal Floriano/ES possui densidade demográfica de 49.975471301422665 hab/km².\nO município Marilândia/ES possui densidade demográfica de 35.942657433175846 hab/km².\nO município Mimoso do Sul/ES possui densidade demográfica de 29.79193264552638 hab/km².\nO município Montanha/ES possui densidade demográfica de 16.242310632257123 hab/km².\nO município Mucurici/ES possui densidade demográfica de 10.468538847442565 hab/km².\nO município Muniz Freire/ES possui densidade demográfica de 27.081493257963846 hab/km².\nO município Muqui/ES possui densidade demográfica de 43.958594155546734 hab/km².\nO município Nova Venécia/ES possui densidade demográfica de 31.918095079602818 hab/km².\nO município Pancas/ES possui densidade demográfica de 25.963322649830108 hab/km².\nO município Pedro Canário/ES possui densidade demográfica de 54.84004793952245 hab/km².\nO município Pinheiros/ES possui densidade demográfica de 24.5545348048585 hab/km².\nO município Piúma/ES possui densidade demográfica de 242.1889616463985 hab/km².\nO município Ponto Belo/ES possui densidade demográfica de 19.350634947041534 hab/km².\nO município Presidente Kennedy/ES possui densidade demográfica de 17.66307605363657 hab/km².\nO município Rio Bananal/ES possui densidade demográfica de 27.29551718231786 hab/km².\nO município Rio Novo do Sul/ES possui densidade demográfica de 55.416911332941865 hab/km².\nO município Santa Leopoldina/ES possui densidade demográfica de 17.04497980782621 hab/km².\nO município Santa Maria de Jetibá/ES possui densidade demográfica de 46.461295848174224 hab/km².\nO município Santa Teresa/ES possui densidade demográfica de 31.94420048012179 hab/km².\nO município São Domingos do Norte/ES possui densidade demográfica de 26.78517625790901 hab/km².\nO município São Gabriel da Palha/ES possui densidade demográfica de 73.25760537147325 hab/km².\nO município São José do Calçado/ES possui densidade demográfica de 38.05623605981937 hab/km².\nO município São Mateus/ES possui densidade demográfica de 46.61846386714157 hab/km².\nO município São Roque do Canaã/ES possui densidade demográfica de 32.96102453144645 hab/km².\nO município Serra/ES possui densidade demográfica de 741.8423389947252 hab/km².\nO município Sooretama/ES possui densidade demográfica de 40.658572354285326 hab/km².\nO município Vargem Alta/ES possui densidade demográfica de 46.249063172400454 hab/km².\nO município Venda Nova do Imigrante/ES possui densidade demográfica de 109.98332526491313 hab/km².\nO município Viana/ES possui densidade demográfica de 207.8369304556355 hab/km².\nO município Vila Pavão/ES possui densidade demográfica de 20.0156949637631 hab/km².\nO município Vila Valério/ES possui densidade demográfica de 29.419272495213782 hab/km².\nO município Vila Velha/ES possui densidade demográfica de 1973.5611938877519 hab/km².\nO município Vitória/ES possui densidade demográfica de 3338.4356859150625 hab/km².\nO município Abadia de Goiás/GO possui densidade demográfica de 46.845619294181766 hab/km².\nO município Abadiânia/GO possui densidade demográfica de 15.076593342455004 hab/km².\nO município Acreúna/GO possui densidade demográfica de 12.94955300127714 hab/km².\nO município Adelândia/GO possui densidade demográfica de 21.473775465973127 hab/km².\nO município Água Fria de Goiás/GO possui densidade demográfica de 2.508105764208493 hab/km².\nO município Água Limpa/GO possui densidade demográfica de 4.4450823654109435 hab/km².\nO município Águas Lindas de Goiás/GO possui densidade demográfica de 846.0003184882426 hab/km².\nO município Alexânia/GO possui densidade demográfica de 28.086190425644837 hab/km².\nO município Aloândia/GO possui densidade demográfica de 20.076350822239625 hab/km².\nO município Alto Horizonte/GO possui densidade demográfica de 8.942750516118787 hab/km².\nO município Alto Paraíso de Goiás/GO possui densidade demográfica de 2.6542940965569355 hab/km².\nO município Alvorada do Norte/GO possui densidade demográfica de 6.419082557151592 hab/km².\nO município Amaralina/GO possui densidade demográfica de 2.5566384002025058 hab/km².\nO município Americano do Brasil/GO possui densidade demográfica de 41.23989218328841 hab/km².\nO município Amorinópolis/GO possui densidade demográfica de 8.83411254987394 hab/km².\nO município Anápolis/GO possui densidade demográfica de 358.58052209696086 hab/km².\nO município Anhanguera/GO possui densidade demográfica de 17.91044776119403 hab/km².\nO município Anicuns/GO possui densidade demográfica de 20.668280179324572 hab/km².\nO município Aparecida de Goiânia/GO possui densidade demográfica de 1580.2767566067837 hab/km².\nO município Aparecida do Rio Doce/GO possui densidade demográfica de 4.030691046783917 hab/km².\nO município Aporé/GO possui densidade demográfica de 1.3113069623744897 hab/km².\nO município Araçu/GO possui densidade demográfica de 25.527057875654627 hab/km².\nO município Aragarças/GO possui densidade demográfica de 27.61351636747624 hab/km².\nO município Aragoiânia/GO possui densidade demográfica de 38.1006604418128 hab/km².\nO município Araguapaz/GO possui densidade demográfica de 3.423439850480923 hab/km².\nO município Arenópolis/GO possui densidade demográfica de 3.0495067932253863 hab/km².\nO município Aruanã/GO possui densidade demográfica de 2.4574551439034065 hab/km².\nO município Aurilândia/GO possui densidade demográfica de 6.456291789011922 hab/km².\nO município Avelinópolis/GO possui densidade demográfica de 14.109652153881596 hab/km².\nO município Baliza/GO possui densidade demográfica de 2.0834735779198925 hab/km².\nO município Barro Alto/GO possui densidade demográfica de 7.972558884061285 hab/km².\nO município Bela Vista de Goiás/GO possui densidade demográfica de 19.558394800146562 hab/km².\nO município Bom Jardim de Goiás/GO possui densidade demográfica de 4.434348167139601 hab/km².\nO município Bom Jesus de Goiás/GO possui densidade demográfica de 14.751053290822137 hab/km².\nO município Bonfinópolis/GO possui densidade demográfica de 61.62400850437484 hab/km².\nO município Bonópolis/GO possui densidade demográfica de 2.151072465903997 hab/km².\nO município Brazabrantes/GO possui densidade demográfica de 26.261477208092955 hab/km².\nO município Britânia/GO possui densidade demográfica de 3.770214688028251 hab/km².\nO município Buriti Alegre/GO possui densidade demográfica de 10.111004399973197 hab/km².\nO município Buriti de Goiás/GO possui densidade demográfica de 12.845601886697777 hab/km².\nO município Buritinópolis/GO possui densidade demográfica de 13.442622950819672 hab/km².\nO município Cabeceiras/GO possui densidade demográfica de 6.52175840937913 hab/km².\nO município Cachoeira Alta/GO possui densidade demográfica de 6.378169290743707 hab/km².\nO município Cachoeira de Goiás/GO possui densidade demográfica de 3.3518628030751034 hab/km².\nO município Cachoeira Dourada/GO possui densidade demográfica de 15.838658300232188 hab/km².\nO município Caçu/GO possui densidade demográfica de 5.900906704101714 hab/km².\nO município Caiapônia/GO possui densidade demográfica de 1.9399458431303085 hab/km².\nO município Caldas Novas/GO possui densidade demográfica de 44.15684505347845 hab/km².\nO município Caldazinha/GO possui densidade demográfica de 13.252819960939057 hab/km².\nO município Campestre de Goiás/GO possui densidade demográfica de 12.369439777956321 hab/km².\nO município Campinaçu/GO possui densidade demográfica de 1.8517205401189234 hab/km².\nO município Campinorte/GO possui densidade demográfica de 10.411356821589205 hab/km².\nO município Campo Alegre de Goiás/GO possui densidade demográfica de 2.4604241186525324 hab/km².\nO município Campo Limpo de Goiás/GO possui densidade demográfica de 39.1138129857107 hab/km².\nO município Campos Belos/GO possui densidade demográfica de 25.425718507879072 hab/km².\nO município Campos Verdes/GO possui densidade demográfica de 11.366466659119213 hab/km².\nO município Carmo do Rio Verde/GO possui densidade demográfica de 21.331294499928322 hab/km².\nO município Castelândia/GO possui densidade demográfica de 12.231449416669468 hab/km².\nO município Catalão/GO possui densidade demográfica de 22.673794832341567 hab/km².\nO município Caturaí/GO possui densidade demográfica de 22.60928302615073 hab/km².\nO município Cavalcante/GO possui densidade demográfica de 1.3506536835944185 hab/km².\nO município Ceres/GO possui densidade demográfica de 96.68719671519224 hab/km².\nO município Cezarina/GO possui densidade demográfica de 18.145306750679396 hab/km².\nO município Chapadão do Céu/GO possui densidade demográfica de 3.203943032876913 hab/km².\nO município Cidade Ocidental/GO possui densidade demográfica de 143.37547116592734 hab/km².\nO município Cocalzinho de Goiás/GO possui densidade demográfica de 9.72979922192908 hab/km².\nO município Colinas do Sul/GO possui densidade demográfica de 2.0624169442509324 hab/km².\nO município Córrego do Ouro/GO possui densidade demográfica de 5.6932727666017735 hab/km².\nO município Corumbá de Goiás/GO possui densidade demográfica de 9.756488003314626 hab/km².\nO município Corumbaíba/GO possui densidade demográfica de 4.343117425026676 hab/km².\nO município Cristalina/GO possui densidade demográfica de 7.559123609035246 hab/km².\nO município Cristianópolis/GO possui densidade demográfica de 13.01029463968761 hab/km².\nO município Crixás/GO possui densidade demográfica de 3.381132593603309 hab/km².\nO município Cromínia/GO possui densidade demográfica de 9.763532998269753 hab/km².\nO município Cumari/GO possui densidade demográfica de 5.19507834682932 hab/km².\nO município Damianópolis/GO possui densidade demográfica de 7.925845672324545 hab/km².\nO município Damolândia/GO possui densidade demográfica de 32.50887573964497 hab/km².\nO município Davinópolis/GO possui densidade demográfica de 4.271763972574278 hab/km².\nO município Diorama/GO possui densidade demográfica de 3.606605077471448 hab/km².\nO município Divinópolis de Goiás/GO possui densidade demográfica de 5.971334705224015 hab/km².\nO município Doverlândia/GO possui densidade demográfica de 2.4486959111866806 hab/km².\nO município Edealina/GO possui densidade demográfica de 6.18404704712996 hab/km².\nO município Edéia/GO possui densidade demográfica de 7.708518645227506 hab/km².\nO município Estrela do Norte/GO possui densidade demográfica de 11.006497811961278 hab/km².\nO município Faina/GO possui densidade demográfica de 3.589013496705488 hab/km².\nO município Fazenda Nova/GO possui densidade demográfica de 4.93358929937101 hab/km².\nO município Firminópolis/GO possui densidade demográfica de 27.333884102443054 hab/km².\nO município Flores de Goiás/GO possui densidade demográfica de 3.2527908600512747 hab/km².\nO município Formosa/GO possui densidade demográfica de 17.22102828904692 hab/km².\nO município Formoso/GO possui densidade demográfica de 5.783557782278601 hab/km².\nO município Gameleira de Goiás/GO possui densidade demográfica de 5.532094594594595 hab/km².\nO município Goianápolis/GO possui densidade demográfica de 65.83969465648855 hab/km².\nO município Goiandira/GO possui densidade demográfica de 9.323699729054878 hab/km².\nO município Goianésia/GO possui densidade demográfica de 38.48649556961616 hab/km².\nO município Goiânia/GO possui densidade demográfica de 1776.7480895196509 hab/km².\nO município Goianira/GO possui densidade demográfica de 162.93532338308458 hab/km².\nO município Goiás/GO possui densidade demográfica de 7.955869009851932 hab/km².\nO município Goiatuba/GO possui densidade demográfica de 13.127497363753529 hab/km².\nO município Gouvelândia/GO possui densidade demográfica de 6.004173440419285 hab/km².\nO município Guapó/GO possui densidade demográfica de 27.041250677192167 hab/km².\nO município Guaraíta/GO possui densidade demográfica de 11.572743655934927 hab/km².\nO município Guarani de Goiás/GO possui densidade demográfica de 3.464182565187324 hab/km².\nO município Guarinos/GO possui densidade demográfica de 3.8582241092855822 hab/km².\nO município Heitoraí/GO possui densidade demográfica de 15.550426754920746 hab/km².\nO município Hidrolândia/GO possui densidade demográfica de 18.432037292086026 hab/km².\nO município Hidrolina/GO possui densidade demográfica de 6.941883905649649 hab/km².\nO município Iaciara/GO possui densidade demográfica de 8.01545427572595 hab/km².\nO município Inaciolândia/GO possui densidade demográfica de 8.278617083091227 hab/km².\nO município Indiara/GO possui densidade demográfica de 14.309760789561727 hab/km².\nO município Inhumas/GO possui densidade demográfica de 78.67521158456044 hab/km².\nO município Ipameri/GO possui densidade demográfica de 5.661491557545337 hab/km².\nO município Ipiranga de Goiás/GO possui densidade demográfica de 11.786646773591944 hab/km².\nO município Iporá/GO possui densidade demográfica de 30.470196223620878 hab/km².\nO município Israelândia/GO possui densidade demográfica de 4.999307335318972 hab/km².\nO município Itaberaí/GO possui densidade demográfica de 24.27193126921388 hab/km².\nO município Itaguari/GO possui densidade demográfica de 30.77605019094381 hab/km².\nO município Itaguaru/GO possui densidade demográfica de 22.684412550066757 hab/km².\nO município Itajá/GO possui densidade demográfica de 2.420388256670173 hab/km².\nO município Itapaci/GO possui densidade demográfica de 19.304906236599624 hab/km².\nO município Itapirapuã/GO possui densidade demográfica de 3.8336954181590435 hab/km².\nO município Itapuranga/GO possui densidade demográfica de 20.466438957132112 hab/km².\nO município Itarumã/GO possui densidade demográfica de 1.8347929159519225 hab/km².\nO município Itauçu/GO possui densidade demográfica de 22.340037515631515 hab/km².\nO município Itumbiara/GO possui densidade demográfica de 37.71239945918074 hab/km².\nO município Ivolândia/GO possui densidade demográfica de 2.1174244231350285 hab/km².\nO município Jandaia/GO possui densidade demográfica de 7.133351077987756 hab/km².\nO município Jaraguá/GO possui densidade demográfica de 22.63793895812495 hab/km².\nO município Jataí/GO possui densidade demográfica de 12.266961053660115 hab/km².\nO município Jaupaci/GO possui densidade demográfica de 5.691519635742743 hab/km².\nO município Jesúpolis/GO possui densidade demográfica de 18.77857609405617 hab/km².\nO município Joviânia/GO possui densidade demográfica de 15.977911962109138 hab/km².\nO município Jussara/GO possui densidade demográfica de 4.689638623837262 hab/km².\nO município Lagoa Santa/GO possui densidade demográfica de 2.7328001394730532 hab/km².\nO município Leopoldo de Bulhões/GO possui densidade demográfica de 16.39044272078854 hab/km².\nO município Luziânia/GO possui densidade demográfica de 44.0610231449691 hab/km².\nO município Mairipotaba/GO possui densidade demográfica de 5.078835333632844 hab/km².\nO município Mambaí/GO possui densidade demográfica de 7.802457359587564 hab/km².\nO município Mara Rosa/GO possui densidade demográfica de 6.308985668667168 hab/km².\nO município Marzagão/GO possui densidade demográfica de 9.315290203659577 hab/km².\nO município Matrinchã/GO possui densidade demográfica de 3.835292686529555 hab/km².\nO município Maurilândia/GO possui densidade demográfica de 29.55921592775041 hab/km².\nO município Mimoso de Goiás/GO possui densidade demográfica de 1.9359443947740316 hab/km².\nO município Minaçu/GO possui densidade demográfica de 10.89018925173207 hab/km².\nO município Mineiros/GO possui densidade demográfica de 5.842657192147098 hab/km².\nO município Moiporá/GO possui densidade demográfica de 3.8274499587512483 hab/km².\nO município Monte Alegre de Goiás/GO possui densidade demográfica de 2.477714989053821 hab/km².\nO município Montes Claros de Goiás/GO possui densidade demográfica de 2.7549169075393736 hab/km².\nO município Montividiu/GO possui densidade demográfica de 5.640957233946056 hab/km².\nO município Montividiu do Norte/GO possui densidade demográfica de 3.0922730682670667 hab/km².\nO município Morrinhos/GO possui densidade demográfica de 14.566790808797696 hab/km².\nO município Morro Agudo de Goiás/GO possui densidade demográfica de 8.336281933338052 hab/km².\nO município Mossâmedes/GO possui densidade demográfica de 7.315362699978084 hab/km².\nO município Mozarlândia/GO possui densidade demográfica de 7.72849927350723 hab/km².\nO município Mundo Novo/GO possui densidade demográfica de 2.999091607854098 hab/km².\nO município Mutunópolis/GO possui densidade demográfica de 4.02665606561493 hab/km².\nO município Nazário/GO possui densidade demográfica de 29.26049795615013 hab/km².\nO município Nerópolis/GO possui densidade demográfica de 118.54862403290569 hab/km².\nO município Niquelândia/GO possui densidade demográfica de 4.303558275975923 hab/km².\nO município Nova América/GO possui densidade demográfica de 10.65415271423855 hab/km².\nO município Nova Aurora/GO possui densidade demográfica de 6.812925394832485 hab/km².\nO município Nova Crixás/GO possui densidade demográfica de 1.634108714059062 hab/km².\nO município Nova Glória/GO possui densidade demográfica de 20.602978568834 hab/km².\nO município Nova Iguaçu de Goiás/GO possui densidade demográfica de 4.496849341225892 hab/km².\nO município Nova Roma/GO possui densidade demográfica de 1.6250304312814847 hab/km².\nO município Nova Veneza/GO possui densidade demográfica de 65.8858810179932 hab/km².\nO município Novo Brasil/GO possui densidade demográfica de 5.414262635587352 hab/km².\nO município Novo Gama/GO possui densidade demográfica de 487.2967844504846 hab/km².\nO município Novo Planalto/GO possui densidade demográfica de 3.18272510780717 hab/km².\nO município Orizona/GO possui densidade demográfica de 7.248286768581971 hab/km².\nO município Ouro Verde de Goiás/GO possui densidade demográfica de 19.32269962159314 hab/km².\nO município Ouvidor/GO possui densidade demográfica de 13.212335057276814 hab/km².\nO município Padre Bernardo/GO possui densidade demográfica de 8.814722316018834 hab/km².\nO município Palestina de Goiás/GO possui densidade demográfica de 2.5524536416570127 hab/km².\nO município Palmeiras de Goiás/GO possui densidade demográfica de 15.157596659067734 hab/km².\nO município Palmelo/GO possui densidade demográfica de 39.603120759837175 hab/km².\nO município Palminópolis/GO possui densidade demográfica de 9.174856199540871 hab/km².\nO município Panamá/GO possui densidade demográfica de 6.183142751752121 hab/km².\nO município Paranaiguara/GO possui densidade demográfica de 7.886777081545809 hab/km².\nO município Paraúna/GO possui densidade demográfica de 2.874273361574222 hab/km².\nO município Perolândia/GO possui densidade demográfica de 2.8651347098929705 hab/km².\nO município Petrolina de Goiás/GO possui densidade demográfica de 19.354413702239793 hab/km².\nO município Pilar de Goiás/GO possui densidade demográfica de 3.05851210500193 hab/km².\nO município Piracanjuba/GO possui densidade demográfica de 9.989522352315062 hab/km².\nO município Piranhas/GO possui densidade demográfica de 5.501594417341791 hab/km².\nO município Pirenópolis/GO possui densidade demográfica de 10.4335127731847 hab/km².\nO município Pires do Rio/GO possui densidade demográfica de 26.796228665126335 hab/km².\nO município Planaltina/GO possui densidade demográfica de 32.09637284924151 hab/km².\nO município Pontalina/GO possui densidade demográfica de 11.914819583144855 hab/km².\nO município Porangatu/GO possui densidade demográfica de 8.786396488345655 hab/km².\nO município Porteirão/GO possui densidade demográfica de 5.541941252442295 hab/km².\nO município Portelândia/GO possui densidade demográfica de 6.897481044953106 hab/km².\nO município Posse/GO possui densidade demográfica de 15.519080877631461 hab/km².\nO município Professor Jamil/GO possui densidade demográfica de 9.321668057674042 hab/km².\nO município Quirinópolis/GO possui densidade demográfica de 11.413662063702072 hab/km².\nO município Rialma/GO possui densidade demográfica de 39.19618579357097 hab/km².\nO município Rianápolis/GO possui densidade demográfica de 28.67009920884089 hab/km².\nO município Rio Quente/GO possui densidade demográfica de 12.939521800281293 hab/km².\nO município Rio Verde/GO possui densidade demográfica de 21.05383750653368 hab/km².\nO município Rubiataba/GO possui densidade demográfica de 25.278646459786707 hab/km².\nO município Sanclerlândia/GO possui densidade demográfica de 15.196344826198096 hab/km².\nO município Santa Bárbara de Goiás/GO possui densidade demográfica de 41.19627507163324 hab/km².\nO município Santa Cruz de Goiás/GO possui densidade demográfica de 2.8332852402250754 hab/km².\nO município Santa Fé de Goiás/GO possui densidade demográfica de 4.072974845403149 hab/km².\nO município Santa Helena de Goiás/GO possui densidade demográfica de 31.953072292851324 hab/km².\nO município Santa Isabel/GO possui densidade demográfica de 4.566402378592666 hab/km².\nO município Santa Rita do Araguaia/GO possui densidade demográfica de 5.084559066509029 hab/km².\nO município Santa Rita do Novo Destino/GO possui densidade demográfica de 3.3188987908455716 hab/km².\nO município Santa Rosa de Goiás/GO possui densidade demográfica de 17.72699573430835 hab/km².\nO município Santa Tereza de Goiás/GO possui densidade demográfica de 5.027939991945228 hab/km².\nO município Santa Terezinha de Goiás/GO possui densidade demográfica de 8.569004524886878 hab/km².\nO município Santo Antônio da Barra/GO possui densidade demográfica de 9.794065544729849 hab/km².\nO município Santo Antônio de Goiás/GO possui densidade demográfica de 35.41149009863715 hab/km².\nO município Santo Antônio do Descoberto/GO possui densidade demográfica de 66.9900650327282 hab/km².\nO município São Domingos/GO possui densidade demográfica de 3.420172707798552 hab/km².\nO município São Francisco de Goiás/GO possui densidade demográfica de 14.71896871016619 hab/km².\nO município São João da Paraúna/GO possui densidade demográfica de 5.868047111141994 hab/km².\nO município São João d`Aliança/GO possui densidade demográfica de 3.0826055334828 hab/km².\nO município São Luís de Montes Belos/GO possui densidade demográfica de 36.360774818401936 hab/km².\nO município São Luíz do Norte/GO possui densidade demográfica de 7.878032965907928 hab/km².\nO município São Miguel do Araguaia/GO possui densidade demográfica de 3.626548358589352 hab/km².\nO município São Miguel do Passa Quatro/GO possui densidade demográfica de 6.9859982521058415 hab/km².\nO município São Patrício/GO possui densidade demográfica de 11.578274017213305 hab/km².\nO município São Simão/GO possui densidade demográfica de 41.27436535349388 hab/km².\nO município Senador Canedo/GO possui densidade demográfica de 344.2718525766471 hab/km².\nO município Serranópolis/GO possui densidade demográfica de 1.3536057553123733 hab/km².\nO município Silvânia/GO possui densidade demográfica de 8.13703675285813 hab/km².\nO município Simolândia/GO possui densidade demográfica de 18.719466636013564 hab/km².\nO município Sítio d`Abadia/GO possui densidade demográfica de 1.7674476804204338 hab/km².\nO município Taquaral de Goiás/GO possui densidade demográfica de 17.339144060327097 hab/km².\nO município Teresina de Goiás/GO possui densidade demográfica de 3.8934214602912323 hab/km².\nO município Terezópolis de Goiás/GO possui densidade demográfica de 61.36937611074736 hab/km².\nO município Três Ranchos/GO possui densidade demográfica de 9.99397312723792 hab/km².\nO município Trindade/GO possui densidade demográfica de 147.0191780050935 hab/km².\nO município Trombas/GO possui densidade demográfica de 4.319697671217449 hab/km².\nO município Turvânia/GO possui densidade demográfica de 10.064894546362162 hab/km².\nO município Turvelândia/GO possui densidade demográfica de 4.710051822347852 hab/km².\nO município Uirapuru/GO possui densidade demográfica de 2.5427402295661823 hab/km².\nO município Uruaçu/GO possui densidade demográfica de 17.241878402480133 hab/km².\nO município Uruana/GO possui densidade demográfica de 26.46073759353888 hab/km².\nO município Urutaí/GO possui densidade demográfica de 4.904901710492724 hab/km².\nO município Valparaíso de Goiás/GO possui densidade demográfica de 2165.4779351897087 hab/km².\nO município Varjão/GO possui densidade demográfica de 7.047516323503919 hab/km².\nO município Vianópolis/GO possui densidade demográfica de 13.149180534015175 hab/km².\nO município Vicentinópolis/GO possui densidade demográfica de 9.99782980224073 hab/km².\nO município Vila Boa/GO possui densidade demográfica de 4.46626484431742 hab/km².\nO município Vila Propício/GO possui densidade demográfica de 2.358382456751529 hab/km².\nO município Açailândia/MA possui densidade demográfica de 17.919241394038345 hab/km².\nO município Afonso Cunha/MA possui densidade demográfica de 15.90186890720095 hab/km².\nO município Água Doce do Maranhão/MA possui densidade demográfica de 26.12628871793715 hab/km².\nO município Alcântara/MA possui densidade demográfica de 14.697850243495573 hab/km².\nO município Aldeias Altas/MA possui densidade demográfica de 12.332978049647034 hab/km².\nO município Altamira do Maranhão/MA possui densidade demográfica de 15.337372280988758 hab/km².\nO município Alto Alegre do Maranhão/MA possui densidade demográfica de 64.175210664997 hab/km².\nO município Alto Alegre do Pindaré/MA possui densidade demográfica de 16.072639200120065 hab/km².\nO município Alto Parnaíba/MA possui densidade demográfica de 0.9671061732742373 hab/km².\nO município Amapá do Maranhão/MA possui densidade demográfica de 12.800557324840765 hab/km².\nO município Amarante do Maranhão/MA possui densidade demográfica de 5.099655156188031 hab/km².\nO município Anajatuba/MA possui densidade demográfica de 25.012609654544914 hab/km².\nO município Anapurus/MA possui densidade demográfica de 22.91468025645241 hab/km².\nO município Apicum-Açu/MA possui densidade demográfica de 42.356372285301696 hab/km².\nO município Araguanã/MA possui densidade demográfica de 17.35345255837059 hab/km².\nO município Araioses/MA possui densidade demográfica de 23.84438460675418 hab/km².\nO município Arame/MA possui densidade demográfica de 10.536811702102908 hab/km².\nO município Arari/MA possui densidade demográfica de 25.891591231322938 hab/km².\nO município Axixá/MA possui densidade demográfica de 56.15062761506276 hab/km².\nO município Bacabal/MA possui densidade demográfica de 59.427437372248896 hab/km².\nO município Bacabeira/MA possui densidade demográfica de 24.24503322016277 hab/km².\nO município Bacuri/MA possui densidade demográfica de 21.074810245475085 hab/km².\nO município Bacurituba/MA possui densidade demográfica de 7.8471779514017586 hab/km².\nO município Balsas/MA possui densidade demográfica de 6.355936394979961 hab/km².\nO município Barão de Grajaú/MA possui densidade demográfica de 7.939071928231965 hab/km².\nO município Barra do Corda/MA possui densidade demográfica de 15.920579699002442 hab/km².\nO município Barreirinhas/MA possui densidade demográfica de 17.651084997059762 hab/km².\nO município Bela Vista do Maranhão/MA possui densidade demográfica de 47.1492858540403 hab/km².\nO município Belágua/MA possui densidade demográfica de 13.062891696534049 hab/km².\nO município Benedito Leite/MA possui densidade demográfica de 3.0694886430603963 hab/km².\nO município Bequimão/MA possui densidade demográfica de 26.45685675271474 hab/km².\nO município Bernardo do Mearim/MA possui densidade demográfica de 22.933639319181488 hab/km².\nO município Boa Vista do Gurupi/MA possui densidade demográfica de 19.702077033658853 hab/km².\nO município Bom Jardim/MA possui densidade demográfica de 5.925016652681954 hab/km².\nO município Bom Jesus das Selvas/MA possui densidade demográfica de 10.622597140830877 hab/km².\nO município Bom Lugar/MA possui densidade demográfica de 33.22570518857348 hab/km².\nO município Brejo/MA possui densidade demográfica de 31.042312237700415 hab/km².\nO município Brejo de Areia/MA possui densidade demográfica de 15.386525409700381 hab/km².\nO município Buriti/MA possui densidade demográfica de 18.326820266492984 hab/km².\nO município Buriti Bravo/MA possui densidade demográfica de 14.469685002053648 hab/km².\nO município Buriticupu/MA possui densidade demográfica de 25.628967879816457 hab/km².\nO município Buritirana/MA possui densidade demográfica de 18.064074680481905 hab/km².\nO município Cachoeira Grande/MA possui densidade demográfica de 11.969106497555446 hab/km².\nO município Cajapió/MA possui densidade demográfica de 11.656927800336733 hab/km².\nO município Cajari/MA possui densidade demográfica de 27.6979775552434 hab/km².\nO município Campestre do Maranhão/MA possui densidade demográfica de 21.724787935909518 hab/km².\nO município Cândido Mendes/MA possui densidade demográfica de 11.33252904324182 hab/km².\nO município Cantanhede/MA possui densidade demográfica de 26.452439166375598 hab/km².\nO município Capinzal do Norte/MA possui densidade demográfica de 18.115929758013987 hab/km².\nO município Carolina/MA possui densidade demográfica de 3.7194175360158965 hab/km².\nO município Carutapera/MA possui densidade demográfica de 17.86085319135121 hab/km².\nO município Caxias/MA possui densidade demográfica de 30.118217629939405 hab/km².\nO município Cedral/MA possui densidade demográfica de 36.36074720152548 hab/km².\nO município Central do Maranhão/MA possui densidade demográfica de 24.697814241873868 hab/km².\nO município Centro do Guilherme/MA possui densidade demográfica de 11.698492649454877 hab/km².\nO município Centro Novo do Maranhão/MA possui densidade demográfica de 2.1338222081221345 hab/km².\nO município Chapadinha/MA possui densidade demográfica de 22.58743972063633 hab/km².\nO município Cidelândia/MA possui densidade demográfica de 9.34475386433338 hab/km².\nO município Codó/MA possui densidade demográfica de 27.064617755093618 hab/km².\nO município Coelho Neto/MA possui densidade demográfica de 47.9216852032187 hab/km².\nO município Colinas/MA possui densidade demográfica de 19.75814798919492 hab/km².\nO município Conceição do Lago-Açu/MA possui densidade demográfica de 19.688228795875784 hab/km².\nO município Coroatá/MA possui densidade demográfica de 27.2663421357199 hab/km².\nO município Cururupu/MA possui densidade demográfica de 26.690208195394693 hab/km².\nO município Davinópolis/MA possui densidade demográfica de 37.46202870927393 hab/km².\nO município Dom Pedro/MA possui densidade demográfica de 63.268152528661886 hab/km².\nO município Duque Bacelar/MA possui densidade demográfica de 33.49584801207851 hab/km².\nO município Esperantinópolis/MA possui densidade demográfica de 38.36812775513599 hab/km².\nO município Estreito/MA possui densidade demográfica de 13.17957469345122 hab/km².\nO município Feira Nova do Maranhão/MA possui densidade demográfica de 5.515060200078728 hab/km².\nO município Fernando Falcão/MA possui densidade demográfica de 1.816741307519001 hab/km².\nO município Formosa da Serra Negra/MA possui densidade demográfica de 4.494839932869767 hab/km².\nO município Fortaleza dos Nogueiras/MA possui densidade demográfica de 6.997410369337812 hab/km².\nO município Fortuna/MA possui densidade demográfica de 21.723741007194246 hab/km².\nO município Godofredo Viana/MA possui densidade demográfica de 15.751588488825037 hab/km².\nO município Gonçalves Dias/MA possui densidade demográfica de 19.78519449065743 hab/km².\nO município Governador Archer/MA possui densidade demográfica de 22.888350603328398 hab/km².\nO município Governador Edison Lobão/MA possui densidade demográfica de 25.809856296176015 hab/km².\nO município Governador Eugênio Barros/MA possui densidade demográfica de 19.573066989804037 hab/km².\nO município Governador Luiz Rocha/MA possui densidade demográfica de 19.661807267659984 hab/km².\nO município Governador Newton Bello/MA possui densidade demográfica de 10.272384940843954 hab/km².\nO município Governador Nunes Freire/MA possui densidade demográfica de 24.491625929247054 hab/km².\nO município Graça Aranha/MA possui densidade demográfica de 22.620100206307104 hab/km².\nO município Grajaú/MA possui densidade demográfica de 7.031285386866208 hab/km².\nO município Guimarães/MA possui densidade demográfica de 20.291242567771842 hab/km².\nO município Humberto de Campos/MA possui densidade demográfica de 12.28809384164223 hab/km².\nO município Icatu/MA possui densidade demográfica de 17.355982274741507 hab/km².\nO município Igarapé do Meio/MA possui densidade demográfica de 34.03943692533022 hab/km².\nO município Igarapé Grande/MA possui densidade demográfica de 29.501670006680026 hab/km².\nO município Imperatriz/MA possui densidade demográfica de 180.79386993330849 hab/km².\nO município Itaipava do Grajaú/MA possui densidade demográfica de 11.540821103953762 hab/km².\nO município Itapecuru Mirim/MA possui densidade demográfica de 42.21035176425814 hab/km².\nO município Itinga do Maranhão/MA possui densidade demográfica de 6.941636978881655 hab/km².\nO município Jatobá/MA possui densidade demográfica de 14.417126044167878 hab/km².\nO município Jenipapo dos Vieiras/MA possui densidade demográfica de 7.865912680218044 hab/km².\nO município João Lisboa/MA possui densidade demográfica de 32.000816467521865 hab/km².\nO município Joselândia/MA possui densidade demográfica de 22.639322859364224 hab/km².\nO município Junco do Maranhão/MA possui densidade demográfica de 7.2420688536994 hab/km².\nO município Lago da Pedra/MA possui densidade demográfica de 37.15022773993309 hab/km².\nO município Lago do Junco/MA possui densidade demográfica de 34.71943563523397 hab/km².\nO município Lago dos Rodrigues/MA possui densidade demográfica de 43.211177025004154 hab/km².\nO município Lago Verde/MA possui densidade demográfica de 24.728836403311725 hab/km².\nO município Lagoa do Mato/MA possui densidade demográfica de 6.477296288617043 hab/km².\nO município Lagoa Grande do Maranhão/MA possui densidade demográfica de 14.130055085315062 hab/km².\nO município Lajeado Novo/MA possui densidade demográfica de 6.607618374963015 hab/km².\nO município Lima Campos/MA possui densidade demográfica de 35.48286894666543 hab/km².\nO município Loreto/MA possui densidade demográfica de 3.166668520145461 hab/km².\nO município Luís Domingues/MA possui densidade demográfica de 14.028358401930785 hab/km².\nO município Magalhães de Almeida/MA possui densidade demográfica de 40.602562622648044 hab/km².\nO município Maracaçumé/MA possui densidade demográfica de 30.438098870191165 hab/km².\nO município Marajá do Sena/MA possui densidade demográfica de 5.561311892130857 hab/km².\nO município Maranhãozinho/MA possui densidade demográfica de 14.460940552322592 hab/km².\nO município Mata Roma/MA possui densidade demográfica de 27.625316825003193 hab/km².\nO município Matinha/MA possui densidade demográfica de 53.543904288894865 hab/km².\nO município Matões/MA possui densidade demográfica de 15.69473822704869 hab/km².\nO município Matões do Norte/MA possui densidade demográfica de 17.35858554080413 hab/km².\nO município Milagres do Maranhão/MA possui densidade demográfica de 12.789488609509405 hab/km².\nO município Mirador/MA possui densidade demográfica de 2.4201115864084675 hab/km².\nO município Miranda do Norte/MA possui densidade demográfica de 71.61033097827679 hab/km².\nO município Mirinzal/MA possui densidade demográfica de 20.673209741912032 hab/km².\nO município Monção/MA possui densidade demográfica de 24.37690576587786 hab/km².\nO município Montes Altos/MA possui densidade demográfica de 6.324495746939544 hab/km².\nO município Morros/MA possui densidade demográfica de 10.368008022481606 hab/km².\nO município Nina Rodrigues/MA possui densidade demográfica de 21.77079876334038 hab/km².\nO município Nova Colinas/MA possui densidade demográfica de 6.573723943965227 hab/km².\nO município Nova Iorque/MA possui densidade demográfica de 4.698776680145365 hab/km².\nO município Nova Olinda do Maranhão/MA possui densidade demográfica de 7.801453139907528 hab/km².\nO município Olho d`Água das Cunhãs/MA possui densidade demográfica de 26.751326708181725 hab/km².\nO município Olinda Nova do Maranhão/MA possui densidade demográfica de 66.69196518923296 hab/km².\nO município Paço do Lumiar/MA possui densidade demográfica de 855.8251241553367 hab/km².\nO município Palmeirândia/MA possui densidade demográfica de 35.70151071197534 hab/km².\nO município Paraibano/MA possui densidade demográfica de 37.893010631078944 hab/km².\nO município Parnarama/MA possui densidade demográfica de 10.056320746213542 hab/km².\nO município Passagem Franca/MA possui densidade demográfica de 12.929111482482167 hab/km².\nO município Pastos Bons/MA possui densidade demográfica de 11.04805816634155 hab/km².\nO município Paulino Neves/MA possui densidade demográfica de 14.82771298433383 hab/km².\nO município Paulo Ramos/MA possui densidade demográfica de 19.0609544242033 hab/km².\nO município Pedreiras/MA possui densidade demográfica de 136.76801997018342 hab/km².\nO município Pedro do Rosário/MA possui densidade demográfica de 12.990530833366668 hab/km².\nO município Penalva/MA possui densidade demográfica de 46.41652556721978 hab/km².\nO município Peri Mirim/MA possui densidade demográfica de 34.056254626202815 hab/km².\nO município Peritoró/MA possui densidade demográfica de 25.706906586477835 hab/km².\nO município Pindaré-Mirim/MA possui densidade demográfica de 113.88878733594123 hab/km².\nO município Pinheiro/MA possui densidade demográfica de 51.671206071343576 hab/km².\nO município Pio XII/MA possui densidade demográfica de 40.38595590123638 hab/km².\nO município Pirapemas/MA possui densidade demográfica de 25.235205296474824 hab/km².\nO município Poção de Pedras/MA possui densidade demográfica de 20.4877643096243 hab/km².\nO município Porto Franco/MA possui densidade demográfica de 15.18881967421287 hab/km².\nO município Porto Rico do Maranhão/MA possui densidade demográfica de 27.555636795686148 hab/km².\nO município Presidente Dutra/MA possui densidade demográfica de 57.97400106276812 hab/km².\nO município Presidente Juscelino/MA possui densidade demográfica de 32.53735551170003 hab/km².\nO município Presidente Médici/MA possui densidade demográfica de 14.56281843313761 hab/km².\nO município Presidente Sarney/MA possui densidade demográfica de 23.70365255817165 hab/km².\nO município Presidente Vargas/MA possui densidade demográfica de 23.33028561476837 hab/km².\nO município Primeira Cruz/MA possui densidade demográfica de 10.202678989237247 hab/km².\nO município Raposa/MA possui densidade demográfica de 397.2088111044055 hab/km².\nO município Riachão/MA possui densidade demográfica de 3.1710240984650917 hab/km².\nO município Ribamar Fiquene/MA possui densidade demográfica de 9.75018319898741 hab/km².\nO município Rosário/MA possui densidade demográfica de 57.77180894546304 hab/km².\nO município Sambaíba/MA possui densidade demográfica de 2.21366038649292 hab/km².\nO município Santa Filomena do Maranhão/MA possui densidade demográfica de 11.722615134309526 hab/km².\nO município Santa Helena/MA possui densidade demográfica de 16.944012407990677 hab/km².\nO município Santa Inês/MA possui densidade demográfica de 202.75474866197922 hab/km².\nO município Santa Luzia/MA possui densidade demográfica de 13.553641249432541 hab/km².\nO município Santa Luzia do Paruá/MA possui densidade demográfica de 25.23992643370674 hab/km².\nO município Santa Quitéria do Maranhão/MA possui densidade demográfica de 15.2227535604587 hab/km².\nO município Santa Rita/MA possui densidade demográfica de 45.81888192075199 hab/km².\nO município Santana do Maranhão/MA possui densidade demográfica de 12.511534087251347 hab/km².\nO município Santo Amaro do Maranhão/MA possui densidade demográfica de 8.631134538278019 hab/km².\nO município Santo Antônio dos Lopes/MA possui densidade demográfica de 18.521687278006794 hab/km².\nO município São Benedito do Rio Preto/MA possui densidade demográfica de 19.108300768669213 hab/km².\nO município São Bento/MA possui densidade demográfica de 88.73592262617902 hab/km².\nO município São Bernardo/MA possui densidade demográfica de 26.29404520716641 hab/km².\nO município São Domingos do Azeitão/MA possui densidade demográfica de 7.266918506030617 hab/km².\nO município São Domingos do Maranhão/MA possui densidade demográfica de 29.173249535582215 hab/km².\nO município São Félix de Balsas/MA possui densidade demográfica de 2.3135664941250567 hab/km².\nO município São Francisco do Brejão/MA possui densidade demográfica de 13.761886240796127 hab/km².\nO município São Francisco do Maranhão/MA possui densidade demográfica de 5.174676209952284 hab/km².\nO município São João Batista/MA possui densidade demográfica de 28.84114206289454 hab/km².\nO município São João do Carú/MA possui densidade demográfica de 19.99187916192951 hab/km².\nO município São João do Paraíso/MA possui densidade demográfica de 5.265259221750477 hab/km².\nO município São João do Soter/MA possui densidade demográfica de 11.986899107832025 hab/km².\nO município São João dos Patos/MA possui densidade demográfica de 16.61168975696874 hab/km².\nO município São José de Ribamar/MA possui densidade demográfica de 419.8187295620156 hab/km².\nO município São José dos Basílios/MA possui densidade demográfica de 20.667787918056742 hab/km².\nO município São Luís/MA possui densidade demográfica de 1215.6793924220462 hab/km².\nO município São Luís Gonzaga do Maranhão/MA possui densidade demográfica de 20.806962842128083 hab/km².\nO município São Mateus do Maranhão/MA possui densidade demográfica de 49.905532718870475 hab/km².\nO município São Pedro da Água Branca/MA possui densidade demográfica de 16.695121104865013 hab/km².\nO município São Pedro dos Crentes/MA possui densidade demográfica de 4.517011524759348 hab/km².\nO município São Raimundo das Mangabeiras/MA possui densidade demográfica de 4.962047746292094 hab/km².\nO município São Raimundo do Doca Bezerra/MA possui densidade demográfica de 14.522475259329914 hab/km².\nO município São Roberto/MA possui densidade demográfica de 26.189220082651893 hab/km².\nO município São Vicente Ferrer/MA possui densidade demográfica de 53.37853396443648 hab/km².\nO município Satubinha/MA possui densidade demográfica de 27.13836264457572 hab/km².\nO município Senador Alexandre Costa/MA possui densidade demográfica de 24.050276709501922 hab/km².\nO município Senador La Rocque/MA possui densidade demográfica de 14.551246291041098 hab/km².\nO município Serrano do Maranhão/MA possui densidade demográfica de 9.063343992842112 hab/km².\nO município Sítio Novo/MA possui densidade demográfica de 5.4583337346341905 hab/km².\nO município Sucupira do Norte/MA possui densidade demográfica de 9.720141092817855 hab/km².\nO município Sucupira do Riachão/MA possui densidade demográfica de 8.165035311609465 hab/km².\nO município Tasso Fragoso/MA possui densidade demográfica de 1.7786985110586864 hab/km².\nO município Timbiras/MA possui densidade demográfica de 18.833033990542113 hab/km².\nO município Timon/MA possui densidade demográfica de 89.17825899899613 hab/km².\nO município Trizidela do Vale/MA possui densidade demográfica de 85.01009194886747 hab/km².\nO município Tufilândia/MA possui densidade demográfica de 20.648684550385596 hab/km².\nO município Tuntum/MA possui densidade demográfica de 11.558407079646019 hab/km².\nO município Turiaçu/MA possui densidade demográfica de 13.159976730657359 hab/km².\nO município Turilândia/MA possui densidade demográfica de 15.111187543820195 hab/km².\nO município Tutóia/MA possui densidade demográfica de 31.960572999285567 hab/km².\nO município Urbano Santos/MA possui densidade demográfica de 20.348119871152587 hab/km².\nO município Vargem Grande/MA possui densidade demográfica de 25.239177627378368 hab/km².\nO município Viana/MA possui densidade demográfica de 42.360754510287215 hab/km².\nO município Vila Nova dos Martírios/MA possui densidade demográfica de 9.470213159709955 hab/km².\nO município Vitória do Mearim/MA possui densidade demográfica de 43.555363321799305 hab/km².\nO município Vitorino Freire/MA possui densidade demográfica de 24.253242524764232 hab/km².\nO município Zé Doca/MA possui densidade demográfica de 20.766454475468326 hab/km².\nO município Acorizal/MT possui densidade demográfica de 6.5620576023983155 hab/km².\nO município Água Boa/MT possui densidade demográfica de 2.787817866843467 hab/km².\nO município Alta Floresta/MT possui densidade demográfica de 5.4771628911184935 hab/km².\nO município Alto Araguaia/MT possui densidade demográfica de 2.8368794326241136 hab/km².\nO município Alto Boa Vista/MT possui densidade demográfica de 2.341940235220603 hab/km².\nO município Alto Garças/MT possui densidade demográfica de 2.7614359466922798 hab/km².\nO município Alto Paraguai/MT possui densidade demográfica de 5.451985051183448 hab/km².\nO município Alto Taquari/MT possui densidade demográfica de 5.69847231242764 hab/km².\nO município Apiacás/MT possui densidade demográfica de 0.42041446673317795 hab/km².\nO município Araguaiana/MT possui densidade demográfica de 0.49724856829118824 hab/km².\nO município Araguainha/MT possui densidade demográfica de 1.593092722066369 hab/km².\nO município Araputanga/MT possui densidade demográfica de 9.587311903214518 hab/km².\nO município Arenápolis/MT possui densidade demográfica de 24.75107368219007 hab/km².\nO município Aripuanã/MT possui densidade demográfica de 0.744548980355816 hab/km².\nO município Barão de Melgaço/MT possui densidade demográfica de 0.6793145107163632 hab/km².\nO município Barra do Bugres/MT possui densidade demográfica de 5.246196495165176 hab/km².\nO município Barra do Garças/MT possui densidade demográfica de 6.229774710374954 hab/km².\nO município Bom Jesus do Araguaia/MT possui densidade demográfica de 1.2432706862788678 hab/km².\nO município Brasnorte/MT possui densidade demográfica de 0.9622698967488224 hab/km².\nO município Cáceres/MT possui densidade demográfica de 3.611371990369346 hab/km².\nO município Campinápolis/MT possui densidade demográfica de 2.3972114925385637 hab/km².\nO município Campo Novo do Parecis/MT possui densidade demográfica de 2.9230201750611062 hab/km².\nO município Campo Verde/MT possui densidade demográfica de 6.6056477043654285 hab/km².\nO município Campos de Júlio/MT possui densidade demográfica de 0.7577339139588289 hab/km².\nO município Canabrava do Norte/MT possui densidade demográfica de 1.3861695842070507 hab/km².\nO município Canarana/MT possui densidade demográfica de 1.723333088289348 hab/km².\nO município Carlinda/MT possui densidade demográfica de 4.59252325513368 hab/km².\nO município Castanheira/MT possui densidade demográfica de 2.1053627792528022 hab/km².\nO município Chapada dos Guimarães/MT possui densidade demográfica de 2.848174601525654 hab/km².\nO município Cláudia/MT possui densidade demográfica de 2.8644230244753883 hab/km².\nO município Cocalinho/MT possui densidade demográfica de 0.33211015168178404 hab/km².\nO município Colíder/MT possui densidade demográfica de 9.946430361085875 hab/km².\nO município Colniza/MT possui densidade demográfica de 0.943971105130707 hab/km².\nO município Comodoro/MT possui densidade demográfica de 0.8350130364561418 hab/km².\nO município Confresa/MT possui densidade demográfica de 4.33068626656715 hab/km².\nO município Conquista d`Oeste/MT possui densidade demográfica de 1.266741760565225 hab/km².\nO município Cotriguaçu/MT possui densidade demográfica de 1.5837479533257863 hab/km².\nO município Cuiabá/MT possui densidade demográfica de 157.662884574672 hab/km².\nO município Curvelândia/MT possui densidade demográfica de 13.525683789192795 hab/km².\nO município Denise/MT possui densidade demográfica de 6.5200927179675485 hab/km².\nO município Diamantino/MT possui densidade demográfica de 2.4715374053778203 hab/km².\nO município Dom Aquino/MT possui densidade demográfica de 3.7070811556329852 hab/km².\nO município Feliz Natal/MT possui densidade demográfica de 0.9538092172186425 hab/km².\nO município Figueirópolis d`Oeste/MT possui densidade demográfica de 4.221295524047818 hab/km².\nO município Gaúcha do Norte/MT possui densidade demográfica de 0.3716923193234527 hab/km².\nO município General Carneiro/MT possui densidade demográfica de 1.3246586243787781 hab/km².\nO município Glória d`Oeste/MT possui densidade demográfica de 3.6716480839501546 hab/km².\nO município Guarantã do Norte/MT possui densidade demográfica de 6.8043906652952 hab/km².\nO município Guiratinga/MT possui densidade demográfica de 2.752835515410861 hab/km².\nO município Indiavaí/MT possui densidade demográfica de 3.973213545724279 hab/km².\nO município Ipiranga do Norte/MT possui densidade demográfica de 1.4776250703047258 hab/km².\nO município Itanhangá/MT possui densidade demográfica de 1.8205156517418428 hab/km².\nO município Itaúba/MT possui densidade demográfica de 1.0100274197607726 hab/km².\nO município Itiquira/MT possui densidade demográfica de 1.3159101539929012 hab/km².\nO município Jaciara/MT possui densidade demográfica de 15.510359592147756 hab/km².\nO município Jangada/MT possui densidade demográfica de 7.556284303233218 hab/km².\nO município Jauru/MT possui densidade demográfica de 8.030632388297013 hab/km².\nO município Juara/MT possui densidade demográfica de 1.4482895996191014 hab/km².\nO município Juína/MT possui densidade demográfica de 1.4988568138324763 hab/km².\nO município Juruena/MT possui densidade demográfica de 4.030644557676253 hab/km².\nO município Juscimeira/MT possui densidade demográfica de 5.181018344340542 hab/km².\nO município Lambari d`Oeste/MT possui densidade demográfica de 3.0789901864628746 hab/km².\nO município Lucas do Rio Verde/MT possui densidade demográfica de 12.433440047598383 hab/km².\nO município Luciára/MT possui densidade demográfica de 0.5241524944379502 hab/km².\nO município Marcelândia/MT possui densidade demográfica de 0.977587786259542 hab/km².\nO município Matupá/MT possui densidade demográfica de 2.7051321934396633 hab/km².\nO município Mirassol d`Oeste/MT possui densidade demográfica de 23.504217919655137 hab/km².\nO município Nobres/MT possui densidade demográfica de 3.8545140619620457 hab/km².\nO município Nortelândia/MT possui densidade demográfica de 4.771365873910207 hab/km².\nO município Nossa Senhora do Livramento/MT possui densidade demográfica de 2.2866856550805825 hab/km².\nO município Nova Bandeirantes/MT possui densidade demográfica de 1.212022160549475 hab/km².\nO município Nova Brasilândia/MT possui densidade demográfica de 1.3976745036381586 hab/km².\nO município Nova Canaã do Norte/MT possui densidade demográfica de 2.0326170761959035 hab/km².\nO município Nova Guarita/MT possui densidade demográfica de 4.4267724592282764 hab/km².\nO município Nova Lacerda/MT possui densidade demográfica de 1.1480246415590833 hab/km².\nO município Nova Marilândia/MT possui densidade demográfica de 1.5212908547272914 hab/km².\nO município Nova Maringá/MT possui densidade demográfica de 0.5702023829095031 hab/km².\nO município Nova Monte verde/MT possui densidade demográfica de 1.5419526192045787 hab/km².\nO município Nova Mutum/MT possui densidade demográfica de 3.309643969355807 hab/km².\nO município Nova Nazaré/MT possui densidade demográfica de 0.7501126778700663 hab/km².\nO município Nova Olímpia/MT possui densidade demográfica de 11.301312410473475 hab/km².\nO município Nova Santa Helena/MT possui densidade demográfica de 1.4696036138349533 hab/km².\nO município Nova Ubiratã/MT possui densidade demográfica de 0.7254417734210349 hab/km².\nO município Nova Xavantina/MT possui densidade demográfica de 3.5242425112313587 hab/km².\nO município Novo Horizonte do Norte/MT possui densidade demográfica de 4.261873905827252 hab/km².\nO município Novo Mundo/MT possui densidade demográfica de 1.2661463010292187 hab/km².\nO município Novo Santo Antônio/MT possui densidade demográfica de 0.4563248213391597 hab/km².\nO município Novo São Joaquim/MT possui densidade demográfica de 1.1999642513132678 hab/km².\nO município Paranaíta/MT possui densidade demográfica de 2.227685096569857 hab/km².\nO município Paranatinga/MT possui densidade demográfica de 0.798226274182656 hab/km².\nO município Pedra Preta/MT possui densidade demográfica de 3.8346488698069168 hab/km².\nO município Peixoto de Azevedo/MT possui densidade demográfica de 2.1611172833271612 hab/km².\nO município Planalto da Serra/MT possui densidade demográfica de 1.1101925121058227 hab/km².\nO município Poconé/MT possui densidade demográfica de 1.8400219095720627 hab/km².\nO município Pontal do Araguaia/MT possui densidade demográfica de 1.9698551909974513 hab/km².\nO município Ponte Branca/MT possui densidade demográfica de 2.5772970451464308 hab/km².\nO município Pontes e Lacerda/MT possui densidade demográfica de 4.837987926060851 hab/km².\nO município Porto Alegre do Norte/MT possui densidade demográfica de 2.705771288312669 hab/km².\nO município Porto dos Gaúchos/MT possui densidade demográfica de 0.7792412086890614 hab/km².\nO município Porto Esperidião/MT possui densidade demográfica de 1.8989433673838272 hab/km².\nO município Porto Estrela/MT possui densidade demográfica de 1.7689891213713664 hab/km².\nO município Poxoréo/MT possui densidade demográfica de 2.547002832254414 hab/km².\nO município Primavera do Leste/MT possui densidade demográfica de 9.51561140718322 hab/km².\nO município Querência/MT possui densidade demográfica de 0.7327591053738292 hab/km².\nO município Reserva do Cabaçal/MT possui densidade demográfica de 1.9236522467540238 hab/km².\nO município Ribeirão Cascalheira/MT possui densidade demográfica de 0.782135500285782 hab/km².\nO município Ribeirãozinho/MT possui densidade demográfica de 3.5151379519805617 hab/km².\nO município Rio Branco/MT possui densidade demográfica de 9.007888565133962 hab/km².\nO município Rondolândia/MT possui densidade demográfica de 0.28444045968230114 hab/km².\nO município Rondonópolis/MT possui densidade demográfica de 46.99936525034142 hab/km².\nO município Rosário Oeste/MT possui densidade demográfica de 2.3649064417916517 hab/km².\nO município Salto do Céu/MT possui densidade demográfica de 2.2301989944701566 hab/km².\nO município Santa Carmem/MT possui densidade demográfica de 1.0595638280212483 hab/km².\nO município Santa Cruz do Xingu/MT possui densidade demográfica de 0.3361790595833149 hab/km².\nO município Santa Rita do Trivelato/MT possui densidade demográfica de 0.5268389662027834 hab/km².\nO município Santa Terezinha/MT possui densidade demográfica de 1.1437415827453818 hab/km².\nO município Santo Afonso/MT possui densidade demográfica de 2.5472879176283225 hab/km².\nO município Santo Antônio do Leste/MT possui densidade demográfica de 1.042572159379678 hab/km².\nO município Santo Antônio do Leverger/MT possui densidade demográfica de 1.5057958828149403 hab/km².\nO município São Félix do Araguaia/MT possui densidade demográfica de 0.6357151660996586 hab/km².\nO município São José do Povo/MT possui densidade demográfica de 8.092277192033883 hab/km².\nO município São José do Rio Claro/MT possui densidade demográfica de 3.774965830430757 hab/km².\nO município São José do Xingu/MT possui densidade demográfica de 0.7024458252062765 hab/km².\nO município São José dos Quatro Marcos/MT possui densidade demográfica de 14.751374351647668 hab/km².\nO município São Pedro da Cipa/MT possui densidade demográfica de 12.12421635806969 hab/km².\nO município Sapezal/MT possui densidade demográfica de 1.3280614076100399 hab/km².\nO município Serra Nova Dourada/MT possui densidade demográfica de 0.90976346150001 hab/km².\nO município Sinop/MT possui densidade demográfica de 28.689092214305102 hab/km².\nO município Sorriso/MT possui densidade demográfica de 7.130102040816326 hab/km².\nO município Tabaporã/MT possui densidade demográfica de 1.1941188564256027 hab/km².\nO município Tangará da Serra/MT possui densidade demográfica de 7.367860511284357 hab/km².\nO município Tapurah/MT possui densidade demográfica de 2.303880815403545 hab/km².\nO município Terra Nova do Norte/MT possui densidade demográfica de 4.406708219012344 hab/km².\nO município Tesouro/MT possui densidade demográfica de 0.8197507650687362 hab/km².\nO município Torixoréu/MT possui densidade demográfica de 1.6966317421419819 hab/km².\nO município União do Sul/MT possui densidade demográfica de 0.8206184757011814 hab/km².\nO município Vale de São Domingos/MT possui densidade demográfica de 1.578852073148651 hab/km².\nO município Várzea Grande/MT possui densidade demográfica de 240.97842989477297 hab/km².\nO município Vera/MT possui densidade demográfica de 3.4546307578585678 hab/km².\nO município Vila Bela da Santíssima Trindade/MT possui densidade demográfica de 1.079876432272457 hab/km².\nO município Vila Rica/MT possui densidade demográfica de 2.8773744866156736 hab/km².\nO município Água Clara/MS possui densidade demográfica de 1.3075734830189498 hab/km².\nO município Alcinópolis/MS possui densidade demográfica de 1.038484617063059 hab/km².\nO município Amambai/MS possui densidade demográfica de 8.2644824763464 hab/km².\nO município Anastácio/MS possui densidade demográfica de 8.082044535167999 hab/km².\nO município Anaurilândia/MS possui densidade demográfica de 2.5012958556181233 hab/km².\nO município Angélica/MS possui densidade demográfica de 7.213709582413785 hab/km².\nO município Antônio João/MS possui densidade demográfica de 7.167432194065562 hab/km².\nO município Aparecida do Taboado/MS possui densidade demográfica de 8.115920949766377 hab/km².\nO município Aquidauana/MS possui densidade demográfica de 2.6898615677197744 hab/km².\nO município Aral Moreira/MS possui densidade demográfica de 6.1914885906526695 hab/km².\nO município Bandeirantes/MS possui densidade demográfica de 2.121206285626252 hab/km².\nO município Bataguassu/MS possui densidade demográfica de 8.213886473729971 hab/km².\nO município Batayporã/MS possui densidade demográfica de 5.982429076268312 hab/km².\nO município Bela Vista/MS possui densidade demográfica de 4.737971630625843 hab/km².\nO município Bodoquena/MS possui densidade demográfica de 3.1846752708070767 hab/km².\nO município Bonito/MS possui densidade demográfica de 3.9694715274977153 hab/km².\nO município Brasilândia/MS possui densidade demográfica de 2.0365427336444575 hab/km².\nO município Caarapó/MS possui densidade demográfica de 12.331068147013783 hab/km².\nO município Camapuã/MS possui densidade demográfica de 2.1871317993713904 hab/km².\nO município Campo Grande/MS possui densidade demográfica de 97.22004954929908 hab/km².\nO município Caracol/MS possui densidade demográfica de 1.8358983079670097 hab/km².\nO município Cassilândia/MS possui densidade demográfica de 5.744550266869788 hab/km².\nO município Chapadão do Sul/MS possui densidade demográfica de 5.102051415216827 hab/km².\nO município Corguinho/MS possui densidade demográfica de 1.8417713127639828 hab/km².\nO município Coronel Sapucaia/MS possui densidade demográfica de 13.720306326520658 hab/km².\nO município Corumbá/MS possui densidade demográfica de 1.5963463352519722 hab/km².\nO município Costa Rica/MS possui densidade demográfica de 3.666368814922372 hab/km².\nO município Coxim/MS possui densidade demográfica de 5.017615248033302 hab/km².\nO município Deodápolis/MS possui densidade demográfica de 14.604011020079161 hab/km².\nO município Dois Irmãos do Buriti/MS possui densidade demográfica de 4.419962552087998 hab/km².\nO município Douradina/MS possui densidade demográfica de 19.103244417536235 hab/km².\nO município Dourados/MS possui densidade demográfica de 47.97442147304123 hab/km².\nO município Eldorado/MS possui densidade demográfica de 11.489600015720336 hab/km².\nO município Fátima do Sul/MS possui densidade demográfica de 60.3978931336464 hab/km².\nO município Figueirão/MS possui densidade demográfica de 0.5996473385529412 hab/km².\nO município Glória de Dourados/MS possui densidade demográfica de 20.187086934417895 hab/km².\nO município Guia Lopes da Laguna/MS possui densidade demográfica de 8.562625453283882 hab/km².\nO município Iguatemi/MS possui densidade demográfica de 5.048328197331089 hab/km².\nO município Inocência/MS possui densidade demográfica de 1.3277285609666156 hab/km².\nO município Itaporã/MS possui densidade demográfica de 15.78517336077046 hab/km².\nO município Itaquiraí/MS possui densidade demográfica de 9.018236080696111 hab/km².\nO município Ivinhema/MS possui densidade demográfica de 11.11398538432073 hab/km².\nO município Japorã/MS possui densidade demográfica de 18.433476394849787 hab/km².\nO município Jaraguari/MS possui densidade demográfica de 2.1769282001634154 hab/km².\nO município Jardim/MS possui densidade demográfica de 11.058773296510122 hab/km².\nO município Jateí/MS possui densidade demográfica de 2.080448144402085 hab/km².\nO município Juti/MS possui densidade demográfica de 3.7234781072109255 hab/km².\nO município Ladário/MS possui densidade demográfica de 57.56668720838102 hab/km².\nO município Laguna Carapã/MS possui densidade demográfica de 3.743216825157001 hab/km².\nO município Maracaju/MS possui densidade demográfica de 7.058639261168708 hab/km².\nO município Miranda/MS possui densidade demográfica de 4.671617845415901 hab/km².\nO município Mundo Novo/MS possui densidade demográfica de 35.67122943614216 hab/km².\nO município Naviraí/MS possui densidade demográfica de 14.536846258384113 hab/km².\nO município Nioaque/MS possui densidade demográfica de 3.667627472418249 hab/km².\nO município Nova Alvorada do Sul/MS possui densidade demográfica de 4.088253734462546 hab/km².\nO município Nova Andradina/MS possui densidade demográfica de 9.544597989949748 hab/km².\nO município Novo Horizonte do Sul/MS possui densidade demográfica de 5.81799338114923 hab/km².\nO município Paranaíba/MS possui densidade demográfica de 7.439312189388541 hab/km².\nO município Paranhos/MS possui densidade demográfica de 9.433529897033212 hab/km².\nO município Pedro Gomes/MS possui densidade demográfica de 2.1820343012395993 hab/km².\nO município Ponta Porã/MS possui densidade demográfica de 14.60889793544635 hab/km².\nO município Porto Murtinho/MS possui densidade demográfica de 0.8663009928197105 hab/km².\nO município Ribas do Rio Pardo/MS possui densidade demográfica de 1.2101862251618896 hab/km².\nO município Rio Brilhante/MS possui densidade demográfica de 7.689973416261223 hab/km².\nO município Rio Negro/MS possui densidade demográfica de 2.785906719699945 hab/km².\nO município Rio Verde de Mato Grosso/MS possui densidade demográfica de 2.316682814358773 hab/km².\nO município Rochedo/MS possui densidade demográfica de 3.1568293339141356 hab/km².\nO município Santa Rita do Pardo/MS possui densidade demográfica de 1.181656728638938 hab/km².\nO município São Gabriel do Oeste/MS possui densidade demográfica de 5.745092103118232 hab/km².\nO município Selvíria/MS possui densidade demográfica de 1.9295160404256169 hab/km².\nO município Sete Quedas/MS possui densidade demográfica de 12.92984539359265 hab/km².\nO município Sidrolândia/MS possui densidade demográfica de 7.9698699117170255 hab/km².\nO município Sonora/MS possui densidade demográfica de 3.6396248730192227 hab/km².\nO município Tacuru/MS possui densidade demográfica de 5.721663343266194 hab/km².\nO município Taquarussu/MS possui densidade demográfica de 3.3790533271861074 hab/km².\nO município Terenos/MS possui densidade demográfica de 6.027751704159943 hab/km².\nO município Três Lagoas/MS possui densidade demográfica de 9.972714669906289 hab/km².\nO município Vicentina/MS possui densidade demográfica de 19.025664173329893 hab/km².\nO município Abadia dos Dourados/MG possui densidade demográfica de 7.609016412049123 hab/km².\nO município Abaeté/MG possui densidade demográfica de 12.487135883592817 hab/km².\nO município Abre Campo/MG possui densidade demográfica de 28.288173414089893 hab/km².\nO município Acaiaca/MG possui densidade demográfica de 38.47286289135342 hab/km².\nO município Açucena/MG possui densidade demográfica de 12.602094626082264 hab/km².\nO município Água Boa/MG possui densidade demográfica de 11.509009520779841 hab/km².\nO município Água Comprida/MG possui densidade demográfica de 4.1140976412506856 hab/km².\nO município Aguanil/MG possui densidade demográfica de 17.467361799301994 hab/km².\nO município Águas Formosas/MG possui densidade demográfica de 22.533167495854062 hab/km².\nO município Águas Vermelhas/MG possui densidade demográfica de 10.10259831014548 hab/km².\nO município Aimorés/MG possui densidade demográfica de 18.504871068669466 hab/km².\nO município Aiuruoca/MG possui densidade demográfica de 9.484669375692649 hab/km².\nO município Alagoa/MG possui densidade demográfica de 16.788547347545858 hab/km².\nO município Albertina/MG possui densidade demográfica de 50.21548008963972 hab/km².\nO município Além Paraíba/MG possui densidade demográfica de 67.30479082982266 hab/km².\nO município Alfenas/MG possui densidade demográfica de 86.7470162854959 hab/km².\nO município Alfredo Vasconcelos/MG possui densidade demográfica de 46.43785353921419 hab/km².\nO município Almenara/MG possui densidade demográfica de 16.899622128371753 hab/km².\nO município Alpercata/MG possui densidade demográfica de 42.95382404024675 hab/km².\nO município Alpinópolis/MG possui densidade demográfica de 40.65530511269929 hab/km².\nO município Alterosa/MG possui densidade demográfica de 37.89121847462777 hab/km².\nO município Alto Caparaó/MG possui densidade demográfica de 51.084964798919856 hab/km².\nO município Alto Jequitibá/MG possui densidade demográfica de 54.62665002955276 hab/km².\nO município Alto Rio Doce/MG possui densidade demográfica de 23.47070746066982 hab/km².\nO município Alvarenga/MG possui densidade demográfica de 15.975842110939352 hab/km².\nO município Alvinópolis/MG possui densidade demográfica de 25.458761510743358 hab/km².\nO município Alvorada de Minas/MG possui densidade demográfica de 9.481029918986124 hab/km².\nO município Amparo do Serra/MG possui densidade demográfica de 34.63093687889795 hab/km².\nO município Andradas/MG possui densidade demográfica de 79.40430790208151 hab/km².\nO município Andrelândia/MG possui densidade demográfica de 12.108943687891056 hab/km².\nO município Angelândia/MG possui densidade demográfica de 43.210409805086115 hab/km².\nO município Antônio Carlos/MG possui densidade demográfica de 20.9729770531401 hab/km².\nO município Antônio Dias/MG possui densidade demográfica de 12.152821894137677 hab/km².\nO município Antônio Prado de Minas/MG possui densidade demográfica de 19.940334128878284 hab/km².\nO município Araçaí/MG possui densidade demográfica de 12.024230727993997 hab/km².\nO município Aracitaba/MG possui densidade demográfica de 19.304005252790546 hab/km².\nO município Araçuaí/MG possui densidade demográfica de 16.103976246266118 hab/km².\nO município Araguari/MG possui densidade demográfica de 40.22736681675465 hab/km².\nO município Arantina/MG possui densidade demográfica de 31.570118541713263 hab/km².\nO município Araponga/MG possui densidade demográfica de 26.834326343855953 hab/km².\nO município Araporã/MG possui densidade demográfica de 20.767982693347758 hab/km².\nO município Arapuá/MG possui densidade demográfica de 15.958364483294037 hab/km².\nO município Araújos/MG possui densidade demográfica de 32.10736396220267 hab/km².\nO município Araxá/MG possui densidade demográfica de 80.44934556322788 hab/km².\nO município Arceburgo/MG possui densidade demográfica de 58.38040275049116 hab/km².\nO município Arcos/MG possui densidade demográfica de 71.77711965795203 hab/km².\nO município Areado/MG possui densidade demográfica de 48.4988697372139 hab/km².\nO município Argirita/MG possui densidade demográfica de 18.201781904881415 hab/km².\nO município Aricanduva/MG possui densidade demográfica de 19.603008260387128 hab/km².\nO município Arinos/MG possui densidade demográfica de 3.3477162264036577 hab/km².\nO município Astolfo Dutra/MG possui densidade demográfica de 82.1259991188873 hab/km².\nO município Ataléia/MG possui densidade demográfica de 7.868893510000109 hab/km².\nO município Augusto de Lima/MG possui densidade demográfica de 3.952726664169649 hab/km².\nO município Baependi/MG possui densidade demográfica de 24.39144627273333 hab/km².\nO município Baldim/MG possui densidade demográfica de 14.22510651302425 hab/km².\nO município Bambuí/MG possui densidade demográfica de 15.615941531233258 hab/km².\nO município Bandeira/MG possui densidade demográfica de 10.308191570722833 hab/km².\nO município Bandeira do Sul/MG possui densidade demográfica de 113.40556617803271 hab/km².\nO município Barão de Cocais/MG possui densidade demográfica de 83.50557839107456 hab/km².\nO município Barão de Monte Alto/MG possui densidade demográfica de 28.843729514396653 hab/km².\nO município Barbacena/MG possui densidade demográfica de 166.340441786641 hab/km².\nO município Barra Longa/MG possui densidade demográfica de 16.01282485728436 hab/km².\nO município Barroso/MG possui densidade demográfica de 238.8083343487267 hab/km².\nO município Bela Vista de Minas/MG possui densidade demográfica de 91.66208539490563 hab/km².\nO município Belmiro Braga/MG possui densidade demográfica de 8.656169714852593 hab/km².\nO município Belo Horizonte/MG possui densidade demográfica de 7167.021726010864 hab/km².\nO município Belo Oriente/MG possui densidade demográfica de 69.86055955331283 hab/km².\nO município Belo Vale/MG possui densidade demográfica de 20.59466550065588 hab/km².\nO município Berilo/MG possui densidade demográfica de 20.95007749825416 hab/km².\nO município Berizal/MG possui densidade demográfica de 8.940993534659137 hab/km².\nO município Bertópolis/MG possui densidade demográfica de 10.514258999532492 hab/km².\nO município Betim/MG possui densidade demográfica de 1102.7825579699577 hab/km².\nO município Bias Fortes/MG possui densidade demográfica de 13.37730126260845 hab/km².\nO município Bicas/MG possui densidade demográfica de 97.46573386636207 hab/km².\nO município Biquinhas/MG possui densidade demográfica de 5.730471728946508 hab/km².\nO município Boa Esperança/MG possui densidade demográfica de 44.75118221850419 hab/km².\nO município Bocaina de Minas/MG possui densidade demográfica de 9.938664919907104 hab/km².\nO município Bocaiúva/MG possui densidade demográfica de 14.454568832239135 hab/km².\nO município Bom Despacho/MG possui densidade demográfica de 37.27846911845213 hab/km².\nO município Bom Jardim de Minas/MG possui densidade demográfica de 15.778360273773119 hab/km².\nO município Bom Jesus da Penha/MG possui densidade demográfica de 18.65610751139909 hab/km².\nO município Bom Jesus do Amparo/MG possui densidade demográfica de 28.07116200603241 hab/km².\nO município Bom Jesus do Galho/MG possui densidade demográfica de 25.93999561025849 hab/km².\nO município Bom Repouso/MG possui densidade demográfica de 45.49488797041549 hab/km².\nO município Bom Sucesso/MG possui densidade demográfica de 24.456421530387917 hab/km².\nO município Bonfim/MG possui densidade demográfica de 22.585881339649518 hab/km².\nO município Bonfinópolis de Minas/MG possui densidade demográfica de 3.169430799409886 hab/km².\nO município Bonito de Minas/MG possui densidade demográfica de 2.4771377573362767 hab/km².\nO município Borda da Mata/MG possui densidade demográfica de 56.84965627179436 hab/km².\nO município Botelhos/MG possui densidade demográfica de 44.65862492142836 hab/km².\nO município Botumirim/MG possui densidade demográfica de 4.141170771505787 hab/km².\nO município Brás Pires/MG possui densidade demográfica de 20.761137228565033 hab/km².\nO município Brasilândia de Minas/MG possui densidade demográfica de 5.668429168542728 hab/km².\nO município Brasília de Minas/MG possui densidade demográfica de 22.303284076942866 hab/km².\nO município Braúnas/MG possui densidade demográfica de 13.295622753224784 hab/km².\nO município Brazópolis/MG possui densidade demográfica de 39.87326280290462 hab/km².\nO município Brumadinho/MG possui densidade demográfica de 53.13013152338802 hab/km².\nO município Bueno Brandão/MG possui densidade demográfica de 30.582619682717958 hab/km².\nO município Buenópolis/MG possui densidade demográfica de 6.432982473685526 hab/km².\nO município Bugre/MG possui densidade demográfica de 24.655672904700143 hab/km².\nO município Buritis/MG possui densidade demográfica de 4.351420713887916 hab/km².\nO município Buritizeiro/MG possui densidade demográfica de 3.7296353762606675 hab/km².\nO município Cabeceira Grande/MG possui densidade demográfica de 6.256483842506859 hab/km².\nO município Cabo Verde/MG possui densidade demográfica de 37.54107710274029 hab/km².\nO município Cachoeira da Prata/MG possui densidade demográfica de 59.530791788856305 hab/km².\nO município Cachoeira de Minas/MG possui densidade demográfica de 36.267420457533525 hab/km².\nO município Cachoeira de Pajeú/MG possui densidade demográfica de 12.878232495292309 hab/km².\nO município Cachoeira Dourada/MG possui densidade demográfica de 12.467028318319812 hab/km².\nO município Caetanópolis/MG possui densidade demográfica de 65.48320943347859 hab/km².\nO município Caeté/MG possui densidade demográfica de 75.10551633890557 hab/km².\nO município Caiana/MG possui densidade demográfica de 46.66103127641589 hab/km².\nO município Cajuri/MG possui densidade demográfica de 48.73554913294797 hab/km².\nO município Caldas/MG possui densidade demográfica de 19.16335165375803 hab/km².\nO município Camacho/MG possui densidade demográfica de 14.143497757847534 hab/km².\nO município Camanducaia/MG possui densidade demográfica de 39.887980623675446 hab/km².\nO município Cambuí/MG possui densidade demográfica de 108.30437093674612 hab/km².\nO município Cambuquira/MG possui densidade demográfica de 51.148632194171604 hab/km².\nO município Campanário/MG possui densidade demográfica de 8.05605786618445 hab/km².\nO município Campanha/MG possui densidade demográfica de 45.98766351798326 hab/km².\nO município Campestre/MG possui densidade demográfica de 35.79883704831787 hab/km².\nO município Campina Verde/MG possui densidade demográfica de 5.293158939943847 hab/km².\nO município Campo Azul/MG possui densidade demográfica de 7.2819276155837995 hab/km².\nO município Campo Belo/MG possui densidade demográfica de 97.57870624538553 hab/km².\nO município Campo do Meio/MG possui densidade demográfica de 41.66575899502596 hab/km².\nO município Campo Florido/MG possui densidade demográfica de 5.4340518093731465 hab/km².\nO município Campos Altos/MG possui densidade demográfica de 19.990149862801662 hab/km².\nO município Campos Gerais/MG possui densidade demográfica de 35.86744639376219 hab/km².\nO município Cana Verde/MG possui densidade demográfica de 26.273975178638587 hab/km².\nO município Canaã/MG possui densidade demográfica de 26.460834762721554 hab/km².\nO município Canápolis/MG possui densidade demográfica de 13.533950984828637 hab/km².\nO município Candeias/MG possui densidade demográfica de 20.256484989798892 hab/km².\nO município Cantagalo/MG possui densidade demográfica de 29.57140843084731 hab/km².\nO município Caparaó/MG possui densidade demográfica de 39.857678475782386 hab/km².\nO município Capela Nova/MG possui densidade demográfica de 42.810840010804 hab/km².\nO município Capelinha/MG possui densidade demográfica de 36.051462133689675 hab/km².\nO município Capetinga/MG possui densidade demográfica de 23.793381217694836 hab/km².\nO município Capim Branco/MG possui densidade demográfica de 93.16060002097976 hab/km².\nO município Capinópolis/MG possui densidade demográfica de 24.632684624307256 hab/km².\nO município Capitão Andrade/MG possui densidade demográfica de 17.646637285463473 hab/km².\nO município Capitão Enéas/MG possui densidade demográfica de 14.62154428868441 hab/km².\nO município Capitólio/MG possui densidade demográfica de 15.682253737064011 hab/km².\nO município Caputira/MG possui densidade demográfica de 48.10868407032499 hab/km².\nO município Caraí/MG possui densidade demográfica de 17.986636612461762 hab/km².\nO município Caranaíba/MG possui densidade demográfica de 20.55642388246327 hab/km².\nO município Carandaí/MG possui densidade demográfica de 48.06373911432277 hab/km².\nO município Carangola/MG possui densidade demográfica de 91.38653084323713 hab/km².\nO município Caratinga/MG possui densidade demográfica de 67.7155658653617 hab/km².\nO município Carbonita/MG possui densidade demográfica de 6.282535540141474 hab/km².\nO município Careaçu/MG possui densidade demográfica de 34.79365780896083 hab/km².\nO município Carlos Chagas/MG possui densidade demográfica de 6.265727541227232 hab/km².\nO município Carmésia/MG possui densidade demográfica de 9.44037051331532 hab/km².\nO município Carmo da Cachoeira/MG possui densidade demográfica de 23.37605909189659 hab/km².\nO município Carmo da Mata/MG possui densidade demográfica de 30.592418388487598 hab/km².\nO município Carmo de Minas/MG possui densidade demográfica de 42.66343975922306 hab/km².\nO município Carmo do Cajuru/MG possui densidade demográfica de 43.90425835326123 hab/km².\nO município Carmo do Paranaíba/MG possui densidade demográfica de 22.73561390362883 hab/km².\nO município Carmo do Rio Claro/MG possui densidade demográfica de 19.166924715442576 hab/km².\nO município Carmópolis de Minas/MG possui densidade demográfica de 42.618934526636835 hab/km².\nO município Carneirinho/MG possui densidade demográfica de 4.590175057674039 hab/km².\nO município Carrancas/MG possui densidade demográfica de 5.423896467872893 hab/km².\nO município Carvalhópolis/MG possui densidade demográfica de 41.1960542540074 hab/km².\nO município Carvalhos/MG possui densidade demográfica de 16.141718334809568 hab/km².\nO município Casa Grande/MG possui densidade demográfica de 14.226843339884613 hab/km².\nO município Cascalho Rico/MG possui densidade demográfica de 7.778171027197735 hab/km².\nO município Cássia/MG possui densidade demográfica de 26.15199759687594 hab/km².\nO município Cataguases/MG possui densidade demográfica de 141.84883177094983 hab/km².\nO município Catas Altas/MG possui densidade demográfica de 20.188301949675054 hab/km².\nO município Catas Altas da Noruega/MG possui densidade demográfica de 24.445699759920913 hab/km².\nO município Catuji/MG possui densidade demográfica de 15.989321383452912 hab/km².\nO município Catuti/MG possui densidade demográfica de 17.726972655571384 hab/km².\nO município Caxambu/MG possui densidade demográfica de 216.01313694267515 hab/km².\nO município Cedro do Abaeté/MG possui densidade demográfica de 4.272448006779422 hab/km².\nO município Central de Minas/MG possui densidade demográfica de 33.14246561934126 hab/km².\nO município Centralina/MG possui densidade demográfica de 31.376264555762706 hab/km².\nO município Chácara/MG possui densidade demográfica de 18.27105555919115 hab/km².\nO município Chalé/MG possui densidade demográfica de 26.54347110546857 hab/km².\nO município Chapada do Norte/MG possui densidade demográfica de 18.27863821798621 hab/km².\nO município Chapada Gaúcha/MG possui densidade demográfica de 3.319314694380359 hab/km².\nO município Chiador/MG possui densidade demográfica de 11.010516327982922 hab/km².\nO município Cipotânea/MG possui densidade demográfica de 42.657023716445146 hab/km².\nO município Claraval/MG possui densidade demográfica de 19.953433203004877 hab/km².\nO município Claro dos Poções/MG possui densidade demográfica de 10.792315593681463 hab/km².\nO município Cláudio/MG possui densidade demográfica de 40.86030029649125 hab/km².\nO município Coimbra/MG possui densidade demográfica de 65.99925149700599 hab/km².\nO município Coluna/MG possui densidade demográfica de 25.894573732388302 hab/km².\nO município Comendador Gomes/MG possui densidade demográfica de 2.8548100475481486 hab/km².\nO município Comercinho/MG possui densidade demográfica de 12.669475998534262 hab/km².\nO município Conceição da Aparecida/MG possui densidade demográfica de 27.856575513446046 hab/km².\nO município Conceição da Barra de Minas/MG possui densidade demográfica de 14.482985971209846 hab/km².\nO município Conceição das Alagoas/MG possui densidade demográfica de 17.19306099608282 hab/km².\nO município Conceição das Pedras/MG possui densidade demográfica de 26.895607083455634 hab/km².\nO município Conceição de Ipanema/MG possui densidade demográfica de 17.547452154052138 hab/km².\nO município Conceição do Mato Dentro/MG possui densidade demográfica de 10.37044758314368 hab/km².\nO município Conceição do Pará/MG possui densidade demográfica de 20.604801661806416 hab/km².\nO município Conceição do Rio Verde/MG possui densidade demográfica de 35.02759143042631 hab/km².\nO município Conceição dos Ouros/MG possui densidade demográfica de 56.77433459037 hab/km².\nO município Cônego Marinho/MG possui densidade demográfica de 4.3246041412911085 hab/km².\nO município Confins/MG possui densidade demográfica de 140.1322001888574 hab/km².\nO município Congonhal/MG possui densidade demográfica de 51.03105347828207 hab/km².\nO município Congonhas/MG possui densidade demográfica de 159.56523169007136 hab/km².\nO município Congonhas do Norte/MG possui densidade demográfica de 12.393130249467218 hab/km².\nO município Conquista/MG possui densidade demográfica de 10.553722750501326 hab/km².\nO município Conselheiro Lafaiete/MG possui densidade demográfica de 314.68467251856856 hab/km².\nO município Conselheiro Pena/MG possui densidade demográfica de 14.989082675148932 hab/km².\nO município Consolação/MG possui densidade demográfica de 19.990739668943164 hab/km².\nO município Contagem/MG possui densidade demográfica de 3090.2954882982535 hab/km².\nO município Coqueiral/MG possui densidade demográfica de 31.36480280929227 hab/km².\nO município Coração de Jesus/MG possui densidade demográfica de 11.69906795732557 hab/km².\nO município Cordisburgo/MG possui densidade demográfica de 10.5226734656711 hab/km².\nO município Cordislândia/MG possui densidade demográfica de 19.132226801826892 hab/km².\nO município Corinto/MG possui densidade demográfica de 9.469390987566326 hab/km².\nO município Coroaci/MG possui densidade demográfica de 17.82150727957381 hab/km².\nO município Coromandel/MG possui densidade demográfica de 8.314519244699861 hab/km².\nO município Coronel Fabriciano/MG possui densidade demográfica de 468.6734463276836 hab/km².\nO município Coronel Murta/MG possui densidade demográfica de 11.180878331146294 hab/km².\nO município Coronel Pacheco/MG possui densidade demográfica de 22.682685727321118 hab/km².\nO município Coronel Xavier Chaves/MG possui densidade demográfica de 23.419652358992554 hab/km².\nO município Córrego Danta/MG possui densidade demográfica de 5.157963585476781 hab/km².\nO município Córrego do Bom Jesus/MG possui densidade demográfica de 30.165790537808327 hab/km².\nO município Córrego Fundo/MG possui densidade demográfica de 57.26436554247849 hab/km².\nO município Córrego Novo/MG possui densidade demográfica de 15.224694483665223 hab/km².\nO município Couto de Magalhães de Minas/MG possui densidade demográfica de 8.65643982291774 hab/km².\nO município Crisólita/MG possui densidade demográfica de 6.258538604843717 hab/km².\nO município Cristais/MG possui densidade demográfica de 17.95904078417644 hab/km².\nO município Cristália/MG possui densidade demográfica de 6.851433329368383 hab/km².\nO município Cristiano Otoni/MG possui densidade demográfica de 37.68344998871077 hab/km².\nO município Cristina/MG possui densidade demográfica de 32.79478367006071 hab/km².\nO município Crucilândia/MG possui densidade demográfica de 28.45776501555396 hab/km².\nO município Cruzeiro da Fortaleza/MG possui densidade demográfica de 20.911072130973263 hab/km².\nO município Cruzília/MG possui densidade demográfica de 27.929635159450253 hab/km².\nO município Cuparaque/MG possui densidade demográfica de 20.63947078280044 hab/km².\nO município Curral de Dentro/MG possui densidade demográfica de 12.165206067645093 hab/km².\nO município Curvelo/MG possui densidade demográfica de 22.498855641007765 hab/km².\nO município Datas/MG possui densidade demográfica de 16.804256691389874 hab/km².\nO município Delfim Moreira/MG possui densidade demográfica de 19.514285014811367 hab/km².\nO município Delfinópolis/MG possui densidade demográfica de 4.954948419204596 hab/km².\nO município Delta/MG possui densidade demográfica de 78.65616491637495 hab/km².\nO município Descoberto/MG possui densidade demográfica de 22.36712482994793 hab/km².\nO município Desterro de Entre Rios/MG possui densidade demográfica de 18.564573004215603 hab/km².\nO município Desterro do Melo/MG possui densidade demográfica de 21.190610064661232 hab/km².\nO município Diamantina/MG possui densidade demográfica de 11.789313557710592 hab/km².\nO município Diogo de Vasconcelos/MG possui densidade demográfica de 23.30849839481495 hab/km².\nO município Dionísio/MG possui densidade demográfica de 25.37161769829288 hab/km².\nO município Divinésia/MG possui densidade demográfica de 28.15251773959135 hab/km².\nO município Divino/MG possui densidade demográfica de 56.643377346201675 hab/km².\nO município Divino das Laranjeiras/MG possui densidade demográfica de 14.425127830533237 hab/km².\nO município Divinolândia de Minas/MG possui densidade demográfica de 52.76442307692307 hab/km².\nO município Divinópolis/MG possui densidade demográfica de 300.819070214088 hab/km².\nO município Divisa Alegre/MG possui densidade demográfica de 49.9490662139219 hab/km².\nO município Divisa Nova/MG possui densidade demográfica de 26.5625 hab/km².\nO município Divisópolis/MG possui densidade demográfica de 15.663344562162917 hab/km².\nO município Dom Bosco/MG possui densidade demográfica de 4.666128361349679 hab/km².\nO município Dom Cavati/MG possui densidade demográfica de 87.51680107526882 hab/km².\nO município Dom Joaquim/MG possui densidade demográfica de 11.371044581515472 hab/km².\nO município Dom Silvério/MG possui densidade demográfica de 26.65025388521311 hab/km².\nO município Dom Viçoso/MG possui densidade demográfica de 26.281601123595504 hab/km².\nO município Dona Eusébia/MG possui densidade demográfica de 85.44781432436281 hab/km².\nO município Dores de Campos/MG possui densidade demográfica de 74.48734380006408 hab/km².\nO município Dores de Guanhães/MG possui densidade demográfica de 13.668481105411912 hab/km².\nO município Dores do Indaiá/MG possui densidade demográfica de 12.399208063354932 hab/km².\nO município Dores do Turvo/MG possui densidade demográfica de 19.301812518925466 hab/km².\nO município Doresópolis/MG possui densidade demográfica de 9.417304296645085 hab/km².\nO município Douradoquara/MG possui densidade demográfica de 5.884045001278445 hab/km².\nO município Durandé/MG possui densidade demográfica de 34.13501333578589 hab/km².\nO município Elói Mendes/MG possui densidade demográfica de 50.48644753172919 hab/km².\nO município Engenheiro Caldas/MG possui densidade demográfica de 54.955629209879184 hab/km².\nO município Engenheiro Navarro/MG possui densidade demográfica de 11.707846328352321 hab/km².\nO município Entre Folhas/MG possui densidade demográfica de 60.73230841450535 hab/km².\nO município Entre Rios de Minas/MG possui densidade demográfica de 31.177758318739052 hab/km².\nO município Ervália/MG possui densidade demográfica de 50.200005594562086 hab/km².\nO município Esmeraldas/MG possui densidade demográfica de 66.2042224126189 hab/km².\nO município Espera Feliz/MG possui densidade demográfica de 71.9556730890316 hab/km².\nO município Espinosa/MG possui densidade demográfica de 16.647137193213375 hab/km².\nO município Espírito Santo do Dourado/MG possui densidade demográfica de 16.784144308018796 hab/km².\nO município Estiva/MG possui densidade demográfica de 44.47041456513716 hab/km².\nO município Estrela Dalva/MG possui densidade demográfica de 18.80185734947096 hab/km².\nO município Estrela do Indaiá/MG possui densidade demográfica de 5.528475738230762 hab/km².\nO município Estrela do Sul/MG possui densidade demográfica de 9.053437898960423 hab/km².\nO município Eugenópolis/MG possui densidade demográfica de 34.065934065934066 hab/km².\nO município Ewbank da Câmara/MG possui densidade demográfica de 36.1456226524126 hab/km².\nO município Extrema/MG possui densidade demográfica de 116.93106550004089 hab/km².\nO município Fama/MG possui densidade demográfica de 27.319228086491513 hab/km².\nO município Faria Lemos/MG possui densidade demográfica de 20.43336157850139 hab/km².\nO município Felício dos Santos/MG possui densidade demográfica de 14.378390470331636 hab/km².\nO município Felisburgo/MG possui densidade demográfica de 11.53433296434202 hab/km².\nO município Felixlândia/MG possui densidade demográfica de 9.083190212462128 hab/km².\nO município Fernandes Tourinho/MG possui densidade demográfica de 19.949960495127733 hab/km².\nO município Ferros/MG possui densidade demográfica de 9.953159441587069 hab/km².\nO município Fervedouro/MG possui densidade demográfica de 28.933683739655557 hab/km².\nO município Florestal/MG possui densidade demográfica de 34.47915578309477 hab/km².\nO município Formiga/MG possui densidade demográfica de 43.36316181953766 hab/km².\nO município Formoso/MG possui densidade demográfica de 2.218574490598801 hab/km².\nO município Fortaleza de Minas/MG possui densidade demográfica de 18.730289318524612 hab/km².\nO município Fortuna de Minas/MG possui densidade demográfica de 13.612802576619194 hab/km².\nO município Francisco Badaró/MG possui densidade demográfica de 22.213070337054297 hab/km².\nO município Francisco Dumont/MG possui densidade demográfica de 3.0854053916872335 hab/km².\nO município Francisco Sá/MG possui densidade demográfica de 9.067845040021258 hab/km².\nO município Franciscópolis/MG possui densidade demográfica de 8.088245547978635 hab/km².\nO município Frei Gaspar/MG possui densidade demográfica de 9.381333078015544 hab/km².\nO município Frei Inocêncio/MG possui densidade demográfica de 18.99650736860039 hab/km².\nO município Frei Lagonegro/MG possui densidade demográfica de 19.878187137994864 hab/km².\nO município Fronteira/MG possui densidade demográfica de 70.20851042552127 hab/km².\nO município Fronteira dos Vales/MG possui densidade demográfica de 14.612171093652575 hab/km².\nO município Fruta de Leite/MG possui densidade demográfica de 7.787202244392297 hab/km².\nO município Frutal/MG possui densidade demográfica de 22.03076263818671 hab/km².\nO município Funilândia/MG possui densidade demográfica de 19.294294294294293 hab/km².\nO município Galiléia/MG possui densidade demográfica de 9.649341995668832 hab/km².\nO município Gameleiras/MG possui densidade demográfica de 2.96503577198246 hab/km².\nO município Glaucilândia/MG possui densidade demográfica de 20.3071438365556 hab/km².\nO município Goiabeira/MG possui densidade demográfica de 27.152258982568483 hab/km².\nO município Goianá/MG possui densidade demográfica de 24.06603525388056 hab/km².\nO município Gonçalves/MG possui densidade demográfica de 22.524686415799305 hab/km².\nO município Gonzaga/MG possui densidade demográfica de 28.28278003343683 hab/km².\nO município Gouveia/MG possui densidade demográfica de 13.47911377798292 hab/km².\nO município Governador Valadares/MG possui densidade demográfica de 112.57599303254892 hab/km².\nO município Grão Mogol/MG possui densidade demográfica de 3.8668928188114657 hab/km².\nO município Grupiara/MG possui densidade demográfica de 7.108832970901937 hab/km².\nO município Guanhães/MG possui densidade demográfica de 29.077684351514254 hab/km².\nO município Guapé/MG possui densidade demográfica de 14.846684861133408 hab/km².\nO município Guaraciaba/MG possui densidade demográfica de 29.325874928284566 hab/km².\nO município Guaraciama/MG possui densidade demográfica de 12.08937631322708 hab/km².\nO município Guaranésia/MG possui densidade demográfica de 63.4738662958315 hab/km².\nO município Guarani/MG possui densidade demográfica de 32.84757182330898 hab/km².\nO município Guarará/MG possui densidade demográfica de 44.31536205729754 hab/km².\nO município Guarda-Mor/MG possui densidade demográfica de 3.1718193633170517 hab/km².\nO município Guaxupé/MG possui densidade demográfica de 172.59078212290504 hab/km².\nO município Guidoval/MG possui densidade demográfica de 45.49816896072737 hab/km².\nO município Guimarânia/MG possui densidade demográfica de 19.804814219120573 hab/km².\nO município Guiricema/MG possui densidade demográfica de 29.6580148511479 hab/km².\nO município Gurinhatã/MG possui densidade demográfica de 3.318840109456288 hab/km².\nO município Heliodora/MG possui densidade demográfica de 39.7596622279961 hab/km².\nO município Iapu/MG possui densidade demográfica de 30.286569968876623 hab/km².\nO município Ibertioga/MG possui densidade demográfica de 14.544824399260628 hab/km².\nO município Ibiá/MG possui densidade demográfica de 8.586125667035239 hab/km².\nO município Ibiaí/MG possui densidade demográfica de 8.961315103571266 hab/km².\nO município Ibiracatu/MG possui densidade demográfica de 17.416032370334737 hab/km².\nO município Ibiraci/MG possui densidade demográfica de 21.662011421658452 hab/km².\nO município Ibirité/MG possui densidade demográfica de 2190.354140829544 hab/km².\nO município Ibitiúra de Minas/MG possui densidade demográfica de 49.50234192037471 hab/km².\nO município Ibituruna/MG possui densidade demográfica de 18.71856834955261 hab/km².\nO município Icaraí de Minas/MG possui densidade demográfica de 17.175462711376788 hab/km².\nO município Igarapé/MG possui densidade demográfica de 316.0801741338654 hab/km².\nO município Igaratinga/MG possui densidade demográfica de 42.42923880186864 hab/km².\nO município Iguatama/MG possui densidade demográfica de 12.780961477236549 hab/km².\nO município Ijaci/MG possui densidade demográfica de 55.66745843230404 hab/km².\nO município Ilicínea/MG possui densidade demográfica de 30.525588563532978 hab/km².\nO município Imbé de Minas/MG possui densidade demográfica de 32.652231371353054 hab/km².\nO município Inconfidentes/MG possui densidade demográfica de 46.17338413207673 hab/km².\nO município Indaiabira/MG possui densidade demográfica de 7.299706219190361 hab/km².\nO município Indianópolis/MG possui densidade demográfica de 7.457561774875607 hab/km².\nO município Ingaí/MG possui densidade demográfica de 8.60303020386793 hab/km².\nO município Inhapim/MG possui densidade demográfica de 28.314025314095243 hab/km².\nO município Inhaúma/MG possui densidade demográfica de 23.510204081632654 hab/km².\nO município Inimutaba/MG possui densidade demográfica de 12.820561709916678 hab/km².\nO município Ipaba/MG possui densidade demográfica de 147.68849995580305 hab/km².\nO município Ipanema/MG possui densidade demográfica de 39.7906447091801 hab/km².\nO município Ipatinga/MG possui densidade demográfica de 1452.3774866569627 hab/km².\nO município Ipiaçu/MG possui densidade demográfica de 8.812926483841895 hab/km².\nO município Ipuiúna/MG possui densidade demográfica de 31.92823608316566 hab/km².\nO município Iraí de Minas/MG possui densidade demográfica de 18.152472913041038 hab/km².\nO município Itabira/MG possui densidade demográfica de 87.56720108478902 hab/km².\nO município Itabirinha/MG possui densidade demográfica de 51.162790697674424 hab/km².\nO município Itabirito/MG possui densidade demográfica de 83.75997493595769 hab/km².\nO município Itacambira/MG possui densidade demográfica de 2.7890072409069306 hab/km².\nO município Itacarambi/MG possui densidade demográfica de 14.462118553461686 hab/km².\nO município Itaguara/MG possui densidade demográfica de 30.14105781177674 hab/km².\nO município Itaipé/MG possui densidade demográfica de 24.53673855624649 hab/km².\nO município Itajubá/MG possui densidade demográfica de 307.48202414869087 hab/km².\nO município Itamarandiba/MG possui densidade demográfica de 11.761716936506833 hab/km².\nO município Itamarati de Minas/MG possui densidade demográfica de 43.132071481442324 hab/km².\nO município Itambacuri/MG possui densidade demográfica de 16.07161730822077 hab/km².\nO município Itambé do Mato Dentro/MG possui densidade demográfica de 6.002524057422307 hab/km².\nO município Itamogi/MG possui densidade demográfica de 42.467889531782184 hab/km².\nO município Itamonte/MG possui densidade demográfica de 32.43011649181315 hab/km².\nO município Itanhandu/MG possui densidade demográfica de 98.87695312499999 hab/km².\nO município Itanhomi/MG possui densidade demográfica de 24.25333442435153 hab/km².\nO município Itaobim/MG possui densidade demográfica de 30.928396807163267 hab/km².\nO município Itapagipe/MG possui densidade demográfica de 7.576396440380817 hab/km².\nO município Itapecerica/MG possui densidade demográfica de 20.544535424595395 hab/km².\nO município Itapeva/MG possui densidade demográfica de 48.85255145193121 hab/km².\nO município Itatiaiuçu/MG possui densidade demográfica de 33.63713366085042 hab/km².\nO município Itaú de Minas/MG possui densidade demográfica de 97.41233216008344 hab/km².\nO município Itaúna/MG possui densidade demográfica de 172.38437178530367 hab/km².\nO município Itaverava/MG possui densidade demográfica de 20.40320878192949 hab/km².\nO município Itinga/MG possui densidade demográfica de 8.733526509135437 hab/km².\nO município Itueta/MG possui densidade demográfica de 12.878854820182028 hab/km².\nO município Ituiutaba/MG possui densidade demográfica de 37.40151267296626 hab/km².\nO município Itumirim/MG possui densidade demográfica de 26.145655877342417 hab/km².\nO município Iturama/MG possui densidade demográfica de 24.529779448407442 hab/km².\nO município Itutinga/MG possui densidade demográfica de 10.518251706897479 hab/km².\nO município Jaboticatubas/MG possui densidade demográfica de 15.367229611559056 hab/km².\nO município Jacinto/MG possui densidade demográfica de 8.706883561398095 hab/km².\nO município Jacuí/MG possui densidade demográfica de 18.331989345844633 hab/km².\nO município Jacutinga/MG possui densidade demográfica de 65.48382458662833 hab/km².\nO município Jaguaraçu/MG possui densidade demográfica de 18.258426966292134 hab/km².\nO município Jaíba/MG possui densidade demográfica de 12.788568077888156 hab/km².\nO município Jampruca/MG possui densidade demográfica de 9.798878360085089 hab/km².\nO município Janaúba/MG possui densidade demográfica de 30.62503438285075 hab/km².\nO município Januária/MG possui densidade demográfica de 9.826815197990895 hab/km².\nO município Japaraíba/MG possui densidade demográfica de 22.88253746950157 hab/km².\nO município Japonvar/MG possui densidade demográfica de 22.114436478959572 hab/km².\nO município Jeceaba/MG possui densidade demográfica de 22.835978835978835 hab/km².\nO município Jenipapo de Minas/MG possui densidade demográfica de 25.016698892599756 hab/km².\nO município Jequeri/MG possui densidade demográfica de 23.449534586603395 hab/km².\nO município Jequitaí/MG possui densidade demográfica de 6.310901579893412 hab/km².\nO município Jequitibá/MG possui densidade demográfica de 11.585735793092601 hab/km².\nO município Jequitinhonha/MG possui densidade demográfica de 6.866673116651775 hab/km².\nO município Jesuânia/MG possui densidade demográfica de 30.991225219369518 hab/km².\nO município Joaíma/MG possui densidade demográfica de 8.977941220653891 hab/km².\nO município Joanésia/MG possui densidade demográfica de 23.254318659179564 hab/km².\nO município João Monlevade/MG possui densidade demográfica de 742.3356192012909 hab/km².\nO município João Pinheiro/MG possui densidade demográfica de 4.219074954299569 hab/km².\nO município Joaquim Felício/MG possui densidade demográfica de 5.442890737603357 hab/km².\nO município Jordânia/MG possui densidade demográfica de 18.8838689616067 hab/km².\nO município José Gonçalves de Minas/MG possui densidade demográfica de 11.939789683476254 hab/km².\nO município José Raydan/MG possui densidade demográfica de 24.200862736422962 hab/km².\nO município Josenópolis/MG possui densidade demográfica de 8.433846210679917 hab/km².\nO município Juatuba/MG possui densidade demográfica de 223.04601165360657 hab/km².\nO município Juiz de Fora/MG possui densidade demográfica de 359.5886212613014 hab/km².\nO município Juramento/MG possui densidade demográfica de 9.52899474086602 hab/km².\nO município Juruaia/MG possui densidade demográfica de 41.92421148173361 hab/km².\nO município Juvenília/MG possui densidade demográfica de 5.361134591903823 hab/km².\nO município Ladainha/MG possui densidade demográfica de 19.61698738297799 hab/km².\nO município Lagamar/MG possui densidade demográfica de 5.154079861111112 hab/km².\nO município Lagoa da Prata/MG possui densidade demográfica de 104.51384153825174 hab/km².\nO município Lagoa dos Patos/MG possui densidade demográfica de 7.0352177170926655 hab/km².\nO município Lagoa Dourada/MG possui densidade demográfica de 25.710629549602466 hab/km².\nO município Lagoa Formosa/MG possui densidade demográfica de 20.407410930885224 hab/km².\nO município Lagoa Grande/MG possui densidade demográfica de 6.9813152147537005 hab/km².\nO município Lagoa Santa/MG possui densidade demográfica de 229.0748898678414 hab/km².\nO município Lajinha/MG possui densidade demográfica de 45.39961103908131 hab/km².\nO município Lambari/MG possui densidade demográfica de 91.75543146731734 hab/km².\nO município Lamim/MG possui densidade demográfica de 29.106239460370997 hab/km².\nO município Laranjal/MG possui densidade demográfica de 31.555056618508395 hab/km².\nO município Lassance/MG possui densidade demográfica de 2.0235814020260783 hab/km².\nO município Lavras/MG possui densidade demográfica de 163.26096964975034 hab/km².\nO município Leandro Ferreira/MG possui densidade demográfica de 9.102269177245747 hab/km².\nO município Leme do Prado/MG possui densidade demográfica de 17.154692186830452 hab/km².\nO município Leopoldina/MG possui densidade demográfica de 54.2159731942147 hab/km².\nO município Liberdade/MG possui densidade demográfica de 13.320376737927942 hab/km².\nO município Lima Duarte/MG possui densidade demográfica de 19.031064391439617 hab/km².\nO município Limeira do Oeste/MG possui densidade demográfica de 5.223495875788452 hab/km².\nO município Lontra/MG possui densidade demográfica de 32.43713060609572 hab/km².\nO município Luisburgo/MG possui densidade demográfica de 42.86893137120067 hab/km².\nO município Luislândia/MG possui densidade demográfica de 15.544922396832723 hab/km².\nO município Luminárias/MG possui densidade demográfica de 10.84096452993162 hab/km².\nO município Luz/MG possui densidade demográfica de 14.924124746086747 hab/km².\nO município Machacalis/MG possui densidade demográfica de 20.988025753655457 hab/km².\nO município Machado/MG possui densidade demográfica de 66.02498464058979 hab/km².\nO município Madre de Deus de Minas/MG possui densidade demográfica de 9.949077924976162 hab/km².\nO município Malacacheta/MG possui densidade demográfica de 25.79510640344008 hab/km².\nO município Mamonas/MG possui densidade demográfica de 21.689599560786466 hab/km².\nO município Manga/MG possui densidade demográfica de 10.159575013588489 hab/km².\nO município Manhuaçu/MG possui densidade demográfica de 126.64565826330531 hab/km².\nO município Manhumirim/MG possui densidade demográfica de 116.90541279387644 hab/km².\nO município Mantena/MG possui densidade demográfica de 39.56597247559142 hab/km².\nO município Mar de Espanha/MG possui densidade demográfica de 31.617330462863293 hab/km².\nO município Maravilhas/MG possui densidade demográfica de 27.38149847094801 hab/km².\nO município Maria da Fé/MG possui densidade demográfica de 70.06407097092163 hab/km².\nO município Mariana/MG possui densidade demográfica de 45.40156253925189 hab/km².\nO município Marilac/MG possui densidade demográfica de 26.56633713242239 hab/km².\nO município Mário Campos/MG possui densidade demográfica de 374.77272727272725 hab/km².\nO município Maripá de Minas/MG possui densidade demográfica de 36.04861649857771 hab/km².\nO município Marliéria/MG possui densidade demográfica de 7.350543229328888 hab/km².\nO município Marmelópolis/MG possui densidade demográfica de 27.506950880444855 hab/km².\nO município Martinho Campos/MG possui densidade demográfica de 12.032248831218396 hab/km².\nO município Martins Soares/MG possui densidade demográfica de 63.326564845060474 hab/km².\nO município Mata Verde/MG possui densidade demográfica de 34.60794655414909 hab/km².\nO município Materlândia/MG possui densidade demográfica de 16.379709834955264 hab/km².\nO município Mateus Leme/MG possui densidade demográfica de 92.02206732516271 hab/km².\nO município Mathias Lobato/MG possui densidade demográfica de 19.558908879860706 hab/km².\nO município Matias Barbosa/MG possui densidade demográfica de 85.51333460632677 hab/km².\nO município Matias Cardoso/MG possui densidade demográfica de 5.118118313210992 hab/km².\nO município Matipó/MG possui densidade demográfica de 66.06614479943069 hab/km².\nO município Mato Verde/MG possui densidade demográfica de 26.85865537321334 hab/km².\nO município Matozinhos/MG possui densidade demográfica de 134.59251625178374 hab/km².\nO município Matutina/MG possui densidade demográfica de 14.412170447578173 hab/km².\nO município Medeiros/MG possui densidade demográfica de 3.638899454799036 hab/km².\nO município Medina/MG possui densidade demográfica de 14.643080994498224 hab/km².\nO município Mendes Pimentel/MG possui densidade demográfica de 20.72272593368466 hab/km².\nO município Mercês/MG possui densidade demográfica de 29.770006029804463 hab/km².\nO município Mesquita/MG possui densidade demográfica de 22.07390703426202 hab/km².\nO município Minas Novas/MG possui densidade demográfica de 16.99073052306334 hab/km².\nO município Minduri/MG possui densidade demográfica de 17.472812485780587 hab/km².\nO município Mirabela/MG possui densidade demográfica de 18.031744276075656 hab/km².\nO município Miradouro/MG possui densidade demográfica de 33.98083999071833 hab/km².\nO município Miraí/MG possui densidade demográfica de 43.05581540380418 hab/km².\nO município Miravânia/MG possui densidade demográfica de 7.554846959958813 hab/km².\nO município Moeda/MG possui densidade demográfica de 30.230159241828378 hab/km².\nO município Moema/MG possui densidade demográfica de 34.67021853879927 hab/km².\nO município Monjolos/MG possui densidade demográfica de 3.625693260204944 hab/km².\nO município Monsenhor Paulo/MG possui densidade demográfica de 37.688186940057264 hab/km².\nO município Montalvânia/MG possui densidade demográfica de 10.54801534788767 hab/km².\nO município Monte Alegre de Minas/MG possui densidade demográfica de 7.5575124424105145 hab/km².\nO município Monte Azul/MG possui densidade demográfica de 22.121641873610734 hab/km².\nO município Monte Belo/MG possui densidade demográfica de 31.003133308013673 hab/km².\nO município Monte Carmelo/MG possui densidade demográfica de 34.08089111269955 hab/km².\nO município Monte Formoso/MG possui densidade demográfica de 12.076254701076383 hab/km².\nO município Monte Santo de Minas/MG possui densidade demográfica de 35.70960092830836 hab/km².\nO município Monte Sião/MG possui densidade demográfica de 72.71511368702632 hab/km².\nO município Montes Claros/MG possui densidade demográfica de 101.40686030025722 hab/km².\nO município Montezuma/MG possui densidade demográfica de 6.602855575803683 hab/km².\nO município Morada Nova de Minas/MG possui densidade demográfica de 3.9606003032222152 hab/km².\nO município Morro da Garça/MG possui densidade demográfica de 6.41319285387082 hab/km².\nO município Morro do Pilar/MG possui densidade demográfica de 7.117579311066904 hab/km².\nO município Munhoz/MG possui densidade demográfica de 32.66339528085195 hab/km².\nO município Muriaé/MG possui densidade demográfica de 119.71747317896137 hab/km².\nO município Mutum/MG possui densidade demográfica de 21.314817479733296 hab/km².\nO município Muzambinho/MG possui densidade demográfica de 49.83534577387486 hab/km².\nO município Nacip Raydan/MG possui densidade demográfica de 13.508073150884405 hab/km².\nO município Nanuque/MG possui densidade demográfica de 26.900931525620248 hab/km².\nO município Naque/MG possui densidade demográfica de 49.862388928206336 hab/km².\nO município Natalândia/MG possui densidade demográfica de 6.998677079332565 hab/km².\nO município Natércia/MG possui densidade demográfica de 24.682068673166597 hab/km².\nO município Nazareno/MG possui densidade demográfica de 24.16674262449488 hab/km².\nO município Nepomuceno/MG possui densidade demográfica de 44.17303235773754 hab/km².\nO município Ninheira/MG possui densidade demográfica de 8.856464813260786 hab/km².\nO município Nova Belém/MG possui densidade demográfica de 25.425807330699005 hab/km².\nO município Nova Era/MG possui densidade demográfica de 48.42925427568867 hab/km².\nO município Nova Lima/MG possui densidade demográfica de 188.73613570696241 hab/km².\nO município Nova Módica/MG possui densidade demográfica de 10.080591536558767 hab/km².\nO município Nova Ponte/MG possui densidade demográfica de 11.531849398295245 hab/km².\nO município Nova Porteirinha/MG possui densidade demográfica de 61.17082851000496 hab/km².\nO município Nova Resende/MG possui densidade demográfica de 39.405356914007434 hab/km².\nO município Nova Serrana/MG possui densidade demográfica de 261.0015228246627 hab/km².\nO município Nova União/MG possui densidade demográfica de 32.27211990937082 hab/km².\nO município Novo Cruzeiro/MG possui densidade demográfica de 18.041903017064204 hab/km².\nO município Novo Oriente de Minas/MG possui densidade demográfica de 13.691319605376416 hab/km².\nO município Novorizonte/MG possui densidade demográfica de 18.25504836870563 hab/km².\nO município Olaria/MG possui densidade demográfica de 11.08617594254937 hab/km².\nO município Olhos-d`Água/MG possui densidade demográfica de 2.517590149516271 hab/km².\nO município Olímpio Noronha/MG possui densidade demográfica de 46.36646531209958 hab/km².\nO município Oliveira/MG possui densidade demográfica de 43.98355046863333 hab/km².\nO município Oliveira Fortes/MG possui densidade demográfica de 19.103752362098444 hab/km².\nO município Onça de Pitangui/MG possui densidade demográfica de 12.36942262531379 hab/km².\nO município Oratórios/MG possui densidade demográfica de 50.443471426967555 hab/km².\nO município Orizânia/MG possui densidade demográfica de 59.80295566502463 hab/km².\nO município Ouro Branco/MG possui densidade demográfica de 136.31198546747575 hab/km².\nO município Ouro Fino/MG possui densidade demográfica de 59.1537683169059 hab/km².\nO município Ouro Preto/MG possui densidade demográfica de 56.411182547135745 hab/km².\nO município Ouro Verde de Minas/MG possui densidade demográfica de 34.28310918623205 hab/km².\nO município Padre Carvalho/MG possui densidade demográfica de 13.071046086976004 hab/km².\nO município Padre Paraíso/MG possui densidade demográfica de 34.624710680039676 hab/km².\nO município Pai Pedro/MG possui densidade demográfica de 7.065883949941059 hab/km².\nO município Paineiras/MG possui densidade demográfica de 7.266479421317727 hab/km².\nO município Pains/MG possui densidade demográfica de 18.99682359076471 hab/km².\nO município Paiva/MG possui densidade demográfica de 26.668948990071893 hab/km².\nO município Palma/MG possui densidade demográfica de 20.67995829252109 hab/km².\nO município Palmópolis/MG possui densidade demográfica de 16.001385201431376 hab/km².\nO município Papagaios/MG possui densidade demográfica de 25.606055132049566 hab/km².\nO município Pará de Minas/MG possui densidade demográfica de 152.77097505668934 hab/km².\nO município Paracatu/MG possui densidade demográfica de 10.294303489841548 hab/km².\nO município Paraguaçu/MG possui densidade demográfica de 47.71388168748527 hab/km².\nO município Paraisópolis/MG possui densidade demográfica de 58.50440768023186 hab/km².\nO município Paraopeba/MG possui densidade demográfica de 36.06502349669128 hab/km².\nO município Passa Quatro/MG possui densidade demográfica de 56.2080657961186 hab/km².\nO município Passa Tempo/MG possui densidade demográfica de 19.099657478388515 hab/km².\nO município Passa-Vinte/MG possui densidade demográfica de 8.432024659312136 hab/km².\nO município Passabém/MG possui densidade demográfica de 18.751327245699724 hab/km².\nO município Passos/MG possui densidade demográfica de 79.43530607516797 hab/km².\nO município Patis/MG possui densidade demográfica de 12.55965781179649 hab/km².\nO município Patos de Minas/MG possui densidade demográfica de 43.48589396727664 hab/km².\nO município Patrocínio/MG possui densidade demográfica de 28.692151937488255 hab/km².\nO município Patrocínio do Muriaé/MG possui densidade demográfica de 48.840646651270205 hab/km².\nO município Paula Cândido/MG possui densidade demográfica de 34.552027429934405 hab/km².\nO município Paulistas/MG possui densidade demográfica de 22.29778745012695 hab/km².\nO município Pavão/MG possui densidade demográfica de 14.286664781516658 hab/km².\nO município Peçanha/MG possui densidade demográfica de 17.31801535142728 hab/km².\nO município Pedra Azul/MG possui densidade demográfica de 14.949361928949925 hab/km².\nO município Pedra Bonita/MG possui densidade demográfica de 38.366009314091876 hab/km².\nO município Pedra do Anta/MG possui densidade demográfica de 20.58733557662894 hab/km².\nO município Pedra do Indaiá/MG possui densidade demográfica de 11.13761784318234 hab/km².\nO município Pedra Dourada/MG possui densidade demográfica de 31.30447206743821 hab/km².\nO município Pedralva/MG possui densidade demográfica de 52.603330428001286 hab/km².\nO município Pedras de Maria da Cruz/MG possui densidade demográfica de 6.7617617945709245 hab/km².\nO município Pedrinópolis/MG possui densidade demográfica de 9.751599653524826 hab/km².\nO município Pedro Leopoldo/MG possui densidade demográfica de 200.5120327700973 hab/km².\nO município Pedro Teixeira/MG possui densidade demográfica de 15.802053824362607 hab/km².\nO município Pequeri/MG possui densidade demográfica de 34.84531542441925 hab/km².\nO município Pequi/MG possui densidade demográfica de 19.9813716358645 hab/km².\nO município Perdigão/MG possui densidade demográfica de 35.745227017487565 hab/km².\nO município Perdizes/MG possui densidade demográfica de 5.877216605054635 hab/km².\nO município Perdões/MG possui densidade demográfica de 74.21488213995418 hab/km².\nO município Periquito/MG possui densidade demográfica de 30.736970861910795 hab/km².\nO município Pescador/MG possui densidade demográfica de 13.003213003213004 hab/km².\nO município Piau/MG possui densidade demográfica de 14.781477627471386 hab/km².\nO município Piedade de Caratinga/MG possui densidade demográfica de 65.02057613168725 hab/km².\nO município Piedade de Ponte Nova/MG possui densidade demográfica de 48.51307774991042 hab/km².\nO município Piedade do Rio Grande/MG possui densidade demográfica de 14.587528267401877 hab/km².\nO município Piedade dos Gerais/MG possui densidade demográfica de 17.870898166692346 hab/km².\nO município Pimenta/MG possui densidade demográfica de 19.84721787117141 hab/km².\nO município Pingo-d`Água/MG possui densidade demográfica de 66.39627459816735 hab/km².\nO município Pintópolis/MG possui densidade demográfica de 5.8686133763041815 hab/km².\nO município Piracema/MG possui densidade demográfica de 22.850823999429267 hab/km².\nO município Pirajuba/MG possui densidade demográfica de 13.775963074738149 hab/km².\nO município Piranga/MG possui densidade demográfica de 26.156251423020297 hab/km².\nO município Piranguçu/MG possui densidade demográfica de 25.621255279442096 hab/km².\nO município Piranguinho/MG possui densidade demográfica de 64.23076923076923 hab/km².\nO município Pirapetinga/MG possui densidade demográfica de 54.35284245856933 hab/km².\nO município Pirapora/MG possui densidade demográfica de 97.11925169696639 hab/km².\nO município Piraúba/MG possui densidade demográfica de 75.27895211033336 hab/km².\nO município Pitangui/MG possui densidade demográfica de 44.43566650866382 hab/km².\nO município Piumhi/MG possui densidade demográfica de 35.32859818054894 hab/km².\nO município Planura/MG possui densidade demográfica de 32.7034517510708 hab/km².\nO município Poço Fundo/MG possui densidade demográfica de 33.6517375168691 hab/km².\nO município Poços de Caldas/MG possui densidade demográfica de 278.5421920111099 hab/km².\nO município Pocrane/MG possui densidade demográfica de 13.003024295657458 hab/km².\nO município Pompéu/MG possui densidade demográfica de 11.408938210241192 hab/km².\nO município Ponte Nova/MG possui densidade demográfica de 121.94033656297808 hab/km².\nO município Ponto Chique/MG possui densidade demográfica de 6.579296615792967 hab/km².\nO município Ponto dos Volantes/MG possui densidade demográfica de 9.357395600498181 hab/km².\nO município Porteirinha/MG possui densidade demográfica de 21.505075213753372 hab/km².\nO município Porto Firme/MG possui densidade demográfica de 36.57911370180491 hab/km².\nO município Poté/MG possui densidade demográfica de 25.06278894914495 hab/km².\nO município Pouso Alegre/MG possui densidade demográfica de 240.51227281934186 hab/km².\nO município Pouso Alto/MG possui densidade demográfica de 23.620879747557314 hab/km².\nO município Prados/MG possui densidade demográfica de 31.769650159018628 hab/km².\nO município Prata/MG possui densidade demográfica de 5.322699761115947 hab/km².\nO município Pratápolis/MG possui densidade demográfica de 40.86395694135115 hab/km².\nO município Pratinha/MG possui densidade demográfica de 5.2451484385040485 hab/km².\nO município Presidente Bernardes/MG possui densidade demográfica de 23.38260135135135 hab/km².\nO município Presidente Juscelino/MG possui densidade demográfica de 5.615910789216532 hab/km².\nO município Presidente Kubitschek/MG possui densidade demográfica de 15.636229127034452 hab/km².\nO município Presidente Olegário/MG possui densidade demográfica de 5.301957874307894 hab/km².\nO município Prudente de Morais/MG possui densidade demográfica de 77.08350108704404 hab/km².\nO município Quartel Geral/MG possui densidade demográfica de 5.935949967651498 hab/km².\nO município Queluzito/MG possui densidade demográfica de 12.119041417035687 hab/km².\nO município Raposos/MG possui densidade demográfica de 212.87637019564315 hab/km².\nO município Raul Soares/MG possui densidade demográfica de 31.201530077551876 hab/km².\nO município Recreio/MG possui densidade demográfica de 43.956466069142124 hab/km².\nO município Reduto/MG possui densidade demográfica de 43.25694718819965 hab/km².\nO município Resende Costa/MG possui densidade demográfica de 17.649722631042682 hab/km².\nO município Resplendor/MG possui densidade demográfica de 15.796820114623776 hab/km².\nO município Ressaquinha/MG possui densidade demográfica de 25.518660960944693 hab/km².\nO município Riachinho/MG possui densidade demográfica de 4.657209164354639 hab/km².\nO município Riacho dos Machados/MG possui densidade demográfica de 7.114948994329325 hab/km².\nO município Ribeirão das Neves/MG possui densidade demográfica de 1905.0855085508551 hab/km².\nO município Ribeirão Vermelho/MG possui densidade demográfica de 77.68527918781726 hab/km².\nO município Rio Acima/MG possui densidade demográfica de 39.554414516339584 hab/km².\nO município Rio Casca/MG possui densidade demográfica de 36.947132896243104 hab/km².\nO município Rio do Prado/MG possui densidade demográfica de 10.872827310241341 hab/km².\nO município Rio Doce/MG possui densidade demográfica de 21.991257025604423 hab/km².\nO município Rio Espera/MG possui densidade demográfica de 25.440067057837386 hab/km².\nO município Rio Manso/MG possui densidade demográfica de 22.786559557743804 hab/km².\nO município Rio Novo/MG possui densidade demográfica de 41.62247384262577 hab/km².\nO município Rio Paranaíba/MG possui densidade demográfica de 8.788405368432729 hab/km².\nO município Rio Pardo de Minas/MG possui densidade demográfica de 9.334261445288442 hab/km².\nO município Rio Piracicaba/MG possui densidade demográfica de 37.92890842805061 hab/km².\nO município Rio Pomba/MG possui densidade demográfica de 67.78385230964267 hab/km².\nO município Rio Preto/MG possui densidade demográfica de 15.20078129488137 hab/km².\nO município Rio Vermelho/MG possui densidade demográfica de 13.830887122932209 hab/km².\nO município Ritápolis/MG possui densidade demográfica de 12.166201427830341 hab/km².\nO município Rochedo de Minas/MG possui densidade demográfica de 26.649874055415616 hab/km².\nO município Rodeiro/MG possui densidade demográfica de 94.49566533645245 hab/km².\nO município Romaria/MG possui densidade demográfica de 8.823240749828246 hab/km².\nO município Rosário da Limeira/MG possui densidade demográfica de 38.20618927671825 hab/km².\nO município Rubelita/MG possui densidade demográfica de 6.999909934252004 hab/km².\nO município Rubim/MG possui densidade demográfica de 10.276946030233017 hab/km².\nO município Sabará/MG possui densidade demográfica de 417.8740444120859 hab/km².\nO município Sabinópolis/MG possui densidade demográfica de 17.073091181874517 hab/km².\nO município Sacramento/MG possui densidade demográfica de 7.775431380906982 hab/km².\nO município Salinas/MG possui densidade demográfica de 20.754906894816305 hab/km².\nO município Salto da Divisa/MG possui densidade demográfica de 7.312990446946435 hab/km².\nO município Santa Bárbara/MG possui densidade demográfica de 40.750811332339275 hab/km².\nO município Santa Bárbara do Leste/MG possui densidade demográfica de 71.5270018621974 hab/km².\nO município Santa Bárbara do Monte Verde/MG possui densidade demográfica de 6.672570184046143 hab/km².\nO município Santa Bárbara do Tugúrio/MG possui densidade demográfica de 23.488898026315788 hab/km².\nO município Santa Cruz de Minas/MG possui densidade demográfica de 2203.0812324929975 hab/km².\nO município Santa Cruz de Salinas/MG possui densidade demográfica de 7.457977848262292 hab/km².\nO município Santa Cruz do Escalvado/MG possui densidade demográfica de 19.294244965794455 hab/km².\nO município Santa Efigênia de Minas/MG possui densidade demográfica de 34.85640675911192 hab/km².\nO município Santa Fé de Minas/MG possui densidade demográfica de 1.3600918610430341 hab/km².\nO município Santa Helena de Minas/MG possui densidade demográfica de 21.904279564446696 hab/km².\nO município Santa Juliana/MG possui densidade demográfica de 15.663599436292797 hab/km².\nO município Santa Luzia/MG possui densidade demográfica de 862.3719882717885 hab/km².\nO município Santa Margarida/MG possui densidade demográfica de 58.69862745864779 hab/km².\nO município Santa Maria de Itabira/MG possui densidade demográfica de 17.662024638457417 hab/km².\nO município Santa Maria do Salto/MG possui densidade demográfica de 11.99246499171603 hab/km².\nO município Santa Maria do Suaçuí/MG possui densidade demográfica de 23.067061934139893 hab/km².\nO município Santa Rita de Caldas/MG possui densidade demográfica de 17.94596528896046 hab/km².\nO município Santa Rita de Ibitipoca/MG possui densidade demográfica de 11.050797273540388 hab/km².\nO município Santa Rita de Jacutinga/MG possui densidade demográfica de 11.861547964080392 hab/km².\nO município Santa Rita de Minas/MG possui densidade demográfica de 96.0674981658107 hab/km².\nO município Santa Rita do Itueto/MG possui densidade demográfica de 11.744454522965285 hab/km².\nO município Santa Rita do Sapucaí/MG possui densidade demográfica de 106.96093152392554 hab/km².\nO município Santa Rosa da Serra/MG possui densidade demográfica de 11.338937150494145 hab/km².\nO município Santa Vitória/MG possui densidade demográfica de 6.043260388623824 hab/km².\nO município Santana da Vargem/MG possui densidade demográfica de 41.93342611922988 hab/km².\nO município Santana de Cataguases/MG possui densidade demográfica de 22.428633351910335 hab/km².\nO município Santana de Pirapama/MG possui densidade demográfica de 6.377455547327266 hab/km².\nO município Santana do Deserto/MG possui densidade demográfica de 21.132158107960144 hab/km².\nO município Santana do Garambéu/MG possui densidade demográfica de 11.00113261436943 hab/km².\nO município Santana do Jacaré/MG possui densidade demográfica de 43.39267212960347 hab/km².\nO município Santana do Manhuaçu/MG possui densidade demográfica de 24.706356517733763 hab/km².\nO município Santana do Paraíso/MG possui densidade demográfica de 98.7611837577426 hab/km².\nO município Santana do Riacho/MG possui densidade demográfica de 5.940550198608999 hab/km².\nO município Santana dos Montes/MG possui densidade demográfica de 19.443455257669026 hab/km².\nO município Santo Antônio do Amparo/MG possui densidade demográfica de 35.478328458344414 hab/km².\nO município Santo Antônio do Aventureiro/MG possui densidade demográfica de 17.512250655843193 hab/km².\nO município Santo Antônio do Grama/MG possui densidade demográfica de 31.372398433300052 hab/km².\nO município Santo Antônio do Itambé/MG possui densidade demográfica de 13.524563354484203 hab/km².\nO município Santo Antônio do Jacinto/MG possui densidade demográfica de 23.391870952362034 hab/km².\nO município Santo Antônio do Monte/MG possui densidade demográfica de 23.07289168398799 hab/km².\nO município Santo Antônio do Retiro/MG possui densidade demográfica de 8.734255108063646 hab/km².\nO município Santo Antônio do Rio Abaixo/MG possui densidade demográfica de 16.56567539852708 hab/km².\nO município Santo Hipólito/MG possui densidade demográfica de 7.518692239817954 hab/km².\nO município Santos Dumont/MG possui densidade demográfica de 72.61716114658675 hab/km².\nO município São Bento Abade/MG possui densidade demográfica de 56.927860696517406 hab/km².\nO município São Brás do Suaçuí/MG possui densidade demográfica de 31.93055808034903 hab/km².\nO município São Domingos das Dores/MG possui densidade demográfica de 88.8450796780023 hab/km².\nO município São Domingos do Prata/MG possui densidade demográfica de 23.336515320596423 hab/km².\nO município São Félix de Minas/MG possui densidade demográfica de 20.804625984251967 hab/km².\nO município São Francisco/MG possui densidade demográfica de 16.271575829025725 hab/km².\nO município São Francisco de Paula/MG possui densidade demográfica de 20.462723312922165 hab/km².\nO município São Francisco de Sales/MG possui densidade demográfica de 5.11666637138352 hab/km².\nO município São Francisco do Glória/MG possui densidade demográfica de 31.45616912702752 hab/km².\nO município São Geraldo/MG possui densidade demográfica de 55.30229550598124 hab/km².\nO município São Geraldo da Piedade/MG possui densidade demográfica de 28.81055533674675 hab/km².\nO município São Geraldo do Baixio/MG possui densidade demográfica de 12.407901761879339 hab/km².\nO município São Gonçalo do Abaeté/MG possui densidade demográfica de 2.3267475679470464 hab/km².\nO município São Gonçalo do Pará/MG possui densidade demográfica de 39.12994392804726 hab/km².\nO município São Gonçalo do Rio Abaixo/MG possui densidade demográfica de 26.873917704296197 hab/km².\nO município São Gonçalo do Rio Preto/MG possui densidade demográfica de 9.718247153851047 hab/km².\nO município São Gonçalo do Sapucaí/MG possui densidade demográfica de 46.268483393976936 hab/km².\nO município São Gotardo/MG possui densidade demográfica de 36.738676119110025 hab/km².\nO município São João Batista do Glória/MG possui densidade demográfica de 12.569582595681773 hab/km².\nO município São João da Lagoa/MG possui densidade demográfica de 4.6652371695958 hab/km².\nO município São João da Mata/MG possui densidade demográfica de 22.65637962502074 hab/km².\nO município São João da Ponte/MG possui densidade demográfica de 13.698881745988873 hab/km².\nO município São João das Missões/MG possui densidade demográfica de 17.271882878499714 hab/km².\nO município São João del Rei/MG possui densidade demográfica de 57.68440174004494 hab/km².\nO município São João do Manhuaçu/MG possui densidade demográfica de 71.59329140461216 hab/km².\nO município São João do Manteninha/MG possui densidade demográfica de 37.613282099615745 hab/km².\nO município São João do Oriente/MG possui densidade demográfica de 65.55111555111554 hab/km².\nO município São João do Pacuí/MG possui densidade demográfica de 9.761492594729756 hab/km².\nO município São João do Paraíso/MG possui densidade demográfica de 11.590793423280259 hab/km².\nO município São João Evangelista/MG possui densidade demográfica de 32.52540884185871 hab/km².\nO município São João Nepomuceno/MG possui densidade demográfica de 61.50013499251405 hab/km².\nO município São Joaquim de Bicas/MG possui densidade demográfica de 356.86137506987143 hab/km².\nO município São José da Barra/MG possui densidade demográfica de 21.56881463802705 hab/km².\nO município São José da Lapa/MG possui densidade demográfica de 413.0815773002295 hab/km².\nO município São José da Safira/MG possui densidade demográfica de 19.052739854123807 hab/km².\nO município São José da Varginha/MG possui densidade demográfica de 20.42822384428224 hab/km².\nO município São José do Alegre/MG possui densidade demográfica de 45.00506813830386 hab/km².\nO município São José do Divino/MG possui densidade demográfica de 11.664131426832979 hab/km².\nO município São José do Goiabal/MG possui densidade demográfica de 30.545769876971438 hab/km².\nO município São José do Jacuri/MG possui densidade demográfica de 18.985948138490514 hab/km².\nO município São José do Mantimento/MG possui densidade demográfica de 47.38574040219378 hab/km².\nO município São Lourenço/MG possui densidade demográfica de 717.9765598069631 hab/km².\nO município São Miguel do Anta/MG possui densidade demográfica de 44.441522582341726 hab/km².\nO município São Pedro da União/MG possui densidade demográfica de 19.32293064448108 hab/km².\nO município São Pedro do Suaçuí/MG possui densidade demográfica de 18.077959170426144 hab/km².\nO município São Pedro dos Ferros/MG possui densidade demográfica de 20.74684675737412 hab/km².\nO município São Romão/MG possui densidade demográfica de 4.221857025472473 hab/km².\nO município São Roque de Minas/MG possui densidade demográfica de 3.1855236389104613 hab/km².\nO município São Sebastião da Bela Vista/MG possui densidade demográfica de 29.600382866714526 hab/km².\nO município São Sebastião da Vargem Alegre/MG possui densidade demográfica de 38.000814885237 hab/km².\nO município São Sebastião do Anta/MG possui densidade demográfica de 71.18580997271148 hab/km².\nO município São Sebastião do Maranhão/MG possui densidade demográfica de 20.560801807543015 hab/km².\nO município São Sebastião do Oeste/MG possui densidade demográfica de 14.224803352201722 hab/km².\nO município São Sebastião do Paraíso/MG possui densidade demográfica de 79.73690991864332 hab/km².\nO município São Sebastião do Rio Preto/MG possui densidade demográfica de 12.6015625 hab/km².\nO município São Sebastião do Rio Verde/MG possui densidade demográfica de 23.225096312603192 hab/km².\nO município São Thomé das Letras/MG possui densidade demográfica de 17.99864773495605 hab/km².\nO município São Tiago/MG possui densidade demográfica de 18.45038434661076 hab/km².\nO município São Tomás de Aquino/MG possui densidade demográfica de 25.520814593602704 hab/km².\nO município São Vicente de Minas/MG possui densidade demográfica de 17.84795619508468 hab/km².\nO município Sapucaí-Mirim/MG possui densidade demográfica de 21.892100463027923 hab/km².\nO município Sardoá/MG possui densidade demográfica de 39.42212825933756 hab/km².\nO município Sarzedo/MG possui densidade demográfica de 415.483663286657 hab/km².\nO município Sem-Peixe/MG possui densidade demográfica de 16.118439676159202 hab/km².\nO município Senador Amaral/MG possui densidade demográfica de 34.54003970880212 hab/km².\nO município Senador Cortes/MG possui densidade demográfica de 20.21557860484035 hab/km².\nO município Senador Firmino/MG possui densidade demográfica de 43.42342342342342 hab/km².\nO município Senador José Bento/MG possui densidade demográfica de 19.895622537011395 hab/km².\nO município Senador Modestino Gonçalves/MG possui densidade demográfica de 4.804319055521711 hab/km².\nO município Senhora de Oliveira/MG possui densidade demográfica de 33.28257686676427 hab/km².\nO município Senhora do Porto/MG possui densidade demográfica de 9.17053470747122 hab/km².\nO município Senhora dos Remédios/MG possui densidade demográfica de 42.872760911613824 hab/km².\nO município Sericita/MG possui densidade demográfica de 42.93717245949039 hab/km².\nO município Seritinga/MG possui densidade demográfica de 15.58769713339723 hab/km².\nO município Serra Azul de Minas/MG possui densidade demográfica de 19.30466605672461 hab/km².\nO município Serra da Saudade/MG possui densidade demográfica de 2.428052195674194 hab/km².\nO município Serra do Salitre/MG possui densidade demográfica de 8.144247917422623 hab/km².\nO município Serra dos Aimorés/MG possui densidade demográfica de 39.39124326855537 hab/km².\nO município Serrania/MG possui densidade demográfica de 36.039566110765996 hab/km².\nO município Serranópolis de Minas/MG possui densidade demográfica de 8.017030528127547 hab/km².\nO município Serranos/MG possui densidade demográfica de 9.358727775953465 hab/km².\nO município Serro/MG possui densidade demográfica de 17.108580156182 hab/km².\nO município Sete Lagoas/MG possui densidade demográfica de 398.31857748679414 hab/km².\nO município Setubinha/MG possui densidade demográfica de 20.35873265252684 hab/km².\nO município Silveirânia/MG possui densidade demográfica de 13.92099580845929 hab/km².\nO município Silvianópolis/MG possui densidade demográfica de 19.306787968094305 hab/km².\nO município Simão Pereira/MG possui densidade demográfica de 18.69702999484118 hab/km².\nO município Simonésia/MG possui densidade demográfica de 37.60841862950631 hab/km².\nO município Sobrália/MG possui densidade demográfica de 28.192852652449346 hab/km².\nO município Soledade de Minas/MG possui densidade demográfica de 28.8312084116422 hab/km².\nO município Tabuleiro/MG possui densidade demográfica de 19.32442675762744 hab/km².\nO município Taiobeiras/MG possui densidade demográfica de 25.882146116045643 hab/km².\nO município Taparuba/MG possui densidade demográfica de 16.247151439817692 hab/km².\nO município Tapira/MG possui densidade demográfica de 3.4869620521517914 hab/km².\nO município Tapiraí/MG possui densidade demográfica de 4.59158658560502 hab/km².\nO município Taquaraçu de Minas/MG possui densidade demográfica de 11.523508686672336 hab/km².\nO município Tarumirim/MG possui densidade demográfica de 19.532627263409633 hab/km².\nO município Teixeiras/MG possui densidade demográfica de 68.10003598416696 hab/km².\nO município Teófilo Otoni/MG possui densidade demográfica de 41.5588461170723 hab/km².\nO município Timóteo/MG possui densidade demográfica de 562.7025903864801 hab/km².\nO município Tiradentes/MG possui densidade demográfica de 83.8169777242625 hab/km².\nO município Tiros/MG possui densidade demográfica de 3.301510204276761 hab/km².\nO município Tocantins/MG possui densidade demográfica de 91.00477368148617 hab/km².\nO município Tocos do Moji/MG possui densidade demográfica de 34.43466131985006 hab/km².\nO município Toledo/MG possui densidade demográfica de 42.14066383974265 hab/km².\nO município Tombos/MG possui densidade demográfica de 33.447900957458 hab/km².\nO município Três Corações/MG possui densidade demográfica de 87.8761895560601 hab/km².\nO município Três Marias/MG possui densidade demográfica de 10.573322131989173 hab/km².\nO município Três Pontas/MG possui densidade demográfica de 78.08173502080344 hab/km².\nO município Tumiritinga/MG possui densidade demográfica de 12.584238206651069 hab/km².\nO município Tupaciguara/MG possui densidade demográfica de 13.261255729292309 hab/km².\nO município Turmalina/MG possui densidade demográfica de 15.657656251355032 hab/km².\nO município Turvolândia/MG possui densidade demográfica de 21.076923076923077 hab/km².\nO município Ubá/MG possui densidade demográfica de 249.1569517732237 hab/km².\nO município Ubaí/MG possui densidade demográfica de 14.236094184175888 hab/km².\nO município Ubaporanga/MG possui densidade demográfica de 63.68685532927797 hab/km².\nO município Uberaba/MG possui densidade demográfica de 65.42675001547317 hab/km².\nO município Uberlândia/MG possui densidade demográfica de 146.77574169969455 hab/km².\nO município Umburatiba/MG possui densidade demográfica de 6.665352487494764 hab/km².\nO município Unaí/MG possui densidade demográfica de 9.18243044070694 hab/km².\nO município União de Minas/MG possui densidade demográfica de 3.8504109254756362 hab/km².\nO município Uruana de Minas/MG possui densidade demográfica de 5.405179615705931 hab/km².\nO município Urucânia/MG possui densidade demográfica de 74.14799337128035 hab/km².\nO município Urucuia/MG possui densidade demográfica de 6.550020703535008 hab/km².\nO município Vargem Alegre/MG possui densidade demográfica de 55.38316475227156 hab/km².\nO município Vargem Bonita/MG possui densidade demográfica de 5.277025543438484 hab/km².\nO município Vargem Grande do Rio Pardo/MG possui densidade demográfica de 9.62950906390511 hab/km².\nO município Varginha/MG possui densidade demográfica de 311.2822458270106 hab/km².\nO município Varjão de Minas/MG possui densidade demográfica de 9.291546442384432 hab/km².\nO município Várzea da Palma/MG possui densidade demográfica de 16.128145999603653 hab/km².\nO município Varzelândia/MG possui densidade demográfica de 23.45550252150333 hab/km².\nO município Vazante/MG possui densidade demográfica de 10.307828995505382 hab/km².\nO município Verdelândia/MG possui densidade demográfica de 5.313960447732685 hab/km².\nO município Veredinha/MG possui densidade demográfica de 8.784372081242381 hab/km².\nO município Veríssimo/MG possui densidade demográfica de 3.3755887654823518 hab/km².\nO município Vermelho Novo/MG possui densidade demográfica de 40.688996876084694 hab/km².\nO município Vespasiano/MG possui densidade demográfica de 1467.6635776467285 hab/km².\nO município Viçosa/MG possui densidade demográfica de 241.19965266181282 hab/km².\nO município Vieiras/MG possui densidade demográfica de 33.108527819682315 hab/km².\nO município Virgem da Lapa/MG possui densidade demográfica de 15.673660102887526 hab/km².\nO município Virgínia/MG possui densidade demográfica de 26.40879578586304 hab/km².\nO município Virginópolis/MG possui densidade demográfica de 24.033827407474767 hab/km².\nO município Virgolândia/MG possui densidade demográfica de 20.13379830617038 hab/km².\nO município Visconde do Rio Branco/MG possui densidade demográfica de 155.91534826381755 hab/km².\nO município Volta Grande/MG possui densidade demográfica de 24.359775140537163 hab/km².\nO município Wenceslau Braz/MG possui densidade demográfica de 24.909747292418775 hab/km².\nO município Abaetetuba/PA possui densidade demográfica de 87.60655900559416 hab/km².\nO município Abel Figueiredo/PA possui densidade demográfica de 11.037491656763313 hab/km².\nO município Acará/PA possui densidade demográfica de 12.332261309771836 hab/km².\nO município Afuá/PA possui densidade demográfica de 4.185218803745462 hab/km².\nO município Água Azul do Norte/PA possui densidade demográfica de 3.5222295317938253 hab/km².\nO município Alenquer/PA possui densidade demográfica de 2.2256290322239582 hab/km².\nO município Almeirim/PA possui densidade demográfica de 0.4607510403702018 hab/km².\nO município Altamira/PA possui densidade demográfica de 0.6210285436189575 hab/km².\nO município Anajás/PA possui densidade demográfica de 3.57698558890454 hab/km².\nO município Ananindeua/PA possui densidade demográfica de 2477.5853018372704 hab/km².\nO município Anapu/PA possui densidade demográfica de 1.7269541196636378 hab/km².\nO município Augusto Corrêa/PA possui densidade demográfica de 37.100793374498416 hab/km².\nO município Aurora do Pará/PA possui densidade demográfica de 14.651404097492053 hab/km².\nO município Aveiro/PA possui densidade demográfica de 0.9282513101761504 hab/km².\nO município Bagre/PA possui densidade demográfica de 5.426941864590251 hab/km².\nO município Baião/PA possui densidade demográfica de 9.813479498709523 hab/km².\nO município Bannach/PA possui densidade demográfica de 1.1604349517190062 hab/km².\nO município Barcarena/PA possui densidade demográfica de 76.20846497855518 hab/km².\nO município Belém/PA possui densidade demográfica de 1315.2594368563634 hab/km².\nO município Belterra/PA possui densidade demográfica de 3.70996857962632 hab/km².\nO município Benevides/PA possui densidade demográfica de 274.9880210828941 hab/km².\nO município Bom Jesus do Tocantins/PA possui densidade demográfica de 5.431602567744134 hab/km².\nO município Bonito/PA possui densidade demográfica de 23.230050789105906 hab/km².\nO município Bragança/PA possui densidade demográfica de 54.12561605789869 hab/km².\nO município Brasil Novo/PA possui densidade demográfica de 2.465980781381138 hab/km².\nO município Brejo Grande do Araguaia/PA possui densidade demográfica de 5.678784303986092 hab/km².\nO município Breu Branco/PA possui densidade demográfica de 13.316539571885924 hab/km².\nO município Breves/PA possui densidade demográfica de 9.723040968492782 hab/km².\nO município Bujaru/PA possui densidade demográfica de 25.562840116597194 hab/km².\nO município Cachoeira do Arari/PA possui densidade demográfica de 6.593963086966899 hab/km².\nO município Cachoeira do Piriá/PA possui densidade demográfica de 10.75723912151651 hab/km².\nO município Cametá/PA possui densidade demográfica de 39.23449634415861 hab/km².\nO município Canaã dos Carajás/PA possui densidade demográfica de 8.490946825111795 hab/km².\nO município Capanema/PA possui densidade demográfica de 103.53023475247684 hab/km².\nO município Capitão Poço/PA possui densidade demográfica de 17.896915038540463 hab/km².\nO município Castanhal/PA possui densidade demográfica de 168.28718327518004 hab/km².\nO município Chaves/PA possui densidade demográfica de 1.605278120834913 hab/km².\nO município Colares/PA possui densidade demográfica de 18.663802292592532 hab/km².\nO município Conceição do Araguaia/PA possui densidade demográfica de 7.8149337505232035 hab/km².\nO município Concórdia do Pará/PA possui densidade demográfica de 40.83652941602142 hab/km².\nO município Cumaru do Norte/PA possui densidade demográfica de 0.612584138132865 hab/km².\nO município Curionópolis/PA possui densidade demográfica de 7.720560297879886 hab/km².\nO município Curralinho/PA possui densidade demográfica de 7.892459741516345 hab/km².\nO município Curuá/PA possui densidade demográfica de 8.562285139327537 hab/km².\nO município Curuçá/PA possui densidade demográfica de 50.98115002675864 hab/km².\nO município Dom Eliseu/PA possui densidade demográfica de 9.740131566460802 hab/km².\nO município Eldorado dos Carajás/PA possui densidade demográfica de 10.750389788719295 hab/km².\nO município Faro/PA possui densidade demográfica de 0.6946951862389694 hab/km².\nO município Floresta do Araguaia/PA possui densidade demográfica de 5.158682921589065 hab/km².\nO município Garrafão do Norte/PA possui densidade demográfica de 15.655741293158979 hab/km².\nO município Goianésia do Pará/PA possui densidade demográfica de 4.333199030169806 hab/km².\nO município Gurupá/PA possui densidade demográfica de 3.4030006639258743 hab/km².\nO município Igarapé-Açu/PA possui densidade demográfica de 45.658922618896156 hab/km².\nO município Igarapé-Miri/PA possui densidade demográfica de 29.08445343642956 hab/km².\nO município Inhangapi/PA possui densidade demográfica de 21.28963834977198 hab/km².\nO município Ipixuna do Pará/PA possui densidade demográfica de 9.83767802498677 hab/km².\nO município Irituia/PA possui densidade demográfica de 22.738081429068554 hab/km².\nO município Itaituba/PA possui densidade demográfica de 1.571435916835897 hab/km².\nO município Itupiranga/PA possui densidade demográfica de 6.499909265225993 hab/km².\nO município Jacareacanga/PA possui densidade demográfica de 0.2645813337615762 hab/km².\nO município Jacundá/PA possui densidade demográfica de 25.573613766730404 hab/km².\nO município Juruti/PA possui densidade demográfica de 5.668729766790629 hab/km².\nO município Limoeiro do Ajuru/PA possui densidade demográfica de 16.790476382206297 hab/km².\nO município Mãe do Rio/PA possui densidade demográfica de 59.4347057445313 hab/km².\nO município Magalhães Barata/PA possui densidade demográfica de 24.948504319488425 hab/km².\nO município Marabá/PA possui densidade demográfica de 15.445697567888782 hab/km².\nO município Maracanã/PA possui densidade demográfica de 33.162704812659236 hab/km².\nO município Marapanim/PA possui densidade demográfica de 33.423786730989086 hab/km².\nO município Marituba/PA possui densidade demográfica de 1047.4743564931293 hab/km².\nO município Medicilândia/PA possui densidade demográfica de 3.3034234578362627 hab/km².\nO município Melgaço/PA possui densidade demográfica de 3.6622271561052373 hab/km².\nO município Mocajuba/PA possui densidade demográfica de 30.696707662980444 hab/km².\nO município Moju/PA possui densidade demográfica de 7.699243688793003 hab/km².\nO município Monte Alegre/PA possui densidade demográfica de 3.055326631615596 hab/km².\nO município Muaná/PA possui densidade demográfica de 9.083400831219874 hab/km².\nO município Nova Esperança do Piriá/PA possui densidade demográfica de 7.175427418120464 hab/km².\nO município Nova Ipixuna/PA possui densidade demográfica de 9.362733189274891 hab/km².\nO município Nova Timboteua/PA possui densidade demográfica de 27.906501990405225 hab/km².\nO município Novo Progresso/PA possui densidade demográfica de 0.658348996767214 hab/km².\nO município Novo Repartimento/PA possui densidade demográfica de 4.029558320144999 hab/km².\nO município Óbidos/PA possui densidade demográfica de 1.7605460394226988 hab/km².\nO município Oeiras do Pará/PA possui densidade demográfica de 7.422857573027991 hab/km².\nO município Oriximiná/PA possui densidade demográfica de 0.5835695172517495 hab/km².\nO município Ourém/PA possui densidade demográfica de 29.003005032095167 hab/km².\nO município Ourilândia do Norte/PA possui densidade demográfica de 1.898536976677536 hab/km².\nO município Pacajá/PA possui densidade demográfica de 3.3787935258736024 hab/km².\nO município Palestina do Pará/PA possui densidade demográfica de 7.593766508188061 hab/km².\nO município Paragominas/PA possui densidade demográfica de 5.057271000012925 hab/km².\nO município Parauapebas/PA possui densidade demográfica de 22.350175205229 hab/km².\nO município Pau d`Arco/PA possui densidade demográfica de 3.6095056897727678 hab/km².\nO município Peixe-Boi/PA possui densidade demográfica de 17.4448047621163 hab/km².\nO município Piçarra/PA possui densidade demográfica de 3.8328714688498065 hab/km².\nO município Placas/PA possui densidade demográfica de 3.336590833367024 hab/km².\nO município Ponta de Pedras/PA possui densidade demográfica de 7.725955752343878 hab/km².\nO município Portel/PA possui densidade demográfica de 2.055232704719645 hab/km².\nO município Porto de Moz/PA possui densidade demográfica de 1.948915859592654 hab/km².\nO município Prainha/PA possui densidade demográfica de 1.9847852740821492 hab/km².\nO município Primavera/PA possui densidade demográfica de 39.706109822119096 hab/km².\nO município Quatipuru/PA possui densidade demográfica de 38.05771058845175 hab/km².\nO município Redenção/PA possui densidade demográfica de 19.75934996770237 hab/km².\nO município Rio Maria/PA possui densidade demográfica de 4.301015163040969 hab/km².\nO município Rondon do Pará/PA possui densidade demográfica de 5.695063566823987 hab/km².\nO município Rurópolis/PA possui densidade demográfica de 5.709325312049586 hab/km².\nO município Salinópolis/PA possui densidade demográfica de 157.40304534365274 hab/km².\nO município Salvaterra/PA possui densidade demográfica de 19.424100397470816 hab/km².\nO município Santa Bárbara do Pará/PA possui densidade demográfica de 61.625022469890354 hab/km².\nO município Santa Cruz do Arari/PA possui densidade demográfica de 7.574420656666511 hab/km².\nO município Santa Isabel do Pará/PA possui densidade demográfica de 82.86096480227405 hab/km².\nO município Santa Luzia do Pará/PA possui densidade demográfica de 14.323216234551516 hab/km².\nO município Santa Maria das Barreiras/PA possui densidade demográfica de 1.66560021529088 hab/km².\nO município Santa Maria do Pará/PA possui densidade demográfica de 50.30476481768728 hab/km².\nO município Santana do Araguaia/PA possui densidade demográfica de 4.844300508300868 hab/km².\nO município Santarém/PA possui densidade demográfica de 12.87127588084217 hab/km².\nO município Santarém Novo/PA possui densidade demográfica de 26.75700405211102 hab/km².\nO município Santo Antônio do Tauá/PA possui densidade demográfica de 49.61404683518405 hab/km².\nO município São Caetano de Odivelas/PA possui densidade demográfica de 22.719141323792485 hab/km².\nO município São Domingos do Araguaia/PA possui densidade demográfica de 16.610890079427776 hab/km².\nO município São Domingos do Capim/PA possui densidade demográfica de 17.79460426293039 hab/km².\nO município São Félix do Xingu/PA possui densidade demográfica de 1.0846270326960308 hab/km².\nO município São Francisco do Pará/PA possui densidade demográfica de 31.403786804570856 hab/km².\nO município São Geraldo do Araguaia/PA possui densidade demográfica de 8.07573586501619 hab/km².\nO município São João da Ponta/PA possui densidade demográfica de 26.873213556553697 hab/km².\nO município São João de Pirabas/PA possui densidade demográfica de 29.26410975990022 hab/km².\nO município São João do Araguaia/PA possui densidade demográfica de 10.278227035135831 hab/km².\nO município São Miguel do Guamá/PA possui densidade demográfica de 46.44922445008917 hab/km².\nO município São Sebastião da Boa Vista/PA possui densidade demográfica de 14.032164190534537 hab/km².\nO município Sapucaia/PA possui densidade demográfica de 3.887720595598487 hab/km².\nO município Senador José Porfírio/PA possui densidade demográfica de 0.9046513434193809 hab/km².\nO município Soure/PA possui densidade demográfica de 6.539353826208591 hab/km².\nO município Tailândia/PA possui densidade demográfica de 17.899111105091844 hab/km².\nO município Terra Alta/PA possui densidade demográfica de 49.71658349886149 hab/km².\nO município Terra Santa/PA possui densidade demográfica de 8.936942067270934 hab/km².\nO município Tomé-Açu/PA possui densidade demográfica de 10.984265435265948 hab/km².\nO município Tracuateua/PA possui densidade demográfica de 29.386579896603767 hab/km².\nO município Trairão/PA possui densidade demográfica de 1.407294916475483 hab/km².\nO município Tucumã/PA possui densidade demográfica de 13.408474920301362 hab/km².\nO município Tucuruí/PA possui densidade demográfica de 46.55760021858028 hab/km².\nO município Ulianópolis/PA possui densidade demográfica de 8.51749150530513 hab/km².\nO município Uruará/PA possui densidade demográfica de 4.150446143538772 hab/km².\nO município Vigia/PA possui densidade demográfica de 88.83468130889663 hab/km².\nO município Viseu/PA possui densidade demográfica de 11.539204935026358 hab/km².\nO município Vitória do Xingu/PA possui densidade demográfica de 4.347249105044764 hab/km².\nO município Xinguara/PA possui densidade demográfica de 10.735415520088058 hab/km².\nO município Água Branca/PB possui densidade demográfica de 39.9349139934914 hab/km².\nO município Aguiar/PB possui densidade demográfica de 16.04247048243451 hab/km².\nO município Alagoa Grande/PB possui densidade demográfica de 88.8414025455453 hab/km².\nO município Alagoa Nova/PB possui densidade demográfica de 160.9766072304924 hab/km².\nO município Alagoinha/PB possui densidade demográfica de 139.98762631470404 hab/km².\nO município Alcantil/PB possui densidade demográfica de 17.155113134025346 hab/km².\nO município Algodão de Jandaíra/PB possui densidade demográfica de 10.742338251986379 hab/km².\nO município Alhandra/PB possui densidade demográfica de 98.58206503887003 hab/km².\nO município Amparo/PB possui densidade demográfica de 17.117560255779637 hab/km².\nO município Aparecida/PB possui densidade demográfica de 25.957864123634643 hab/km².\nO município Araçagi/PB possui densidade demográfica de 74.51116110053643 hab/km².\nO município Arara/PB possui densidade demográfica de 127.66622944203411 hab/km².\nO município Araruna/PB possui densidade demográfica de 76.83135275923816 hab/km².\nO município Areia/PB possui densidade demográfica de 88.42257597684515 hab/km².\nO município Areia de Baraúnas/PB possui densidade demográfica de 20.002075980900976 hab/km².\nO município Areial/PB possui densidade demográfica de 195.2323476161738 hab/km².\nO município Aroeiras/PB possui densidade demográfica de 50.92607419268749 hab/km².\nO município Assunção/PB possui densidade demográfica de 27.857312346753144 hab/km².\nO município Baía da Traição/PB possui densidade demográfica de 78.26511673341798 hab/km².\nO município Bananeiras/PB possui densidade demográfica de 84.71678362346373 hab/km².\nO município Baraúna/PB possui densidade demográfica de 83.43218663503362 hab/km².\nO município Barra de Santa Rosa/PB possui densidade demográfica de 18.25155351571565 hab/km².\nO município Barra de Santana/PB possui densidade demográfica de 21.771775755485393 hab/km².\nO município Barra de São Miguel/PB possui densidade demográfica de 9.42692495085768 hab/km².\nO município Bayeux/PB possui densidade demográfica de 3119.0491085392555 hab/km².\nO município Belém/PB possui densidade demográfica de 170.67398901647528 hab/km².\nO município Belém do Brejo do Cruz/PB possui densidade demográfica de 11.844985407269833 hab/km².\nO município Bernardino Batista/PB possui densidade demográfica de 60.734742247679236 hab/km².\nO município Boa Ventura/PB possui densidade demográfica de 33.71438621174815 hab/km².\nO município Boa Vista/PB possui densidade demográfica de 13.067108742183237 hab/km².\nO município Bom Jesus/PB possui densidade demográfica de 50.38841066554692 hab/km².\nO município Bom Sucesso/PB possui densidade demográfica de 27.34926670287887 hab/km².\nO município Bonito de Santa Fé/PB possui densidade demográfica de 47.317479087285946 hab/km².\nO município Boqueirão/PB possui densidade demográfica de 45.400290338190224 hab/km².\nO município Borborema/PB possui densidade demográfica de 196.72825250192454 hab/km².\nO município Brejo do Cruz/PB possui densidade demográfica de 32.89632006417327 hab/km².\nO município Brejo dos Santos/PB possui densidade demográfica de 66.0415556739478 hab/km².\nO município Caaporã/PB possui densidade demográfica de 135.59299460611308 hab/km².\nO município Cabaceiras/PB possui densidade demográfica de 11.116753510553739 hab/km².\nO município Cabedelo/PB possui densidade demográfica de 1815.2882205513783 hab/km².\nO município Cachoeira dos Índios/PB possui densidade demográfica de 49.44320712694878 hab/km².\nO município Cacimba de Areia/PB possui densidade demográfica de 16.140303112805157 hab/km².\nO município Cacimba de Dentro/PB possui densidade demográfica de 102.31535219011546 hab/km².\nO município Cacimbas/PB possui densidade demográfica de 53.8485854275328 hab/km².\nO município Caiçara/PB possui densidade demográfica de 56.44593855054335 hab/km².\nO município Cajazeiras/PB possui densidade demográfica de 103.27973140130766 hab/km².\nO município Cajazeirinhas/PB possui densidade demográfica de 10.535273889332732 hab/km².\nO município Caldas Brandão/PB possui densidade demográfica de 100.93106535362578 hab/km².\nO município Camalaú/PB possui densidade demográfica de 10.574040353878129 hab/km².\nO município Campina Grande/PB possui densidade demográfica de 648.3102763472349 hab/km².\nO município Capim/PB possui densidade demográfica de 71.65152871945759 hab/km².\nO município Caraúbas/PB possui densidade demográfica de 7.841914722445696 hab/km².\nO município Carrapateira/PB possui densidade demográfica de 43.61702127659574 hab/km².\nO município Casserengue/PB possui densidade demográfica de 35.04816764326149 hab/km².\nO município Catingueira/PB possui densidade demográfica de 9.088676928888468 hab/km².\nO município Catolé do Rocha/PB possui densidade demográfica de 52.089257575483146 hab/km².\nO município Caturité/PB possui densidade demográfica de 38.47391598915989 hab/km².\nO município Conceição/PB possui densidade demográfica de 31.69094297942841 hab/km².\nO município Condado/PB possui densidade demográfica de 23.43727751673074 hab/km².\nO município Conde/PB possui densidade demográfica de 123.7351835790691 hab/km².\nO município Congo/PB possui densidade demográfica de 14.055237352685397 hab/km².\nO município Coremas/PB possui densidade demográfica de 39.919365464175605 hab/km².\nO município Coxixola/PB possui densidade demográfica de 10.425005886508124 hab/km².\nO município Cruz do Espírito Santo/PB possui densidade demográfica de 83.11349693251535 hab/km².\nO município Cubati/PB possui densidade demográfica de 50.12776520405929 hab/km².\nO município Cuité/PB possui densidade demográfica de 26.930335382292675 hab/km².\nO município Cuité de Mamanguape/PB possui densidade demográfica de 57.187644075610876 hab/km².\nO município Cuitegi/PB possui densidade demográfica de 175.29262086513995 hab/km².\nO município Curral de Cima/PB possui densidade demográfica de 61.21034077555817 hab/km².\nO município Curral Velho/PB possui densidade demográfica de 11.235199138858988 hab/km².\nO município Damião/PB possui densidade demográfica de 26.38806613172492 hab/km².\nO município Desterro/PB possui densidade demográfica de 44.545403868666035 hab/km².\nO município Diamante/PB possui densidade demográfica de 24.58474229868827 hab/km².\nO município Dona Inês/PB possui densidade demográfica de 63.29060600589758 hab/km².\nO município Duas Estradas/PB possui densidade demográfica de 138.53769992383852 hab/km².\nO município Emas/PB possui densidade demográfica de 13.769198837691988 hab/km².\nO município Esperança/PB possui densidade demográfica de 189.85834656246183 hab/km².\nO município Fagundes/PB possui densidade demográfica de 60.334338464793944 hab/km².\nO município Frei Martinho/PB possui densidade demográfica de 12.004747871643746 hab/km².\nO município Gado Bravo/PB possui densidade demográfica de 43.53204095421236 hab/km².\nO município Guarabira/PB possui densidade demográfica de 333.811994690479 hab/km².\nO município Gurinhém/PB possui densidade demográfica de 40.084375993296156 hab/km².\nO município Gurjão/PB possui densidade demográfica de 9.204545454545455 hab/km².\nO município Ibiara/PB possui densidade demográfica de 24.667675569552944 hab/km².\nO município Igaracy/PB possui densidade demográfica de 32.01914074690524 hab/km².\nO município Imaculada/PB possui densidade demográfica de 35.81298504637516 hab/km².\nO município Ingá/PB possui densidade demográfica de 63.12719191638598 hab/km².\nO município Itabaiana/PB possui densidade demográfica de 111.86200594014166 hab/km².\nO município Itaporanga/PB possui densidade demográfica de 49.54920309362048 hab/km².\nO município Itapororoca/PB possui densidade demográfica de 116.36201821044705 hab/km².\nO município Itatuba/PB possui densidade demográfica de 41.76971582998935 hab/km².\nO município Jacaraú/PB possui densidade demográfica de 55.104541322477374 hab/km².\nO município Jericó/PB possui densidade demográfica de 42.038926997936535 hab/km².\nO município João Pessoa/PB possui densidade demográfica de 3421.198222054095 hab/km².\nO município Joca Claudino/PB possui densidade demográfica de 35.33306309958113 hab/km².\nO município Juarez Távora/PB possui densidade demográfica de 105.2936194240542 hab/km².\nO município Juazeirinho/PB possui densidade demográfica de 35.88218937822172 hab/km².\nO município Junco do Seridó/PB possui densidade demográfica de 38.98016664710715 hab/km².\nO município Juripiranga/PB possui densidade demográfica de 129.8287888395688 hab/km².\nO município Juru/PB possui densidade demográfica de 24.365205316405476 hab/km².\nO município Lagoa/PB possui densidade demográfica de 26.312535132096684 hab/km².\nO município Lagoa de Dentro/PB possui densidade demográfica de 87.20861436516388 hab/km².\nO município Lagoa Seca/PB possui densidade demográfica de 240.72869225764475 hab/km².\nO município Lastro/PB possui densidade demográfica de 27.671179507158858 hab/km².\nO município Livramento/PB possui densidade demográfica de 27.5305510721697 hab/km².\nO município Logradouro/PB possui densidade demográfica de 103.73684210526316 hab/km².\nO município Lucena/PB possui densidade demográfica de 131.88666516752866 hab/km².\nO município Mãe d`Água/PB possui densidade demográfica de 16.488205128205127 hab/km².\nO município Malta/PB possui densidade demográfica de 35.925499231950845 hab/km².\nO município Mamanguape/PB possui densidade demográfica de 124.22694035767775 hab/km².\nO município Manaíra/PB possui densidade demográfica de 30.51592591542105 hab/km².\nO município Marcação/PB possui densidade demográfica de 61.912123677786816 hab/km².\nO município Mari/PB possui densidade demográfica de 136.77819403177884 hab/km².\nO município Marizópolis/PB possui densidade demográfica de 97.0444898600849 hab/km².\nO município Massaranduba/PB possui densidade demográfica de 62.643231695474846 hab/km².\nO município Mataraca/PB possui densidade demográfica de 40.18990775908844 hab/km².\nO município Matinhas/PB possui densidade demográfica de 113.35257082896118 hab/km².\nO município Mato Grosso/PB possui densidade demográfica de 32.35153256704981 hab/km².\nO município Maturéia/PB possui densidade demográfica de 70.96427291193692 hab/km².\nO município Mogeiro/PB possui densidade demográfica de 64.40651747963288 hab/km².\nO município Montadas/PB possui densidade demográfica de 157.96138018360242 hab/km².\nO município Monte Horebe/PB possui densidade demográfica de 38.805199276921755 hab/km².\nO município Monteiro/PB possui densidade demográfica de 31.278640658583072 hab/km².\nO município Mulungu/PB possui densidade demográfica de 48.481900568327276 hab/km².\nO município Natuba/PB possui densidade demográfica de 51.53140850565743 hab/km².\nO município Nazarezinho/PB possui densidade demográfica de 38.01765105227427 hab/km².\nO município Nova Floresta/PB possui densidade demográfica de 222.3089911355002 hab/km².\nO município Nova Olinda/PB possui densidade demográfica de 72.04747774480713 hab/km².\nO município Nova Palmeira/PB possui densidade demográfica de 14.051876913162557 hab/km².\nO município Olho d`Água/PB possui densidade demográfica de 11.626658614731687 hab/km².\nO município Olivedos/PB possui densidade demográfica de 11.408530447911424 hab/km².\nO município Ouro Velho/PB possui densidade demográfica de 22.627511591962904 hab/km².\nO município Parari/PB possui densidade demográfica de 9.775840597758407 hab/km².\nO município Passagem/PB possui densidade demográfica de 19.958884519127636 hab/km².\nO município Patos/PB possui densidade demográfica de 212.81444214264576 hab/km².\nO município Paulista/PB possui densidade demográfica de 20.433350667360028 hab/km².\nO município Pedra Branca/PB possui densidade demográfica de 32.9496148056318 hab/km².\nO município Pedra Lavrada/PB possui densidade demográfica de 21.25511828935396 hab/km².\nO município Pedras de Fogo/PB possui densidade demográfica de 67.51417368066136 hab/km².\nO município Pedro Régis/PB possui densidade demográfica de 78.37139749864056 hab/km².\nO município Piancó/PB possui densidade demográfica de 27.384283032900097 hab/km².\nO município Picuí/PB possui densidade demográfica de 27.53982407883203 hab/km².\nO município Pilar/PB possui densidade demográfica de 109.287109375 hab/km².\nO município Pilões/PB possui densidade demográfica de 108.2699767261443 hab/km².\nO município Pilõezinhos/PB possui densidade demográfica de 117.42596810933941 hab/km².\nO município Pirpirituba/PB possui densidade demográfica de 129.33366733466934 hab/km².\nO município Pitimbu/PB possui densidade demográfica de 124.7727939020815 hab/km².\nO município Pocinhos/PB possui densidade demográfica de 27.117564641446947 hab/km².\nO município Poço Dantas/PB possui densidade demográfica de 38.5706940874036 hab/km².\nO município Poço de José de Moura/PB possui densidade demográfica de 39.397840942854316 hab/km².\nO município Pombal/PB possui densidade demográfica de 36.12695626736873 hab/km².\nO município Prata/PB possui densidade demográfica de 20.071871256705382 hab/km².\nO município Princesa Isabel/PB possui densidade demográfica de 57.83738246643839 hab/km².\nO município Puxinanã/PB possui densidade demográfica de 177.8068244358833 hab/km².\nO município Queimadas/PB possui densidade demográfica de 102.16785305391011 hab/km².\nO município Quixabá/PB possui densidade demográfica de 10.84375797804442 hab/km².\nO município Remígio/PB possui densidade demográfica de 98.76966292134831 hab/km².\nO município Riachão/PB possui densidade demográfica de 36.228508042151965 hab/km².\nO município Riachão do Bacamarte/PB possui densidade demográfica de 111.12848579619495 hab/km².\nO município Riachão do Poço/PB possui densidade demográfica de 104.33475319468806 hab/km².\nO município Riacho de Santo Antônio/PB possui densidade demográfica de 18.856767411300922 hab/km².\nO município Riacho dos Cavalos/PB possui densidade demográfica de 31.488845964473736 hab/km².\nO município Rio Tinto/PB possui densidade demográfica de 49.42244401901525 hab/km².\nO município Salgadinho/PB possui densidade demográfica de 19.040382110290924 hab/km².\nO município Salgado de São Félix/PB possui densidade demográfica de 59.331186524647016 hab/km².\nO município Santa Cecília/PB possui densidade demográfica de 29.217131823766895 hab/km².\nO município Santa Cruz/PB possui densidade demográfica de 30.78936099348147 hab/km².\nO município Santa Helena/PB possui densidade demográfica de 25.52776721186763 hab/km².\nO município Santa Inês/PB possui densidade demográfica de 10.908362358598156 hab/km².\nO município Santa Luzia/PB possui densidade demográfica de 32.29975861312267 hab/km².\nO município Santa Rita/PB possui densidade demográfica de 165.5224599298342 hab/km².\nO município Santa Teresinha/PB possui densidade demográfica de 12.797876798435537 hab/km².\nO município Santana de Mangueira/PB possui densidade demográfica de 13.256247668780306 hab/km².\nO município Santana dos Garrotes/PB possui densidade demográfica de 20.53586569442089 hab/km².\nO município Santo André/PB possui densidade demográfica de 11.715592663321047 hab/km².\nO município São Bentinho/PB possui densidade demográfica de 21.115476858702863 hab/km².\nO município São Bento/PB possui densidade demográfica de 124.41176470588236 hab/km².\nO município São Domingos/PB possui densidade demográfica de 16.882502513157114 hab/km².\nO município São Domingos do Cariri/PB possui densidade demográfica de 11.060329067641682 hab/km².\nO município São Francisco/PB possui densidade demográfica de 35.388175888912265 hab/km².\nO município São João do Cariri/PB possui densidade demográfica de 6.646266829865361 hab/km².\nO município São João do Rio do Peixe/PB possui densidade demográfica de 38.36393145458761 hab/km².\nO município São João do Tigre/PB possui densidade demográfica de 5.386462775082096 hab/km².\nO município São José da Lagoa Tapada/PB possui densidade demográfica de 22.129253093824055 hab/km².\nO município São José de Caiana/PB possui densidade demográfica de 34.08382011002098 hab/km².\nO município São José de Espinharas/PB possui densidade demográfica de 6.559545792795524 hab/km².\nO município São José de Piranhas/PB possui densidade demográfica de 28.193884631852477 hab/km².\nO município São José de Princesa/PB possui densidade demográfica de 26.699152006075177 hab/km².\nO município São José do Bonfim/PB possui densidade demográfica de 23.9979216152019 hab/km².\nO município São José do Brejo do Cruz/PB possui densidade demográfica de 6.655600347798592 hab/km².\nO município São José do Sabugi/PB possui densidade demográfica de 19.37947032669631 hab/km².\nO município São José dos Cordeiros/PB possui densidade demográfica de 9.539198084979054 hab/km².\nO município São José dos Ramos/PB possui densidade demográfica de 56.07248294818283 hab/km².\nO município São Mamede/PB possui densidade demográfica de 14.59876019821755 hab/km².\nO município São Miguel de Taipu/PB possui densidade demográfica de 72.3657192261969 hab/km².\nO município São Sebastião de Lagoa de Roça/PB possui densidade demográfica de 221.1738782051282 hab/km².\nO município São Sebastião do Umbuzeiro/PB possui densidade demográfica de 7.023905160996157 hab/km².\nO município São Vicente do Seridó/PB possui densidade demográfica de 37.00220638767316 hab/km².\nO município Sapé/PB possui densidade demográfica de 158.91674325737648 hab/km².\nO município Serra Branca/PB possui densidade demográfica de 18.885750888021896 hab/km².\nO município Serra da Raiz/PB possui densidade demográfica de 110.17881705639616 hab/km².\nO município Serra Grande/PB possui densidade demográfica de 35.64154786150713 hab/km².\nO município Serra Redonda/PB possui densidade demográfica de 126.09551064210339 hab/km².\nO município Serraria/PB possui densidade demográfica de 95.52833078101072 hab/km².\nO município Sertãozinho/PB possui densidade demográfica de 133.9939024390244 hab/km².\nO município Sobrado/PB possui densidade demográfica de 119.42014901198574 hab/km².\nO município Solânea/PB possui densidade demográfica de 115.00646273158122 hab/km².\nO município Soledade/PB possui densidade demográfica de 24.53217627312335 hab/km².\nO município Sossêgo/PB possui densidade demográfica de 20.478190630048466 hab/km².\nO município Sousa/PB possui densidade demográfica de 89.09755602193488 hab/km².\nO município Sumé/PB possui densidade demográfica de 19.16307706993449 hab/km².\nO município Tacima/PB possui densidade demográfica de 41.60382713046299 hab/km².\nO município Taperoá/PB possui densidade demográfica de 22.530961970704922 hab/km².\nO município Tavares/PB possui densidade demográfica de 59.42358740993553 hab/km².\nO município Teixeira/PB possui densidade demográfica de 87.96146674953387 hab/km².\nO município Tenório/PB possui densidade demográfica de 26.72176308539945 hab/km².\nO município Triunfo/PB possui densidade demográfica de 41.93387001409924 hab/km².\nO município Uiraúna/PB possui densidade demográfica de 49.521222410865875 hab/km².\nO município Umbuzeiro/PB possui densidade demográfica de 51.27667788010809 hab/km².\nO município Várzea/PB possui densidade demográfica de 13.147807823575743 hab/km².\nO município Vieirópolis/PB possui densidade demográfica de 34.37116773402371 hab/km².\nO município Vista Serrana/PB possui densidade demográfica de 57.23598435462842 hab/km².\nO município Zabelê/PB possui densidade demográfica de 18.968827132279003 hab/km².\nO município Abatiá/PR possui densidade demográfica de 33.945435466946485 hab/km².\nO município Adrianópolis/PR possui densidade demográfica de 4.725308115879733 hab/km².\nO município Agudos do Sul/PR possui densidade demográfica de 43.01466763757412 hab/km².\nO município Almirante Tamandaré/PR possui densidade demográfica de 529.957892574715 hab/km².\nO município Altamira do Paraná/PR possui densidade demográfica de 11.128052719989663 hab/km².\nO município Alto Paraíso/PR possui densidade demográfica de 3.3127705963193734 hab/km².\nO município Alto Paraná/PR possui densidade demográfica de 33.510742666535855 hab/km².\nO município Alto Piquiri/PR possui densidade demográfica de 22.737730917863605 hab/km².\nO município Altônia/PR possui densidade demográfica de 31.011548461212893 hab/km².\nO município Alvorada do Sul/PR possui densidade demográfica de 24.238067177371832 hab/km².\nO município Amaporã/PR possui densidade demográfica de 14.147216301918178 hab/km².\nO município Ampére/PR possui densidade demográfica de 58.012401541813304 hab/km².\nO município Anahy/PR possui densidade demográfica de 27.9980516317584 hab/km².\nO município Andirá/PR possui densidade demográfica de 87.30091494408674 hab/km².\nO município Ângulo/PR possui densidade demográfica de 26.966610073571026 hab/km².\nO município Antonina/PR possui densidade demográfica de 21.410599329041617 hab/km².\nO município Antônio Olinto/PR possui densidade demográfica de 15.653081214599037 hab/km².\nO município Apucarana/PR possui densidade demográfica de 216.5493651390605 hab/km².\nO município Arapongas/PR possui densidade demográfica de 272.4870493433101 hab/km².\nO município Arapoti/PR possui densidade demográfica de 19.004182316665318 hab/km².\nO município Arapuã/PR possui densidade demográfica de 16.33711061155205 hab/km².\nO município Araruna/PR possui densidade demográfica de 27.208580871469415 hab/km².\nO município Araucária/PR possui densidade demográfica de 253.86369448469867 hab/km².\nO município Ariranha do Ivaí/PR possui densidade demográfica de 10.23960594423109 hab/km².\nO município Assaí/PR possui densidade demográfica de 37.13863971840581 hab/km².\nO município Assis Chateaubriand/PR possui densidade demográfica de 34.060788580740315 hab/km².\nO município Astorga/PR possui densidade demográfica de 56.80443432461648 hab/km².\nO município Atalaia/PR possui densidade demográfica de 28.425105331977335 hab/km².\nO município Balsa Nova/PR possui densidade demográfica de 32.38471899807984 hab/km².\nO município Bandeirantes/PR possui densidade demográfica de 72.2927289471911 hab/km².\nO município Barbosa Ferraz/PR possui densidade demográfica de 23.496212683796227 hab/km².\nO município Barra do Jacaré/PR possui densidade demográfica de 23.565502938126514 hab/km².\nO município Barracão/PR possui densidade demográfica de 56.77709086667444 hab/km².\nO município Bela Vista da Caroba/PR possui densidade demográfica de 26.635608669232326 hab/km².\nO município Bela Vista do Paraíso/PR possui densidade demográfica de 62.132761959701675 hab/km².\nO município Bituruna/PR possui densidade demográfica de 13.070927064556221 hab/km².\nO município Boa Esperança/PR possui densidade demográfica de 14.861084000260265 hab/km².\nO município Boa Esperança do Iguaçu/PR possui densidade demográfica de 18.20816864295125 hab/km².\nO município Boa Ventura de São Roque/PR possui densidade demográfica de 10.53392908804526 hab/km².\nO município Boa Vista da Aparecida/PR possui densidade demográfica de 30.866172454155286 hab/km².\nO município Bocaiúva do Sul/PR possui densidade demográfica de 13.295979863010382 hab/km².\nO município Bom Jesus do Sul/PR possui densidade demográfica de 21.819853997815716 hab/km².\nO município Bom Sucesso/PR possui densidade demográfica de 20.327797744454084 hab/km².\nO município Bom Sucesso do Sul/PR possui densidade demográfica de 16.807022916347673 hab/km².\nO município Borrazópolis/PR possui densidade demográfica de 23.5600215323883 hab/km².\nO município Braganey/PR possui densidade demográfica de 16.704532214843294 hab/km².\nO município Brasilândia do Sul/PR possui densidade demográfica de 11.0259758108851 hab/km².\nO município Cafeara/PR possui densidade demográfica de 14.504843918191604 hab/km².\nO município Cafelândia/PR possui densidade demográfica de 53.95995878109819 hab/km².\nO município Cafezal do Sul/PR possui densidade demográfica de 12.791079042308954 hab/km².\nO município Califórnia/PR possui densidade demográfica de 56.896065435058524 hab/km².\nO município Cambará/PR possui densidade demográfica de 65.23199606740039 hab/km².\nO município Cambé/PR possui densidade demográfica de 195.47153797967144 hab/km².\nO município Cambira/PR possui densidade demográfica de 44.28667605116593 hab/km².\nO município Campina da Lagoa/PR possui densidade demográfica de 19.324387090295126 hab/km².\nO município Campina do Simão/PR possui densidade demográfica de 9.089692698809152 hab/km².\nO município Campina Grande do Sul/PR possui densidade demográfica de 71.89563088791633 hab/km².\nO município Campo Bonito/PR possui densidade demográfica de 10.158356960099578 hab/km².\nO município Campo do Tenente/PR possui densidade demográfica de 23.399783244113106 hab/km².\nO município Campo Largo/PR possui densidade demográfica de 89.92534028983651 hab/km².\nO município Campo Magro/PR possui densidade demográfica de 90.22335209733066 hab/km².\nO município Campo Mourão/PR possui densidade demográfica de 115.04987596981053 hab/km².\nO município Cândido de Abreu/PR possui densidade demográfica de 11.028632727657996 hab/km².\nO município Candói/PR possui densidade demográfica de 9.904216712167585 hab/km².\nO município Cantagalo/PR possui densidade demográfica de 22.195564999828633 hab/km².\nO município Capanema/PR possui densidade demográfica de 44.24542045807361 hab/km².\nO município Capitão Leônidas Marques/PR possui densidade demográfica de 54.2883046237534 hab/km².\nO município Carambeí/PR possui densidade demográfica de 29.496059598571605 hab/km².\nO município Carlópolis/PR possui densidade demográfica de 30.361968898143633 hab/km².\nO município Cascavel/PR possui densidade demográfica de 136.23425027251133 hab/km².\nO município Castro/PR possui densidade demográfica de 26.499703732964644 hab/km².\nO município Catanduvas/PR possui densidade demográfica de 17.536441144114413 hab/km².\nO município Centenário do Sul/PR possui densidade demográfica de 30.094397977570395 hab/km².\nO município Cerro Azul/PR possui densidade demográfica de 12.629083127670203 hab/km².\nO município Céu Azul/PR possui densidade demográfica de 9.353512230276824 hab/km².\nO município Chopinzinho/PR possui densidade demográfica de 20.50557992685138 hab/km².\nO município Cianorte/PR possui densidade demográfica de 86.19020045092218 hab/km².\nO município Cidade Gaúcha/PR possui densidade demográfica de 27.445726336682792 hab/km².\nO município Clevelândia/PR possui densidade demográfica de 24.501165368654426 hab/km².\nO município Colombo/PR possui densidade demográfica de 1076.7328985287427 hab/km².\nO município Colorado/PR possui densidade demográfica de 55.41090115558201 hab/km².\nO município Congonhinhas/PR possui densidade demográfica de 15.44704828718561 hab/km².\nO município Conselheiro Mairinck/PR possui densidade demográfica de 17.76171168970739 hab/km².\nO município Contenda/PR possui densidade demográfica de 53.140048154093094 hab/km².\nO município Corbélia/PR possui densidade demográfica de 30.813404359817145 hab/km².\nO município Cornélio Procópio/PR possui densidade demográfica de 73.89072586994173 hab/km².\nO município Coronel Domingos Soares/PR possui densidade demográfica de 4.591998578878583 hab/km².\nO município Coronel Vivida/PR possui densidade demográfica de 31.777271266181586 hab/km².\nO município Corumbataí do Sul/PR possui densidade demográfica de 24.35195326761592 hab/km².\nO município Cruz Machado/PR possui densidade demográfica de 12.202793655088444 hab/km².\nO município Cruzeiro do Iguaçu/PR possui densidade demográfica de 26.43024836278265 hab/km².\nO município Cruzeiro do Oeste/PR possui densidade demográfica de 26.200559533892868 hab/km².\nO município Cruzeiro do Sul/PR possui densidade demográfica de 17.610961018911617 hab/km².\nO município Cruzmaltina/PR possui densidade demográfica de 10.124879923150816 hab/km².\nO município Curitiba/PR possui densidade demográfica de 4027.002114748069 hab/km².\nO município Curiúva/PR possui densidade demográfica de 24.16096900704543 hab/km².\nO município Diamante do Norte/PR possui densidade demográfica de 22.709868664827702 hab/km².\nO município Diamante do Sul/PR possui densidade demográfica de 9.751354354771497 hab/km².\nO município Diamante d`Oeste/PR possui densidade demográfica de 16.262819061175634 hab/km².\nO município Dois Vizinhos/PR possui densidade demográfica de 86.41824913412158 hab/km².\nO município Douradina/PR possui densidade demográfica de 17.73252352030487 hab/km².\nO município Doutor Camargo/PR possui densidade demográfica de 49.27291173486642 hab/km².\nO município Doutor Ulysses/PR possui densidade demográfica de 7.328683856932625 hab/km².\nO município Enéas Marques/PR possui densidade demográfica de 31.753381893860563 hab/km².\nO município Engenheiro Beltrão/PR possui densidade demográfica de 29.747363467174363 hab/km².\nO município Entre Rios do Oeste/PR possui densidade demográfica de 32.1618743343983 hab/km².\nO município Esperança Nova/PR possui densidade demográfica de 14.217667436489608 hab/km².\nO município Espigão Alto do Iguaçu/PR possui densidade demográfica de 14.327288322509496 hab/km².\nO município Farol/PR possui densidade demográfica de 12.0042872454448 hab/km².\nO município Faxinal/PR possui densidade demográfica de 22.78682571165181 hab/km².\nO município Fazenda Rio Grande/PR possui densidade demográfica de 699.9914295509084 hab/km².\nO município Fênix/PR possui densidade demográfica de 20.51260145237078 hab/km².\nO município Fernandes Pinheiro/PR possui densidade demográfica de 14.592865928659286 hab/km².\nO município Figueira/PR possui densidade demográfica de 63.90537104107266 hab/km².\nO município Flor da Serra do Sul/PR possui densidade demográfica de 19.781507680716587 hab/km².\nO município Floraí/PR possui densidade demográfica de 26.421807146968032 hab/km².\nO município Floresta/PR possui densidade demográfica de 37.483410225620936 hab/km².\nO município Florestópolis/PR possui densidade demográfica de 45.55677343401128 hab/km².\nO município Flórida/PR possui densidade demográfica de 30.62010836845274 hab/km².\nO município Formosa do Oeste/PR possui densidade demográfica de 27.3512023502956 hab/km².\nO município Foz do Iguaçu/PR possui densidade demográfica de 414.58313096972637 hab/km².\nO município Foz do Jordão/PR possui densidade demográfica de 23.02659529271816 hab/km².\nO município Francisco Alves/PR possui densidade demográfica de 19.937868903386146 hab/km².\nO município Francisco Beltrão/PR possui densidade demográfica de 107.3893702983227 hab/km².\nO município General Carneiro/PR possui densidade demográfica de 12.76069381429825 hab/km².\nO município Godoy Moreira/PR possui densidade demográfica de 25.471338065796505 hab/km².\nO município Goioerê/PR possui densidade demográfica de 51.435762904140674 hab/km².\nO município Goioxim/PR possui densidade demográfica de 10.680883169388016 hab/km².\nO município Grandes Rios/PR possui densidade demográfica de 21.085295989815403 hab/km².\nO município Guaíra/PR possui densidade demográfica de 54.78063836999768 hab/km².\nO município Guairaçá/PR possui densidade demográfica de 12.546058225695429 hab/km².\nO município Guamiranga/PR possui densidade demográfica de 32.27124183006536 hab/km².\nO município Guapirama/PR possui densidade demográfica de 20.576414595452142 hab/km².\nO município Guaporema/PR possui densidade demográfica de 11.084469753733952 hab/km².\nO município Guaraci/PR possui densidade demográfica de 24.688267523143775 hab/km².\nO município Guaraniaçu/PR possui densidade demográfica de 11.897748876069876 hab/km².\nO município Guarapuava/PR possui densidade demográfica de 53.68221468651046 hab/km².\nO município Guaraqueçaba/PR possui densidade demográfica de 3.896361053220401 hab/km².\nO município Guaratuba/PR possui densidade demográfica de 24.189962239691287 hab/km².\nO município Honório Serpa/PR possui densidade demográfica de 11.856881172347881 hab/km².\nO município Ibaiti/PR possui densidade demográfica de 32.02597634058859 hab/km².\nO município Ibema/PR possui densidade demográfica de 41.705053282915095 hab/km².\nO município Ibiporã/PR possui densidade demográfica de 161.87949217438032 hab/km².\nO município Icaraíma/PR possui densidade demográfica de 13.090160535513299 hab/km².\nO município Iguaraçu/PR possui densidade demográfica de 24.136258940477635 hab/km².\nO município Iguatu/PR possui densidade demográfica de 20.890218814288385 hab/km².\nO município Imbaú/PR possui densidade demográfica de 34.09132143937103 hab/km².\nO município Imbituva/PR possui densidade demográfica de 37.6120231580617 hab/km².\nO município Inácio Martins/PR possui densidade demográfica de 11.688616870146655 hab/km².\nO município Inajá/PR possui densidade demográfica de 15.346687211093991 hab/km².\nO município Indianópolis/PR possui densidade demográfica de 35.059533518186264 hab/km².\nO município Ipiranga/PR possui densidade demográfica de 15.262811593264948 hab/km².\nO município Iporã/PR possui densidade demográfica de 23.12275231906651 hab/km².\nO município Iracema do Oeste/PR possui densidade demográfica de 31.616384596517044 hab/km².\nO município Irati/PR possui densidade demográfica de 56.23399231631183 hab/km².\nO município Iretama/PR possui densidade demográfica de 18.620061003400764 hab/km².\nO município Itaguajé/PR possui densidade demográfica de 23.995377422913272 hab/km².\nO município Itaipulândia/PR possui densidade demográfica de 27.24501192308853 hab/km².\nO município Itambaracá/PR possui densidade demográfica de 32.59863026912318 hab/km².\nO município Itambé/PR possui densidade demográfica de 24.522188499712904 hab/km².\nO município Itapejara d`Oeste/PR possui densidade demográfica de 41.458997677256804 hab/km².\nO município Itaperuçu/PR possui densidade demográfica de 75.9619665458246 hab/km².\nO município Itaúna do Sul/PR possui densidade demográfica de 27.803212539768758 hab/km².\nO município Ivaí/PR possui densidade demográfica de 21.08250390721395 hab/km².\nO município Ivaiporã/PR possui densidade demográfica de 73.7334878331402 hab/km².\nO município Ivaté/PR possui densidade demográfica de 18.286242729551482 hab/km².\nO município Ivatuba/PR possui densidade demográfica de 31.140078626112146 hab/km².\nO município Jaboti/PR possui densidade demográfica de 35.19529006318208 hab/km².\nO município Jacarezinho/PR possui densidade demográfica de 64.92788740809587 hab/km².\nO município Jaguapitã/PR possui densidade demográfica de 25.736842105263158 hab/km².\nO município Jaguariaíva/PR possui densidade demográfica de 22.439386953140595 hab/km².\nO município Jandaia do Sul/PR possui densidade demográfica de 108.04371002132197 hab/km².\nO município Janiópolis/PR possui densidade demográfica de 19.460747802770744 hab/km².\nO município Japira/PR possui densidade demográfica de 26.039619735514368 hab/km².\nO município Japurá/PR possui densidade demográfica de 51.752527392699314 hab/km².\nO município Jardim Alegre/PR possui densidade demográfica de 30.388361484403895 hab/km².\nO município Jardim Olinda/PR possui densidade demográfica de 10.963274198568316 hab/km².\nO município Jataizinho/PR possui densidade demográfica de 74.60108053775599 hab/km².\nO município Jesuítas/PR possui densidade demográfica de 36.36767676767677 hab/km².\nO município Joaquim Távora/PR possui densidade demográfica de 37.1269495452502 hab/km².\nO município Jundiaí do Sul/PR possui densidade demográfica de 10.700704444860046 hab/km².\nO município Juranda/PR possui densidade demográfica de 21.84890769758664 hab/km².\nO município Jussara/PR possui densidade demográfica de 31.34632712097501 hab/km².\nO município Kaloré/PR possui densidade demográfica de 23.310915675116398 hab/km².\nO município Lapa/PR possui densidade demográfica de 21.458932306839998 hab/km².\nO município Laranjal/PR possui densidade demográfica de 11.368511368511367 hab/km².\nO município Laranjeiras do Sul/PR possui densidade demográfica de 45.79365551720033 hab/km².\nO município Leópolis/PR possui densidade demográfica de 12.017279369129072 hab/km².\nO município Lidianópolis/PR possui densidade demográfica de 25.036234167244313 hab/km².\nO município Lindoeste/PR possui densidade demográfica de 14.835210449124165 hab/km².\nO município Loanda/PR possui densidade demográfica de 29.343944636678202 hab/km².\nO município Lobato/PR possui densidade demográfica de 18.268991282689914 hab/km².\nO município Londrina/PR possui densidade demográfica de 306.51934570619693 hab/km².\nO município Luiziana/PR possui densidade demográfica de 8.050847457627118 hab/km².\nO município Lunardelli/PR possui densidade demográfica de 25.902314140856383 hab/km².\nO município Lupionópolis/PR possui densidade demográfica de 37.92847113240274 hab/km².\nO município Mallet/PR possui densidade demográfica de 17.942795496666758 hab/km².\nO município Mamborê/PR possui densidade demográfica de 17.71565616831206 hab/km².\nO município Mandaguaçu/PR possui densidade demográfica de 67.2777362084212 hab/km².\nO município Mandaguari/PR possui densidade demográfica de 97.25142193502278 hab/km².\nO município Mandirituba/PR possui densidade demográfica de 58.600137138034704 hab/km².\nO município Manfrinópolis/PR possui densidade demográfica de 14.44875704648369 hab/km².\nO município Mangueirinha/PR possui densidade demográfica de 16.15219904117636 hab/km².\nO município Manoel Ribas/PR possui densidade demográfica de 23.0573939839619 hab/km².\nO município Marechal Cândido Rondon/PR possui densidade demográfica de 62.592245989304814 hab/km².\nO município Maria Helena/PR possui densidade demográfica de 12.249598946978733 hab/km².\nO município Marialva/PR possui densidade demográfica de 67.20287660862982 hab/km².\nO município Marilândia do Sul/PR possui densidade demográfica de 23.0555122001977 hab/km².\nO município Marilena/PR possui densidade demográfica de 29.513276240478547 hab/km².\nO município Mariluz/PR possui densidade demográfica de 23.60274257220029 hab/km².\nO município Maringá/PR possui densidade demográfica de 733.1423878451905 hab/km².\nO município Mariópolis/PR possui densidade demográfica de 27.20958499739538 hab/km².\nO município Maripá/PR possui densidade demográfica de 20.02889460516579 hab/km².\nO município Marmeleiro/PR possui densidade demográfica de 35.88207961175073 hab/km².\nO município Marquinho/PR possui densidade demográfica de 9.744693338550329 hab/km².\nO município Marumbi/PR possui densidade demográfica de 22.07991557538255 hab/km².\nO município Matelândia/PR possui densidade demográfica de 25.131692067213756 hab/km².\nO município Matinhos/PR possui densidade demográfica de 249.9405469678954 hab/km².\nO município Mato Rico/PR possui densidade demográfica de 9.677337591564648 hab/km².\nO município Mauá da Serra/PR possui densidade demográfica de 78.97895125553914 hab/km².\nO município Medianeira/PR possui densidade demográfica de 127.20773887384783 hab/km².\nO município Mercedes/PR possui densidade demográfica de 25.121975505327093 hab/km².\nO município Mirador/PR possui densidade demográfica de 10.495692571377024 hab/km².\nO município Miraselva/PR possui densidade demográfica de 20.622438808284414 hab/km².\nO município Missal/PR possui densidade demográfica de 32.287299630086316 hab/km².\nO município Moreira Sales/PR possui densidade demográfica de 35.63332108432032 hab/km².\nO município Morretes/PR possui densidade demográfica de 22.96006310438517 hab/km².\nO município Munhoz de Melo/PR possui densidade demográfica de 26.799007444168733 hab/km².\nO município Nossa Senhora das Graças/PR possui densidade demográfica de 20.65363699994616 hab/km².\nO município Nova Aliança do Ivaí/PR possui densidade demográfica de 10.901196008227316 hab/km².\nO município Nova América da Colina/PR possui densidade demográfica de 26.861291319122646 hab/km².\nO município Nova Aurora/PR possui densidade demográfica de 25.0332271471066 hab/km².\nO município Nova Cantu/PR possui densidade demográfica de 13.366577256116221 hab/km².\nO município Nova Esperança/PR possui densidade demográfica de 66.27406060907892 hab/km².\nO município Nova Esperança do Sudoeste/PR possui densidade demográfica de 24.45435794119058 hab/km².\nO município Nova Fátima/PR possui densidade demográfica de 28.745324959424174 hab/km².\nO município Nova Laranjeiras/PR possui densidade demográfica de 9.813267684571668 hab/km².\nO município Nova Londrina/PR possui densidade demográfica de 48.505883663090685 hab/km².\nO município Nova Olímpia/PR possui densidade demográfica de 40.35936927026036 hab/km².\nO município Nova Prata do Iguaçu/PR possui densidade demográfica de 29.43245312987492 hab/km².\nO município Nova Santa Bárbara/PR possui densidade demográfica de 54.45930880713489 hab/km².\nO município Nova Santa Rosa/PR possui densidade demográfica de 37.25997947916158 hab/km².\nO município Nova Tebas/PR possui densidade demográfica de 13.557147831186203 hab/km².\nO município Novo Itacolomi/PR possui densidade demográfica de 17.514404312000497 hab/km².\nO município Ortigueira/PR possui densidade demográfica de 9.623141638815259 hab/km².\nO município Ourizona/PR possui densidade demográfica de 19.15448260228947 hab/km².\nO município Ouro Verde do Oeste/PR possui densidade demográfica de 19.42396942396942 hab/km².\nO município Paiçandu/PR possui densidade demográfica de 209.68607772202125 hab/km².\nO município Palmas/PR possui densidade demográfica de 27.529543164151512 hab/km².\nO município Palmeira/PR possui densidade demográfica de 22.043423960034584 hab/km².\nO município Palmital/PR possui densidade demográfica de 18.180150431113557 hab/km².\nO município Palotina/PR possui densidade demográfica de 44.04367053620785 hab/km².\nO município Paraíso do Norte/PR possui densidade demográfica de 57.54790770434102 hab/km².\nO município Paranacity/PR possui densidade demográfica de 29.40079740699309 hab/km².\nO município Paranaguá/PR possui densidade demográfica de 169.9214922520474 hab/km².\nO município Paranapoema/PR possui densidade demográfica de 15.868774164202867 hab/km².\nO município Paranavaí/PR possui densidade demográfica de 67.86329193941461 hab/km².\nO município Pato Bragado/PR possui densidade demográfica de 35.64195432034888 hab/km².\nO município Pato Branco/PR possui densidade demográfica de 134.24474577528798 hab/km².\nO município Paula Freitas/PR possui densidade demográfica de 12.894805533803185 hab/km².\nO município Paulo Frontin/PR possui densidade demográfica de 18.690855999567404 hab/km².\nO município Peabiru/PR possui densidade demográfica de 29.073836961160904 hab/km².\nO município Perobal/PR possui densidade demográfica de 13.869669758084303 hab/km².\nO município Pérola/PR possui densidade demográfica de 42.42021276595745 hab/km².\nO município Pérola d`Oeste/PR possui densidade demográfica de 32.81242416889104 hab/km².\nO município Piên/PR possui densidade demográfica de 44.09906197260489 hab/km².\nO município Pinhais/PR possui densidade demográfica de 1922.260555281748 hab/km².\nO município Pinhal de São Bento/PR possui densidade demográfica de 26.934126821260005 hab/km².\nO município Pinhalão/PR possui densidade demográfica de 28.16933327290033 hab/km².\nO município Pinhão/PR possui densidade demográfica de 15.092001858522476 hab/km².\nO município Piraí do Sul/PR possui densidade demográfica de 16.694819217857983 hab/km².\nO município Piraquara/PR possui densidade demográfica de 410.513102840784 hab/km².\nO município Pitanga/PR possui densidade demográfica de 19.617129977460557 hab/km².\nO município Pitangueiras/PR possui densidade demográfica de 22.83534853525927 hab/km².\nO município Planaltina do Paraná/PR possui densidade demográfica de 11.496673123894551 hab/km².\nO município Planalto/PR possui densidade demográfica de 39.49210389309886 hab/km².\nO município Ponta Grossa/PR possui densidade demográfica de 150.71509757926046 hab/km².\nO município Pontal do Paraná/PR possui densidade demográfica de 104.66803422224446 hab/km².\nO município Porecatu/PR possui densidade demográfica de 48.647444029211094 hab/km².\nO município Porto Amazonas/PR possui densidade demográfica de 24.19337549576589 hab/km².\nO município Porto Barreiro/PR possui densidade demográfica de 10.146252285191956 hab/km².\nO município Porto Rico/PR possui densidade demográfica de 11.622565233370084 hab/km².\nO município Porto Vitória/PR possui densidade demográfica de 18.872353410638 hab/km².\nO município Prado Ferreira/PR possui densidade demográfica de 22.38591916558018 hab/km².\nO município Pranchita/PR possui densidade demográfica de 24.92029755579171 hab/km².\nO município Presidente Castelo Branco/PR possui densidade demográfica de 30.7198356129198 hab/km².\nO município Primeiro de Maio/PR possui densidade demográfica de 26.136473313386738 hab/km².\nO município Prudentópolis/PR possui densidade demográfica de 21.135802469135804 hab/km².\nO município Quarto Centenário/PR possui densidade demográfica de 15.086367590406363 hab/km².\nO município Quatiguá/PR possui densidade demográfica de 62.51663856597746 hab/km².\nO município Quatro Barras/PR possui densidade demográfica de 109.99612123898709 hab/km².\nO município Quatro Pontes/PR possui densidade demográfica de 33.245913104292335 hab/km².\nO município Quedas do Iguaçu/PR possui densidade demográfica de 37.255021302495436 hab/km².\nO município Querência do Norte/PR possui densidade demográfica de 12.821942367396913 hab/km².\nO município Quinta do Sol/PR possui densidade demográfica de 15.598749156907228 hab/km².\nO município Quitandinha/PR possui densidade demográfica de 38.22871459889938 hab/km².\nO município Ramilândia/PR possui densidade demográfica de 17.4283305227656 hab/km².\nO município Rancho Alegre/PR possui densidade demográfica de 23.59081419624217 hab/km².\nO município Rancho Alegre d`Oeste/PR possui densidade demográfica de 11.794191971498405 hab/km².\nO município Realeza/PR possui densidade demográfica de 46.22828362854394 hab/km².\nO município Rebouças/PR possui densidade demográfica de 29.42055454092645 hab/km².\nO município Renascença/PR possui densidade demográfica de 16.01805911538552 hab/km².\nO município Reserva/PR possui densidade demográfica de 15.39082371355899 hab/km².\nO município Reserva do Iguaçu/PR possui densidade demográfica de 8.758975342531436 hab/km².\nO município Ribeirão Claro/PR possui densidade demográfica de 16.970217094180096 hab/km².\nO município Ribeirão do Pinhal/PR possui densidade demográfica de 36.08998478904811 hab/km².\nO município Rio Azul/PR possui densidade demográfica de 22.378721714966257 hab/km².\nO município Rio Bom/PR possui densidade demográfica de 18.747188484030588 hab/km².\nO município Rio Bonito do Iguaçu/PR possui densidade demográfica de 18.309387229936203 hab/km².\nO município Rio Branco do Ivaí/PR possui densidade demográfica de 10.195380953626449 hab/km².\nO município Rio Branco do Sul/PR possui densidade demográfica de 37.732829408216276 hab/km².\nO município Rio Negro/PR possui densidade demográfica de 51.766146919588174 hab/km².\nO município Rolândia/PR possui densidade demográfica de 126.05550956385343 hab/km².\nO município Roncador/PR possui densidade demográfica de 15.54600334177761 hab/km².\nO município Rondon/PR possui densidade demográfica de 16.17723749752738 hab/km².\nO município Rosário do Ivaí/PR possui densidade demográfica de 15.051851851851852 hab/km².\nO município Sabáudia/PR possui densidade demográfica de 32.028581936636364 hab/km².\nO município Salgado Filho/PR possui densidade demográfica de 23.256919501373336 hab/km².\nO município Salto do Itararé/PR possui densidade demográfica de 25.8228605625374 hab/km².\nO município Salto do Lontra/PR possui densidade demográfica de 43.77398311588641 hab/km².\nO município Santa Amélia/PR possui densidade demográfica de 48.725176169122356 hab/km².\nO município Santa Cecília do Pavão/PR possui densidade demográfica de 33.08529945553539 hab/km².\nO município Santa Cruz de Monte Castelo/PR possui densidade demográfica de 18.307278115879733 hab/km².\nO município Santa Fé/PR possui densidade demográfica de 37.764262959745146 hab/km².\nO município Santa Helena/PR possui densidade demográfica de 30.878493333157483 hab/km².\nO município Santa Inês/PR possui densidade demográfica de 13.128249566724438 hab/km².\nO município Santa Isabel do Ivaí/PR possui densidade demográfica de 25.064377682403432 hab/km².\nO município Santa Izabel do Oeste/PR possui densidade demográfica de 40.886730182452204 hab/km².\nO município Santa Lúcia/PR possui densidade demográfica de 33.58719835700838 hab/km².\nO município Santa Maria do Oeste/PR possui densidade demográfica de 13.575087942961021 hab/km².\nO município Santa Mariana/PR possui densidade demográfica de 29.108827453826166 hab/km².\nO município Santa Mônica/PR possui densidade demográfica de 13.736728727496539 hab/km².\nO município Santa Tereza do Oeste/PR possui densidade demográfica de 31.67479076611791 hab/km².\nO município Santa Terezinha de Itaipu/PR possui densidade demográfica de 80.34619684644744 hab/km².\nO município Santana do Itararé/PR possui densidade demográfica de 20.889879412584072 hab/km².\nO município Santo Antônio da Platina/PR possui densidade demográfica de 59.19442249850999 hab/km².\nO município Santo Antônio do Caiuá/PR possui densidade demográfica de 12.44807595745652 hab/km².\nO município Santo Antônio do Paraíso/PR possui densidade demográfica de 14.514767932489452 hab/km².\nO município Santo Antônio do Sudoeste/PR possui densidade demográfica de 58.00024559464603 hab/km².\nO município Santo Inácio/PR possui densidade demográfica de 17.17013719164467 hab/km².\nO município São Carlos do Ivaí/PR possui densidade demográfica de 28.229962679936023 hab/km².\nO município São Jerônimo da Serra/PR possui densidade demográfica de 13.762336574529298 hab/km².\nO município São João/PR possui densidade demográfica de 27.31278668247178 hab/km².\nO município São João do Caiuá/PR possui densidade demográfica de 19.417890345258037 hab/km².\nO município São João do Ivaí/PR possui densidade demográfica de 32.61823224747403 hab/km².\nO município São João do Triunfo/PR possui densidade demográfica de 19.022501075776294 hab/km².\nO município São Jorge do Ivaí/PR possui densidade demográfica de 17.50928306198229 hab/km².\nO município São Jorge do Patrocínio/PR possui densidade demográfica de 14.92747535150362 hab/km².\nO município São Jorge d`Oeste/PR possui densidade demográfica de 23.93624028454749 hab/km².\nO município São José da Boa Vista/PR possui densidade demográfica de 16.290940025521053 hab/km².\nO município São José das Palmeiras/PR possui densidade demográfica de 20.995504878851005 hab/km².\nO município São José dos Pinhais/PR possui densidade demográfica de 279.1619120071003 hab/km².\nO município São Manoel do Paraná/PR possui densidade demográfica de 21.996225623820507 hab/km².\nO município São Mateus do Sul/PR possui densidade demográfica de 30.74956585253147 hab/km².\nO município São Miguel do Iguaçu/PR possui densidade demográfica de 30.270175026430167 hab/km².\nO município São Pedro do Iguaçu/PR possui densidade demográfica de 21.052802283341983 hab/km².\nO município São Pedro do Ivaí/PR possui densidade demográfica de 31.507019120518144 hab/km².\nO município São Pedro do Paraná/PR possui densidade demográfica de 9.938160781966886 hab/km².\nO município São Sebastião da Amoreira/PR possui densidade demográfica de 37.836652337924384 hab/km².\nO município São Tomé/PR possui densidade demográfica de 24.467111883633702 hab/km².\nO município Sapopema/PR possui densidade demográfica de 9.940821416448989 hab/km².\nO município Sarandi/PR possui densidade demográfica de 800.7635801275856 hab/km².\nO município Saudade do Iguaçu/PR possui densidade demográfica de 33.0593727398251 hab/km².\nO município Sengés/PR possui densidade demográfica de 12.81098680915011 hab/km².\nO município Serranópolis do Iguaçu/PR possui densidade demográfica de 9.44465120125708 hab/km².\nO município Sertaneja/PR possui densidade demográfica de 13.086908591869333 hab/km².\nO município Sertanópolis/PR possui densidade demográfica de 30.933871382509448 hab/km².\nO município Siqueira Campos/PR possui densidade demográfica de 66.37174507265141 hab/km².\nO município Sulina/PR possui densidade demográfica de 19.87584914499883 hab/km².\nO município Tamarana/PR possui densidade demográfica de 25.970010166045405 hab/km².\nO município Tamboara/PR possui densidade demográfica de 24.122058443237652 hab/km².\nO município Tapejara/PR possui densidade demográfica de 24.683801149814002 hab/km².\nO município Tapira/PR possui densidade demográfica de 13.435550337270069 hab/km².\nO município Teixeira Soares/PR possui densidade demográfica de 11.39024579359541 hab/km².\nO município Telêmaco Borba/PR possui densidade demográfica de 50.52716833229684 hab/km².\nO município Terra Boa/PR possui densidade demográfica de 49.16939379772479 hab/km².\nO município Terra Rica/PR possui densidade demográfica de 21.725973822064258 hab/km².\nO município Terra Roxa/PR possui densidade demográfica de 20.9275608446448 hab/km².\nO município Tibagi/PR possui densidade demográfica de 6.553800180920662 hab/km².\nO município Tijucas do Sul/PR possui densidade demográfica de 21.635982080400066 hab/km².\nO município Toledo/PR possui densidade demográfica de 99.67669172932331 hab/km².\nO município Tomazina/PR possui densidade demográfica de 14.863722440146082 hab/km².\nO município Três Barras do Paraná/PR possui densidade demográfica de 23.4524069262352 hab/km².\nO município Tunas do Paraná/PR possui densidade demográfica de 9.358544758257539 hab/km².\nO município Tuneiras do Oeste/PR possui densidade demográfica de 12.441512727688984 hab/km².\nO município Tupãssi/PR possui densidade demográfica de 25.721269820848477 hab/km².\nO município Turvo/PR possui densidade demográfica de 15.069449748496982 hab/km².\nO município Ubiratã/PR possui densidade demográfica de 33.03503018786969 hab/km².\nO município Umuarama/PR possui densidade demográfica de 81.66649091071328 hab/km².\nO município União da Vitória/PR possui densidade demográfica de 73.24305555555556 hab/km².\nO município Uniflor/PR possui densidade demográfica de 26.007171482809536 hab/km².\nO município Uraí/PR possui densidade demográfica de 48.24019174971616 hab/km².\nO município Ventania/PR possui densidade demográfica de 13.112185100807247 hab/km².\nO município Vera Cruz do Oeste/PR possui densidade demográfica de 27.43281665596625 hab/km².\nO município Verê/PR possui densidade demográfica de 25.266196279666453 hab/km².\nO município Virmond/PR possui densidade demográfica de 16.24378007155488 hab/km².\nO município Vitorino/PR possui densidade demográfica de 21.13101031730582 hab/km².\nO município Wenceslau Braz/PR possui densidade demográfica de 48.497185363892235 hab/km².\nO município Xambrê/PR possui densidade demográfica de 16.713463623474468 hab/km².\nO município Abreu e Lima/PE possui densidade demográfica de 748.3081068230447 hab/km².\nO município Afogados da Ingazeira/PE possui densidade demográfica de 92.89912629070692 hab/km².\nO município Afrânio/PE possui densidade demográfica de 11.79793371796592 hab/km².\nO município Agrestina/PE possui densidade demográfica de 112.57880367336809 hab/km².\nO município Água Preta/PE possui densidade demográfica de 62.05351283445521 hab/km².\nO município Águas Belas/PE possui densidade demográfica de 45.41247643878599 hab/km².\nO município Alagoinha/PE possui densidade demográfica de 63.16393517880916 hab/km².\nO município Aliança/PE possui densidade demográfica de 137.15678727226071 hab/km².\nO município Altinho/PE possui densidade demográfica de 49.18368245027284 hab/km².\nO município Amaraji/PE possui densidade demográfica de 93.37334014300306 hab/km².\nO município Angelim/PE possui densidade demográfica de 86.42832937987123 hab/km².\nO município Araçoiaba/PE possui densidade demográfica de 188.37933181157916 hab/km².\nO município Araripina/PE possui densidade demográfica de 40.84434111803868 hab/km².\nO município Arcoverde/PE possui densidade demográfica de 196.0473069250499 hab/km².\nO município Barra de Guabiraba/PE possui densidade demográfica de 111.43480156999563 hab/km².\nO município Barreiros/PE possui densidade demográfica de 174.53828684063933 hab/km².\nO município Belém de Maria/PE possui densidade demográfica de 153.95985896392733 hab/km².\nO município Belém de São Francisco/PE possui densidade demográfica de 11.062377102905835 hab/km².\nO município Belo Jardim/PE possui densidade demográfica de 111.82955071792496 hab/km².\nO município Betânia/PE possui densidade demográfica de 9.64817092285804 hab/km².\nO município Bezerros/PE possui densidade demográfica de 119.53058147589748 hab/km².\nO município Bodocó/PE possui densidade demográfica de 21.749458707083203 hab/km².\nO município Bom Conselho/PE possui densidade demográfica de 57.43950314949696 hab/km².\nO município Bom Jardim/PE possui densidade demográfica de 169.48651312841653 hab/km².\nO município Bonito/PE possui densidade demográfica de 94.95715477364071 hab/km².\nO município Brejão/PE possui densidade demográfica de 55.34764378246449 hab/km².\nO município Brejinho/PE possui densidade demográfica de 68.75235227700414 hab/km².\nO município Brejo da Madre de Deus/PE possui densidade demográfica de 59.26411753131764 hab/km².\nO município Buenos Aires/PE possui densidade demográfica de 134.53160210322997 hab/km².\nO município Buíque/PE possui densidade demográfica de 39.184351828176936 hab/km².\nO município Cabo de Santo Agostinho/PE possui densidade demográfica de 412.3211659312742 hab/km².\nO município Cabrobó/PE possui densidade demográfica de 18.62388475668241 hab/km².\nO município Cachoeirinha/PE possui densidade demográfica de 104.98159098516122 hab/km².\nO município Caetés/PE possui densidade demográfica de 80.66346970984581 hab/km².\nO município Calçado/PE possui densidade demográfica de 91.2259122591226 hab/km².\nO município Calumbi/PE possui densidade demográfica de 31.498522112542524 hab/km².\nO município Camaragibe/PE possui densidade demográfica de 2818.298868513461 hab/km².\nO município Camocim de São Félix/PE possui densidade demográfica de 235.98233995584988 hab/km².\nO município Camutanga/PE possui densidade demográfica de 217.3773987206823 hab/km².\nO município Canhotinho/PE possui densidade demográfica de 57.95830575777631 hab/km².\nO município Capoeiras/PE possui densidade demográfica de 58.255284987958255 hab/km².\nO município Carnaíba/PE possui densidade demográfica de 43.4174848059841 hab/km².\nO município Carnaubeira da Penha/PE possui densidade demográfica de 11.727233818069616 hab/km².\nO município Carpina/PE possui densidade demográfica de 516.5114193058718 hab/km².\nO município Caruaru/PE possui densidade demográfica de 342.0688456566842 hab/km².\nO município Casinhas/PE possui densidade demográfica de 118.80555795287822 hab/km².\nO município Catende/PE possui densidade demográfica de 182.49372707971435 hab/km².\nO município Cedro/PE possui densidade demográfica de 72.45227211616026 hab/km².\nO município Chã de Alegria/PE possui densidade demográfica de 255.48918640576727 hab/km².\nO município Chã Grande/PE possui densidade demográfica de 237.32469063052446 hab/km².\nO município Condado/PE possui densidade demográfica de 270.8533184606804 hab/km².\nO município Correntes/PE possui densidade demográfica de 53.000060853161315 hab/km².\nO município Cortês/PE possui densidade demográfica de 122.89774970390842 hab/km².\nO município Cumaru/PE possui densidade demográfica de 58.79957567669301 hab/km².\nO município Cupira/PE possui densidade demográfica de 221.5801439939371 hab/km².\nO município Custódia/PE possui densidade demográfica de 24.11101536182547 hab/km².\nO município Dormentes/PE possui densidade demográfica de 11.001925027964933 hab/km².\nO município Escada/PE possui densidade demográfica de 183.06721235877336 hab/km².\nO município Exu/PE possui densidade demográfica de 23.653084112149532 hab/km².\nO município Feira Nova/PE possui densidade demográfica de 190.9495962127541 hab/km².\nO município Fernando de Noronha/PE possui densidade demográfica de 154.52408930669802 hab/km².\nO município Ferreiros/PE possui densidade demográfica de 127.92389479574707 hab/km².\nO município Flores/PE possui densidade demográfica de 22.26786933986902 hab/km².\nO município Floresta/PE possui densidade demográfica de 8.036123451979464 hab/km².\nO município Frei Miguelinho/PE possui densidade demográfica de 67.19477222509519 hab/km².\nO município Gameleira/PE possui densidade demográfica de 109.04828879512424 hab/km².\nO município Garanhuns/PE possui densidade demográfica de 282.2113182859012 hab/km².\nO município Glória do Goitá/PE possui densidade demográfica de 125.17361859983608 hab/km².\nO município Goiana/PE possui densidade demográfica de 150.72128795728062 hab/km².\nO município Granito/PE possui densidade demográfica de 13.133693527991722 hab/km².\nO município Gravatá/PE possui densidade demográfica de 150.86722311016396 hab/km².\nO município Iati/PE possui densidade demográfica de 28.907012627137323 hab/km².\nO município Ibimirim/PE possui densidade demográfica de 14.138394074820082 hab/km².\nO município Ibirajuba/PE possui densidade demográfica de 39.73628691983122 hab/km².\nO município Igarassu/PE possui densidade demográfica de 333.8820526246891 hab/km².\nO município Iguaraci/PE possui densidade demográfica de 14.053905718683259 hab/km².\nO município Ilha de Itamaracá/PE possui densidade demográfica de 328.1943611277744 hab/km².\nO município Inajá/PE possui densidade demográfica de 16.135469958986935 hab/km².\nO município Ingazeira/PE possui densidade demográfica de 18.45118397833135 hab/km².\nO município Ipojuca/PE possui densidade demográfica de 152.9794540039081 hab/km².\nO município Ipubi/PE possui densidade demográfica de 32.64377423324279 hab/km².\nO município Itacuruba/PE possui densidade demográfica de 10.159756296072368 hab/km².\nO município Itaíba/PE possui densidade demográfica de 24.20398606168993 hab/km².\nO município Itambé/PE possui densidade demográfica de 116.131360519668 hab/km².\nO município Itapetim/PE possui densidade demográfica de 34.28677287884401 hab/km².\nO município Itapissuma/PE possui densidade demográfica de 320.16433189655174 hab/km².\nO município Itaquitinga/PE possui densidade demográfica de 151.73080642042157 hab/km².\nO município Jaboatão dos Guararapes/PE possui densidade demográfica de 2491.862847423557 hab/km².\nO município Jaqueira/PE possui densidade demográfica de 131.87707831670681 hab/km².\nO município Jataúba/PE possui densidade demográfica de 23.53387485494957 hab/km².\nO município Jatobá/PE possui densidade demográfica de 50.251925430072696 hab/km².\nO município João Alfredo/PE possui densidade demográfica de 227.52368265245707 hab/km².\nO município Joaquim Nabuco/PE possui densidade demográfica de 129.3929450369155 hab/km².\nO município Jucati/PE possui densidade demográfica de 87.92703150912106 hab/km².\nO município Jupi/PE possui densidade demográfica de 130.53624154681398 hab/km².\nO município Jurema/PE possui densidade demográfica de 98.08431703204047 hab/km².\nO município Lagoa do Carro/PE possui densidade demográfica de 229.75455719822017 hab/km².\nO município Lagoa do Itaenga/PE possui densidade demográfica de 360.6668994413408 hab/km².\nO município Lagoa do Ouro/PE possui densidade demográfica de 61.03843831756893 hab/km².\nO município Lagoa dos Gatos/PE possui densidade demográfica de 70.06326558083187 hab/km².\nO município Lagoa Grande/PE possui densidade demográfica de 12.310022175347504 hab/km².\nO município Lajedo/PE possui densidade demográfica de 193.69645690111054 hab/km².\nO município Limoeiro/PE possui densidade demográfica de 202.524293124863 hab/km².\nO município Macaparana/PE possui densidade demográfica de 221.4252660805183 hab/km².\nO município Machados/PE possui densidade demográfica de 226.44903397734845 hab/km².\nO município Manari/PE possui densidade demográfica de 47.55805696552087 hab/km².\nO município Maraial/PE possui densidade demográfica de 61.18977335267924 hab/km².\nO município Mirandiba/PE possui densidade demográfica de 17.413104858339015 hab/km².\nO município Moreilândia/PE possui densidade demográfica de 27.515633882888004 hab/km².\nO município Moreno/PE possui densidade demográfica de 289.1620339674606 hab/km².\nO município Nazaré da Mata/PE possui densidade demográfica de 204.9514175429256 hab/km².\nO município Olinda/PE possui densidade demográfica de 9063.795585412669 hab/km².\nO município Orobó/PE possui densidade demográfica de 164.9935093033319 hab/km².\nO município Orocó/PE possui densidade demográfica de 23.758021486769053 hab/km².\nO município Ouricuri/PE possui densidade demográfica de 26.562493551089815 hab/km².\nO município Palmares/PE possui densidade demográfica de 175.4428365115388 hab/km².\nO município Palmeirina/PE possui densidade demográfica de 51.82255410707505 hab/km².\nO município Panelas/PE possui densidade demográfica de 69.13517010837332 hab/km².\nO município Paranatama/PE possui densidade demográfica de 47.64606522586514 hab/km².\nO município Parnamirim/PE possui densidade demográfica de 7.787117218754692 hab/km².\nO município Passira/PE possui densidade demográfica de 87.61170277879789 hab/km².\nO município Paudalho/PE possui densidade demográfica de 185.06360131166446 hab/km².\nO município Paulista/PE possui densidade demográfica de 3087.7196588223205 hab/km².\nO município Pedra/PE possui densidade demográfica de 26.075047932073403 hab/km².\nO município Pesqueira/PE possui densidade demográfica de 63.21292966631175 hab/km².\nO município Petrolândia/PE possui densidade demográfica de 30.751466969524895 hab/km².\nO município Petrolina/PE possui densidade demográfica de 64.4389252652969 hab/km².\nO município Poção/PE possui densidade demográfica de 45.56028368794326 hab/km².\nO município Pombos/PE possui densidade demográfica de 118.34826262427404 hab/km².\nO município Primavera/PE possui densidade demográfica de 121.96206552318722 hab/km².\nO município Quipapá/PE possui densidade demográfica de 104.873818402567 hab/km².\nO município Quixaba/PE possui densidade demográfica de 31.98234540363533 hab/km².\nO município Recife/PE possui densidade demográfica de 7039.479948727339 hab/km².\nO município Riacho das Almas/PE possui densidade demográfica de 61.02547770700637 hab/km².\nO município Ribeirão/PE possui densidade demográfica de 154.3556790552275 hab/km².\nO município Rio Formoso/PE possui densidade demográfica de 97.38415545590433 hab/km².\nO município Sairé/PE possui densidade demográfica de 59.354702434387704 hab/km².\nO município Salgadinho/PE possui densidade demográfica de 106.76450355423069 hab/km².\nO município Salgueiro/PE possui densidade demográfica de 33.57165300182 hab/km².\nO município Saloá/PE possui densidade demográfica de 60.73072040622024 hab/km².\nO município Sanharó/PE possui densidade demográfica de 81.71126577096283 hab/km².\nO município Santa Cruz/PE possui densidade demográfica de 10.823765466503177 hab/km².\nO município Santa Cruz da Baixa Verde/PE possui densidade demográfica de 102.39276081092838 hab/km².\nO município Santa Cruz do Capibaribe/PE possui densidade demográfica de 261.1971011899436 hab/km².\nO município Santa Filomena/PE possui densidade demográfica de 13.30381573056067 hab/km².\nO município Santa Maria da Boa Vista/PE possui densidade demográfica de 13.139831666211292 hab/km².\nO município Santa Maria do Cambucá/PE possui densidade demográfica de 141.30222463374932 hab/km².\nO município Santa Terezinha/PE possui densidade demográfica de 56.19407945191472 hab/km².\nO município São Benedito do Sul/PE possui densidade demográfica de 86.87063808574278 hab/km².\nO município São Bento do Una/PE possui densidade demográfica de 74.03462420913579 hab/km².\nO município São Caitano/PE possui densidade demográfica de 92.2268413208879 hab/km².\nO município São João/PE possui densidade demográfica de 82.49912902101963 hab/km².\nO município São Joaquim do Monte/PE possui densidade demográfica de 88.38654012079378 hab/km².\nO município São José da Coroa Grande/PE possui densidade demográfica de 262.18632823766944 hab/km².\nO município São José do Belmonte/PE possui densidade demográfica de 22.12687149359944 hab/km².\nO município São José do Egito/PE possui densidade demográfica de 39.84202884037653 hab/km².\nO município São Lourenço da Mata/PE possui densidade demográfica de 392.56419060699704 hab/km².\nO município São Vicente Ferrer/PE possui densidade demográfica de 149.1358891130801 hab/km².\nO município Serra Talhada/PE possui densidade demográfica de 26.587830242180395 hab/km².\nO município Serrita/PE possui densidade demográfica de 11.924463005607379 hab/km².\nO município Sertânia/PE possui densidade demográfica de 13.95274888190524 hab/km².\nO município Sirinhaém/PE possui densidade demográfica de 107.56787058540883 hab/km².\nO município Solidão/PE possui densidade demográfica de 41.5028901734104 hab/km².\nO município Surubim/PE possui densidade demográfica de 231.41263940520446 hab/km².\nO município Tabira/PE possui densidade demográfica de 68.10906935388263 hab/km².\nO município Tacaimbó/PE possui densidade demográfica de 55.90949033391916 hab/km².\nO município Tacaratu/PE possui densidade demográfica de 17.451543261132596 hab/km².\nO município Tamandaré/PE possui densidade demográfica de 96.6590453081984 hab/km².\nO município Taquaritinga do Norte/PE possui densidade demográfica de 52.40750873353255 hab/km².\nO município Terezinha/PE possui densidade demográfica de 44.48332783096732 hab/km².\nO município Terra Nova/PE possui densidade demográfica de 28.94851794071763 hab/km².\nO município Timbaúba/PE possui densidade demográfica de 184.63570252469813 hab/km².\nO município Toritama/PE possui densidade demográfica de 1383.4241245136186 hab/km².\nO município Tracunhaém/PE possui densidade demográfica de 110.27113776501393 hab/km².\nO município Trindade/PE possui densidade demográfica de 113.7753768406378 hab/km².\nO município Triunfo/PE possui densidade demográfica de 78.35213032581453 hab/km².\nO município Tupanatinga/PE possui densidade demográfica de 25.697812661104507 hab/km².\nO município Tuparetama/PE possui densidade demográfica de 44.38035504284034 hab/km².\nO município Venturosa/PE possui densidade demográfica de 50.04832725345306 hab/km².\nO município Verdejante/PE possui densidade demográfica de 19.20426854886144 hab/km².\nO município Vertente do Lério/PE possui densidade demográfica de 106.92652451446422 hab/km².\nO município Vertentes/PE possui densidade demográfica de 92.81312076605714 hab/km².\nO município Vicência/PE possui densidade demográfica de 134.7776510832383 hab/km².\nO município Vitória de Santo Antão/PE possui densidade demográfica de 348.79240017174754 hab/km².\nO município Xexéu/PE possui densidade demográfica de 127.18166230484613 hab/km².\nO município Acauã/PI possui densidade demográfica de 5.274345688853461 hab/km².\nO município Agricolândia/PI possui densidade demográfica de 45.34376945655074 hab/km².\nO município Água Branca/PI possui densidade demográfica de 169.52802967848308 hab/km².\nO município Alagoinha do Piauí/PI possui densidade demográfica de 13.773499943712709 hab/km².\nO município Alegrete do Piauí/PI possui densidade demográfica de 18.227158572388667 hab/km².\nO município Alto Longá/PI possui densidade demográfica de 7.852276389080698 hab/km².\nO município Altos/PI possui densidade demográfica de 40.53839567278575 hab/km².\nO município Alvorada do Gurguéia/PI possui densidade demográfica de 2.368756801380915 hab/km².\nO município Amarante/PI possui densidade demográfica de 14.832929362880886 hab/km².\nO município Angical do Piauí/PI possui densidade demográfica de 29.860365198711065 hab/km².\nO município Anísio de Abreu/PI possui densidade demográfica de 26.926719545400733 hab/km².\nO município Antônio Almeida/PI possui densidade demográfica de 4.706155632984902 hab/km².\nO município Aroazes/PI possui densidade demográfica de 7.033322785580411 hab/km².\nO município Aroeiras do Itaim/PI possui densidade demográfica de 9.488994322159135 hab/km².\nO município Arraial/PI possui densidade demográfica de 6.86624875505302 hab/km².\nO município Assunção do Piauí/PI possui densidade demográfica de 4.437806825575206 hab/km².\nO município Avelino Lopes/PI possui densidade demográfica de 8.47708192903977 hab/km².\nO município Baixa Grande do Ribeiro/PI possui densidade demográfica de 1.3466668203372814 hab/km².\nO município Barra d`Alcântara/PI possui densidade demográfica de 14.62525628369656 hab/km².\nO município Barras/PI possui densidade demográfica de 26.078613792301432 hab/km².\nO município Barreiras do Piauí/PI possui densidade demográfica de 1.5944465535007322 hab/km².\nO município Barro Duro/PI possui densidade demográfica de 50.388956680902986 hab/km².\nO município Batalha/PI possui densidade demográfica de 16.221285165838 hab/km².\nO município Bela Vista do Piauí/PI possui densidade demográfica de 7.565229580087707 hab/km².\nO município Belém do Piauí/PI possui densidade demográfica de 13.498849062808286 hab/km².\nO município Beneditinos/PI possui densidade demográfica de 12.568160491009156 hab/km².\nO município Bertolínia/PI possui densidade demográfica de 4.340836012861737 hab/km².\nO município Betânia do Piauí/PI possui densidade demográfica de 10.651484832923092 hab/km².\nO município Boa Hora/PI possui densidade demográfica de 18.650946470361703 hab/km².\nO município Bocaina/PI possui densidade demográfica de 16.267034030828803 hab/km².\nO município Bom Jesus/PI possui densidade demográfica de 4.137548956150647 hab/km².\nO município Bom Princípio do Piauí/PI possui densidade demográfica de 10.169296546963974 hab/km².\nO município Bonfim do Piauí/PI possui densidade demográfica de 18.64734967670551 hab/km².\nO município Boqueirão do Piauí/PI possui densidade demográfica de 22.25296442687747 hab/km².\nO município Brasileira/PI possui densidade demográfica de 9.042921524332792 hab/km².\nO município Brejo do Piauí/PI possui densidade demográfica de 1.7633372416825441 hab/km².\nO município Buriti dos Lopes/PI possui densidade demográfica de 27.596284614716865 hab/km².\nO município Buriti dos Montes/PI possui densidade demográfica de 3.0066626195746027 hab/km².\nO município Cabeceiras do Piauí/PI possui densidade demográfica de 16.3147256503377 hab/km².\nO município Cajazeiras do Piauí/PI possui densidade demográfica de 6.499338984368924 hab/km².\nO município Cajueiro da Praia/PI possui densidade demográfica de 26.362666077803542 hab/km².\nO município Caldeirão Grande do Piauí/PI possui densidade demográfica de 11.459112125926975 hab/km².\nO município Campinas do Piauí/PI possui densidade demográfica de 6.506256015399422 hab/km².\nO município Campo Alegre do Fidalgo/PI possui densidade demográfica de 7.134387351778656 hab/km².\nO município Campo Grande do Piauí/PI possui densidade demográfica de 17.932848026168106 hab/km².\nO município Campo Largo do Piauí/PI possui densidade demográfica de 14.238174968606112 hab/km².\nO município Campo Maior/PI possui densidade demográfica de 26.95991549850511 hab/km².\nO município Canavieira/PI possui densidade demográfica de 1.8128690120071942 hab/km².\nO município Canto do Buriti/PI possui densidade demográfica de 4.628216865018818 hab/km².\nO município Capitão de Campos/PI possui densidade demográfica de 18.496377729368255 hab/km².\nO município Capitão Gervásio Oliveira/PI possui densidade demográfica de 3.4192405018647998 hab/km².\nO município Caracol/PI possui densidade demográfica de 6.339077320355564 hab/km².\nO município Caraúbas do Piauí/PI possui densidade demográfica de 11.719164280411496 hab/km².\nO município Caridade do Piauí/PI possui densidade demográfica de 9.625817775650232 hab/km².\nO município Castelo do Piauí/PI possui densidade demográfica de 9.009478230533759 hab/km².\nO município Caxingó/PI possui densidade demográfica de 10.322223815474118 hab/km².\nO município Cocal/PI possui densidade demográfica de 20.508861756597085 hab/km².\nO município Cocal de Telha/PI possui densidade demográfica de 16.039842614582962 hab/km².\nO município Cocal dos Alves/PI possui densidade demográfica de 15.5777349101177 hab/km².\nO município Coivaras/PI possui densidade demográfica de 7.849639546858908 hab/km².\nO município Colônia do Gurguéia/PI possui densidade demográfica de 14.016998745994147 hab/km².\nO município Colônia do Piauí/PI possui densidade demográfica de 7.841792650890945 hab/km².\nO município Conceição do Canindé/PI possui densidade demográfica de 5.382422631433348 hab/km².\nO município Coronel José Dias/PI possui densidade demográfica de 2.371502282198849 hab/km².\nO município Corrente/PI possui densidade demográfica de 8.33439944890026 hab/km².\nO município Cristalândia do Piauí/PI possui densidade demográfica de 6.510100590240253 hab/km².\nO município Cristino Castro/PI possui densidade demográfica de 5.405829912150525 hab/km².\nO município Curimatá/PI possui densidade demográfica de 4.603557586180344 hab/km².\nO município Currais/PI possui densidade demográfica de 1.4901826614206155 hab/km².\nO município Curral Novo do Piauí/PI possui densidade demográfica de 6.47206603660725 hab/km².\nO município Curralinhos/PI possui densidade demográfica de 12.094838802949255 hab/km².\nO município Demerval Lobão/PI possui densidade demográfica de 61.24256261242562 hab/km².\nO município Dirceu Arcoverde/PI possui densidade demográfica de 6.563034629225415 hab/km².\nO município Dom Expedito Lopes/PI possui densidade demográfica de 29.985849271922216 hab/km².\nO município Dom Inocêncio/PI possui densidade demográfica de 2.388783955226773 hab/km².\nO município Domingos Mourão/PI possui densidade demográfica de 5.03518964621416 hab/km².\nO município Elesbão Veloso/PI possui densidade demográfica de 10.76973313147505 hab/km².\nO município Eliseu Martins/PI possui densidade demográfica de 4.278050346187354 hab/km².\nO município Esperantina/PI possui densidade demográfica de 41.44663198788437 hab/km².\nO município Fartura do Piauí/PI possui densidade demográfica de 7.1172081018908155 hab/km².\nO município Flores do Piauí/PI possui densidade demográfica de 4.611663304215563 hab/km².\nO município Floresta do Piauí/PI possui densidade demográfica de 12.747817154596817 hab/km².\nO município Floriano/PI possui densidade demográfica de 16.919625181470238 hab/km².\nO município Francinópolis/PI possui densidade demográfica de 19.482694454782287 hab/km².\nO município Francisco Ayres/PI possui densidade demográfica de 6.81970509383378 hab/km².\nO município Francisco Macedo/PI possui densidade demográfica de 18.540700669757857 hab/km².\nO município Francisco Santos/PI possui densidade demográfica de 17.46838531289391 hab/km².\nO município Fronteiras/PI possui densidade demográfica de 14.331941006600662 hab/km².\nO município Geminiano/PI possui densidade demográfica de 11.837325953472282 hab/km².\nO município Gilbués/PI possui densidade demográfica de 2.9762858516263417 hab/km².\nO município Guadalupe/PI possui densidade demográfica de 10.031360212585117 hab/km².\nO município Guaribas/PI possui densidade demográfica de 1.4113776084509482 hab/km².\nO município Hugo Napoleão/PI possui densidade demográfica de 16.800320769847634 hab/km².\nO município Ilha Grande/PI possui densidade demográfica de 66.36390708755212 hab/km².\nO município Inhuma/PI possui densidade demográfica de 15.175522888511786 hab/km².\nO município Ipiranga do Piauí/PI possui densidade demográfica de 17.673810471263714 hab/km².\nO município Isaías Coelho/PI possui densidade demográfica de 10.593389601185491 hab/km².\nO município Itainópolis/PI possui densidade demográfica de 13.414236551349394 hab/km².\nO município Itaueira/PI possui densidade demográfica de 4.180598078443963 hab/km².\nO município Jacobina do Piauí/PI possui densidade demográfica de 4.174509374772014 hab/km².\nO município Jaicós/PI possui densidade demográfica de 20.846337009039 hab/km².\nO município Jardim do Mulato/PI possui densidade demográfica de 8.451505344709227 hab/km².\nO município Jatobá do Piauí/PI possui densidade demográfica de 7.127657945899607 hab/km².\nO município Jerumenha/PI possui densidade demográfica de 2.3509754673835626 hab/km².\nO município João Costa/PI possui densidade demográfica de 1.6442252144158556 hab/km².\nO município Joaquim Pires/PI possui densidade demográfica de 18.682225046648096 hab/km².\nO município Joca Marques/PI possui densidade demográfica de 30.64167267483778 hab/km².\nO município José de Freitas/PI possui densidade demográfica de 24.10966206815847 hab/km².\nO município Juazeiro do Piauí/PI possui densidade demográfica de 5.750447270441468 hab/km².\nO município Júlio Borges/PI possui densidade demográfica de 4.142285542475195 hab/km².\nO município Jurema/PI possui densidade demográfica de 3.551407747525336 hab/km².\nO município Lagoa Alegre/PI possui densidade demográfica de 20.290883291947498 hab/km².\nO município Lagoa de São Francisco/PI possui densidade demográfica de 41.26188640452326 hab/km².\nO município Lagoa do Barro do Piauí/PI possui densidade demográfica de 3.5841640648525286 hab/km².\nO município Lagoa do Piauí/PI possui densidade demográfica de 9.054684386939503 hab/km².\nO município Lagoa do Sítio/PI possui densidade demográfica de 6.027090841307319 hab/km².\nO município Lagoinha do Piauí/PI possui densidade demográfica de 39.34814814814815 hab/km².\nO município Landri Sales/PI possui densidade demográfica de 4.851274136949053 hab/km².\nO município Luís Correia/PI possui densidade demográfica de 26.524609451598142 hab/km².\nO município Luzilândia/PI possui densidade demográfica de 35.09760772343295 hab/km².\nO município Madeiro/PI possui densidade demográfica de 44.12080158058143 hab/km².\nO município Manoel Emídio/PI possui densidade demográfica de 3.2199285970178755 hab/km².\nO município Marcolândia/PI possui densidade demográfica de 54.295246038365306 hab/km².\nO município Marcos Parente/PI possui densidade demográfica de 6.577995600891632 hab/km².\nO município Massapê do Piauí/PI possui densidade demográfica de 11.935601481396198 hab/km².\nO município Matias Olímpio/PI possui densidade demográfica de 46.26496443875072 hab/km².\nO município Miguel Alves/PI possui densidade demográfica de 23.167660417159954 hab/km².\nO município Miguel Leão/PI possui densidade demográfica de 13.398203592814372 hab/km².\nO município Milton Brandão/PI possui densidade demográfica de 4.934608599297243 hab/km².\nO município Monsenhor Gil/PI possui densidade demográfica de 18.168550982012555 hab/km².\nO município Monsenhor Hipólito/PI possui densidade demográfica de 18.41167825025534 hab/km².\nO município Monte Alegre do Piauí/PI possui densidade demográfica de 4.278453056953675 hab/km².\nO município Morro Cabeça no Tempo/PI possui densidade demográfica de 1.9216416147836026 hab/km².\nO município Morro do Chapéu do Piauí/PI possui densidade demográfica de 19.796521368302415 hab/km².\nO município Murici dos Portelas/PI possui densidade demográfica de 17.570737580702083 hab/km².\nO município Nazaré do Piauí/PI possui densidade demográfica de 5.563746352140078 hab/km².\nO município Nazária/PI possui densidade demográfica de 22.18982920322341 hab/km².\nO município Nossa Senhora de Nazaré/PI possui densidade demográfica de 12.788412956829282 hab/km².\nO município Nossa Senhora dos Remédios/PI possui densidade demográfica de 22.89045719545873 hab/km².\nO município Nova Santa Rita/PI possui densidade demográfica de 4.602413876492185 hab/km².\nO município Novo Oriente do Piauí/PI possui densidade demográfica de 12.369367825937982 hab/km².\nO município Novo Santo Antônio/PI possui densidade demográfica de 6.767557243984971 hab/km².\nO município Oeiras/PI possui densidade demográfica de 13.18783788284138 hab/km².\nO município Olho d`Água do Piauí/PI possui densidade demográfica de 11.958105646630237 hab/km².\nO município Padre Marcos/PI possui densidade demográfica de 24.470666078517862 hab/km².\nO município Paes Landim/PI possui densidade demográfica de 10.112611490358264 hab/km².\nO município Pajeú do Piauí/PI possui densidade demográfica de 3.116283810706376 hab/km².\nO município Palmeira do Piauí/PI possui densidade demográfica de 2.4674946009656487 hab/km².\nO município Palmeirais/PI possui densidade demográfica de 9.168345362131298 hab/km².\nO município Paquetá/PI possui densidade demográfica de 9.247201534139053 hab/km².\nO município Parnaguá/PI possui densidade demográfica de 2.99654738020809 hab/km².\nO município Parnaíba/PI possui densidade demográfica de 334.51569208164017 hab/km².\nO município Passagem Franca do Piauí/PI possui densidade demográfica de 5.35069031673356 hab/km².\nO município Patos do Piauí/PI possui densidade demográfica de 8.12267163384779 hab/km².\nO município Pau d`Arco do Piauí/PI possui densidade demográfica de 8.72057936028968 hab/km².\nO município Paulistana/PI possui densidade demográfica de 10.043351134033179 hab/km².\nO município Pavussu/PI possui densidade demográfica de 3.3583936921243236 hab/km².\nO município Pedro II/PI possui densidade demográfica de 24.697180269129184 hab/km².\nO município Pedro Laurentino/PI possui densidade demográfica de 2.7655858629960703 hab/km².\nO município Picos/PI possui densidade demográfica de 137.29428485936563 hab/km².\nO município Pimenteiras/PI possui densidade demográfica de 2.5712613929473846 hab/km².\nO município Pio IX/PI possui densidade demográfica de 9.075268596314633 hab/km².\nO município Piracuruca/PI possui densidade demográfica de 11.574896761482266 hab/km².\nO município Piripiri/PI possui densidade demográfica de 43.88720518407586 hab/km².\nO município Porto/PI possui densidade demográfica de 47.07581513137069 hab/km².\nO município Porto Alegre do Piauí/PI possui densidade demográfica de 2.188226843617458 hab/km².\nO município Prata do Piauí/PI possui densidade demográfica de 15.713339785055773 hab/km².\nO município Queimada Nova/PI possui densidade demográfica de 6.324312333629104 hab/km².\nO município Redenção do Gurguéia/PI possui densidade demográfica de 3.4035518494657637 hab/km².\nO município Regeneração/PI possui densidade demográfica de 14.033124440465533 hab/km².\nO município Riacho Frio/PI possui densidade demográfica de 1.9085549705233789 hab/km².\nO município Ribeira do Piauí/PI possui densidade demográfica de 4.2450434661382355 hab/km².\nO município Ribeiro Gonçalves/PI possui densidade demográfica de 1.7202987715382914 hab/km².\nO município Rio Grande do Piauí/PI possui densidade demográfica de 9.863983017532824 hab/km².\nO município Santa Cruz do Piauí/PI possui densidade demográfica de 9.854157810405153 hab/km².\nO município Santa Cruz dos Milagres/PI possui densidade demográfica de 3.8727721862687057 hab/km².\nO município Santa Filomena/PI possui densidade demográfica de 1.1533571471816917 hab/km².\nO município Santa Luz/PI possui densidade demográfica de 4.6451080179299655 hab/km².\nO município Santa Rosa do Piauí/PI possui densidade demográfica de 15.135214579659024 hab/km².\nO município Santana do Piauí/PI possui densidade demográfica de 34.84268707482993 hab/km².\nO município Santo Antônio de Lisboa/PI possui densidade demográfica de 15.50593701600413 hab/km².\nO município Santo Antônio dos Milagres/PI possui densidade demográfica de 62.11161387631976 hab/km².\nO município Santo Inácio do Piauí/PI possui densidade demográfica de 4.277222150570414 hab/km².\nO município São Braz do Piauí/PI possui densidade demográfica de 6.5710890365043575 hab/km².\nO município São Félix do Piauí/PI possui densidade demográfica de 4.669527113383239 hab/km².\nO município São Francisco de Assis do Piauí/PI possui densidade demográfica de 5.059069429298437 hab/km².\nO município São Francisco do Piauí/PI possui densidade demográfica de 4.697651174412793 hab/km².\nO município São Gonçalo do Gurguéia/PI possui densidade demográfica de 2.039269472316466 hab/km².\nO município São Gonçalo do Piauí/PI possui densidade demográfica de 31.646917853814404 hab/km².\nO município São João da Canabrava/PI possui densidade demográfica de 9.255017906221372 hab/km².\nO município São João da Fronteira/PI possui densidade demográfica de 7.331964909069515 hab/km².\nO município São João da Serra/PI possui densidade demográfica de 6.117237953303527 hab/km².\nO município São João da Varjota/PI possui densidade demográfica de 11.76544990007842 hab/km².\nO município São João do Arraial/PI possui densidade demográfica de 34.38320209973753 hab/km².\nO município São João do Piauí/PI possui densidade demográfica de 12.795119684245666 hab/km².\nO município São José do Divino/PI possui densidade demográfica de 16.131357127189546 hab/km².\nO município São José do Peixe/PI possui densidade demográfica de 2.8745231787564967 hab/km².\nO município São José do Piauí/PI possui densidade demográfica de 18.06000822030415 hab/km².\nO município São Julião/PI possui densidade demográfica de 22.06539912127221 hab/km².\nO município São Lourenço do Piauí/PI possui densidade demográfica de 6.580844643308409 hab/km².\nO município São Luis do Piauí/PI possui densidade demográfica de 11.620836736545966 hab/km².\nO município São Miguel da Baixa Grande/PI possui densidade demográfica de 5.492074234102918 hab/km².\nO município São Miguel do Fidalgo/PI possui densidade demográfica de 3.6585365853658534 hab/km².\nO município São Miguel do Tapuio/PI possui densidade demográfica de 3.4826061739728287 hab/km².\nO município São Pedro do Piauí/PI possui densidade demográfica de 26.315383279631096 hab/km².\nO município São Raimundo Nonato/PI possui densidade demográfica de 13.382596456366949 hab/km².\nO município Sebastião Barros/PI possui densidade demográfica de 3.9833504900863805 hab/km².\nO município Sebastião Leal/PI possui densidade demográfica de 1.3060074438616698 hab/km².\nO município Sigefredo Pacheco/PI possui densidade demográfica de 9.947362433944509 hab/km².\nO município Simões/PI possui densidade demográfica de 13.233290404464602 hab/km².\nO município Simplício Mendes/PI possui densidade demográfica de 8.973911234293611 hab/km².\nO município Socorro do Piauí/PI possui densidade demográfica de 5.935551617772528 hab/km².\nO município Sussuapara/PI possui densidade demográfica de 29.704339532665713 hab/km².\nO município Tamboril do Piauí/PI possui densidade demográfica de 1.7343917343917346 hab/km².\nO município Tanque do Piauí/PI possui densidade demográfica de 6.5710272873194215 hab/km².\nO município Teresina/PI possui densidade demográfica de 584.9437491917987 hab/km².\nO município União/PI possui densidade demográfica de 36.34922663939665 hab/km².\nO município Uruçuí/PI possui densidade demográfica de 2.3952942910706367 hab/km².\nO município Valença do Piauí/PI possui densidade demográfica de 15.229689127323676 hab/km².\nO município Várzea Branca/PI possui densidade demográfica de 10.899369952968321 hab/km².\nO município Várzea Grande/PI possui densidade demográfica de 18.29458672629847 hab/km².\nO município Vera Mendes/PI possui densidade demográfica de 8.731760095914845 hab/km².\nO município Vila Nova do Piauí/PI possui densidade demográfica de 14.089410040307806 hab/km².\nO município Wall Ferraz/PI possui densidade demográfica de 15.852438979221452 hab/km².\nO município Angra dos Reis/RJ possui densidade demográfica de 205.44546655516368 hab/km².\nO município Aperibé/RJ possui densidade demográfica de 107.91420118343196 hab/km².\nO município Araruama/RJ possui densidade demográfica de 175.5556252155105 hab/km².\nO município Areal/RJ possui densidade demográfica de 102.9841327082582 hab/km².\nO município Armação dos Búzios/RJ possui densidade demográfica de 392.145702902675 hab/km².\nO município Arraial do Cabo/RJ possui densidade demográfica de 172.90535903674592 hab/km².\nO município Barra do Piraí/RJ possui densidade demográfica de 163.70105532238284 hab/km².\nO município Barra Mansa/RJ possui densidade demográfica de 324.9328435941012 hab/km².\nO município Belford Roxo/RJ possui densidade demográfica de 6030.994602929839 hab/km².\nO município Bom Jardim/RJ possui densidade demográfica de 65.86158485856906 hab/km².\nO município Bom Jesus do Itabapoana/RJ possui densidade demográfica de 59.133643939014405 hab/km².\nO município Cabo Frio/RJ possui densidade demográfica de 453.7473807319331 hab/km².\nO município Cachoeiras de Macacu/RJ possui densidade demográfica de 56.90186621933319 hab/km².\nO município Cambuci/RJ possui densidade demográfica de 26.396653017625063 hab/km².\nO município Campos dos Goytacazes/RJ possui densidade demográfica de 115.16403009908859 hab/km².\nO município Cantagalo/RJ possui densidade demográfica de 26.465406790518898 hab/km².\nO município Carapebus/RJ possui densidade demográfica de 43.355077402395096 hab/km².\nO município Cardoso Moreira/RJ possui densidade demográfica de 24.016926214665574 hab/km².\nO município Carmo/RJ possui densidade demográfica de 54.15294775423992 hab/km².\nO município Casimiro de Abreu/RJ possui densidade demográfica de 76.71289363456823 hab/km².\nO município Comendador Levy Gasparian/RJ possui densidade demográfica de 76.5272710262887 hab/km².\nO município Conceição de Macabu/RJ possui densidade demográfica de 61.07927549169234 hab/km².\nO município Cordeiro/RJ possui densidade demográfica de 175.59088955737002 hab/km².\nO município Duas Barras/RJ possui densidade demográfica de 29.136565990456642 hab/km².\nO município Duque de Caxias/RJ possui densidade demográfica de 1828.5103288995338 hab/km².\nO município Engenheiro Paulo de Frontin/RJ possui densidade demográfica de 99.57123514367385 hab/km².\nO município Guapimirim/RJ possui densidade demográfica de 142.7031072428417 hab/km².\nO município Iguaba Grande/RJ possui densidade demográfica de 439.8652550529355 hab/km².\nO município Itaboraí/RJ possui densidade demográfica de 506.55947208216185 hab/km².\nO município Itaguaí/RJ possui densidade demográfica de 395.4435059992025 hab/km².\nO município Italva/RJ possui densidade demográfica de 47.862636988632495 hab/km².\nO município Itaocara/RJ possui densidade demográfica de 53.088051189317014 hab/km².\nO município Itaperuna/RJ possui densidade demográfica de 86.70725749543128 hab/km².\nO município Itatiaia/RJ possui densidade demográfica de 117.40974913318377 hab/km².\nO município Japeri/RJ possui densidade demográfica de 1166.385733479907 hab/km².\nO município Laje do Muriaé/RJ possui densidade demográfica de 29.951594191302956 hab/km².\nO município Macaé/RJ possui densidade demográfica de 169.88782512224188 hab/km².\nO município Macuco/RJ possui densidade demográfica de 67.7946474523932 hab/km².\nO município Magé/RJ possui densidade demográfica de 585.1274131274131 hab/km².\nO município Mangaratiba/RJ possui densidade demográfica de 102.28669229258438 hab/km².\nO município Maricá/RJ possui densidade demográfica de 351.5486664644069 hab/km².\nO município Mendes/RJ possui densidade demográfica de 184.82069249793898 hab/km².\nO município Mesquita/RJ possui densidade demográfica de 4310.701484895033 hab/km².\nO município Miguel Pereira/RJ possui densidade demográfica de 85.21336191991148 hab/km².\nO município Miracema/RJ possui densidade demográfica de 88.15145643821221 hab/km².\nO município Natividade/RJ possui densidade demográfica de 38.99777628380824 hab/km².\nO município Nilópolis/RJ possui densidade demográfica de 8118.875709128416 hab/km².\nO município Niterói/RJ possui densidade demográfica de 3640.6959378733577 hab/km².\nO município Nova Friburgo/RJ possui densidade demográfica de 195.07183338511481 hab/km².\nO município Nova Iguaçu/RJ possui densidade demográfica de 1527.5913669064748 hab/km².\nO município Paracambi/RJ possui densidade demográfica de 262.26625111308994 hab/km².\nO município Paraíba do Sul/RJ possui densidade demográfica de 70.7698137908463 hab/km².\nO município Paraty/RJ possui densidade demográfica de 40.574023025782395 hab/km².\nO município Paty do Alferes/RJ possui densidade demográfica de 82.68193224592221 hab/km².\nO município Petrópolis/RJ possui densidade demográfica de 371.848454385524 hab/km².\nO município Pinheiral/RJ possui densidade demográfica de 296.8639749117993 hab/km².\nO município Piraí/RJ possui densidade demográfica de 52.06775099924809 hab/km².\nO município Porciúncula/RJ possui densidade demográfica de 58.80210575108433 hab/km².\nO município Porto Real/RJ possui densidade demográfica de 326.935960591133 hab/km².\nO município Quatis/RJ possui densidade demográfica de 44.71669754273131 hab/km².\nO município Queimados/RJ possui densidade demográfica de 1822.4834874504622 hab/km².\nO município Quissamã/RJ possui densidade demográfica de 28.3950790466705 hab/km².\nO município Resende/RJ possui densidade demográfica de 109.35311572700297 hab/km².\nO município Rio Bonito/RJ possui densidade demográfica de 121.69960127941113 hab/km².\nO município Rio Claro/RJ possui densidade demográfica de 20.811685597238647 hab/km².\nO município Rio das Flores/RJ possui densidade demográfica de 17.898434069954632 hab/km².\nO município Rio das Ostras/RJ possui densidade demográfica de 461.38665735242756 hab/km².\nO município Rio de Janeiro/RJ possui densidade demográfica de 5265.809644416303 hab/km².\nO município Santa Maria Madalena/RJ possui densidade demográfica de 12.667533997741666 hab/km².\nO município Santo Antônio de Pádua/RJ possui densidade demográfica de 67.27161230442853 hab/km².\nO município São Fidélis/RJ possui densidade demográfica de 36.39439295823801 hab/km².\nO município São Francisco de Itabapoana/RJ possui densidade demográfica de 36.84294928904885 hab/km².\nO município São Gonçalo/RJ possui densidade demográfica de 4035.8806669088854 hab/km².\nO município São João da Barra/RJ possui densidade demográfica de 71.96510196905766 hab/km².\nO município São João de Meriti/RJ possui densidade demográfica de 13023.083475298126 hab/km².\nO município São José de Ubá/RJ possui densidade demográfica de 27.980661658941983 hab/km².\nO município São José do Vale do Rio Preto/RJ possui densidade demográfica de 91.87043505874881 hab/km².\nO município São Pedro da Aldeia/RJ possui densidade demográfica de 264.05541031881967 hab/km².\nO município São Sebastião do Alto/RJ possui densidade demográfica de 22.35486303091229 hab/km².\nO município Sapucaia/RJ possui densidade demográfica de 32.351258053201896 hab/km².\nO município Saquarema/RJ possui densidade demográfica de 209.95559578018498 hab/km².\nO município Seropédica/RJ possui densidade demográfica de 275.5356639413589 hab/km².\nO município Silva Jardim/RJ possui densidade demográfica de 22.77105221054877 hab/km².\nO município Sumidouro/RJ possui densidade demográfica de 37.67192556634304 hab/km².\nO município Tanguá/RJ possui densidade demográfica de 211.2164948453608 hab/km².\nO município Teresópolis/RJ possui densidade demográfica de 212.49156501427458 hab/km².\nO município Trajano de Moraes/RJ possui densidade demográfica de 17.444600803648633 hab/km².\nO município Três Rios/RJ possui densidade demográfica de 237.41951309253696 hab/km².\nO município Valença/RJ possui densidade demográfica de 55.06012369617033 hab/km².\nO município Varre-Sai/RJ possui densidade demográfica de 49.85267810165211 hab/km².\nO município Vassouras/RJ possui densidade demográfica de 63.943656737219634 hab/km².\nO município Volta Redonda/RJ possui densidade demográfica de 1412.7740026304252 hab/km².\nO município Acari/RN possui densidade demográfica de 18.132671672938198 hab/km².\nO município Açu/RN possui densidade demográfica de 40.83578837537593 hab/km².\nO município Afonso Bezerra/RN possui densidade demográfica de 18.820507480301295 hab/km².\nO município Água Nova/RN possui densidade demográfica de 58.80031570639306 hab/km².\nO município Alexandria/RN possui densidade demográfica de 35.43191416804386 hab/km².\nO município Almino Afonso/RN possui densidade demográfica de 38.042799125273355 hab/km².\nO município Alto do Rodrigues/RN possui densidade demográfica de 64.3129671248628 hab/km².\nO município Angicos/RN possui densidade demográfica de 15.57350521858734 hab/km².\nO município Antônio Martins/RN possui densidade demográfica de 28.23563077426212 hab/km².\nO município Apodi/RN possui densidade demográfica de 21.693250461784235 hab/km².\nO município Areia Branca/RN possui densidade demográfica de 70.78544864804407 hab/km².\nO município Arês/RN possui densidade demográfica de 111.88641676045364 hab/km².\nO município Augusto Severo/RN possui densidade demográfica de 10.356207146440715 hab/km².\nO município Baía Formosa/RN possui densidade demográfica de 34.89782626394204 hab/km².\nO município Baraúna/RN possui densidade demográfica de 29.28737525433582 hab/km².\nO município Barcelona/RN possui densidade demográfica de 25.87957806460067 hab/km².\nO município Bento Fernandes/RN possui densidade demográfica de 16.982761484040257 hab/km².\nO município Bodó/RN possui densidade demográfica de 9.565320290312401 hab/km².\nO município Bom Jesus/RN possui densidade demográfica de 77.35168797115699 hab/km².\nO município Brejinho/RN possui densidade demográfica de 188.06042884990254 hab/km².\nO município Caiçara do Norte/RN possui densidade demográfica de 31.73832761804273 hab/km².\nO município Caiçara do Rio do Vento/RN possui densidade demográfica de 12.665109690263792 hab/km².\nO município Caicó/RN possui densidade demográfica de 51.041853196373054 hab/km².\nO município Campo Redondo/RN possui densidade demográfica de 48.0325644504749 hab/km².\nO município Canguaretama/RN possui densidade demográfica de 125.9769365551526 hab/km².\nO município Caraúbas/RN possui densidade demográfica de 17.877462306280307 hab/km².\nO município Carnaúba dos Dantas/RN possui densidade demográfica de 30.242214532871973 hab/km².\nO município Carnaubais/RN possui densidade demográfica de 17.993475015206535 hab/km².\nO município Ceará-Mirim/RN possui densidade demográfica de 94.06803059167841 hab/km².\nO município Cerro Corá/RN possui densidade demográfica de 27.735853850649185 hab/km².\nO município Coronel Ezequiel/RN possui densidade demográfica de 29.098250336473754 hab/km².\nO município Coronel João Pessoa/RN possui densidade demográfica de 40.73757896534062 hab/km².\nO município Cruzeta/RN possui densidade demográfica de 26.931007673325897 hab/km².\nO município Currais Novos/RN possui densidade demográfica de 49.345751142477006 hab/km².\nO município Doutor Severiano/RN possui densidade demográfica de 59.95567048393055 hab/km².\nO município Encanto/RN possui densidade demográfica de 41.59840954274354 hab/km².\nO município Equador/RN possui densidade demográfica de 21.970640401524584 hab/km².\nO município Espírito Santo/RN possui densidade demográfica de 77.11277974087162 hab/km².\nO município Extremoz/RN possui densidade demográfica de 176.02091990256483 hab/km².\nO município Felipe Guerra/RN possui densidade demográfica de 21.348523772292342 hab/km².\nO município Fernando Pedroza/RN possui densidade demográfica de 8.846046554877104 hab/km².\nO município Florânia/RN possui densidade demográfica de 17.747622820919176 hab/km².\nO município Francisco Dantas/RN possui densidade demográfica de 15.829477858559153 hab/km².\nO município Frutuoso Gomes/RN possui densidade demográfica de 66.89317319848293 hab/km².\nO município Galinhos/RN possui densidade demográfica de 6.308807200046753 hab/km².\nO município Goianinha/RN possui densidade demográfica de 116.9180361972124 hab/km².\nO município Governador Dix-Sept Rosado/RN possui densidade demográfica de 10.956453983601621 hab/km².\nO município Grossos/RN possui densidade demográfica de 74.27645105171597 hab/km².\nO município Guamaré/RN possui densidade demográfica de 47.89928946555453 hab/km².\nO município Ielmo Marinho/RN possui densidade demográfica de 39.00586482069032 hab/km².\nO município Ipanguaçu/RN possui densidade demográfica de 37.02338009352037 hab/km².\nO município Ipueira/RN possui densidade demográfica de 16.309383588535532 hab/km².\nO município Itajá/RN possui densidade demográfica de 34.04380709164129 hab/km².\nO município Itaú/RN possui densidade demográfica de 41.82515222130346 hab/km².\nO município Jaçanã/RN possui densidade demográfica de 145.25293255131965 hab/km².\nO município Jandaíra/RN possui densidade demográfica de 15.600412891386627 hab/km².\nO município Janduís/RN possui densidade demográfica de 17.53033781567727 hab/km².\nO município Januário Cicco/RN possui densidade demográfica de 48.133112547406654 hab/km².\nO município Japi/RN possui densidade demográfica de 29.21847716810413 hab/km².\nO município Jardim de Angicos/RN possui densidade demográfica de 10.262971419573262 hab/km².\nO município Jardim de Piranhas/RN possui densidade demográfica de 40.86164644661604 hab/km².\nO município Jardim do Seridó/RN possui densidade demográfica de 32.857724128577246 hab/km².\nO município João Câmara/RN possui densidade demográfica de 45.07524896497706 hab/km².\nO município João Dias/RN possui densidade demográfica de 29.499829874106837 hab/km².\nO município José da Penha/RN possui densidade demográfica de 49.88099285957158 hab/km².\nO município Jucurutu/RN possui densidade demográfica de 18.947661529564222 hab/km².\nO município Jundiá/RN possui densidade demográfica de 80.24193548387096 hab/km².\nO município Lagoa d`Anta/RN possui densidade demográfica de 58.939895882631326 hab/km².\nO município Lagoa de Pedras/RN possui densidade demográfica de 59.39996600373959 hab/km².\nO município Lagoa de Velhos/RN possui densidade demográfica de 23.642002658396102 hab/km².\nO município Lagoa Nova/RN possui densidade demográfica de 79.31366988088485 hab/km².\nO município Lagoa Salgada/RN possui densidade demográfica de 95.34854405647296 hab/km².\nO município Lajes/RN possui densidade demográfica de 15.342437409476515 hab/km².\nO município Lajes Pintadas/RN possui densidade demográfica de 35.41970662775516 hab/km².\nO município Lucrécia/RN possui densidade demográfica de 117.45877788554802 hab/km².\nO município Luís Gomes/RN possui densidade demográfica de 57.66922707633222 hab/km².\nO município Macaíba/RN possui densidade demográfica de 136.00446384869903 hab/km².\nO município Macau/RN possui densidade demográfica de 36.74178975686514 hab/km².\nO município Major Sales/RN possui densidade demográfica de 110.60369096027526 hab/km².\nO município Marcelino Vieira/RN possui densidade demográfica de 23.907321165138413 hab/km².\nO município Martins/RN possui densidade demográfica de 48.495220110940636 hab/km².\nO município Maxaranguape/RN possui densidade demográfica de 79.50807188547061 hab/km².\nO município Messias Targino/RN possui densidade demográfica de 30.999259807549965 hab/km².\nO município Montanhas/RN possui densidade demográfica de 138.82739326116047 hab/km².\nO município Monte Alegre/RN possui densidade demográfica de 98.0703584297364 hab/km².\nO município Monte das Gameleiras/RN possui densidade demográfica de 31.424600416956217 hab/km².\nO município Mossoró/RN possui densidade demográfica de 123.7609141964341 hab/km².\nO município Natal/RN possui densidade demográfica de 4805.327035752721 hab/km².\nO município Nísia Floresta/RN possui densidade demográfica de 77.26091476091477 hab/km².\nO município Nova Cruz/RN possui densidade demográfica de 127.8181949146438 hab/km².\nO município Olho-d`Água do Borges/RN possui densidade demográfica de 30.424311114259407 hab/km².\nO município Ouro Branco/RN possui densidade demográfica de 18.551125148045795 hab/km².\nO município Paraná/RN possui densidade demográfica de 48.556333701928985 hab/km².\nO município Paraú/RN possui densidade demográfica de 10.070196498003707 hab/km².\nO município Parazinho/RN possui densidade demográfica de 17.63934903702625 hab/km².\nO município Parelhas/RN possui densidade demográfica de 39.671773281877364 hab/km².\nO município Parnamirim/RN possui densidade demográfica de 1639.7181501579332 hab/km².\nO município Passa e Fica/RN possui densidade demográfica de 263.4076886568581 hab/km².\nO município Passagem/RN possui densidade demográfica de 70.23289665211063 hab/km².\nO município Patu/RN possui densidade demográfica de 37.48942437251277 hab/km².\nO município Pau dos Ferros/RN possui densidade demográfica de 106.7279581474073 hab/km².\nO município Pedra Grande/RN possui densidade demográfica de 15.901905880227623 hab/km².\nO município Pedra Preta/RN possui densidade demográfica de 8.779958642665854 hab/km².\nO município Pedro Avelino/RN possui densidade demográfica de 7.526554431336328 hab/km².\nO município Pedro Velho/RN possui densidade demográfica de 73.23958279279746 hab/km².\nO município Pendências/RN possui densidade demográfica de 32.046571551271654 hab/km².\nO município Pilões/RN possui densidade demográfica de 41.75837465231588 hab/km².\nO município Poço Branco/RN possui densidade demográfica de 60.54253472222222 hab/km².\nO município Portalegre/RN possui densidade demográfica de 66.51522035438437 hab/km².\nO município Porto do Mangue/RN possui densidade demográfica de 16.355770135122423 hab/km².\nO município Presidente Juscelino/RN possui densidade demográfica de 52.393187929489095 hab/km².\nO município Pureza/RN possui densidade demográfica de 16.70434265318263 hab/km².\nO município Rafael Fernandes/RN possui densidade demográfica de 59.97699092419788 hab/km².\nO município Rafael Godeiro/RN possui densidade demográfica de 30.608573998201262 hab/km².\nO município Riacho da Cruz/RN possui densidade demográfica de 24.878163810721585 hab/km².\nO município Riacho de Santana/RN possui densidade demográfica de 32.44087112637577 hab/km².\nO município Riachuelo/RN possui densidade demográfica de 26.88196584122637 hab/km².\nO município Rio do Fogo/RN possui densidade demográfica de 66.94396379608679 hab/km².\nO município Rodolfo Fernandes/RN possui densidade demográfica de 28.532678894342546 hab/km².\nO município Ruy Barbosa/RN possui densidade demográfica de 28.57483506875447 hab/km².\nO município Santa Cruz/RN possui densidade demográfica de 57.33390992376193 hab/km².\nO município Santa Maria/RN possui densidade demográfica de 21.687844423190782 hab/km².\nO município Santana do Matos/RN possui densidade demográfica de 9.72779914620229 hab/km².\nO município Santana do Seridó/RN possui densidade demográfica de 13.40764331210191 hab/km².\nO município Santo Antônio/RN possui densidade demográfica de 73.78769762189452 hab/km².\nO município São Bento do Norte/RN possui densidade demográfica de 10.30374398226717 hab/km².\nO município São Bento do Trairí/RN possui densidade demográfica de 20.464311916989836 hab/km².\nO município São Fernando/RN possui densidade demográfica de 8.409366268575527 hab/km².\nO município São Francisco do Oeste/RN possui densidade demográfica de 51.25016536578912 hab/km².\nO município São Gonçalo do Amarante/RN possui densidade demográfica de 351.91072575465637 hab/km².\nO município São João do Sabugi/RN possui densidade demográfica de 21.378289592433486 hab/km².\nO município São José de Mipibu/RN possui densidade demográfica de 137.00272104157338 hab/km².\nO município São José do Campestre/RN possui densidade demográfica de 36.2218574108818 hab/km².\nO município São José do Seridó/RN possui densidade demográfica de 24.245028938169735 hab/km².\nO município São Miguel/RN possui densidade demográfica de 129.05236181489894 hab/km².\nO município São Miguel do Gostoso/RN possui densidade demográfica de 25.221818181818183 hab/km².\nO município São Paulo do Potengi/RN possui densidade demográfica de 65.89443912989228 hab/km².\nO município São Pedro/RN possui densidade demográfica de 31.935054292153247 hab/km².\nO município São Rafael/RN possui densidade demográfica de 17.29055638456619 hab/km².\nO município São Tomé/RN possui densidade demográfica de 12.551733732132298 hab/km².\nO município São Vicente/RN possui densidade demográfica de 30.472146395713274 hab/km².\nO município Senador Elói de Souza/RN possui densidade demográfica de 33.63164488992303 hab/km².\nO município Senador Georgino Avelino/RN possui densidade demográfica de 151.33050520632472 hab/km².\nO município Serra de São Bento/RN possui densidade demográfica de 59.4328883369554 hab/km².\nO município Serra do Mel/RN possui densidade demográfica de 16.685860732186015 hab/km².\nO município Serra Negra do Norte/RN possui densidade demográfica de 13.81578947368421 hab/km².\nO município Serrinha/RN possui densidade demográfica de 34.036720972329974 hab/km².\nO município Serrinha dos Pintos/RN possui densidade demográfica de 37.01589889930697 hab/km².\nO município Severiano Melo/RN possui densidade demográfica de 36.439657903072536 hab/km².\nO município Sítio Novo/RN possui densidade demográfica de 23.517286611074674 hab/km².\nO município Taboleiro Grande/RN possui densidade demográfica de 18.67193166250302 hab/km².\nO município Taipu/RN possui densidade demográfica de 33.546851085539366 hab/km².\nO município Tangará/RN possui densidade demográfica de 39.7247989238573 hab/km².\nO município Tenente Ananias/RN possui densidade demográfica de 44.18563061653329 hab/km².\nO município Tenente Laurentino Cruz/RN possui densidade demográfica de 72.68082817961817 hab/km².\nO município Tibau/RN possui densidade demográfica de 21.785629874734106 hab/km².\nO município Tibau do Sul/RN possui densidade demográfica de 111.81496758986448 hab/km².\nO município Timbaúba dos Batistas/RN possui densidade demográfica de 16.943521594684388 hab/km².\nO município Touros/RN possui densidade demográfica de 37.06940751427856 hab/km².\nO município Triunfo Potiguar/RN possui densidade demográfica de 12.533025713541472 hab/km².\nO município Umarizal/RN possui densidade demográfica de 49.90635827324655 hab/km².\nO município Upanema/RN possui densidade demográfica de 14.866179213438148 hab/km².\nO município Várzea/RN possui densidade demográfica de 72.04182718767198 hab/km².\nO município Venha-Ver/RN possui densidade demográfica de 53.35101926836079 hab/km².\nO município Vera Cruz/RN possui densidade demográfica de 127.77446656335678 hab/km².\nO município Viçosa/RN possui densidade demográfica de 42.680031653917176 hab/km².\nO município Vila Flor/RN possui densidade demográfica de 60.260176248426355 hab/km².\nO município Aceguá/RS possui densidade demográfica de 2.835973098916986 hab/km².\nO município Água Santa/RS possui densidade demográfica de 12.755748997566743 hab/km².\nO município Agudo/RS possui densidade demográfica de 31.191359982093225 hab/km².\nO município Ajuricaba/RS possui densidade demográfica de 22.44462319019923 hab/km².\nO município Alecrim/RS possui densidade demográfica de 22.383554680053376 hab/km².\nO município Alegrete/RS possui densidade demográfica de 9.950473798525106 hab/km².\nO município Alegria/RS possui densidade demográfica de 24.9059007470033 hab/km².\nO município Almirante Tamandaré do Sul/RS possui densidade demográfica de 7.789124618457248 hab/km².\nO município Alpestre/RS possui densidade demográfica de 24.72585017249877 hab/km².\nO município Alto Alegre/RS possui densidade demográfica de 16.146788990825687 hab/km².\nO município Alto Feliz/RS possui densidade demográfica de 36.844764430971324 hab/km².\nO município Alvorada/RS possui densidade demográfica de 2743.977001823026 hab/km².\nO município Amaral Ferrador/RS possui densidade demográfica de 12.543932393476286 hab/km².\nO município Ametista do Sul/RS possui densidade demográfica de 78.32923307305595 hab/km².\nO município André da Rocha/RS possui densidade demográfica de 3.7492677211482133 hab/km².\nO município Anta Gorda/RS possui densidade demográfica de 24.995884096147513 hab/km².\nO município Antônio Prado/RS possui densidade demográfica de 36.916748173292675 hab/km².\nO município Arambaré/RS possui densidade demográfica de 7.113962089690245 hab/km².\nO município Araricá/RS possui densidade demográfica de 137.82941343156702 hab/km².\nO município Aratiba/RS possui densidade demográfica de 19.16788321167883 hab/km².\nO município Arroio do Meio/RS possui densidade demográfica de 118.90985059508736 hab/km².\nO município Arroio do Padre/RS possui densidade demográfica de 21.95945945945946 hab/km².\nO município Arroio do Sal/RS possui densidade demográfica de 64.01455628153172 hab/km².\nO município Arroio do Tigre/RS possui densidade demográfica de 39.74483863872042 hab/km².\nO município Arroio dos Ratos/RS possui densidade demográfica de 31.944216185758222 hab/km².\nO município Arroio Grande/RS possui densidade demográfica de 7.348026734563972 hab/km².\nO município Arvorezinha/RS possui densidade demográfica de 37.64173170372552 hab/km².\nO município Augusto Pestana/RS possui densidade demográfica de 20.423670274004145 hab/km².\nO município Áurea/RS possui densidade demográfica de 23.15370522458778 hab/km².\nO município Bagé/RS possui densidade demográfica de 28.517432420223997 hab/km².\nO município Balneário Pinhal/RS possui densidade demográfica de 104.6260601387818 hab/km².\nO município Barão/RS possui densidade demográfica de 46.116153907944415 hab/km².\nO município Barão de Cotegipe/RS possui densidade demográfica de 25.098988967054936 hab/km².\nO município Barão do Triunfo/RS possui densidade demográfica de 16.081576535288725 hab/km².\nO município Barra do Guarita/RS possui densidade demográfica de 47.98073936004971 hab/km².\nO município Barra do Quaraí/RS possui densidade demográfica de 3.7987388035677085 hab/km².\nO município Barra do Ribeiro/RS possui densidade demográfica de 17.246724741065915 hab/km².\nO município Barra do Rio Azul/RS possui densidade demográfica de 13.612885687100722 hab/km².\nO município Barra Funda/RS possui densidade demográfica de 39.430284857571216 hab/km².\nO município Barracão/RS possui densidade demográfica de 10.36711628897103 hab/km².\nO município Barros Cassal/RS possui densidade demográfica de 17.156726768377254 hab/km².\nO município Benjamin Constant do Sul/RS possui densidade demográfica de 17.424471299093653 hab/km².\nO município Bento Gonçalves/RS possui densidade demográfica de 280.8618703529165 hab/km².\nO município Boa Vista das Missões/RS possui densidade demográfica de 10.851041987475618 hab/km².\nO município Boa Vista do Buricá/RS possui densidade demográfica de 60.461694104662925 hab/km².\nO município Boa Vista do Cadeado/RS possui densidade demográfica de 3.4816716588218513 hab/km².\nO município Boa Vista do Incra/RS possui densidade demográfica de 4.816572983494548 hab/km².\nO município Boa Vista do Sul/RS possui densidade demográfica de 29.4223635400106 hab/km².\nO município Bom Jesus/RS possui densidade demográfica de 4.388742203781809 hab/km².\nO município Bom Princípio/RS possui densidade demográfica de 133.2090395480226 hab/km².\nO município Bom Progresso/RS possui densidade demográfica de 26.233941852603113 hab/km².\nO município Bom Retiro do Sul/RS possui densidade demográfica de 112.10788625036646 hab/km².\nO município Boqueirão do Leão/RS possui densidade demográfica de 28.907809968729985 hab/km².\nO município Bossoroca/RS possui densidade demográfica de 4.274263149071447 hab/km².\nO município Bozano/RS possui densidade demográfica de 10.943095901313171 hab/km².\nO município Braga/RS possui densidade demográfica de 28.699899216993565 hab/km².\nO município Brochier/RS possui densidade demográfica de 43.80211749273869 hab/km².\nO município Butiá/RS possui densidade demográfica de 27.126620139581256 hab/km².\nO município Caçapava do Sul/RS possui densidade demográfica de 11.05637801063959 hab/km².\nO município Cacequi/RS possui densidade demográfica de 5.770585877339185 hab/km².\nO município Cachoeira do Sul/RS possui densidade demográfica de 22.4426798316538 hab/km².\nO município Cachoeirinha/RS possui densidade demográfica de 2686.9150386188094 hab/km².\nO município Cacique Doble/RS possui densidade demográfica de 23.873277426315532 hab/km².\nO município Caibaté/RS possui densidade demográfica de 19.07879534776246 hab/km².\nO município Caiçara/RS possui densidade demográfica de 26.80232558139535 hab/km².\nO município Camaquã/RS possui densidade demográfica de 37.37220366433849 hab/km².\nO município Camargo/RS possui densidade demográfica de 18.773086115738394 hab/km².\nO município Cambará do Sul/RS possui densidade demográfica de 5.412650477805816 hab/km².\nO município Campestre da Serra/RS possui densidade demográfica de 6.0353159851301115 hab/km².\nO município Campina das Missões/RS possui densidade demográfica de 27.095145287030476 hab/km².\nO município Campinas do Sul/RS possui densidade demográfica de 19.937717265353417 hab/km².\nO município Campo Bom/RS possui densidade demográfica de 992.7945794083623 hab/km².\nO município Campo Novo/RS possui densidade demográfica de 24.58233890214797 hab/km².\nO município Campos Borges/RS possui densidade demográfica de 15.420601994880395 hab/km².\nO município Candelária/RS possui densidade demográfica de 31.962498013665975 hab/km².\nO município Cândido Godói/RS possui densidade demográfica de 26.534838395322396 hab/km².\nO município Candiota/RS possui densidade demográfica de 9.392501847231294 hab/km².\nO município Canela/RS possui densidade demográfica de 154.58486030657681 hab/km².\nO município Canguçu/RS possui densidade demográfica de 15.107693267787898 hab/km².\nO município Canoas/RS possui densidade demográfica de 2470.0762776506485 hab/km².\nO município Canudos do Vale/RS possui densidade demográfica de 22.06079843730924 hab/km².\nO município Capão Bonito do Sul/RS possui densidade demográfica de 3.32751555623008 hab/km².\nO município Capão da Canoa/RS possui densidade demográfica de 432.95571575695163 hab/km².\nO município Capão do Cipó/RS possui densidade demográfica de 3.077380657314232 hab/km².\nO município Capão do Leão/RS possui densidade demográfica de 30.93828386620319 hab/km².\nO município Capela de Santana/RS possui densidade demográfica de 63.1911188506748 hab/km².\nO município Capitão/RS possui densidade demográfica de 35.63606867649047 hab/km².\nO município Capivari do Sul/RS possui densidade demográfica de 9.423677899173914 hab/km².\nO município Caraá/RS possui densidade demográfica de 24.8437075292199 hab/km².\nO município Carazinho/RS possui densidade demográfica de 89.18642589724699 hab/km².\nO município Carlos Barbosa/RS possui densidade demográfica de 110.16749026982114 hab/km².\nO município Carlos Gomes/RS possui densidade demográfica de 19.324194324194323 hab/km².\nO município Casca/RS possui densidade demográfica de 31.83440662373505 hab/km².\nO município Caseiros/RS possui densidade demográfica de 12.757201646090534 hab/km².\nO município Catuípe/RS possui densidade demográfica de 15.984295168535473 hab/km².\nO município Caxias do Sul/RS possui densidade demográfica de 264.8932676518883 hab/km².\nO município Centenário/RS possui densidade demográfica de 22.072508002679964 hab/km².\nO município Cerrito/RS possui densidade demográfica de 14.17312375470445 hab/km².\nO município Cerro Branco/RS possui densidade demográfica de 28.053158657177047 hab/km².\nO município Cerro Grande/RS possui densidade demográfica de 32.91122004357299 hab/km².\nO município Cerro Grande do Sul/RS possui densidade demográfica de 31.614273838480248 hab/km².\nO município Cerro Largo/RS possui densidade demográfica de 74.79176046825754 hab/km².\nO município Chapada/RS possui densidade demográfica de 13.708262674697387 hab/km².\nO município Charqueadas/RS possui densidade demográfica de 163.13334257078196 hab/km².\nO município Charrua/RS possui densidade demográfica de 17.519685039370078 hab/km².\nO município Chiapetta/RS possui densidade demográfica de 10.197957382423402 hab/km².\nO município Chuí/RS possui densidade demográfica de 29.21254011355221 hab/km².\nO município Chuvisca/RS possui densidade demográfica de 22.424819703361003 hab/km².\nO município Cidreira/RS possui densidade demográfica de 51.51897189800317 hab/km².\nO município Ciríaco/RS possui densidade demográfica de 17.972030525431773 hab/km².\nO município Colinas/RS possui densidade demográfica de 41.45965393181429 hab/km².\nO município Colorado/RS possui densidade demográfica de 12.44478721166655 hab/km².\nO município Condor/RS possui densidade demográfica de 14.084567596036027 hab/km².\nO município Constantina/RS possui densidade demográfica de 48.039408866995075 hab/km².\nO município Coqueiro Baixo/RS possui densidade demográfica de 13.608835055219094 hab/km².\nO município Coqueiros do Sul/RS possui densidade demográfica de 8.916712030484485 hab/km².\nO município Coronel Barros/RS possui densidade demográfica de 15.090518563976682 hab/km².\nO município Coronel Bicaco/RS possui densidade demográfica de 15.744127448589774 hab/km².\nO município Coronel Pilar/RS possui densidade demográfica de 16.35846372688478 hab/km².\nO município Cotiporã/RS possui densidade demográfica de 22.72305371852883 hab/km².\nO município Coxilha/RS possui densidade demográfica de 6.684169445824168 hab/km².\nO município Crissiumal/RS possui densidade demográfica de 38.889962722628745 hab/km².\nO município Cristal/RS possui densidade demográfica de 10.680281090914425 hab/km².\nO município Cristal do Sul/RS possui densidade demográfica de 28.91936144085141 hab/km².\nO município Cruz Alta/RS possui densidade demográfica de 46.17934826554541 hab/km².\nO município Cruzaltense/RS possui densidade demográfica de 12.829578139980825 hab/km².\nO município Cruzeiro do Sul/RS possui densidade demográfica de 79.20282867245258 hab/km².\nO município David Canabarro/RS possui densidade demográfica de 26.76917800388705 hab/km².\nO município Derrubadas/RS possui densidade demográfica de 8.831672203765228 hab/km².\nO município Dezesseis de Novembro/RS possui densidade demográfica de 13.21650910767812 hab/km².\nO município Dilermando de Aguiar/RS possui densidade demográfica de 5.101989842644243 hab/km².\nO município Dois Irmãos/RS possui densidade demográfica de 423.1430325352978 hab/km².\nO município Dois Irmãos das Missões/RS possui densidade demográfica de 9.55778092874867 hab/km².\nO município Dois Lajeados/RS possui densidade demográfica de 24.578240983729472 hab/km².\nO município Dom Feliciano/RS possui densidade demográfica de 10.603390430403268 hab/km².\nO município Dom Pedrito/RS possui densidade demográfica de 7.491766337320159 hab/km².\nO município Dom Pedro de Alcântara/RS possui densidade demográfica de 32.62538382804504 hab/km².\nO município Dona Francisca/RS possui densidade demográfica de 29.742020113686053 hab/km².\nO município Doutor Maurício Cardoso/RS possui densidade demográfica de 21.025762792354268 hab/km².\nO município Doutor Ricardo/RS possui densidade demográfica de 18.721755971594575 hab/km².\nO município Eldorado do Sul/RS possui densidade demográfica de 67.3748847429031 hab/km².\nO município Encantado/RS possui densidade demográfica de 147.38430583501005 hab/km².\nO município Encruzilhada do Sul/RS possui densidade demográfica de 7.327256654083241 hab/km².\nO município Engenho Velho/RS possui densidade demográfica de 21.449641803624104 hab/km².\nO município Entre-Ijuís/RS possui densidade demográfica de 16.17444806369888 hab/km².\nO município Entre Rios do Sul/RS possui densidade demográfica de 25.651703173149 hab/km².\nO município Erebango/RS possui densidade demográfica de 19.39655172413793 hab/km².\nO município Erechim/RS possui densidade demográfica de 223.11050224069473 hab/km².\nO município Ernestina/RS possui densidade demográfica de 12.912398076521011 hab/km².\nO município Erval Grande/RS possui densidade demográfica de 18.069506177160257 hab/km².\nO município Erval Seco/RS possui densidade demográfica de 21.649399543818188 hab/km².\nO município Esmeralda/RS possui densidade demográfica de 3.8179254492208687 hab/km².\nO município Esperança do Sul/RS possui densidade demográfica de 22.051489419059173 hab/km².\nO município Espumoso/RS possui densidade demográfica de 19.461861647106897 hab/km².\nO município Estação/RS possui densidade demográfica de 59.94814002194076 hab/km².\nO município Estância Velha/RS possui densidade demográfica de 816.3758389261745 hab/km².\nO município Esteio/RS possui densidade demográfica de 2917.449421965318 hab/km².\nO município Estrela/RS possui densidade demográfica de 166.2449777391682 hab/km².\nO município Estrela Velha/RS possui densidade demográfica de 12.88032094294742 hab/km².\nO município Eugênio de Castro/RS possui densidade demográfica de 6.672708194219212 hab/km².\nO município Fagundes Varela/RS possui densidade demográfica de 19.203276247207743 hab/km².\nO município Farroupilha/RS possui densidade demográfica de 176.57260190349345 hab/km².\nO município Faxinal do Soturno/RS possui densidade demográfica de 39.27015891701001 hab/km².\nO município Faxinalzinho/RS possui densidade demográfica de 17.903473287766776 hab/km².\nO município Fazenda Vilanova/RS possui densidade demográfica de 43.60183983960373 hab/km².\nO município Feliz/RS possui densidade demográfica de 129.59001782531195 hab/km².\nO município Flores da Cunha/RS possui densidade demográfica de 99.19912232583654 hab/km².\nO município Floriano Peixoto/RS possui densidade demográfica de 11.981238496704862 hab/km².\nO município Fontoura Xavier/RS possui densidade demográfica de 18.37112447940768 hab/km².\nO município Formigueiro/RS possui densidade demográfica de 12.051753466554407 hab/km².\nO município Forquetinha/RS possui densidade demográfica de 26.49353425243134 hab/km².\nO município Fortaleza dos Valos/RS possui densidade demográfica de 7.034889978933772 hab/km².\nO município Frederico Westphalen/RS possui densidade demográfica de 108.84972450750999 hab/km².\nO município Garibaldi/RS possui densidade demográfica de 181.33419995272985 hab/km².\nO município Garruchos/RS possui densidade demográfica de 4.043258110895793 hab/km².\nO município Gaurama/RS possui densidade demográfica de 28.698717321061395 hab/km².\nO município General Câmara/RS possui densidade demográfica de 16.562420344699124 hab/km².\nO município Gentil/RS possui densidade demográfica de 9.113635128525624 hab/km².\nO município Getúlio Vargas/RS possui densidade demográfica de 56.37017133684615 hab/km².\nO município Giruá/RS possui densidade demográfica de 19.949294326572577 hab/km².\nO município Glorinha/RS possui densidade demográfica de 21.292176492398962 hab/km².\nO município Gramado/RS possui densidade demográfica de 135.69776731278643 hab/km².\nO município Gramado dos Loureiros/RS possui densidade demográfica de 17.267884322678842 hab/km².\nO município Gramado Xavier/RS possui densidade demográfica de 18.250356272698017 hab/km².\nO município Gravataí/RS possui densidade demográfica de 551.5857605177994 hab/km².\nO município Guabiju/RS possui densidade demográfica de 10.768919738526856 hab/km².\nO município Guaíba/RS possui densidade demográfica de 252.56400053057436 hab/km².\nO município Guaporé/RS possui densidade demográfica de 76.64449371766445 hab/km².\nO município Guarani das Missões/RS possui densidade demográfica de 27.934595524956972 hab/km².\nO município Harmonia/RS possui densidade demográfica de 95.0402144772118 hab/km².\nO município Herval/RS possui densidade demográfica de 3.841646566240386 hab/km².\nO município Herveiras/RS possui densidade demográfica de 24.974636455867433 hab/km².\nO município Horizontina/RS possui densidade demográfica de 78.92291810048177 hab/km².\nO município Hulha Negra/RS possui densidade demográfica de 7.343541135010329 hab/km².\nO município Humaitá/RS possui densidade demográfica de 36.569771764181105 hab/km².\nO município Ibarama/RS possui densidade demográfica de 22.634767748951372 hab/km².\nO município Ibiaçá/RS possui densidade demográfica de 13.502666131529155 hab/km².\nO município Ibiraiaras/RS possui densidade demográfica de 23.851654748045902 hab/km².\nO município Ibirapuitã/RS possui densidade demográfica de 13.226720515910499 hab/km².\nO município Ibirubá/RS possui densidade demográfica de 31.78862457815458 hab/km².\nO município Igrejinha/RS possui densidade demográfica de 233.03400559399378 hab/km².\nO município Ijuí/RS possui densidade demográfica de 114.51395237473336 hab/km².\nO município Ilópolis/RS possui densidade demográfica de 35.21634615384615 hab/km².\nO município Imbé/RS possui densidade demográfica de 448.4771573604061 hab/km².\nO município Imigrante/RS possui densidade demográfica de 41.20774263904035 hab/km².\nO município Independência/RS possui densidade demográfica de 18.514995523724263 hab/km².\nO município Inhacorá/RS possui densidade demográfica de 19.866795197616334 hab/km².\nO município Ipê/RS possui densidade demográfica de 10.03921568627451 hab/km².\nO município Ipiranga do Sul/RS possui densidade demográfica de 12.313149227261212 hab/km².\nO município Iraí/RS possui densidade demográfica de 44.63969938107869 hab/km².\nO município Itaara/RS possui densidade demográfica de 28.961211630730098 hab/km².\nO município Itacurubi/RS possui densidade demográfica de 3.069936745563714 hab/km².\nO município Itapuca/RS possui densidade demográfica de 12.721845318860245 hab/km².\nO município Itaqui/RS possui densidade demográfica de 11.20991527714128 hab/km².\nO município Itati/RS possui densidade demográfica de 12.488521579430671 hab/km².\nO município Itatiba do Sul/RS possui densidade demográfica de 19.652280437240858 hab/km².\nO município Ivorá/RS possui densidade demográfica de 17.53843650858212 hab/km².\nO município Ivoti/RS possui densidade demográfica de 314.7110055423595 hab/km².\nO município Jaboticaba/RS possui densidade demográfica de 32.003123779773524 hab/km².\nO município Jacuizinho/RS possui densidade demográfica de 7.405328764695456 hab/km².\nO município Jacutinga/RS possui densidade demográfica de 20.262130507529278 hab/km².\nO município Jaguarão/RS possui densidade demográfica de 13.595829398650688 hab/km².\nO município Jaguari/RS possui densidade demográfica de 17.037422037422036 hab/km².\nO município Jaquirana/RS possui densidade demográfica de 4.600524263717866 hab/km².\nO município Jari/RS possui densidade demográfica de 4.174158746468019 hab/km².\nO município Jóia/RS possui densidade demográfica de 6.740945722885717 hab/km².\nO município Júlio de Castilhos/RS possui densidade demográfica de 10.147819506784563 hab/km².\nO município Lagoa Bonita do Sul/RS possui densidade demográfica de 24.534562211981566 hab/km².\nO município Lagoa dos Três Cantos/RS possui densidade demográfica de 11.526255049047895 hab/km².\nO município Lagoa Vermelha/RS possui densidade demográfica de 21.78472497032054 hab/km².\nO município Lagoão/RS possui densidade demográfica de 16.12356621480709 hab/km².\nO município Lajeado/RS possui densidade demográfica de 793.040293040293 hab/km².\nO município Lajeado do Bugre/RS possui densidade demográfica de 36.61121742970705 hab/km².\nO município Lavras do Sul/RS possui densidade demográfica de 2.952780127662847 hab/km².\nO município Liberato Salzano/RS possui densidade demográfica de 23.531327606562716 hab/km².\nO município Lindolfo Collor/RS possui densidade demográfica de 158.44195210669898 hab/km².\nO município Linha Nova/RS possui densidade demográfica de 25.482504315079243 hab/km².\nO município Maçambara/RS possui densidade demográfica de 2.815512057142178 hab/km².\nO município Machadinho/RS possui densidade demográfica de 16.44628839208429 hab/km².\nO município Mampituba/RS possui densidade demográfica de 19.01595744680851 hab/km².\nO município Manoel Viana/RS possui densidade demográfica de 5.0852088876105555 hab/km².\nO município Maquiné/RS possui densidade demográfica de 11.106821727870804 hab/km².\nO município Maratá/RS possui densidade demográfica de 31.12835673811283 hab/km².\nO município Marau/RS possui densidade demográfica de 56.00492838441399 hab/km².\nO município Marcelino Ramos/RS possui densidade demográfica de 22.345055710306408 hab/km².\nO município Mariana Pimentel/RS possui densidade demográfica de 11.15485952810918 hab/km².\nO município Mariano Moro/RS possui densidade demográfica de 22.32774297837947 hab/km².\nO município Marques de Souza/RS possui densidade demográfica de 32.49720402620227 hab/km².\nO município Mata/RS possui densidade demográfica de 16.3877132230345 hab/km².\nO município Mato Castelhano/RS possui densidade demográfica de 10.362042203297394 hab/km².\nO município Mato Leitão/RS possui densidade demográfica de 84.20479302832244 hab/km².\nO município Mato Queimado/RS possui densidade demográfica de 15.692602930914166 hab/km².\nO município Maximiliano de Almeida/RS possui densidade demográfica de 23.560736902705816 hab/km².\nO município Minas do Leão/RS possui densidade demográfica de 17.983221002026678 hab/km².\nO município Miraguaí/RS possui densidade demográfica de 37.234450494669844 hab/km².\nO município Montauri/RS possui densidade demográfica de 18.78654970760234 hab/km².\nO município Monte Alegre dos Campos/RS possui densidade demográfica de 5.642667442791137 hab/km².\nO município Monte Belo do Sul/RS possui densidade demográfica de 39.052215884159715 hab/km².\nO município Montenegro/RS possui densidade demográfica de 140.12641211292186 hab/km².\nO município Mormaço/RS possui densidade demográfica de 18.814591745944835 hab/km².\nO município Morrinhos do Sul/RS possui densidade demográfica de 19.233558994197292 hab/km².\nO município Morro Redondo/RS possui densidade demográfica de 25.45268751277335 hab/km².\nO município Morro Reuter/RS possui densidade demográfica de 64.76494751255134 hab/km².\nO município Mostardas/RS possui densidade demográfica de 6.113999566311479 hab/km².\nO município Muçum/RS possui densidade demográfica de 43.20497790603301 hab/km².\nO município Muitos Capões/RS possui densidade demográfica de 2.4943026721093884 hab/km².\nO município Muliterno/RS possui densidade demográfica de 16.314226581481147 hab/km².\nO município Não-Me-Toque/RS possui densidade demográfica de 44.06226670721929 hab/km².\nO município Nicolau Vergueiro/RS possui densidade demográfica de 11.044795276601207 hab/km².\nO município Nonoai/RS possui densidade demográfica de 25.74907764816276 hab/km².\nO município Nova Alvorada/RS possui densidade demográfica de 21.304231387252276 hab/km².\nO município Nova Araçá/RS possui densidade demográfica de 53.80580957504034 hab/km².\nO município Nova Bassano/RS possui densidade demográfica de 41.77496337602192 hab/km².\nO município Nova Boa Vista/RS possui densidade demográfica de 20.797962648556876 hab/km².\nO município Nova Bréscia/RS possui densidade demográfica de 30.96673798871815 hab/km².\nO município Nova Candelária/RS possui densidade demográfica de 28.120208524992336 hab/km².\nO município Nova Esperança do Sul/RS possui densidade demográfica de 24.455497382198953 hab/km².\nO município Nova Hartz/RS possui densidade demográfica de 293.2544757033248 hab/km².\nO município Nova Pádua/RS possui densidade demográfica de 23.731111972103836 hab/km².\nO município Nova Palma/RS possui densidade demográfica de 20.229019807980606 hab/km².\nO município Nova Petrópolis/RS possui densidade demográfica de 65.37933401991074 hab/km².\nO município Nova Prata/RS possui densidade demográfica de 88.23529411764706 hab/km².\nO município Nova Ramada/RS possui densidade demográfica de 9.565865913016172 hab/km².\nO município Nova Roma do Sul/RS possui densidade demográfica de 22.42871519624287 hab/km².\nO município Nova Santa Rita/RS possui densidade demográfica de 104.26401064855189 hab/km².\nO município Novo Barreiro/RS possui densidade demográfica de 32.189674704644766 hab/km².\nO município Novo Cabrais/RS possui densidade demográfica de 20.047844401684955 hab/km².\nO município Novo Hamburgo/RS possui densidade demográfica de 1067.5542846930568 hab/km².\nO município Novo Machado/RS possui densidade demográfica de 17.949421502720995 hab/km².\nO município Novo Tiradentes/RS possui densidade demográfica de 30.19893899204244 hab/km².\nO município Novo Xingu/RS possui densidade demográfica de 21.801712371261942 hab/km².\nO município Osório/RS possui densidade demográfica de 61.647200663099994 hab/km².\nO município Paim Filho/RS possui densidade demográfica de 23.290152596333296 hab/km².\nO município Palmares do Sul/RS possui densidade demográfica de 11.555925453798421 hab/km².\nO município Palmeira das Missões/RS possui densidade demográfica de 24.184355692073577 hab/km².\nO município Palmitinho/RS possui densidade demográfica de 48.03887539048941 hab/km².\nO município Panambi/RS possui densidade demográfica de 77.5333088864442 hab/km².\nO município Pantano Grande/RS possui densidade demográfica de 11.762538188129287 hab/km².\nO município Paraí/RS possui densidade demográfica de 56.568676299618005 hab/km².\nO município Paraíso do Sul/RS possui densidade demográfica de 21.714421027705423 hab/km².\nO município Pareci Novo/RS possui densidade demográfica de 61.156592928061315 hab/km².\nO município Parobé/RS possui densidade demográfica de 474.01748734468475 hab/km².\nO município Passa Sete/RS possui densidade demográfica de 16.9238852039141 hab/km².\nO município Passo do Sobrado/RS possui densidade demográfica de 22.67360718192448 hab/km².\nO município Passo Fundo/RS possui densidade demográfica de 235.92198309974216 hab/km².\nO município Paulo Bento/RS possui densidade demográfica de 14.801833378269073 hab/km².\nO município Paverama/RS possui densidade demográfica de 46.8055393925288 hab/km².\nO município Pedras Altas/RS possui densidade demográfica de 1.605959183080799 hab/km².\nO município Pedro Osório/RS possui densidade demográfica de 12.830368435749602 hab/km².\nO município Pejuçara/RS possui densidade demográfica de 9.591058323677094 hab/km².\nO município Pelotas/RS possui densidade demográfica de 203.88738447778994 hab/km².\nO município Picada Café/RS possui densidade demográfica de 60.85731062830299 hab/km².\nO município Pinhal/RS possui densidade demográfica de 36.8421052631579 hab/km².\nO município Pinhal da Serra/RS possui densidade demográfica de 4.863013698630137 hab/km².\nO município Pinhal Grande/RS possui densidade demográfica de 9.370611782952235 hab/km².\nO município Pinheirinho do Vale/RS possui densidade demográfica de 42.58119496259824 hab/km².\nO município Pinheiro Machado/RS possui densidade demográfica de 5.681110972812461 hab/km².\nO município Pirapó/RS possui densidade demográfica de 9.450195379447454 hab/km².\nO município Piratini/RS possui densidade demográfica de 5.605293118888942 hab/km².\nO município Planalto/RS possui densidade demográfica de 45.673118652894715 hab/km².\nO município Poço das Antas/RS possui densidade demográfica de 31.0021518598217 hab/km².\nO município Pontão/RS possui densidade demográfica de 7.626900792944573 hab/km².\nO município Ponte Preta/RS possui densidade demográfica de 17.522779613497548 hab/km².\nO município Portão/RS possui densidade demográfica de 193.38295077866036 hab/km².\nO município Porto Alegre/RS possui densidade demográfica de 2837.543287428525 hab/km².\nO município Porto Lucena/RS possui densidade demográfica de 21.645073576455534 hab/km².\nO município Porto Mauá/RS possui densidade demográfica de 24.081091322470634 hab/km².\nO município Porto Vera Cruz/RS possui densidade demográfica de 16.29564452265728 hab/km².\nO município Porto Xavier/RS possui densidade demográfica de 37.6385868596485 hab/km².\nO município Pouso Novo/RS possui densidade demográfica de 17.600675865953253 hab/km².\nO município Presidente Lucena/RS possui densidade demográfica de 50.25288286465709 hab/km².\nO município Progresso/RS possui densidade demográfica de 24.087391542249666 hab/km².\nO município Protásio Alves/RS possui densidade demográfica de 11.57273463719477 hab/km².\nO município Putinga/RS possui densidade demográfica de 20.195074372104365 hab/km².\nO município Quaraí/RS possui densidade demográfica de 7.313756699485009 hab/km².\nO município Quatro Irmãos/RS possui densidade demográfica de 6.623381469457815 hab/km².\nO município Quevedos/RS possui densidade demográfica de 4.987485276796231 hab/km².\nO município Quinze de Novembro/RS possui densidade demográfica de 16.334287247361832 hab/km².\nO município Redentora/RS possui densidade demográfica de 33.77164001585833 hab/km².\nO município Relvado/RS possui densidade demográfica de 17.45787427090084 hab/km².\nO município Restinga Seca/RS possui densidade demográfica de 16.577584854348622 hab/km².\nO município Rio dos Índios/RS possui densidade demográfica de 15.366309705932348 hab/km².\nO município Rio Grande/RS possui densidade demográfica de 72.79075260562756 hab/km².\nO município Rio Pardo/RS possui densidade demográfica de 18.331797190077 hab/km².\nO município Riozinho/RS possui densidade demográfica de 18.074803806979464 hab/km².\nO município Roca Sales/RS possui densidade demográfica de 49.29300675837607 hab/km².\nO município Rodeio Bonito/RS possui densidade demográfica de 69.0264423076923 hab/km².\nO município Rolador/RS possui densidade demográfica de 8.630215924883903 hab/km².\nO município Rolante/RS possui densidade demográfica de 65.90786091191991 hab/km².\nO município Ronda Alta/RS possui densidade demográfica de 24.374016311346402 hab/km².\nO município Rondinha/RS possui densidade demográfica de 21.878593235795567 hab/km².\nO município Roque Gonzales/RS possui densidade demográfica de 20.78068201488662 hab/km².\nO município Rosário do Sul/RS possui densidade demográfica de 9.086997814470267 hab/km².\nO município Sagrada Família/RS possui densidade demográfica de 33.1629392971246 hab/km².\nO município Saldanha Marinho/RS possui densidade demográfica de 12.946166689228825 hab/km².\nO município Salto do Jacuí/RS possui densidade demográfica de 23.412557644554806 hab/km².\nO município Salvador das Missões/RS possui densidade demográfica de 28.381539770310503 hab/km².\nO município Salvador do Sul/RS possui densidade demográfica de 67.5916649969946 hab/km².\nO município Sananduva/RS possui densidade demográfica de 30.46873451590526 hab/km².\nO município Santa Bárbara do Sul/RS possui densidade demográfica de 9.050650429006366 hab/km².\nO município Santa Cecília do Sul/RS possui densidade demográfica de 8.299899699097292 hab/km².\nO município Santa Clara do Sul/RS possui densidade demográfica de 65.75484764542936 hab/km².\nO município Santa Cruz do Sul/RS possui densidade demográfica de 161.40221704094571 hab/km².\nO município Santa Margarida do Sul/RS possui densidade demográfica de 2.462053805087407 hab/km².\nO município Santa Maria/RS possui densidade demográfica de 145.98069480795473 hab/km².\nO município Santa Maria do Herval/RS possui densidade demográfica de 43.3595988538682 hab/km².\nO município Santa Rosa/RS possui densidade demográfica de 140.03062474479378 hab/km².\nO município Santa Tereza/RS possui densidade demográfica de 23.760187871252935 hab/km².\nO município Santa Vitória do Palmar/RS possui densidade demográfica de 5.90921658546817 hab/km².\nO município Santana da Boa Vista/RS possui densidade demográfica de 5.801692218890344 hab/km².\nO município Santana do Livramento/RS possui densidade demográfica de 11.864726236808218 hab/km².\nO município Santiago/RS possui densidade demográfica de 20.335000600879354 hab/km².\nO município Santo Ângelo/RS possui densidade demográfica de 112.08670095518002 hab/km².\nO município Santo Antônio da Patrulha/RS possui densidade demográfica de 37.80207847134243 hab/km².\nO município Santo Antônio das Missões/RS possui densidade demográfica de 6.552221968939779 hab/km².\nO município Santo Antônio do Palma/RS possui densidade demográfica de 16.96407328098977 hab/km².\nO município Santo Antônio do Planalto/RS possui densidade demográfica de 9.767007471490366 hab/km².\nO município Santo Augusto/RS possui densidade demográfica de 29.839777825251012 hab/km².\nO município Santo Cristo/RS possui densidade demográfica de 39.188857695767126 hab/km².\nO município Santo Expedito do Sul/RS possui densidade demográfica de 19.57213297280102 hab/km².\nO município São Borja/RS possui densidade demográfica de 17.054938855426684 hab/km².\nO município São Domingos do Sul/RS possui densidade demográfica de 37.06143128562381 hab/km².\nO município São Francisco de Assis/RS possui densidade demográfica de 7.675656281767626 hab/km².\nO município São Francisco de Paula/RS possui densidade demográfica de 6.274709897402367 hab/km².\nO município São Gabriel/RS possui densidade demográfica de 12.027700037023621 hab/km².\nO município São Jerônimo/RS possui densidade demográfica de 23.637839338730004 hab/km².\nO município São João da Urtiga/RS possui densidade demográfica de 27.60836546325505 hab/km².\nO município São João do Polêsine/RS possui densidade demográfica de 30.93812375249501 hab/km².\nO município São Jorge/RS possui densidade demográfica de 23.498517577297754 hab/km².\nO município São José das Missões/RS possui densidade demográfica de 27.735291118588766 hab/km².\nO município São José do Herval/RS possui densidade demográfica de 21.379377243185566 hab/km².\nO município São José do Hortêncio/RS possui densidade demográfica de 63.8589923568866 hab/km².\nO município São José do Inhacorá/RS possui densidade demográfica de 28.27400077110911 hab/km².\nO município São José do Norte/RS possui densidade demográfica de 22.809229943654415 hab/km².\nO município São José do Ouro/RS possui densidade demográfica de 20.62311437703498 hab/km².\nO município São José do Sul/RS possui densidade demográfica de 35.270201592410636 hab/km².\nO município São José dos Ausentes/RS possui densidade demográfica de 2.802504365603305 hab/km².\nO município São Leopoldo/RS possui densidade demográfica de 2083.774576601129 hab/km².\nO município São Lourenço do Sul/RS possui densidade demográfica de 21.17300958190292 hab/km².\nO município São Luiz Gonzaga/RS possui densidade demográfica de 26.670165472956285 hab/km².\nO município São Marcos/RS possui densidade demográfica de 78.45073170731708 hab/km².\nO município São Martinho/RS possui densidade demográfica de 33.63043224979611 hab/km².\nO município São Martinho da Serra/RS possui densidade demográfica de 4.780822940781122 hab/km².\nO município São Miguel das Missões/RS possui densidade demográfica de 6.034118259285761 hab/km².\nO município São Nicolau/RS possui densidade demográfica de 11.800461551141515 hab/km².\nO município São Paulo das Missões/RS possui densidade demográfica de 28.42467283040779 hab/km².\nO município São Pedro da Serra/RS possui densidade demográfica de 93.6705283978525 hab/km².\nO município São Pedro das Missões/RS possui densidade demográfica de 23.583843941478055 hab/km².\nO município São Pedro do Butiá/RS possui densidade demográfica de 26.693301124221872 hab/km².\nO município São Pedro do Sul/RS possui densidade demográfica de 18.736478210602225 hab/km².\nO município São Sebastião do Caí/RS possui densidade demográfica de 196.805455850682 hab/km².\nO município São Sepé/RS possui densidade demográfica de 10.813881100927436 hab/km².\nO município São Valentim/RS possui densidade demográfica de 23.55535378429211 hab/km².\nO município São Valentim do Sul/RS possui densidade demográfica de 23.503902862098872 hab/km².\nO município São Valério do Sul/RS possui densidade demográfica de 24.516069278503288 hab/km².\nO município São Vendelino/RS possui densidade demográfica de 60.579619819258326 hab/km².\nO município São Vicente do Sul/RS possui densidade demográfica de 7.181572968695489 hab/km².\nO município Sapiranga/RS possui densidade demográfica de 542.1516882365701 hab/km².\nO município Sapucaia do Sul/RS possui densidade demográfica de 2245.8754930543646 hab/km².\nO município Sarandi/RS possui densidade demográfica de 60.23090636407369 hab/km².\nO município Seberi/RS possui densidade demográfica de 36.152212859133435 hab/km².\nO município Sede Nova/RS possui densidade demográfica de 25.23889354568315 hab/km².\nO município Segredo/RS possui densidade demográfica de 28.928225024248302 hab/km².\nO município Selbach/RS possui densidade demográfica de 27.747129024994372 hab/km².\nO município Senador Salgado Filho/RS possui densidade demográfica de 19.11554921540656 hab/km².\nO município Sentinela do Sul/RS possui densidade demográfica de 18.435239040998724 hab/km².\nO município Serafina Corrêa/RS possui densidade demográfica de 87.29176874081332 hab/km².\nO município Sério/RS possui densidade demográfica de 22.894710428585768 hab/km².\nO município Sertão/RS possui densidade demográfica de 14.321796709673015 hab/km².\nO município Sertão Santana/RS possui densidade demográfica de 23.228111971411554 hab/km².\nO município Sete de Setembro/RS possui densidade demográfica de 16.339718439879988 hab/km².\nO município Severiano de Almeida/RS possui densidade demográfica de 22.923627684964202 hab/km².\nO município Silveira Martins/RS possui densidade demográfica de 20.680628272251308 hab/km².\nO município Sinimbu/RS possui densidade demográfica de 19.73653258056928 hab/km².\nO município Sobradinho/RS possui densidade demográfica de 109.54060894240357 hab/km².\nO município Soledade/RS possui densidade demográfica de 24.759973957689486 hab/km².\nO município Tabaí/RS possui densidade demográfica de 43.598944591029024 hab/km².\nO município Tapejara/RS possui densidade demográfica de 80.61139028475712 hab/km².\nO município Tapera/RS possui densidade demográfica de 58.15429143938551 hab/km².\nO município Tapes/RS possui densidade demográfica de 20.623837281408907 hab/km².\nO município Taquara/RS possui densidade demográfica de 119.34434106495435 hab/km².\nO município Taquari/RS possui densidade demográfica de 74.55496185387318 hab/km².\nO município Taquaruçu do Sul/RS possui densidade demográfica de 38.5946649316851 hab/km².\nO município Tavares/RS possui densidade demográfica de 8.855606123293338 hab/km².\nO município Tenente Portela/RS possui densidade demográfica de 40.57915286322764 hab/km².\nO município Terra de Areia/RS possui densidade demográfica de 69.67623615715596 hab/km².\nO município Teutônia/RS possui densidade demográfica de 152.6816705856007 hab/km².\nO município Tio Hugo/RS possui densidade demográfica de 23.844537815126053 hab/km².\nO município Tiradentes do Sul/RS possui densidade demográfica de 27.554588877516206 hab/km².\nO município Toropi/RS possui densidade demográfica de 14.543304759089565 hab/km².\nO município Torres/RS possui densidade demográfica de 215.83110170019307 hab/km².\nO município Tramandaí/RS possui densidade demográfica de 287.96482238072156 hab/km².\nO município Travesseiro/RS possui densidade demográfica de 28.525641025641026 hab/km².\nO município Três Arroios/RS possui densidade demográfica de 19.215237582447166 hab/km².\nO município Três Cachoeiras/RS possui densidade demográfica de 40.69545128654505 hab/km².\nO município Três Coroas/RS possui densidade demográfica de 128.5329309043872 hab/km².\nO município Três de Maio/RS possui densidade demográfica de 56.196115585030796 hab/km².\nO município Três Forquilhas/RS possui densidade demográfica de 13.41250115069502 hab/km².\nO município Três Palmeiras/RS possui densidade demográfica de 24.258028792912516 hab/km².\nO município Três Passos/RS possui densidade demográfica de 89.28837555886737 hab/km².\nO município Trindade do Sul/RS possui densidade demográfica de 21.55949631175024 hab/km².\nO município Triunfo/RS possui densidade demográfica de 31.500977039570106 hab/km².\nO município Tucunduva/RS possui densidade demográfica de 32.61987721918035 hab/km².\nO município Tunas/RS possui densidade demográfica de 20.154078965469804 hab/km².\nO município Tupanci do Sul/RS possui densidade demográfica de 11.641503848431023 hab/km².\nO município Tupanciretã/RS possui densidade demográfica de 9.894487223894913 hab/km².\nO município Tupandi/RS possui densidade demográfica de 65.90527376553578 hab/km².\nO município Tuparendi/RS possui densidade demográfica de 27.81136245449818 hab/km².\nO município Turuçu/RS possui densidade demográfica de 13.88582242548494 hab/km².\nO município Ubiretama/RS possui densidade demográfica de 18.12297734627832 hab/km².\nO município União da Serra/RS possui densidade demográfica de 11.352011603939232 hab/km².\nO município Unistalda/RS possui densidade demográfica de 4.067132588522386 hab/km².\nO município Uruguaiana/RS possui densidade demográfica de 21.945463070527804 hab/km².\nO município Vacaria/RS possui densidade demográfica de 28.872530100066836 hab/km².\nO município Vale do Sol/RS possui densidade demográfica de 33.747676933857356 hab/km².\nO município Vale Real/RS possui densidade demográfica de 113.50632069194943 hab/km².\nO município Vale Verde/RS possui densidade demográfica de 9.865647651108482 hab/km².\nO município Vanini/RS possui densidade demográfica de 30.584245413904732 hab/km².\nO município Venâncio Aires/RS possui densidade demográfica de 85.2852930526098 hab/km².\nO município Vera Cruz/RS possui densidade demográfica de 77.45946644273626 hab/km².\nO município Veranópolis/RS possui densidade demográfica de 78.8345890647681 hab/km².\nO município Vespasiano Correa/RS possui densidade demográfica de 17.332513829133376 hab/km².\nO município Viadutos/RS possui densidade demográfica de 19.790579818154715 hab/km².\nO município Viamão/RS possui densidade demográfica de 159.90701527033707 hab/km².\nO município Vicente Dutra/RS possui densidade demográfica de 27.374909354604785 hab/km².\nO município Victor Graeff/RS possui densidade demográfica de 12.741847483946783 hab/km².\nO município Vila Flores/RS possui densidade demográfica de 29.71921045315541 hab/km².\nO município Vila Lângaro/RS possui densidade demográfica de 14.142077939147008 hab/km².\nO município Vila Maria/RS possui densidade demográfica de 23.26388888888889 hab/km².\nO município Vila Nova do Sul/RS possui densidade demográfica de 8.310036618498248 hab/km².\nO município Vista Alegre/RS possui densidade demográfica de 36.56080557707204 hab/km².\nO município Vista Alegre do Prata/RS possui densidade demográfica de 13.148411966814715 hab/km².\nO município Vista Gaúcha/RS possui densidade demográfica de 31.097835888187557 hab/km².\nO município Vitória das Missões/RS possui densidade demográfica de 13.423982127036709 hab/km².\nO município Westfália/RS possui densidade demográfica de 43.640625 hab/km².\nO município Xangri-lá/RS possui densidade demográfica de 204.87724501565333 hab/km².\nO município Alta Floresta d`Oeste/RO possui densidade demográfica de 3.451520652947561 hab/km².\nO município Alto Alegre dos Parecis/RO possui densidade demográfica de 3.2377781202393976 hab/km².\nO município Alto Paraíso/RO possui densidade demográfica de 6.4615999577648555 hab/km².\nO município Alvorada d`Oeste/RO possui densidade demográfica de 5.563533485849352 hab/km².\nO município Ariquemes/RO possui densidade demográfica de 20.411515010493453 hab/km².\nO município Buritis/RO possui densidade demográfica de 9.915763623725814 hab/km².\nO município Cabixi/RO possui densidade demográfica de 4.803098085760371 hab/km².\nO município Cacaulândia/RO possui densidade demográfica de 2.9238752561449295 hab/km².\nO município Cacoal/RO possui densidade demográfica de 20.716620966040917 hab/km².\nO município Campo Novo de Rondônia/RO possui densidade demográfica de 3.6795360850200898 hab/km².\nO município Candeias do Jamari/RO possui densidade demográfica de 2.8900315172555877 hab/km².\nO município Castanheiras/RO possui densidade demográfica de 4.004076878276063 hab/km².\nO município Cerejeiras/RO possui densidade demográfica de 6.118276865591204 hab/km².\nO município Chupinguaia/RO possui densidade demográfica de 1.6191639098682977 hab/km².\nO município Colorado do Oeste/RO possui densidade demográfica de 12.812013286838587 hab/km².\nO município Corumbiara/RO possui densidade demográfica de 2.8699613112354263 hab/km².\nO município Costa Marques/RO possui densidade demográfica de 2.742632108726775 hab/km².\nO município Cujubim/RO possui densidade demográfica de 4.1030657825949675 hab/km².\nO município Espigão d`Oeste/RO possui densidade demográfica de 6.358744851185141 hab/km².\nO município Governador Jorge Teixeira/RO possui densidade demográfica de 2.074444782116202 hab/km².\nO município Guajará-Mirim/RO possui densidade demográfica de 1.675912023469849 hab/km².\nO município Itapuã do Oeste/RO possui densidade demográfica de 2.0986970731922443 hab/km².\nO município Jaru/RO possui densidade demográfica de 17.66396184951072 hab/km².\nO município Ji-Paraná/RO possui densidade demográfica de 16.907988411916357 hab/km².\nO município Machadinho d`Oeste/RO possui densidade demográfica de 3.658933568056635 hab/km².\nO município Ministro Andreazza/RO possui densidade demográfica de 12.97113071371291 hab/km².\nO município Mirante da Serra/RO possui densidade demográfica de 9.965768365942878 hab/km².\nO município Monte Negro/RO possui densidade demográfica de 7.295819569427041 hab/km².\nO município Nova Brasilândia d`Oeste/RO possui densidade demográfica de 11.6699256023159 hab/km².\nO município Nova Mamoré/RO possui densidade demográfica de 2.2385629351327094 hab/km².\nO município Nova União/RO possui densidade demográfica de 9.283510710790084 hab/km².\nO município Novo Horizonte do Oeste/RO possui densidade demográfica de 12.140612958681604 hab/km².\nO município Ouro Preto do Oeste/RO possui densidade demográfica de 19.25425793842171 hab/km².\nO município Parecis/RO possui densidade demográfica de 1.887251439961078 hab/km².\nO município Pimenta Bueno/RO possui densidade demográfica de 5.419384610947406 hab/km².\nO município Pimenteiras do Oeste/RO possui densidade demográfica de 0.3848884322322033 hab/km².\nO município Porto Velho/RO possui densidade demográfica de 12.56810471724426 hab/km².\nO município Presidente Médici/RO possui densidade demográfica de 12.6922836329309 hab/km².\nO município Primavera de Rondônia/RO possui densidade demográfica de 5.818157803496838 hab/km².\nO município Rio Crespo/RO possui densidade demográfica de 1.930555878996763 hab/km².\nO município Rolim de Moura/RO possui densidade demográfica de 34.74061829081755 hab/km².\nO município Santa Luzia d`Oeste/RO possui densidade demográfica de 7.418600768074804 hab/km².\nO município São Felipe d`Oeste/RO possui densidade demográfica de 11.110495707560233 hab/km².\nO município São Francisco do Guaporé/RO possui densidade demográfica de 1.4630781485377886 hab/km².\nO município São Miguel do Guaporé/RO possui densidade demográfica de 2.925919074772594 hab/km².\nO município Seringueiras/RO possui densidade demográfica de 3.081746172661527 hab/km².\nO município Teixeirópolis/RO possui densidade demográfica de 10.626548980390451 hab/km².\nO município Theobroma/RO possui densidade demográfica de 4.8461597972158135 hab/km².\nO município Urupá/RO possui densidade demográfica de 15.596374389921381 hab/km².\nO município Vale do Anari/RO possui densidade demográfica de 2.993167769222427 hab/km².\nO município Vale do Paraíso/RO possui densidade demográfica de 8.501781128324083 hab/km².\nO município Vilhena/RO possui densidade demográfica de 6.615365649964319 hab/km².\nO município Alto Alegre/RR possui densidade demográfica de 0.6433287884157012 hab/km².\nO município Amajari/RR possui densidade demográfica de 0.32758119900970517 hab/km².\nO município Boa Vista/RR possui densidade demográfica de 49.99314230249831 hab/km².\nO município Bonfim/RR possui densidade demográfica de 1.3517519782790763 hab/km².\nO município Cantá/RR possui densidade demográfica de 1.813738856569552 hab/km².\nO município Caracaraí/RR possui densidade demográfica de 0.38805315978159516 hab/km².\nO município Caroebe/RR possui densidade demográfica de 0.6724820255682407 hab/km².\nO município Iracema/RR possui densidade demográfica de 0.6034874021310822 hab/km².\nO município Mucajaí/RR possui densidade demográfica de 1.187043633804422 hab/km².\nO município Normandia/RR possui densidade demográfica de 1.2832271871918424 hab/km².\nO município Pacaraima/RR possui densidade demográfica de 1.2994987843277932 hab/km².\nO município Rorainópolis/RR possui densidade demográfica de 0.7227172668969654 hab/km².\nO município São João da Baliza/RR possui densidade demográfica de 1.5798772788486897 hab/km².\nO município São Luiz/RR possui densidade demográfica de 4.420750676211122 hab/km².\nO município Uiramutã/RR possui densidade demográfica de 1.038365593957518 hab/km².\nO município Abdon Batista/SC possui densidade demográfica de 11.249628970020778 hab/km².\nO município Abelardo Luz/SC possui densidade demográfica de 17.942207206261937 hab/km².\nO município Agrolândia/SC possui densidade demográfica de 44.91929655504698 hab/km².\nO município Agronômica/SC possui densidade demográfica de 37.5699073010036 hab/km².\nO município Água Doce/SC possui densidade demográfica de 5.296476370913131 hab/km².\nO município Águas de Chapecó/SC possui densidade demográfica de 43.69591646999928 hab/km².\nO município Águas Frias/SC possui densidade demográfica de 31.836091410559494 hab/km².\nO município Águas Mornas/SC possui densidade demográfica de 16.947702834799607 hab/km².\nO município Alfredo Wagner/SC possui densidade demográfica de 12.841682929159218 hab/km².\nO município Alto Bela Vista/SC possui densidade demográfica de 19.28255433737257 hab/km².\nO município Anchieta/SC possui densidade demográfica de 27.94079004992555 hab/km².\nO município Angelina/SC possui densidade demográfica de 10.499160067194625 hab/km².\nO município Anita Garibaldi/SC possui densidade demográfica de 14.67070452728108 hab/km².\nO município Anitápolis/SC possui densidade demográfica de 5.9285766988858555 hab/km².\nO município Antônio Carlos/SC possui densidade demográfica de 32.61753772140826 hab/km².\nO município Apiúna/SC possui densidade demográfica de 19.45919649734463 hab/km².\nO município Arabutã/SC possui densidade demográfica de 31.564287865100873 hab/km².\nO município Araquari/SC possui densidade demográfica de 64.61105757962447 hab/km².\nO município Araranguá/SC possui densidade demográfica de 202.14309264754368 hab/km².\nO município Armazém/SC possui densidade demográfica de 44.66528401889618 hab/km².\nO município Arroio Trinta/SC possui densidade demográfica de 37.13679745493107 hab/km².\nO município Arvoredo/SC possui densidade demográfica de 24.898094083948443 hab/km².\nO município Ascurra/SC possui densidade demográfica de 66.83498647430118 hab/km².\nO município Atalanta/SC possui densidade demográfica de 35.035566408323604 hab/km².\nO município Aurora/SC possui densidade demográfica de 26.85736411596728 hab/km².\nO município Balneário Arroio do Silva/SC possui densidade demográfica de 100.62985513331932 hab/km².\nO município Balneário Barra do Sul/SC possui densidade demográfica de 75.76166082502023 hab/km².\nO município Balneário Camboriú/SC possui densidade demográfica de 2337.5648788927333 hab/km².\nO município Balneário Gaivota/SC possui densidade demográfica de 56.490120746432495 hab/km².\nO município Balneário Piçarras/SC possui densidade demográfica de 171.79358213459412 hab/km².\nO município Bandeirante/SC possui densidade demográfica de 19.719074438488157 hab/km².\nO município Barra Bonita/SC possui densidade demográfica de 20.089858793324776 hab/km².\nO município Barra Velha/SC possui densidade demográfica de 159.78586723768737 hab/km².\nO município Bela Vista do Toldo/SC possui densidade demográfica de 11.157155334212923 hab/km².\nO município Belmonte/SC possui densidade demográfica de 28.52040264097846 hab/km².\nO município Benedito Novo/SC possui densidade demográfica de 26.584362139917694 hab/km².\nO município Biguaçu/SC possui densidade demográfica de 156.9444818939251 hab/km².\nO município Blumenau/SC possui densidade demográfica de 595.9710703953713 hab/km².\nO município Bocaina do Sul/SC possui densidade demográfica de 6.415131129960027 hab/km².\nO município Bom Jardim da Serra/SC possui densidade demográfica de 4.696165065660829 hab/km².\nO município Bom Jesus/SC possui densidade demográfica de 39.79832991964708 hab/km².\nO município Bom Jesus do Oeste/SC possui densidade demográfica de 31.77820837680727 hab/km².\nO município Bom Retiro/SC possui densidade demográfica de 8.471413007436881 hab/km².\nO município Bombinhas/SC possui densidade demográfica de 398.0228348649402 hab/km².\nO município Botuverá/SC possui densidade demográfica de 15.084911712076707 hab/km².\nO município Braço do Norte/SC possui densidade demográfica de 136.96780893042575 hab/km².\nO município Braço do Trombudo/SC possui densidade demográfica de 38.275022143489814 hab/km².\nO município Brunópolis/SC possui densidade demográfica de 8.455969617849513 hab/km².\nO município Brusque/SC possui densidade demográfica de 372.5125344255349 hab/km².\nO município Caçador/SC possui densidade demográfica de 71.89141411575856 hab/km².\nO município Caibi/SC possui densidade demográfica de 35.56966369251887 hab/km².\nO município Calmon/SC possui densidade demográfica de 5.307280077721019 hab/km².\nO município Camboriú/SC possui densidade demográfica de 293.68465668267874 hab/km².\nO município Campo Alegre/SC possui densidade demográfica de 23.53978399823672 hab/km².\nO município Campo Belo do Sul/SC possui densidade demográfica de 7.281662044470393 hab/km².\nO município Campo Erê/SC possui densidade demográfica de 19.55791187459559 hab/km².\nO município Campos Novos/SC possui densidade demográfica de 19.09071345899952 hab/km².\nO município Canelinha/SC possui densidade demográfica de 69.50052438384898 hab/km².\nO município Canoinhas/SC possui densidade demográfica de 46.268853034023145 hab/km².\nO município Capão Alto/SC possui densidade demográfica de 2.060875553958558 hab/km².\nO município Capinzal/SC possui densidade demográfica de 85.04914004914005 hab/km².\nO município Capivari de Baixo/SC possui densidade demográfica de 406.33670791151104 hab/km².\nO município Catanduvas/SC possui densidade demográfica de 48.428788646730865 hab/km².\nO município Caxambu do Sul/SC possui densidade demográfica de 31.348162888209792 hab/km².\nO município Celso Ramos/SC possui densidade demográfica de 13.304844672780524 hab/km².\nO município Cerro Negro/SC possui densidade demográfica de 8.580533857286625 hab/km².\nO município Chapadão do Lageado/SC possui densidade demográfica de 22.138505931388263 hab/km².\nO município Chapecó/SC possui densidade demográfica de 293.1508162156982 hab/km².\nO município Cocal do Sul/SC possui densidade demográfica de 213.11682834247154 hab/km².\nO município Concórdia/SC possui densidade demográfica de 85.78911836775517 hab/km².\nO município Cordilheira Alta/SC possui densidade demográfica de 45.46222544050205 hab/km².\nO município Coronel Freitas/SC possui densidade demográfica de 43.65089541394196 hab/km².\nO município Coronel Martins/SC possui densidade demográfica de 22.907735321528424 hab/km².\nO município Correia Pinto/SC possui densidade demográfica de 22.707027890404227 hab/km².\nO município Corupá/SC possui densidade demográfica de 34.390128851262446 hab/km².\nO município Criciúma/SC possui densidade demográfica de 815.8669551567604 hab/km².\nO município Cunha Porã/SC possui densidade demográfica de 48.701358296622615 hab/km².\nO município Cunhataí/SC possui densidade demográfica de 33.74574143804913 hab/km².\nO município Curitibanos/SC possui densidade demográfica de 39.78750764171427 hab/km².\nO município Descanso/SC possui densidade demográfica de 30.174040679387712 hab/km².\nO município Dionísio Cerqueira/SC possui densidade demográfica de 39.059574355863816 hab/km².\nO município Dona Emma/SC possui densidade demográfica de 20.53872053872054 hab/km².\nO município Doutor Pedrinho/SC possui densidade demográfica de 9.620158556442357 hab/km².\nO município Entre Rios/SC possui densidade demográfica de 28.866571018651364 hab/km².\nO município Ermo/SC possui densidade demográfica de 32.313997477931906 hab/km².\nO município Erval Velho/SC possui densidade demográfica de 20.98765432098765 hab/km².\nO município Faxinal dos Guedes/SC possui densidade demográfica de 31.38357374153665 hab/km².\nO município Flor do Sertão/SC possui densidade demográfica de 26.96552895228392 hab/km².\nO município Florianópolis/SC possui densidade demográfica de 623.6804311455264 hab/km².\nO município Formosa do Sul/SC possui densidade demográfica de 25.98142043751873 hab/km².\nO município Forquilhinha/SC possui densidade demográfica de 123.1256484464588 hab/km².\nO município Fraiburgo/SC possui densidade demográfica de 63.070183444373455 hab/km².\nO município Frei Rogério/SC possui densidade demográfica de 15.538248963698027 hab/km².\nO município Galvão/SC possui densidade demográfica de 28.46835027877993 hab/km².\nO município Garopaba/SC possui densidade demográfica de 157.16142448661296 hab/km².\nO município Garuva/SC possui densidade demográfica de 29.40613980915194 hab/km².\nO município Gaspar/SC possui densidade demográfica de 149.90692383266975 hab/km².\nO município Governador Celso Ramos/SC possui densidade demográfica de 110.93189964157706 hab/km².\nO município Grão Pará/SC possui densidade demográfica de 18.40253134610835 hab/km².\nO município Gravatal/SC possui densidade demográfica de 64.55235204855842 hab/km².\nO município Guabiruba/SC possui densidade demográfica de 105.50721318983284 hab/km².\nO município Guaraciaba/SC possui densidade demográfica de 31.77649302297424 hab/km².\nO município Guaramirim/SC possui densidade demográfica de 130.99441340782124 hab/km².\nO município Guarujá do Sul/SC possui densidade demográfica de 48.972261025743364 hab/km².\nO município Guatambú/SC possui densidade demográfica de 22.72683116378473 hab/km².\nO município Herval d`Oeste/SC possui densidade demográfica de 97.72695900243868 hab/km².\nO município Ibiam/SC possui densidade demográfica de 13.256543075245366 hab/km².\nO município Ibicaré/SC possui densidade demográfica de 21.6509403684447 hab/km².\nO município Ibirama/SC possui densidade demográfica de 70.06266424095412 hab/km².\nO município Içara/SC possui densidade demográfica de 200.41900868676544 hab/km².\nO município Ilhota/SC possui densidade demográfica de 48.85716545397026 hab/km².\nO município Imaruí/SC possui densidade demográfica de 21.510052890551574 hab/km².\nO município Imbituba/SC possui densidade demográfica de 219.59219373530857 hab/km².\nO município Imbuia/SC possui densidade demográfica de 46.3832899869961 hab/km².\nO município Indaial/SC possui densidade demográfica de 127.33350356322106 hab/km².\nO município Iomerê/SC possui densidade demográfica de 24.07912087912088 hab/km².\nO município Ipira/SC possui densidade demográfica de 30.743352526363463 hab/km².\nO município Iporã do Oeste/SC possui densidade demográfica de 42.10394552373322 hab/km².\nO município Ipuaçu/SC possui densidade demográfica de 26.056958871555064 hab/km².\nO município Ipumirim/SC possui densidade demográfica de 29.187047742248453 hab/km².\nO município Iraceminha/SC possui densidade demográfica de 26.055259449856035 hab/km².\nO município Irani/SC possui densidade demográfica de 29.259532142199298 hab/km².\nO município Irati/SC possui densidade demográfica de 26.77567705671947 hab/km².\nO município Irineópolis/SC possui densidade demográfica de 17.721690752425538 hab/km².\nO município Itá/SC possui densidade demográfica de 38.74819102749638 hab/km².\nO município Itaiópolis/SC possui densidade demográfica de 15.671244297260369 hab/km².\nO município Itajaí/SC possui densidade demográfica de 636.1154473236896 hab/km².\nO município Itapema/SC possui densidade demográfica de 792.3356401384084 hab/km².\nO município Itapiranga/SC possui densidade demográfica de 54.50654403961797 hab/km².\nO município Itapoá/SC possui densidade demográfica de 59.42997463870215 hab/km².\nO município Ituporanga/SC possui densidade demográfica de 66.03745585136379 hab/km².\nO município Jaborá/SC possui densidade demográfica de 21.054551138435887 hab/km².\nO município Jacinto Machado/SC possui densidade demográfica de 24.59316611803978 hab/km².\nO município Jaguaruna/SC possui densidade demográfica de 52.657225521547126 hab/km².\nO município Jaraguá do Sul/SC possui densidade demográfica de 270.27797711221064 hab/km².\nO município Jardinópolis/SC possui densidade demográfica de 26.093380614657207 hab/km².\nO município Joaçaba/SC possui densidade demográfica de 116.350170089997 hab/km².\nO município Joinville/SC possui densidade demográfica de 457.58229657848705 hab/km².\nO município José Boiteux/SC possui densidade demográfica de 11.650173975273301 hab/km².\nO município Jupiá/SC possui densidade demográfica de 23.335143943508964 hab/km².\nO município Lacerdópolis/SC possui densidade demográfica de 31.920452895921034 hab/km².\nO município Lages/SC possui densidade demográfica de 59.558046741402244 hab/km².\nO município Laguna/SC possui densidade demográfica de 116.76970808705302 hab/km².\nO município Lajeado Grande/SC possui densidade demográfica de 22.824754901960784 hab/km².\nO município Laurentino/SC possui densidade demográfica de 75.43661263977886 hab/km².\nO município Lauro Muller/SC possui densidade demográfica de 53.0578329270995 hab/km².\nO município Lebon Régis/SC possui densidade demográfica de 12.573686390721091 hab/km².\nO município Leoberto Leal/SC possui densidade demográfica de 11.555235053741287 hab/km².\nO município Lindóia do Sul/SC possui densidade demográfica de 24.60771840542833 hab/km².\nO município Lontras/SC possui densidade demográfica de 51.97098067069149 hab/km².\nO município Luiz Alves/SC possui densidade demográfica de 40.164691396028935 hab/km².\nO município Luzerna/SC possui densidade demográfica de 47.30528805541477 hab/km².\nO município Macieira/SC possui densidade demográfica de 7.032814666461254 hab/km².\nO município Mafra/SC possui densidade demográfica de 37.685804434378184 hab/km².\nO município Major Gercino/SC possui densidade demográfica de 11.476270474590507 hab/km².\nO município Major Vieira/SC possui densidade demográfica de 14.232159847764034 hab/km².\nO município Maracajá/SC possui densidade demográfica de 102.52961895613193 hab/km².\nO município Maravilha/SC possui densidade demográfica de 129.03432975245212 hab/km².\nO município Marema/SC possui densidade demográfica de 21.16844431632555 hab/km².\nO município Massaranduba/SC possui densidade demográfica de 39.226903336184776 hab/km².\nO município Matos Costa/SC possui densidade demográfica de 6.555522201953495 hab/km².\nO município Meleiro/SC possui densidade demográfica de 37.42114829466481 hab/km².\nO município Mirim Doce/SC possui densidade demográfica de 7.485181544693652 hab/km².\nO município Modelo/SC possui densidade demográfica de 44.39688288881572 hab/km².\nO município Mondaí/SC possui densidade demográfica de 50.61093247588424 hab/km².\nO município Monte Carlo/SC possui densidade demográfica de 48.11905746176105 hab/km².\nO município Monte Castelo/SC possui densidade demográfica de 14.55046287417842 hab/km².\nO município Morro da Fumaça/SC possui densidade demográfica de 194.0086621751684 hab/km².\nO município Morro Grande/SC possui densidade demográfica de 11.193740800991556 hab/km².\nO município Navegantes/SC possui densidade demográfica de 540.5820389216211 hab/km².\nO município Nova Erechim/SC possui densidade demográfica de 65.88072122052705 hab/km².\nO município Nova Itaberaba/SC possui densidade demográfica de 31.021446746637583 hab/km².\nO município Nova Trento/SC possui densidade demográfica de 30.256397527861203 hab/km².\nO município Nova Veneza/SC possui densidade demográfica de 45.109137744034705 hab/km².\nO município Novo Horizonte/SC possui densidade demográfica de 18.109976950938428 hab/km².\nO município Orleans/SC possui densidade demográfica de 38.9821243098453 hab/km².\nO município Otacílio Costa/SC possui densidade demográfica de 19.333499011846015 hab/km².\nO município Ouro/SC possui densidade demográfica de 34.501801843965 hab/km².\nO município Ouro Verde/SC possui densidade demográfica de 12.00190254729944 hab/km².\nO município Paial/SC possui densidade demográfica de 20.557369402985074 hab/km².\nO município Painel/SC possui densidade demográfica de 3.1789564700478263 hab/km².\nO município Palhoça/SC possui densidade demográfica de 347.5666236428517 hab/km².\nO município Palma Sola/SC possui densidade demográfica de 23.523174795516507 hab/km².\nO município Palmeira/SC possui densidade demográfica de 8.202557898375389 hab/km².\nO município Palmitos/SC possui densidade demográfica de 45.445519276048906 hab/km².\nO município Papanduva/SC possui densidade demográfica de 23.97240125157115 hab/km².\nO município Paraíso/SC possui densidade demográfica de 22.51158684617082 hab/km².\nO município Passo de Torres/SC possui densidade demográfica de 69.67721585532541 hab/km².\nO município Passos Maia/SC possui densidade demográfica de 7.1467795077201375 hab/km².\nO município Paulo Lopes/SC possui densidade demográfica de 14.881693648816936 hab/km².\nO município Pedras Grandes/SC possui densidade demográfica de 25.779925930575608 hab/km².\nO município Penha/SC possui densidade demográfica de 427.85908781484005 hab/km².\nO município Peritiba/SC possui densidade demográfica de 31.176961602671117 hab/km².\nO município Petrolândia/SC possui densidade demográfica de 20.044463334096186 hab/km².\nO município Pinhalzinho/SC possui densidade demográfica de 127.43445692883896 hab/km².\nO município Pinheiro Preto/SC possui densidade demográfica de 47.78317643486183 hab/km².\nO município Piratuba/SC possui densidade demográfica de 32.78531305658309 hab/km².\nO município Planalto Alegre/SC possui densidade demográfica de 42.49119436439321 hab/km².\nO município Pomerode/SC possui densidade demográfica de 129.273971964793 hab/km².\nO município Ponte Alta/SC possui densidade demográfica de 8.60165916760405 hab/km².\nO município Ponte Alta do Norte/SC possui densidade demográfica de 8.273219116321009 hab/km².\nO município Ponte Serrada/SC possui densidade demográfica de 19.541533065244735 hab/km².\nO município Porto Belo/SC possui densidade demográfica de 171.77186799102853 hab/km².\nO município Porto União/SC possui densidade demográfica de 39.62074431589656 hab/km².\nO município Pouso Redondo/SC possui densidade demográfica de 41.208714766688 hab/km².\nO município Praia Grande/SC possui densidade demográfica de 25.576320698271918 hab/km².\nO município Presidente Castello Branco/SC possui densidade demográfica de 26.291723822588022 hab/km².\nO município Presidente Getúlio/SC possui densidade demográfica de 50.589594590002385 hab/km².\nO município Presidente Nereu/SC possui densidade demográfica de 10.121421607728442 hab/km².\nO município Princesa/SC possui densidade demográfica de 32.01392919326756 hab/km².\nO município Quilombo/SC possui densidade demográfica de 36.56604581460073 hab/km².\nO município Rancho Queimado/SC possui densidade demográfica de 9.598658702714031 hab/km².\nO município Rio das Antas/SC possui densidade demográfica de 19.31761006289308 hab/km².\nO município Rio do Campo/SC possui densidade demográfica de 12.231111111111112 hab/km².\nO município Rio do Oeste/SC possui densidade demográfica de 28.610629111012468 hab/km².\nO município Rio do Sul/SC possui densidade demográfica de 235.05146719926256 hab/km².\nO município Rio dos Cedros/SC possui densidade demográfica de 18.560496679179902 hab/km².\nO município Rio Fortuna/SC possui densidade demográfica de 14.679565490144286 hab/km².\nO município Rio Negrinho/SC possui densidade demográfica de 43.91663268342683 hab/km².\nO município Rio Rufino/SC possui densidade demográfica de 8.623008849557522 hab/km².\nO município Riqueza/SC possui densidade demográfica de 25.201854456425483 hab/km².\nO município Rodeio/SC possui densidade demográfica de 84.06064804125297 hab/km².\nO município Romelândia/SC possui densidade demográfica de 24.578259907017934 hab/km².\nO município Salete/SC possui densidade demográfica de 41.09283523836075 hab/km².\nO município Saltinho/SC possui densidade demográfica de 25.305053344406822 hab/km².\nO município Salto Veloso/SC possui densidade demográfica de 40.93461501855906 hab/km².\nO município Sangão/SC possui densidade demográfica de 125.46748703100495 hab/km².\nO município Santa Cecília/SC possui densidade demográfica de 13.751843673907542 hab/km².\nO município Santa Helena/SC possui densidade demográfica de 29.15544675642595 hab/km².\nO município Santa Rosa de Lima/SC possui densidade demográfica de 10.222772277227723 hab/km².\nO município Santa Rosa do Sul/SC possui densidade demográfica de 53.32715354565318 hab/km².\nO município Santa Terezinha/SC possui densidade demográfica de 12.257081341050807 hab/km².\nO município Santa Terezinha do Progresso/SC possui densidade demográfica de 24.375052604999578 hab/km².\nO município Santiago do Sul/SC possui densidade demográfica de 19.840195016251354 hab/km².\nO município Santo Amaro da Imperatriz/SC possui densidade demográfica de 57.616625490481034 hab/km².\nO município São Bento do Sul/SC possui densidade demográfica de 149.11588222394994 hab/km².\nO município São Bernardino/SC possui densidade demográfica de 18.47991163882369 hab/km².\nO município São Bonifácio/SC possui densidade demográfica de 6.534016856373273 hab/km².\nO município São Carlos/SC possui densidade demográfica de 63.80432760865522 hab/km².\nO município São Cristovão do Sul/SC possui densidade demográfica de 14.275135289091425 hab/km².\nO município São Domingos/SC possui densidade demográfica de 24.678228763098367 hab/km².\nO município São Francisco do Sul/SC possui densidade demográfica de 85.27022961997393 hab/km².\nO município São João Batista/SC possui densidade demográfica de 118.79665234109929 hab/km².\nO município São João do Itaperiú/SC possui densidade demográfica de 22.685246334698192 hab/km².\nO município São João do Oeste/SC possui densidade demográfica de 36.96264543784446 hab/km².\nO município São João do Sul/SC possui densidade demográfica de 38.187172774869104 hab/km².\nO município São Joaquim/SC possui densidade demográfica de 13.112362994514497 hab/km².\nO município São José/SC possui densidade demográfica de 1376.7570050528252 hab/km².\nO município São José do Cedro/SC possui densidade demográfica de 48.69231042949152 hab/km².\nO município São José do Cerrito/SC possui densidade demográfica de 9.813529187656098 hab/km².\nO município São Lourenço do Oeste/SC possui densidade demográfica de 60.452729693741674 hab/km².\nO município São Ludgero/SC possui densidade demográfica de 102.10848968976407 hab/km².\nO município São Martinho/SC possui densidade demográfica de 14.332931350216626 hab/km².\nO município São Miguel da Boa Vista/SC possui densidade demográfica de 26.662932362414228 hab/km².\nO município São Miguel do Oeste/SC possui densidade demográfica de 155.11407331453472 hab/km².\nO município São Pedro de Alcântara/SC possui densidade demográfica de 33.59520068561634 hab/km².\nO município Saudades/SC possui densidade demográfica de 43.63988383349468 hab/km².\nO município Schroeder/SC possui densidade demográfica de 93.1743521109624 hab/km².\nO município Seara/SC possui densidade demográfica de 54.38838755258679 hab/km².\nO município Serra Alta/SC possui densidade demográfica de 35.5711965349215 hab/km².\nO município Siderópolis/SC possui densidade demográfica de 49.6751509592601 hab/km².\nO município Sombrio/SC possui densidade demográfica de 185.6764110793274 hab/km².\nO município Sul Brasil/SC possui densidade demográfica de 24.506068928856205 hab/km².\nO município Taió/SC possui densidade demográfica de 24.91051841588731 hab/km².\nO município Tangará/SC possui densidade demográfica de 22.34185040181331 hab/km².\nO município Tigrinhos/SC possui densidade demográfica de 30.324473593372456 hab/km².\nO município Tijucas/SC possui densidade demográfica de 110.7375348737392 hab/km².\nO município Timbé do Sul/SC possui densidade demográfica de 16.08046290405647 hab/km².\nO município Timbó/SC possui densidade demográfica de 288.62726630562753 hab/km².\nO município Timbó Grande/SC possui densidade demográfica de 11.97553762093338 hab/km².\nO município Três Barras/SC possui densidade demográfica de 41.432032178444096 hab/km².\nO município Treviso/SC possui densidade demográfica de 22.45352686529157 hab/km².\nO município Treze de Maio/SC possui densidade demográfica de 42.53108183336426 hab/km².\nO município Treze Tílias/SC possui densidade demográfica de 33.974496356622375 hab/km².\nO município Trombudo Central/SC possui densidade demográfica de 60.329589394218374 hab/km².\nO município Tubarão/SC possui densidade demográfica de 322.2262725344645 hab/km².\nO município Tunápolis/SC possui densidade demográfica de 34.774450198904155 hab/km².\nO município Turvo/SC possui densidade demográfica de 50.33118206521739 hab/km².\nO município União do Oeste/SC possui densidade demográfica de 31.418700064780822 hab/km².\nO município Urubici/SC possui densidade demográfica de 10.51354113438937 hab/km².\nO município Urupema/SC possui densidade demográfica de 7.090618215061135 hab/km².\nO município Urussanga/SC possui densidade demográfica de 79.34633342488327 hab/km².\nO município Vargeão/SC possui densidade demográfica de 21.194119411941195 hab/km².\nO município Vargem/SC possui densidade demográfica de 8.019420248464945 hab/km².\nO município Vargem Bonita/SC possui densidade demográfica de 16.056951423785595 hab/km².\nO município Vidal Ramos/SC possui densidade demográfica de 18.34407535944472 hab/km².\nO município Videira/SC possui densidade demográfica de 124.09077760538565 hab/km².\nO município Vitor Meireles/SC possui densidade demográfica de 14.053222498110763 hab/km².\nO município Witmarsum/SC possui densidade demográfica de 23.68732727990525 hab/km².\nO município Xanxerê/SC possui densidade demográfica de 116.81490893689116 hab/km².\nO município Xavantina/SC possui densidade demográfica de 19.114864553048132 hab/km².\nO município Xaxim/SC possui densidade demográfica de 87.6738952536825 hab/km².\nO município Zortéa/SC possui densidade demográfica de 15.765338393421885 hab/km².\nO município Adamantina/SP possui densidade demográfica de 82.15318797248354 hab/km².\nO município Adolfo/SP possui densidade demográfica de 16.851430737161266 hab/km².\nO município Aguaí/SP possui densidade demográfica de 67.71706618359524 hab/km².\nO município Águas da Prata/SP possui densidade demográfica de 53.04980414101846 hab/km².\nO município Águas de Lindóia/SP possui densidade demográfica de 287.14452020621985 hab/km².\nO município Águas de Santa Bárbara/SP possui densidade demográfica de 13.831678767224775 hab/km².\nO município Águas de São Pedro/SP possui densidade demográfica de 488.6281588447653 hab/km².\nO município Agudos/SP possui densidade demográfica de 35.73321189037013 hab/km².\nO município Alambari/SP possui densidade demográfica de 30.664908645695984 hab/km².\nO município Alfredo Marcondes/SP possui densidade demográfica de 32.86317567567568 hab/km².\nO município Altair/SP possui densidade demográfica de 12.155101000446058 hab/km².\nO município Altinópolis/SP possui densidade demográfica de 16.80050809507406 hab/km².\nO município Alto Alegre/SP possui densidade demográfica de 12.857321965897691 hab/km².\nO município Alumínio/SP possui densidade demográfica de 201.27898637341622 hab/km².\nO município Álvares Florence/SP possui densidade demográfica de 10.737311952388824 hab/km².\nO município Álvares Machado/SP possui densidade demográfica de 67.68668317116702 hab/km².\nO município Álvaro de Carvalho/SP possui densidade demográfica de 30.358425279101656 hab/km².\nO município Alvinlândia/SP possui densidade demográfica de 35.37735849056604 hab/km².\nO município Americana/SP possui densidade demográfica de 1572.746957365788 hab/km².\nO município Américo Brasiliense/SP possui densidade demográfica de 280.90272119928306 hab/km².\nO município Américo de Campos/SP possui densidade demográfica de 22.544448834452787 hab/km².\nO município Amparo/SP possui densidade demográfica de 147.74772752777466 hab/km².\nO município Analândia/SP possui densidade demográfica de 13.182055454908342 hab/km².\nO município Andradina/SP possui densidade demográfica de 57.389103807340874 hab/km².\nO município Angatuba/SP possui densidade demográfica de 21.605478705811397 hab/km².\nO município Anhembi/SP possui densidade demográfica de 7.674866949060498 hab/km².\nO município Anhumas/SP possui densidade demográfica de 11.664846309876737 hab/km².\nO município Aparecida/SP possui densidade demográfica de 289.1228939544103 hab/km².\nO município Aparecida d`Oeste/SP possui densidade demográfica de 24.857557814769297 hab/km².\nO município Apiaí/SP possui densidade demográfica de 25.854955250841613 hab/km².\nO município Araçariguama/SP possui densidade demográfica de 117.63085399449037 hab/km².\nO município Araçatuba/SP possui densidade demográfica de 155.53604467895565 hab/km².\nO município Araçoiaba da Serra/SP possui densidade demográfica de 106.87468190893787 hab/km².\nO município Aramina/SP possui densidade demográfica de 25.393070136527182 hab/km².\nO município Arandu/SP possui densidade demográfica de 21.415830156342903 hab/km².\nO município Arapeí/SP possui densidade demográfica de 15.889101338432122 hab/km².\nO município Araraquara/SP possui densidade demográfica de 207.8990106309843 hab/km².\nO município Araras/SP possui densidade demográfica de 184.30128871175347 hab/km².\nO município Arco-Íris/SP possui densidade demográfica de 7.271559702338231 hab/km².\nO município Arealva/SP possui densidade demográfica de 15.52765510822425 hab/km².\nO município Areias/SP possui densidade demográfica de 12.108901484126724 hab/km².\nO município Areiópolis/SP possui densidade demográfica de 123.34149469511485 hab/km².\nO município Ariranha/SP possui densidade demográfica de 64.19076229815997 hab/km².\nO município Artur Nogueira/SP possui densidade demográfica de 248.14357130820648 hab/km².\nO município Arujá/SP possui densidade demográfica de 779.3673915305379 hab/km².\nO município Aspásia/SP possui densidade demográfica de 26.0888376117681 hab/km².\nO município Assis/SP possui densidade demográfica de 206.69548782342335 hab/km².\nO município Atibaia/SP possui densidade demográfica de 264.5720137089359 hab/km².\nO município Auriflama/SP possui densidade demográfica de 32.724256319270026 hab/km².\nO município Avaí/SP possui densidade demográfica de 9.175517152055656 hab/km².\nO município Avanhandava/SP possui densidade demográfica de 33.39829907866761 hab/km².\nO município Avaré/SP possui densidade demográfica de 68.36759929434653 hab/km².\nO município Bady Bassitt/SP possui densidade demográfica de 132.321493294672 hab/km².\nO município Balbinos/SP possui densidade demográfica de 40.397206460061106 hab/km².\nO município Bálsamo/SP possui densidade demográfica de 54.18326693227092 hab/km².\nO município Bananal/SP possui densidade demográfica de 16.584202585857277 hab/km².\nO município Barão de Antonina/SP possui densidade demográfica de 20.34739454094293 hab/km².\nO município Barbosa/SP possui densidade demográfica de 32.13746039483305 hab/km².\nO município Bariri/SP possui densidade demográfica de 71.14418897921499 hab/km².\nO município Barra Bonita/SP possui densidade demográfica de 235.11440197451805 hab/km².\nO município Barra do Chapéu/SP possui densidade demográfica de 12.926444488266615 hab/km².\nO município Barra do Turvo/SP possui densidade demográfica de 7.669028199480065 hab/km².\nO município Barretos/SP possui densidade demográfica de 71.60075113052808 hab/km².\nO município Barrinha/SP possui densidade demográfica de 195.66053282065369 hab/km².\nO município Barueri/SP possui densidade demográfica de 3664.9261683665704 hab/km².\nO município Bastos/SP possui densidade demográfica de 118.94234684972949 hab/km².\nO município Batatais/SP possui densidade demográfica de 66.47911197956518 hab/km².\nO município Bauru/SP possui densidade demográfica de 515.1225137790559 hab/km².\nO município Bebedouro/SP possui densidade demográfica de 109.81267378896533 hab/km².\nO município Bento de Abreu/SP possui densidade demográfica de 8.87193098871931 hab/km².\nO município Bernardino de Campos/SP possui densidade demográfica de 44.123669123669124 hab/km².\nO município Bertioga/SP possui densidade demográfica de 97.20493726410282 hab/km².\nO município Bilac/SP possui densidade demográfica de 44.6358454718176 hab/km².\nO município Birigui/SP possui densidade demográfica de 204.7916823626912 hab/km².\nO município Biritiba-Mirim/SP possui densidade demográfica de 90.02551904476859 hab/km².\nO município Boa Esperança do Sul/SP possui densidade demográfica de 19.753604725230183 hab/km².\nO município Bocaina/SP possui densidade demográfica de 29.838155689280907 hab/km².\nO município Bofete/SP possui densidade demográfica de 14.716773265599658 hab/km².\nO município Boituva/SP possui densidade demográfica de 194.07109861417956 hab/km².\nO município Bom Jesus dos Perdões/SP possui densidade demográfica de 181.85844790993818 hab/km².\nO município Bom Sucesso de Itararé/SP possui densidade demográfica de 26.73304386884264 hab/km².\nO município Borá/SP possui densidade demográfica de 6.796116504854369 hab/km².\nO município Boracéia/SP possui densidade demográfica de 34.95209237572681 hab/km².\nO município Borborema/SP possui densidade demográfica de 26.308260601890414 hab/km².\nO município Borebi/SP possui densidade demográfica de 6.589269806603638 hab/km².\nO município Botucatu/SP possui densidade demográfica de 85.87924243241784 hab/km².\nO município Bragança Paulista/SP possui densidade demográfica de 286.2627287269322 hab/km².\nO município Braúna/SP possui densidade demográfica de 25.705216812573592 hab/km².\nO município Brejo Alegre/SP possui densidade demográfica de 24.41176470588235 hab/km².\nO município Brodowski/SP possui densidade demográfica de 75.79903756374345 hab/km².\nO município Brotas/SP possui densidade demográfica de 19.59360075541593 hab/km².\nO município Buri/SP possui densidade demográfica de 15.522071058858943 hab/km².\nO município Buritama/SP possui densidade demográfica de 47.18447790427225 hab/km².\nO município Buritizal/SP possui densidade demográfica de 15.212821860220703 hab/km².\nO município Cabrália Paulista/SP possui densidade demográfica de 18.194322871076654 hab/km².\nO município Cabreúva/SP possui densidade demográfica de 159.87395765284555 hab/km².\nO município Caçapava/SP possui densidade demográfica de 229.66154513183213 hab/km².\nO município Cachoeira Paulista/SP possui densidade demográfica de 104.48626688426681 hab/km².\nO município Caconde/SP possui densidade demográfica de 39.44423166943274 hab/km².\nO município Cafelândia/SP possui densidade demográfica de 18.049125095098358 hab/km².\nO município Caiabu/SP possui densidade demográfica de 16.105046669830724 hab/km².\nO município Caieiras/SP possui densidade demográfica de 900.4058272632675 hab/km².\nO município Caiuá/SP possui densidade demográfica de 9.163650912000582 hab/km².\nO município Cajamar/SP possui densidade demográfica de 488.1900555851671 hab/km².\nO município Cajati/SP possui densidade demográfica de 62.432884429187574 hab/km².\nO município Cajobi/SP possui densidade demográfica de 55.21763708309779 hab/km².\nO município Cajuru/SP possui densidade demográfica de 35.4057779999697 hab/km².\nO município Campina do Monte Alegre/SP possui densidade demográfica de 30.0870129168243 hab/km².\nO município Campinas/SP possui densidade demográfica de 1359.607517339476 hab/km².\nO município Campo Limpo Paulista/SP possui densidade demográfica de 932.9219143576826 hab/km².\nO município Campos do Jordão/SP possui densidade demográfica de 164.7555678135558 hab/km².\nO município Campos Novos Paulista/SP possui densidade demográfica de 9.378486714327037 hab/km².\nO município Cananéia/SP possui densidade demográfica de 9.864609724216946 hab/km².\nO município Canas/SP possui densidade demográfica de 82.33195644010515 hab/km².\nO município Cândido Mota/SP possui densidade demográfica de 50.123278710521454 hab/km².\nO município Cândido Rodrigues/SP possui densidade demográfica de 37.946238088465364 hab/km².\nO município Canitar/SP possui densidade demográfica de 76.34107985322383 hab/km².\nO município Capão Bonito/SP possui densidade demográfica de 28.153368734872547 hab/km².\nO município Capela do Alto/SP possui densidade demográfica de 103.19618576726118 hab/km².\nO município Capivari/SP possui densidade demográfica de 150.44598612487613 hab/km².\nO município Caraguatatuba/SP possui densidade demográfica de 207.87466501752215 hab/km².\nO município Carapicuíba/SP possui densidade demográfica de 10697.076700434154 hab/km².\nO município Cardoso/SP possui densidade demográfica de 18.453097400465822 hab/km².\nO município Casa Branca/SP possui densidade demográfica de 32.75590733412021 hab/km².\nO município Cássia dos Coqueiros/SP possui densidade demográfica de 13.741652754590984 hab/km².\nO município Castilho/SP possui densidade demográfica de 16.89153687370989 hab/km².\nO município Catanduva/SP possui densidade demográfica de 388.2312456985547 hab/km².\nO município Catiguá/SP possui densidade demográfica de 48.028842913942995 hab/km².\nO município Cedral/SP possui densidade demográfica de 40.32576255753958 hab/km².\nO município Cerqueira César/SP possui densidade demográfica de 34.26762049958954 hab/km².\nO município Cerquilho/SP possui densidade demográfica de 309.9921752738654 hab/km².\nO município Cesário Lange/SP possui densidade demográfica de 81.45934895423808 hab/km².\nO município Charqueada/SP possui densidade demográfica de 85.78333807222064 hab/km².\nO município Chavantes/SP possui densidade demográfica de 64.40191387559808 hab/km².\nO município Clementina/SP possui densidade demográfica de 41.84434968017057 hab/km².\nO município Colina/SP possui densidade demográfica de 41.107982109472985 hab/km².\nO município Colômbia/SP possui densidade demográfica de 8.21940349674323 hab/km².\nO município Conchal/SP possui densidade demográfica de 138.0217736200011 hab/km².\nO município Conchas/SP possui densidade demográfica de 34.951289644221276 hab/km².\nO município Cordeirópolis/SP possui densidade demográfica de 153.2199447594127 hab/km².\nO município Coroados/SP possui densidade demográfica de 21.261568436434484 hab/km².\nO município Coronel Macedo/SP possui densidade demográfica de 16.454446747606358 hab/km².\nO município Corumbataí/SP possui densidade demográfica de 13.904242337233509 hab/km².\nO município Cosmópolis/SP possui densidade demográfica de 380.3633777317988 hab/km².\nO município Cosmorama/SP possui densidade demográfica de 16.331982522469495 hab/km².\nO município Cotia/SP possui densidade demográfica de 620.8141724020863 hab/km².\nO município Cravinhos/SP possui densidade demográfica de 101.7694283879255 hab/km².\nO município Cristais Paulista/SP possui densidade demográfica de 19.69732367676453 hab/km².\nO município Cruzália/SP possui densidade demográfica de 15.256625293525662 hab/km².\nO município Cruzeiro/SP possui densidade demográfica de 252.0085050703304 hab/km².\nO município Cubatão/SP possui densidade demográfica de 830.9070548712207 hab/km².\nO município Cunha/SP possui densidade demográfica de 15.537333371230424 hab/km².\nO município Descalvado/SP possui densidade demográfica de 41.20417667272558 hab/km².\nO município Diadema/SP possui densidade demográfica de 12535.357142857143 hab/km².\nO município Dirce Reis/SP possui densidade demográfica de 19.117147707979626 hab/km².\nO município Divinolândia/SP possui densidade demográfica de 50.456939629946426 hab/km².\nO município Dobrada/SP possui densidade demográfica de 53.022106458291596 hab/km².\nO município Dois Córregos/SP possui densidade demográfica de 39.11875760304596 hab/km².\nO município Dolcinópolis/SP possui densidade demográfica de 26.755169772785294 hab/km².\nO município Dourado/SP possui densidade demográfica de 41.81765191625783 hab/km².\nO município Dracena/SP possui densidade demográfica de 88.63617736251126 hab/km².\nO município Duartina/SP possui densidade demográfica de 46.30707589960689 hab/km².\nO município Dumont/SP possui densidade demográfica de 73.1232040229885 hab/km².\nO município Echaporã/SP possui densidade demográfica de 12.257726558407544 hab/km².\nO município Eldorado/SP possui densidade demográfica de 8.850482995417892 hab/km².\nO município Elias Fausto/SP possui densidade demográfica de 77.82821056786226 hab/km².\nO município Elisiário/SP possui densidade demográfica de 33.19855288359225 hab/km².\nO município Embaúba/SP possui densidade demográfica de 29.147118970287504 hab/km².\nO município Embu das Artes/SP possui densidade demográfica de 3412.8427333428044 hab/km².\nO município Embu-Guaçu/SP possui densidade demográfica de 403.3219816230804 hab/km².\nO município Emilianópolis/SP possui densidade demográfica de 13.452715042986323 hab/km².\nO município Engenheiro Coelho/SP possui densidade demográfica de 142.99617973440058 hab/km².\nO município Espírito Santo do Pinhal/SP possui densidade demográfica de 107.6138873196035 hab/km².\nO município Espírito Santo do Turvo/SP possui densidade demográfica de 21.914695858721473 hab/km².\nO município Estiva Gerbi/SP possui densidade demográfica de 135.3456407492252 hab/km².\nO município Estrela do Norte/SP possui densidade demográfica de 10.090350011388656 hab/km².\nO município Estrela d`Oeste/SP possui densidade demográfica de 27.691373435444145 hab/km².\nO município Euclides da Cunha Paulista/SP possui densidade demográfica de 16.663479424905685 hab/km².\nO município Fartura/SP possui densidade demográfica de 35.69681012186313 hab/km².\nO município Fernando Prestes/SP possui densidade demográfica de 32.425147946329176 hab/km².\nO município Fernandópolis/SP possui densidade demográfica de 117.62267512681127 hab/km².\nO município Fernão/SP possui densidade demográfica de 15.512107979356887 hab/km².\nO município Ferraz de Vasconcelos/SP possui densidade demográfica de 5691.782211701048 hab/km².\nO município Flora Rica/SP possui densidade demográfica de 7.7762982689747 hab/km².\nO município Floreal/SP possui densidade demográfica de 14.698972099853156 hab/km².\nO município Flórida Paulista/SP possui densidade demográfica de 24.468652395825398 hab/km².\nO município Florínia/SP possui densidade demográfica de 12.53822629969419 hab/km².\nO município Franca/SP possui densidade demográfica de 526.0863822480518 hab/km².\nO município Francisco Morato/SP possui densidade demográfica de 3147.9926635418788 hab/km².\nO município Franco da Rocha/SP possui densidade demográfica de 980.948121645796 hab/km².\nO município Gabriel Monteiro/SP possui densidade demográfica de 19.54529050884157 hab/km².\nO município Gália/SP possui densidade demográfica de 19.69326704306059 hab/km².\nO município Garça/SP possui densidade demográfica de 77.59660205532458 hab/km².\nO município Gastão Vidigal/SP possui densidade demográfica de 23.173427655576436 hab/km².\nO município Gavião Peixoto/SP possui densidade demográfica de 18.127743364647003 hab/km².\nO município General Salgado/SP possui densidade demográfica de 21.625620756055536 hab/km².\nO município Getulina/SP possui densidade demográfica de 15.861205245321939 hab/km².\nO município Glicério/SP possui densidade demográfica de 16.687381196081297 hab/km².\nO município Guaiçara/SP possui densidade demográfica de 39.35236409235082 hab/km².\nO município Guaimbê/SP possui densidade demográfica de 24.884179624787855 hab/km².\nO município Guaíra/SP possui densidade demográfica de 29.721568876740193 hab/km².\nO município Guapiaçu/SP possui densidade demográfica de 54.99507571094423 hab/km².\nO município Guapiara/SP possui densidade demográfica de 44.08141272135002 hab/km².\nO município Guará/SP possui densidade demográfica de 54.783712204811295 hab/km².\nO município Guaraçaí/SP possui densidade demográfica de 14.801621422429676 hab/km².\nO município Guaraci/SP possui densidade demográfica de 15.551052221356196 hab/km².\nO município Guarani d`Oeste/SP possui densidade demográfica de 23.032853969367473 hab/km².\nO município Guarantã/SP possui densidade demográfica de 13.887021576493549 hab/km².\nO município Guararapes/SP possui densidade demográfica de 31.99385155906895 hab/km².\nO município Guararema/SP possui densidade demográfica de 95.42869802821062 hab/km².\nO município Guaratinguetá/SP possui densidade demográfica de 148.90518707482994 hab/km².\nO município Guareí/SP possui densidade demográfica de 25.717312615873574 hab/km².\nO município Guariba/SP possui densidade demográfica de 131.28861593103701 hab/km².\nO município Guarujá/SP possui densidade demográfica de 2026.852561868247 hab/km².\nO município Guarulhos/SP possui densidade demográfica de 3834.50169448977 hab/km².\nO município Guatapará/SP possui densidade demográfica de 16.836660704790447 hab/km².\nO município Guzolândia/SP possui densidade demográfica de 18.864330780524583 hab/km².\nO município Herculândia/SP possui densidade demográfica de 23.848179025888548 hab/km².\nO município Holambra/SP possui densidade demográfica de 172.29338212869777 hab/km².\nO município Hortolândia/SP possui densidade demográfica de 3093.9627488760434 hab/km².\nO município Iacanga/SP possui densidade demográfica de 18.2922596320722 hab/km².\nO município Iacri/SP possui densidade demográfica de 19.895855934042093 hab/km².\nO município Iaras/SP possui densidade demográfica de 15.887966908375072 hab/km².\nO município Ibaté/SP possui densidade demográfica de 105.73866373081951 hab/km².\nO município Ibirá/SP possui densidade demográfica de 40.072082674414325 hab/km².\nO município Ibirarema/SP possui densidade demográfica de 29.45427470217239 hab/km².\nO município Ibitinga/SP possui densidade demográfica de 77.12441059122234 hab/km².\nO município Ibiúna/SP possui densidade demográfica de 67.30776500831696 hab/km².\nO município Icém/SP possui densidade demográfica de 20.579718138944816 hab/km².\nO município Iepê/SP possui densidade demográfica de 12.809618969252213 hab/km².\nO município Igaraçu do Tietê/SP possui densidade demográfica de 239.07081457224723 hab/km².\nO município Igarapava/SP possui densidade demográfica de 59.69460758142018 hab/km².\nO município Igaratá/SP possui densidade demográfica de 30.145075951527566 hab/km².\nO município Iguape/SP possui densidade demográfica de 14.581258373568593 hab/km².\nO município Ilha Comprida/SP possui densidade demográfica de 47.01255404490285 hab/km².\nO município Ilha Solteira/SP possui densidade demográfica de 38.415204230209206 hab/km².\nO município Ilhabela/SP possui densidade demográfica de 81.1302296138574 hab/km².\nO município Indaiatuba/SP possui densidade demográfica de 646.1112001281846 hab/km².\nO município Indiana/SP possui densidade demográfica de 38.10614436897804 hab/km².\nO município Indiaporã/SP possui densidade demográfica de 13.959227467811157 hab/km².\nO município Inúbia Paulista/SP possui densidade demográfica de 41.52842924150555 hab/km².\nO município Ipaussu/SP possui densidade demográfica de 65.16741390823238 hab/km².\nO município Iperó/SP possui densidade demográfica de 166.19685224336388 hab/km².\nO município Ipeúna/SP possui densidade demográfica de 31.661491500447347 hab/km².\nO município Ipiguá/SP possui densidade demográfica de 32.891148942442335 hab/km².\nO município Iporanga/SP possui densidade demográfica de 3.7316088711427455 hab/km².\nO município Ipuã/SP possui densidade demográfica de 30.36833519361209 hab/km².\nO município Iracemápolis/SP possui densidade demográfica de 173.98366921473246 hab/km².\nO município Irapuã/SP possui densidade demográfica de 28.20751424915668 hab/km².\nO município Irapuru/SP possui densidade demográfica de 36.24476500697999 hab/km².\nO município Itaberá/SP possui densidade demográfica de 16.081044574515985 hab/km².\nO município Itaí/SP possui densidade demográfica de 22.17255582851549 hab/km².\nO município Itajobi/SP possui densidade demográfica de 28.99197323082439 hab/km².\nO município Itaju/SP possui densidade demográfica de 14.124097119484814 hab/km².\nO município Itanhaém/SP possui densidade demográfica de 144.6922731730018 hab/km².\nO município Itaóca/SP possui densidade demográfica de 17.63741667577314 hab/km².\nO município Itapecerica da Serra/SP possui densidade demográfica de 1011.5596208656459 hab/km².\nO município Itapetininga/SP possui densidade demográfica de 80.64808039280308 hab/km².\nO município Itapeva/SP possui densidade demográfica de 48.050660913561046 hab/km².\nO município Itapevi/SP possui densidade demográfica de 2428.853133317203 hab/km².\nO município Itapira/SP possui densidade demográfica de 132.21126950751366 hab/km².\nO município Itapirapuã Paulista/SP possui densidade demográfica de 9.545365085613067 hab/km².\nO município Itápolis/SP possui densidade demográfica de 40.17755931183227 hab/km².\nO município Itaporanga/SP possui densidade demográfica de 28.65612258966733 hab/km².\nO município Itapuí/SP possui densidade demográfica de 86.4559659090909 hab/km².\nO município Itapura/SP possui densidade demográfica de 14.457311610312905 hab/km².\nO município Itaquaquecetuba/SP possui densidade demográfica de 3895.049025541702 hab/km².\nO município Itararé/SP possui densidade demográfica de 47.76300842982124 hab/km².\nO município Itariri/SP possui densidade demográfica de 56.531589140205355 hab/km².\nO município Itatiba/SP possui densidade demográfica de 314.90239890761256 hab/km².\nO município Itatinga/SP possui densidade demográfica de 18.423792125084198 hab/km².\nO município Itirapina/SP possui densidade demográfica de 27.487782420851335 hab/km².\nO município Itirapuã/SP possui densidade demográfica de 36.70556107249255 hab/km².\nO município Itobi/SP possui densidade demográfica de 54.205876014654116 hab/km².\nO município Itu/SP possui densidade demográfica de 241.0128521842459 hab/km².\nO município Itupeva/SP possui densidade demográfica de 223.37914550343592 hab/km².\nO município Ituverava/SP possui densidade demográfica de 54.867846406896945 hab/km².\nO município Jaborandi/SP possui densidade demográfica de 24.107665301345815 hab/km².\nO município Jaboticabal/SP possui densidade demográfica de 101.41805830738748 hab/km².\nO município Jacareí/SP possui densidade demográfica de 454.93785943524244 hab/km².\nO município Jaci/SP possui densidade demográfica de 38.874381528312256 hab/km².\nO município Jacupiranga/SP possui densidade demográfica de 24.440057379028246 hab/km².\nO município Jaguariúna/SP possui densidade demográfica de 313.37340876944836 hab/km².\nO município Jales/SP possui densidade demográfica de 127.5697384131119 hab/km².\nO município Jambeiro/SP possui densidade demográfica de 29.006019196355947 hab/km².\nO município Jandira/SP possui densidade demográfica de 6208.8252148997135 hab/km².\nO município Jardinópolis/SP possui densidade demográfica de 74.98904862410895 hab/km².\nO município Jarinu/SP possui densidade demográfica de 114.8478135234059 hab/km².\nO município Jaú/SP possui densidade demográfica de 191.08726084927673 hab/km².\nO município Jeriquara/SP possui densidade demográfica de 22.25822356835951 hab/km².\nO município Joanópolis/SP possui densidade demográfica de 31.441701400021376 hab/km².\nO município João Ramalho/SP possui densidade demográfica de 9.993979530403372 hab/km².\nO município José Bonifácio/SP possui densidade demográfica de 38.09872667015524 hab/km².\nO município Júlio Mesquita/SP possui densidade demográfica de 34.5499922009047 hab/km².\nO município Jumirim/SP possui densidade demográfica de 49.356147468689365 hab/km².\nO município Jundiaí/SP possui densidade demográfica de 858.4224319873831 hab/km².\nO município Junqueirópolis/SP possui densidade demográfica de 32.122272540139974 hab/km².\nO município Juquiá/SP possui densidade demográfica de 23.680098431251924 hab/km².\nO município Juquitiba/SP possui densidade demográfica de 55.03274732850742 hab/km².\nO município Lagoinha/SP possui densidade demográfica de 18.949387403609034 hab/km².\nO município Laranjal Paulista/SP possui densidade demográfica de 65.75438779230248 hab/km².\nO município Lavínia/SP possui densidade demográfica de 16.326037230580404 hab/km².\nO município Lavrinhas/SP possui densidade demográfica de 39.44454420302867 hab/km².\nO município Leme/SP possui densidade demográfica de 227.75585176359618 hab/km².\nO município Lençóis Paulista/SP possui densidade demográfica de 75.88481636585999 hab/km².\nO município Limeira/SP possui densidade demográfica de 475.3181450293606 hab/km².\nO município Lindóia/SP possui densidade demográfica de 137.6538146021329 hab/km².\nO município Lins/SP possui densidade demográfica de 124.98162858242644 hab/km².\nO município Lorena/SP possui densidade demográfica de 199.28771489279504 hab/km².\nO município Lourdes/SP possui densidade demográfica de 18.709337084578866 hab/km².\nO município Louveira/SP possui densidade demográfica de 673.4083076364956 hab/km².\nO município Lucélia/SP possui densidade demográfica de 63.16558647858686 hab/km².\nO município Lucianópolis/SP possui densidade demográfica de 11.848066589400485 hab/km².\nO município Luís Antônio/SP possui densidade demográfica de 18.84863971140839 hab/km².\nO município Luiziânia/SP possui densidade demográfica de 30.20114079855899 hab/km².\nO município Lupércio/SP possui densidade demográfica de 28.176581008479513 hab/km².\nO município Lutécia/SP possui densidade demográfica de 5.714526351251763 hab/km².\nO município Macatuba/SP possui densidade demográfica de 72.1948403712091 hab/km².\nO município Macaubal/SP possui densidade demográfica de 30.88300487647604 hab/km².\nO município Macedônia/SP possui densidade demográfica de 11.180275845233735 hab/km².\nO município Magda/SP possui densidade demográfica de 10.26595232748388 hab/km².\nO município Mairinque/SP possui densidade demográfica de 205.52042223384527 hab/km².\nO município Mairiporã/SP possui densidade demográfica de 252.43529778609292 hab/km².\nO município Manduri/SP possui densidade demográfica de 39.257803972931676 hab/km².\nO município Marabá Paulista/SP possui densidade demográfica de 5.237437008173972 hab/km².\nO município Maracaí/SP possui densidade demográfica de 24.969097651421507 hab/km².\nO município Marapoama/SP possui densidade demográfica de 23.66316167879932 hab/km².\nO município Mariápolis/SP possui densidade demográfica de 21.06508875739645 hab/km².\nO município Marília/SP possui densidade demográfica de 185.21256141850031 hab/km².\nO município Marinópolis/SP possui densidade demográfica de 27.148914300398303 hab/km².\nO município Martinópolis/SP possui densidade demográfica de 19.33328543717221 hab/km².\nO município Matão/SP possui densidade demográfica de 146.29806043516365 hab/km².\nO município Mauá/SP possui densidade demográfica de 6740.9730079198325 hab/km².\nO município Mendonça/SP possui densidade demográfica de 23.789991796554553 hab/km².\nO município Meridiano/SP possui densidade demográfica de 16.815703380588875 hab/km².\nO município Mesópolis/SP possui densidade demográfica de 12.66962246406019 hab/km².\nO município Miguelópolis/SP possui densidade demográfica de 24.880772786996932 hab/km².\nO município Mineiros do Tietê/SP possui densidade demográfica de 56.45282311011067 hab/km².\nO município Mira Estrela/SP possui densidade demográfica de 13.005580408615044 hab/km².\nO município Miracatu/SP possui densidade demográfica de 20.560337080895422 hab/km².\nO município Mirandópolis/SP possui densidade demográfica de 29.91184153243361 hab/km².\nO município Mirante do Paranapanema/SP possui densidade demográfica de 13.767472640991704 hab/km².\nO município Mirassol/SP possui densidade demográfica de 221.10238809651034 hab/km².\nO município Mirassolândia/SP possui densidade demográfica de 25.847024131913102 hab/km².\nO município Mococa/SP possui densidade demográfica de 77.54486114685446 hab/km².\nO município Mogi das Cruzes/SP possui densidade demográfica de 544.12140261271 hab/km².\nO município Mogi Guaçu/SP possui densidade demográfica de 168.98763790386133 hab/km².\nO município Moji Mirim/SP possui densidade demográfica de 173.77460827641622 hab/km².\nO município Mombuca/SP possui densidade demográfica de 24.42782348541511 hab/km².\nO município Monções/SP possui densidade demográfica de 20.452801227935534 hab/km².\nO município Mongaguá/SP possui densidade demográfica de 325.9840856277727 hab/km².\nO município Monte Alegre do Sul/SP possui densidade demográfica de 64.83546369322818 hab/km².\nO município Monte Alto/SP possui densidade demográfica de 134.6089466089466 hab/km².\nO município Monte Aprazível/SP possui densidade demográfica de 43.76245195306997 hab/km².\nO município Monte Azul Paulista/SP possui densidade demográfica de 71.86076525964167 hab/km².\nO município Monte Castelo/SP possui densidade demográfica de 17.470009029539494 hab/km².\nO município Monte Mor/SP possui densidade demográfica de 203.6063391705836 hab/km².\nO município Monteiro Lobato/SP possui densidade demográfica de 12.382040031255634 hab/km².\nO município Morro Agudo/SP possui densidade demográfica de 20.973923065840655 hab/km².\nO município Morungaba/SP possui densidade demográfica de 80.19761499148211 hab/km².\nO município Motuca/SP possui densidade demográfica de 18.75819851333625 hab/km².\nO município Murutinga do Sul/SP possui densidade demográfica de 16.68792856003827 hab/km².\nO município Nantes/SP possui densidade demográfica de 9.45974280123008 hab/km².\nO município Narandiba/SP possui densidade demográfica de 11.976650001396532 hab/km².\nO município Natividade da Serra/SP possui densidade demográfica de 8.013247417113647 hab/km².\nO município Nazaré Paulista/SP possui densidade demográfica de 50.30494345520855 hab/km².\nO município Neves Paulista/SP possui densidade demográfica de 40.17587249244298 hab/km².\nO município Nhandeara/SP possui densidade demográfica de 24.611607040411226 hab/km².\nO município Nipoã/SP possui densidade demográfica de 31.01146422870411 hab/km².\nO município Nova Aliança/SP possui densidade demográfica de 27.10873866826193 hab/km².\nO município Nova Campina/SP possui densidade demográfica de 22.095074990918054 hab/km².\nO município Nova Canaã Paulista/SP possui densidade demográfica de 16.990837485934737 hab/km².\nO município Nova Castilho/SP possui densidade demográfica de 6.139824264585494 hab/km².\nO município Nova Europa/SP possui densidade demográfica de 57.998129092609915 hab/km².\nO município Nova Granada/SP possui densidade demográfica de 36.06076558622246 hab/km².\nO município Nova Guataporanga/SP possui densidade demográfica de 63.8042203985932 hab/km².\nO município Nova Independência/SP possui densidade demográfica de 11.543381744299799 hab/km².\nO município Nova Luzitânia/SP possui densidade demográfica de 46.46232784229004 hab/km².\nO município Nova Odessa/SP possui densidade demográfica de 689.4779332615716 hab/km².\nO município Novais/SP possui densidade demográfica de 38.991254139424306 hab/km².\nO município Novo Horizonte/SP possui densidade demográfica de 39.27678255176189 hab/km².\nO município Nuporanga/SP possui densidade demográfica de 19.57389381801476 hab/km².\nO município Ocauçu/SP possui densidade demográfica de 13.860496087897452 hab/km².\nO município Óleo/SP possui densidade demográfica de 13.490461289996972 hab/km².\nO município Olímpia/SP possui densidade demográfica de 62.32355322992587 hab/km².\nO município Onda Verde/SP possui densidade demográfica de 16.029053691552143 hab/km².\nO município Oriente/SP possui densidade demográfica de 27.889849503682356 hab/km².\nO município Orindiúva/SP possui densidade demográfica de 22.87291926967877 hab/km².\nO município Orlândia/SP possui densidade demográfica de 136.3436953764952 hab/km².\nO município Osasco/SP possui densidade demográfica de 10265.43494996151 hab/km².\nO município Oscar Bressane/SP possui densidade demográfica de 11.46200415650131 hab/km².\nO município Osvaldo Cruz/SP possui densidade demográfica de 124.46958412174403 hab/km².\nO município Ourinhos/SP possui densidade demográfica de 347.77398994160734 hab/km².\nO município Ouro Verde/SP possui densidade demográfica de 29.14689286648481 hab/km².\nO município Ouroeste/SP possui densidade demográfica de 29.099155241656284 hab/km².\nO município Pacaembu/SP possui densidade demográfica de 39.07237813884786 hab/km².\nO município Palestina/SP possui densidade demográfica de 15.890202168349006 hab/km².\nO município Palmares Paulista/SP possui densidade demográfica de 133.13040301960308 hab/km².\nO município Palmeira d`Oeste/SP possui densidade demográfica de 30.023181504918234 hab/km².\nO município Palmital/SP possui densidade demográfica de 38.67399280772531 hab/km².\nO município Panorama/SP possui densidade demográfica de 40.92784373158205 hab/km².\nO município Paraguaçu Paulista/SP possui densidade demográfica de 42.22310995705583 hab/km².\nO município Paraibuna/SP possui densidade demográfica de 21.477803305417623 hab/km².\nO município Paraíso/SP possui densidade demográfica de 37.846509240246405 hab/km².\nO município Paranapanema/SP possui densidade demográfica de 17.480760169624627 hab/km².\nO município Paranapuã/SP possui densidade demográfica de 27.156890660592257 hab/km².\nO município Parapuã/SP possui densidade demográfica de 29.653531679838114 hab/km².\nO município Pardinho/SP possui densidade demográfica de 26.594883033970177 hab/km².\nO município Pariquera-Açu/SP possui densidade demográfica de 51.338714166434734 hab/km².\nO município Parisi/SP possui densidade demográfica de 24.041646947468056 hab/km².\nO município Patrocínio Paulista/SP possui densidade demográfica de 21.564236543087002 hab/km².\nO município Paulicéia/SP possui densidade demográfica de 16.968707337312953 hab/km².\nO município Paulínia/SP possui densidade demográfica de 592.1712802768166 hab/km².\nO município Paulistânia/SP possui densidade demográfica de 6.931618936294565 hab/km².\nO município Paulo de Faria/SP possui densidade demográfica de 11.633639897601213 hab/km².\nO município Pederneiras/SP possui densidade demográfica de 56.92318244170096 hab/km².\nO município Pedra Bela/SP possui densidade demográfica de 36.44618197868718 hab/km².\nO município Pedranópolis/SP possui densidade demográfica de 9.831277143625812 hab/km².\nO município Pedregulho/SP possui densidade demográfica de 22.03199550940219 hab/km².\nO município Pedreira/SP possui densidade demográfica de 382.70558983331796 hab/km².\nO município Pedrinhas Paulista/SP possui densidade demográfica de 19.276160503540517 hab/km².\nO município Pedro de Toledo/SP possui densidade demográfica de 15.21985561720661 hab/km².\nO município Penápolis/SP possui densidade demográfica de 82.31222655205886 hab/km².\nO município Pereira Barreto/SP possui densidade demográfica de 25.50057208237986 hab/km².\nO município Pereiras/SP possui densidade demográfica de 33.385586957495406 hab/km².\nO município Peruíbe/SP possui densidade demográfica de 184.40488677731844 hab/km².\nO município Piacatu/SP possui densidade demográfica de 22.75348597004648 hab/km².\nO município Piedade/SP possui densidade demográfica de 69.81536278067134 hab/km².\nO município Pilar do Sul/SP possui densidade demográfica de 38.768498942917546 hab/km².\nO município Pindamonhangaba/SP possui densidade demográfica de 201.39336064338463 hab/km².\nO município Pindorama/SP possui densidade demográfica de 81.36666125628956 hab/km².\nO município Pinhalzinho/SP possui densidade demográfica de 84.80553937746716 hab/km².\nO município Piquerobi/SP possui densidade demográfica de 7.329506600078745 hab/km².\nO município Piquete/SP possui densidade demográfica de 80.1534090909091 hab/km².\nO município Piracaia/SP possui densidade demográfica de 65.14668119212513 hab/km².\nO município Piracicaba/SP possui densidade demográfica de 264.46935074356185 hab/km².\nO município Piraju/SP possui densidade demográfica de 56.4420218037661 hab/km².\nO município Pirajuí/SP possui densidade demográfica de 27.546711963115747 hab/km².\nO município Pirangi/SP possui densidade demográfica de 49.303815093288776 hab/km².\nO município Pirapora do Bom Jesus/SP possui densidade demográfica de 144.97788426096574 hab/km².\nO município Pirapozinho/SP possui densidade demográfica de 51.662168664616416 hab/km².\nO município Pirassununga/SP possui densidade demográfica de 96.38161513917923 hab/km².\nO município Piratininga/SP possui densidade demográfica de 29.99925449168758 hab/km².\nO município Pitangueiras/SP possui densidade demográfica de 81.98727475385473 hab/km².\nO município Planalto/SP possui densidade demográfica de 15.384350224060668 hab/km².\nO município Platina/SP possui densidade demográfica de 9.769534478009366 hab/km².\nO município Poá/SP possui densidade demográfica de 6142.120509849362 hab/km².\nO município Poloni/SP possui densidade demográfica de 40.39988018571215 hab/km².\nO município Pompéia/SP possui densidade demográfica de 25.462337066040867 hab/km².\nO município Pongaí/SP possui densidade demográfica de 18.98761795669012 hab/km².\nO município Pontal/SP possui densidade demográfica de 112.94342164346655 hab/km².\nO município Pontalinda/SP possui densidade demográfica de 19.382463485417954 hab/km².\nO município Pontes Gestal/SP possui densidade demográfica de 11.58340233692152 hab/km².\nO município Populina/SP possui densidade demográfica de 13.366038930210477 hab/km².\nO município Porangaba/SP possui densidade demográfica de 31.337272761488954 hab/km².\nO município Porto Feliz/SP possui densidade demográfica de 87.82489985809487 hab/km².\nO município Porto Ferreira/SP possui densidade demográfica de 209.8730145767833 hab/km².\nO município Potim/SP possui densidade demográfica de 436.1816955250731 hab/km².\nO município Potirendaba/SP possui densidade demográfica de 45.122378643612365 hab/km².\nO município Pracinha/SP possui densidade demográfica de 45.48058561425843 hab/km².\nO município Pradópolis/SP possui densidade demográfica de 103.81766041343052 hab/km².\nO município Praia Grande/SP possui densidade demográfica de 1781.8113823349427 hab/km².\nO município Pratânia/SP possui densidade demográfica de 26.26499143346659 hab/km².\nO município Presidente Alves/SP possui densidade demográfica de 14.356349455064592 hab/km².\nO município Presidente Bernardes/SP possui densidade demográfica de 18.118699512651045 hab/km².\nO município Presidente Epitácio/SP possui densidade demográfica de 32.785818574239826 hab/km².\nO município Presidente Prudente/SP possui densidade demográfica de 368.8942589598252 hab/km².\nO município Presidente Venceslau/SP possui densidade demográfica de 50.09646642175648 hab/km².\nO município Promissão/SP possui densidade demográfica de 45.77815419361462 hab/km².\nO município Quadra/SP possui densidade demográfica de 15.73317775184753 hab/km².\nO município Quatá/SP possui densidade demográfica de 19.679567015698755 hab/km².\nO município Queiroz/SP possui densidade demográfica de 12.010778904144745 hab/km².\nO município Queluz/SP possui densidade demográfica de 45.26678141135972 hab/km².\nO município Quintana/SP possui densidade demográfica de 18.78774603373283 hab/km².\nO município Rafard/SP possui densidade demográfica de 70.79325935059597 hab/km².\nO município Rancharia/SP possui densidade demográfica de 18.144594858485515 hab/km².\nO município Redenção da Serra/SP possui densidade demográfica de 12.518990205902318 hab/km².\nO município Regente Feijó/SP possui densidade demográfica de 69.77024936809146 hab/km².\nO município Reginópolis/SP possui densidade demográfica de 17.825324959836426 hab/km².\nO município Registro/SP possui densidade demográfica de 75.11108650212483 hab/km².\nO município Restinga/SP possui densidade demográfica de 26.803662258392677 hab/km².\nO município Ribeira/SP possui densidade demográfica de 10.001489203276247 hab/km².\nO município Ribeirão Bonito/SP possui densidade demográfica de 25.734280564097126 hab/km².\nO município Ribeirão Branco/SP possui densidade demográfica de 26.192114695340504 hab/km².\nO município Ribeirão Corrente/SP possui densidade demográfica de 28.807388930088315 hab/km².\nO município Ribeirão do Sul/SP possui densidade demográfica de 21.82728656291423 hab/km².\nO município Ribeirão dos Índios/SP possui densidade demográfica de 11.138840786390954 hab/km².\nO município Ribeirão Grande/SP possui densidade demográfica de 22.264218862491 hab/km².\nO município Ribeirão Pires/SP possui densidade demográfica de 1140.7183212267958 hab/km².\nO município Ribeirão Preto/SP possui densidade demográfica de 928.9080742288312 hab/km².\nO município Rifaina/SP possui densidade demográfica de 21.14331425758415 hab/km².\nO município Rincão/SP possui densidade demográfica de 32.96091153663554 hab/km².\nO município Rinópolis/SP possui densidade demográfica de 27.725839310133118 hab/km².\nO município Rio Claro/SP possui densidade demográfica de 373.6868504474138 hab/km².\nO município Rio das Pedras/SP possui densidade demográfica de 130.15529868525545 hab/km².\nO município Rio Grande da Serra/SP possui densidade demográfica de 1210.0715465052283 hab/km².\nO município Riolândia/SP possui densidade demográfica de 16.696138179292053 hab/km².\nO município Riversul/SP possui densidade demográfica de 15.958052822371828 hab/km².\nO município Rosana/SP possui densidade demográfica de 26.506656615558576 hab/km².\nO município Roseira/SP possui densidade demográfica de 73.47110600841944 hab/km².\nO município Rubiácea/SP possui densidade demográfica de 11.518169923606129 hab/km².\nO município Rubinéia/SP possui densidade demográfica de 11.782626595306711 hab/km².\nO município Sabino/SP possui densidade demográfica de 16.780315213895143 hab/km².\nO município Sagres/SP possui densidade demográfica de 16.204330175913395 hab/km².\nO município Sales/SP possui densidade demográfica de 17.671659210270377 hab/km².\nO município Sales Oliveira/SP possui densidade demográfica de 34.57662609606073 hab/km².\nO município Salesópolis/SP possui densidade demográfica de 36.78823529411765 hab/km².\nO município Salmourão/SP possui densidade demográfica de 27.964478495559813 hab/km².\nO município Saltinho/SP possui densidade demográfica de 70.77401243232404 hab/km².\nO município Salto/SP possui densidade demográfica de 792.1026949928684 hab/km².\nO município Salto de Pirapora/SP possui densidade demográfica de 143.016998681444 hab/km².\nO município Salto Grande/SP possui densidade demográfica de 46.64012738853503 hab/km².\nO município Sandovalina/SP possui densidade demográfica de 8.127526806117068 hab/km².\nO município Santa Adélia/SP possui densidade demográfica de 43.31520096705954 hab/km².\nO município Santa Albertina/SP possui densidade demográfica de 20.981046302745906 hab/km².\nO município Santa Bárbara d`Oeste/SP possui densidade demográfica de 664.485049833887 hab/km².\nO município Santa Branca/SP possui densidade demográfica de 50.55465765501028 hab/km².\nO município Santa Clara d`Oeste/SP possui densidade demográfica de 11.361282233004415 hab/km².\nO município Santa Cruz da Conceição/SP possui densidade demográfica de 26.656897355625127 hab/km².\nO município Santa Cruz da Esperança/SP possui densidade demográfica de 13.1905984060516 hab/km².\nO município Santa Cruz das Palmeiras/SP possui densidade demográfica de 101.34759937698924 hab/km².\nO município Santa Cruz do Rio Pardo/SP possui densidade demográfica de 39.44409519533004 hab/km².\nO município Santa Ernestina/SP possui densidade demográfica de 41.42240737985419 hab/km².\nO município Santa Fé do Sul/SP possui densidade demográfica de 141.80610116882488 hab/km².\nO município Santa Gertrudes/SP possui densidade demográfica de 220.1037745447146 hab/km².\nO município Santa Isabel/SP possui densidade demográfica de 138.87420864299477 hab/km².\nO município Santa Lúcia/SP possui densidade demográfica de 53.548010127897165 hab/km².\nO município Santa Maria da Serra/SP possui densidade demográfica de 21.42744042435278 hab/km².\nO município Santa Mercedes/SP possui densidade demográfica de 16.96530233115599 hab/km².\nO município Santa Rita do Passa Quatro/SP possui densidade demográfica de 35.11019174158644 hab/km².\nO município Santa Rita d`Oeste/SP possui densidade demográfica de 12.104912414318354 hab/km².\nO município Santa Rosa de Viterbo/SP possui densidade demográfica de 82.68764294129878 hab/km².\nO município Santa Salete/SP possui densidade demográfica de 18.226476886257714 hab/km².\nO município Santana da Ponte Pensa/SP possui densidade demográfica de 12.597881160755414 hab/km².\nO município Santana de Parnaíba/SP possui densidade demográfica de 604.7518479408659 hab/km².\nO município Santo Anastácio/SP possui densidade demográfica de 37.05614073189272 hab/km².\nO município Santo André/SP possui densidade demográfica de 3848.03163044715 hab/km².\nO município Santo Antônio da Alegria/SP possui densidade demográfica de 20.316478133359116 hab/km².\nO município Santo Antônio de Posse/SP possui densidade demográfica de 134.0909090909091 hab/km².\nO município Santo Antônio do Aracanguá/SP possui densidade demográfica de 5.829205650339388 hab/km².\nO município Santo Antônio do Jardim/SP possui densidade demográfica de 54.04692615496545 hab/km².\nO município Santo Antônio do Pinhal/SP possui densidade demográfica de 48.76325088339223 hab/km².\nO município Santo Expedito/SP possui densidade demográfica de 29.68022024565862 hab/km².\nO município Santópolis do Aguapeí/SP possui densidade demográfica de 33.43757329372215 hab/km².\nO município Santos/SP possui densidade demográfica de 1494.28154059928 hab/km².\nO município São Bento do Sapucaí/SP possui densidade demográfica de 41.36731871171705 hab/km².\nO município São Bernardo do Campo/SP possui densidade demográfica de 1869.3538145941193 hab/km².\nO município São Caetano do Sul/SP possui densidade demográfica de 9736.660143509458 hab/km².\nO município São Carlos/SP possui densidade demográfica de 195.15004440224035 hab/km².\nO município São Francisco/SP possui densidade demográfica de 36.93467336683417 hab/km².\nO município São João da Boa Vista/SP possui densidade demográfica de 161.95925796832037 hab/km².\nO município São João das Duas Pontes/SP possui densidade demográfica de 19.839183547239834 hab/km².\nO município São João de Iracema/SP possui densidade demográfica de 9.965847376966574 hab/km².\nO município São João do Pau d`Alho/SP possui densidade demográfica de 17.86442405708461 hab/km².\nO município São Joaquim da Barra/SP possui densidade demográfica de 113.27812956648806 hab/km².\nO município São José da Bela Vista/SP possui densidade demográfica de 30.352049106336885 hab/km².\nO município São José do Barreiro/SP possui densidade demográfica de 7.143983598801451 hab/km².\nO município São José do Rio Pardo/SP possui densidade demográfica de 123.81020539612109 hab/km².\nO município São José do Rio Preto/SP possui densidade demográfica de 945.1291786276507 hab/km².\nO município São José dos Campos/SP possui densidade demográfica de 572.9627709407773 hab/km².\nO município São Lourenço da Serra/SP possui densidade demográfica de 74.99060806096709 hab/km².\nO município São Luís do Paraitinga/SP possui densidade demográfica de 16.842156418065183 hab/km².\nO município São Manuel/SP possui densidade demográfica de 58.91789726016872 hab/km².\nO município São Miguel Arcanjo/SP possui densidade demográfica de 33.80484554034009 hab/km².\nO município São Paulo/SP possui densidade demográfica de 7398.266386167906 hab/km².\nO município São Pedro/SP possui densidade demográfica de 51.98246564547111 hab/km².\nO município São Pedro do Turvo/SP possui densidade demográfica de 9.836558434459386 hab/km².\nO município São Roque/SP possui densidade demográfica de 256.8212179466293 hab/km².\nO município São Sebastião/SP possui densidade demográfica de 185.00300240192152 hab/km².\nO município São Sebastião da Grama/SP possui densidade demográfica de 47.939614866471196 hab/km².\nO município São Simão/SP possui densidade demográfica de 23.24179829890644 hab/km².\nO município São Vicente/SP possui densidade demográfica de 2247.9207519102038 hab/km².\nO município Sarapuí/SP possui densidade demográfica de 25.594714905441037 hab/km².\nO município Sarutaiá/SP possui densidade demográfica de 25.57728973942518 hab/km².\nO município Sebastianópolis do Sul/SP possui densidade demográfica de 18.033079485959068 hab/km².\nO município Serra Azul/SP possui densidade demográfica de 39.75418520873067 hab/km².\nO município Serra Negra/SP possui densidade demográfica de 129.51310493766564 hab/km².\nO município Serrana/SP possui densidade demográfica de 308.4331614438715 hab/km².\nO município Sertãozinho/SP possui densidade demográfica de 273.22461339886314 hab/km².\nO município Sete Barras/SP possui densidade demográfica de 12.237696433612497 hab/km².\nO município Severínia/SP possui densidade demográfica de 110.38239692373423 hab/km².\nO município Silveiras/SP possui densidade demográfica de 13.964029123872898 hab/km².\nO município Socorro/SP possui densidade demográfica de 81.70055452865066 hab/km².\nO município Sorocaba/SP possui densidade demográfica de 1304.1907514450866 hab/km².\nO município Sud Mennucci/SP possui densidade demográfica de 12.573989514628785 hab/km².\nO município Sumaré/SP possui densidade demográfica de 1572.0586319218241 hab/km².\nO município Suzanápolis/SP possui densidade demográfica de 10.244995608854971 hab/km².\nO município Suzano/SP possui densidade demográfica de 1272.9388942774005 hab/km².\nO município Tabapuã/SP possui densidade demográfica de 32.88095375889809 hab/km².\nO município Tabatinga/SP possui densidade demográfica de 39.73914925857777 hab/km².\nO município Taboão da Serra/SP possui densidade demográfica de 11992.545365375183 hab/km².\nO município Taciba/SP possui densidade demográfica de 9.408703956793072 hab/km².\nO município Taguaí/SP possui densidade demográfica de 74.50629601596366 hab/km².\nO município Taiaçu/SP possui densidade demográfica de 55.27006751687922 hab/km².\nO município Taiúva/SP possui densidade demográfica de 41.121848105088326 hab/km².\nO município Tambaú/SP possui densidade demográfica de 39.88323038857936 hab/km².\nO município Tanabi/SP possui densidade demográfica de 32.253955484043985 hab/km².\nO município Tapiraí/SP possui densidade demográfica de 10.610515163554496 hab/km².\nO município Tapiratiba/SP possui densidade demográfica de 57.23465444414487 hab/km².\nO município Taquaral/SP possui densidade demográfica de 50.58452403043236 hab/km².\nO município Taquaritinga/SP possui densidade demográfica de 90.95319923178005 hab/km².\nO município Taquarituba/SP possui densidade demográfica de 49.708984679883145 hab/km².\nO município Taquarivaí/SP possui densidade demográfica de 22.222701583329737 hab/km².\nO município Tarabai/SP possui densidade demográfica de 32.78257417882306 hab/km².\nO município Tarumã/SP possui densidade demográfica de 42.499505244409264 hab/km².\nO município Tatuí/SP possui densidade demográfica de 205.02406968747613 hab/km².\nO município Taubaté/SP possui densidade demográfica de 445.97609179215544 hab/km².\nO município Tejupá/SP possui densidade demográfica de 16.231267719724585 hab/km².\nO município Teodoro Sampaio/SP possui densidade demográfica de 13.744304269307642 hab/km².\nO município Terra Roxa/SP possui densidade demográfica de 38.39035840028889 hab/km².\nO município Tietê/SP possui densidade demográfica de 91.08555885262118 hab/km².\nO município Timburi/SP possui densidade demográfica de 13.445805173027086 hab/km².\nO município Torre de Pedra/SP possui densidade demográfica de 31.59074982480729 hab/km².\nO município Torrinha/SP possui densidade demográfica de 29.59368160624227 hab/km².\nO município Trabiju/SP possui densidade demográfica de 24.34563229265216 hab/km².\nO município Tremembé/SP possui densidade demográfica de 214.17224080267556 hab/km².\nO município Três Fronteiras/SP possui densidade demográfica de 35.89523116608241 hab/km².\nO município Tuiuti/SP possui densidade demográfica de 46.8034727703236 hab/km².\nO município Tupã/SP possui densidade demográfica de 100.9944153633196 hab/km².\nO município Tupi Paulista/SP possui densidade demográfica de 58.16010434499062 hab/km².\nO município Turiúba/SP possui densidade demográfica de 12.603670084242147 hab/km².\nO município Turmalina/SP possui densidade demográfica de 13.370285250777343 hab/km².\nO município Ubarana/SP possui densidade demográfica de 25.23016743786672 hab/km².\nO município Ubatuba/SP possui densidade demográfica de 108.86672284928781 hab/km².\nO município Ubirajara/SP possui densidade demográfica de 15.67801112016149 hab/km².\nO município Uchoa/SP possui densidade demográfica de 37.5148538382318 hab/km².\nO município União Paulista/SP possui densidade demográfica de 20.212362533181647 hab/km².\nO município Urânia/SP possui densidade demográfica de 42.28965253182732 hab/km².\nO município Uru/SP possui densidade demográfica de 8.511941212492346 hab/km².\nO município Urupês/SP possui densidade demográfica de 39.27104247104247 hab/km².\nO município Valentim Gentil/SP possui densidade demográfica de 73.7256997795444 hab/km².\nO município Valinhos/SP possui densidade demográfica de 718.709199811562 hab/km².\nO município Valparaíso/SP possui densidade demográfica de 26.327696793002914 hab/km².\nO município Vargem/SP possui densidade demográfica de 61.7137648131267 hab/km².\nO município Vargem Grande do Sul/SP possui densidade demográfica de 146.93709538599708 hab/km².\nO município Vargem Grande Paulista/SP possui densidade demográfica de 1012.1704331450095 hab/km².\nO município Várzea Paulista/SP possui densidade demográfica de 3049.231207289294 hab/km².\nO município Vera Cruz/SP possui densidade demográfica de 43.41113395412585 hab/km².\nO município Vinhedo/SP possui densidade demográfica de 779.546568627451 hab/km².\nO município Viradouro/SP possui densidade demográfica de 79.4424286960915 hab/km².\nO município Vista Alegre do Alto/SP possui densidade demográfica de 72.49947357338387 hab/km².\nO município Vitória Brasil/SP possui densidade demográfica de 34.94969818913481 hab/km².\nO município Votorantim/SP possui densidade demográfica de 591.0320478001087 hab/km².\nO município Votuporanga/SP possui densidade demográfica de 201.1543120442724 hab/km².\nO município Zacarias/SP possui densidade demográfica de 7.316538196402833 hab/km².\nO município Amparo de São Francisco/SE possui densidade demográfica de 64.75946484486194 hab/km².\nO município Aquidabã/SE possui densidade demográfica de 55.82120292799688 hab/km².\nO município Aracaju/SE possui densidade demográfica de 3140.5971626525898 hab/km².\nO município Arauá/SE possui densidade demográfica de 54.73207547169811 hab/km².\nO município Areia Branca/SE possui densidade demográfica de 114.92364330515407 hab/km².\nO município Barra dos Coqueiros/SE possui densidade demográfica de 276.52790079716567 hab/km².\nO município Boquim/SE possui densidade demográfica de 123.9827134116733 hab/km².\nO município Brejo Grande/SE possui densidade demográfica de 52.00859868332661 hab/km².\nO município Campo do Brito/SE possui densidade demográfica de 83.02681802409161 hab/km².\nO município Canhoba/SE possui densidade demográfica de 23.23095895237536 hab/km².\nO município Canindé de São Francisco/SE possui densidade demográfica de 27.3604876697146 hab/km².\nO município Capela/SE possui densidade demográfica de 69.47870081763563 hab/km².\nO município Carira/SE possui densidade demográfica de 31.437774984286612 hab/km².\nO município Carmópolis/SE possui densidade demográfica de 294.11892833805274 hab/km².\nO município Cedro de São João/SE possui densidade demográfica de 67.2918408792259 hab/km².\nO município Cristinápolis/SE possui densidade demográfica de 69.93945552309582 hab/km².\nO município Cumbe/SE possui densidade demográfica de 29.65007776049767 hab/km².\nO município Divina Pastora/SE possui densidade demográfica de 47.12931691905436 hab/km².\nO município Estância/SE possui densidade demográfica de 100.00155260216121 hab/km².\nO município Feira Nova/SE possui densidade demográfica de 28.78927161628724 hab/km².\nO município Frei Paulo/SE possui densidade demográfica de 34.65381156958737 hab/km².\nO município Gararu/SE possui densidade demográfica de 17.412479579840912 hab/km².\nO município General Maynard/SE possui densidade demográfica de 146.59659659659658 hab/km².\nO município Gracho Cardoso/SE possui densidade demográfica de 23.320664298107907 hab/km².\nO município Ilha das Flores/SE possui densidade demográfica de 152.78184480234262 hab/km².\nO município Indiaroba/SE possui densidade demográfica de 50.492775810927185 hab/km².\nO município Itabaiana/SE possui densidade demográfica de 258.2999198075381 hab/km².\nO município Itabaianinha/SE possui densidade demográfica de 78.87535221260465 hab/km².\nO município Itabi/SE possui densidade demográfica de 26.96019954451795 hab/km².\nO município Itaporanga d`Ajuda/SE possui densidade demográfica de 41.11064560161097 hab/km².\nO município Japaratuba/SE possui densidade demográfica de 46.2154014798575 hab/km².\nO município Japoatã/SE possui densidade demográfica de 31.755927544057727 hab/km².\nO município Lagarto/SE possui densidade demográfica de 97.8372078631985 hab/km².\nO município Laranjeiras/SE possui densidade demográfica de 165.7752033522307 hab/km².\nO município Macambira/SE possui densidade demográfica de 46.74309916751862 hab/km².\nO município Malhada dos Bois/SE possui densidade demográfica de 54.68354430379747 hab/km².\nO município Malhador/SE possui densidade demográfica de 119.29859322369725 hab/km².\nO município Maruim/SE possui densidade demográfica de 174.28815186093635 hab/km².\nO município Moita Bonita/SE possui densidade demográfica de 114.80901690670007 hab/km².\nO município Monte Alegre de Sergipe/SE possui densidade demográfica de 33.447878058957805 hab/km².\nO município Muribeca/SE possui densidade demográfica de 96.80991299762721 hab/km².\nO município Neópolis/SE possui densidade demográfica de 69.58450836623426 hab/km².\nO município Nossa Senhora Aparecida/SE possui densidade demográfica de 24.99559316058523 hab/km².\nO município Nossa Senhora da Glória/SE possui densidade demográfica de 42.95760684212613 hab/km².\nO município Nossa Senhora das Dores/SE possui densidade demográfica de 50.85341884762594 hab/km².\nO município Nossa Senhora de Lourdes/SE possui densidade demográfica de 76.9553417221811 hab/km².\nO município Nossa Senhora do Socorro/SE possui densidade demográfica de 1025.8786757670473 hab/km².\nO município Pacatuba/SE possui densidade demográfica de 35.142581991332726 hab/km².\nO município Pedra Mole/SE possui densidade demográfica de 36.255028648055585 hab/km².\nO município Pedrinhas/SE possui densidade demográfica de 260.2533883323512 hab/km².\nO município Pinhão/SE possui densidade demográfica de 38.315478863301045 hab/km².\nO município Pirambu/SE possui densidade demográfica de 40.64989314163591 hab/km².\nO município Poço Redondo/SE possui densidade demográfica de 25.06249391293056 hab/km².\nO município Poço Verde/SE possui densidade demográfica de 49.946606684388705 hab/km².\nO município Porto da Folha/SE possui densidade demográfica de 30.94266499487063 hab/km².\nO município Propriá/SE possui densidade demográfica de 319.2437163375224 hab/km².\nO município Riachão do Dantas/SE possui densidade demográfica de 36.47618868421548 hab/km².\nO município Riachuelo/SE possui densidade demográfica de 118.50772738788955 hab/km².\nO município Ribeirópolis/SE possui densidade demográfica de 66.42555989633699 hab/km².\nO município Rosário do Catete/SE possui densidade demográfica de 87.2704902517509 hab/km².\nO município Salgado/SE possui densidade demográfica de 78.13823992252753 hab/km².\nO município Santa Luzia do Itanhy/SE possui densidade demográfica de 39.81518435514076 hab/km².\nO município Santa Rosa de Lima/SE possui densidade demográfica de 55.450377163141546 hab/km².\nO município Santana do São Francisco/SE possui densidade demográfica de 154.27444103463395 hab/km².\nO município Santo Amaro das Brotas/SE possui densidade demográfica de 48.727365903655624 hab/km².\nO município São Cristóvão/SE possui densidade demográfica de 180.52465320697706 hab/km².\nO município São Domingos/SE possui densidade demográfica de 100.23421489216356 hab/km².\nO município São Francisco/SE possui densidade demográfica de 40.46511627906977 hab/km².\nO município São Miguel do Aleixo/SE possui densidade demográfica de 25.664515233534594 hab/km².\nO município Simão Dias/SE possui densidade demográfica de 68.5367192618959 hab/km².\nO município Siriri/SE possui densidade demográfica de 48.27211869006695 hab/km².\nO município Telha/SE possui densidade demográfica de 60.31001427697328 hab/km².\nO município Tobias Barreto/SE possui densidade demográfica de 47.03762814424612 hab/km².\nO município Tomar do Geru/SE possui densidade demográfica de 42.161364381764514 hab/km².\nO município Umbaúba/SE possui densidade demográfica de 188.74305906108026 hab/km².\nO município Abreulândia/TO possui densidade demográfica de 1.2616016167073834 hab/km².\nO município Aguiarnópolis/TO possui densidade demográfica de 21.929563702791114 hab/km².\nO município Aliança do Tocantins/TO possui densidade demográfica de 3.5898085140053806 hab/km².\nO município Almas/TO possui densidade demográfica de 1.890243294694561 hab/km².\nO município Alvorada/TO possui densidade demográfica de 6.9082719420543315 hab/km².\nO município Ananás/TO possui densidade demográfica de 6.255667514283721 hab/km².\nO município Angico/TO possui densidade demográfica de 7.02853474420561 hab/km².\nO município Aparecida do Rio Negro/TO possui densidade demográfica de 3.630738471349656 hab/km².\nO município Aragominas/TO possui densidade demográfica de 5.014236270949483 hab/km².\nO município Araguacema/TO possui densidade demográfica de 2.273545247761366 hab/km².\nO município Araguaçu/TO possui densidade demográfica de 1.700093847657195 hab/km².\nO município Araguaína/TO possui densidade demográfica de 37.61705020972798 hab/km².\nO município Araguanã/TO possui densidade demográfica de 6.016530507278447 hab/km².\nO município Araguatins/TO possui densidade demográfica de 11.933538770954828 hab/km².\nO município Arapoema/TO possui densidade demográfica de 4.343456468799526 hab/km².\nO município Arraias/TO possui densidade demográfica de 1.839509095590535 hab/km².\nO município Augustinópolis/TO possui densidade demográfica de 40.38179148311307 hab/km².\nO município Aurora do Tocantins/TO possui densidade demográfica de 4.577394630925973 hab/km².\nO município Axixá do Tocantins/TO possui densidade demográfica de 61.74688769056654 hab/km².\nO município Babaçulândia/TO possui densidade demográfica de 5.828478132024199 hab/km².\nO município Bandeirantes do Tocantins/TO possui densidade demográfica de 2.0248534218855396 hab/km².\nO município Barra do Ouro/TO possui densidade demográfica de 3.7266687757038914 hab/km².\nO município Barrolândia/TO possui densidade demográfica de 7.4989485489976175 hab/km².\nO município Bernardo Sayão/TO possui densidade demográfica de 4.807474457594752 hab/km².\nO município Bom Jesus do Tocantins/TO possui densidade demográfica de 2.827406634800813 hab/km².\nO município Brasilândia do Tocantins/TO possui densidade demográfica de 3.2176095530578204 hab/km².\nO município Brejinho de Nazaré/TO possui densidade demográfica de 3.006755777204326 hab/km².\nO município Buriti do Tocantins/TO possui densidade demográfica de 38.77421403620197 hab/km².\nO município Cachoeirinha/TO possui densidade demográfica de 6.096211153682417 hab/km².\nO município Campos Lindos/TO possui densidade demográfica de 2.5118974871766384 hab/km².\nO município Cariri do Tocantins/TO possui densidade demográfica de 3.328017012227539 hab/km².\nO município Carmolândia/TO possui densidade demográfica de 6.8236056686603215 hab/km².\nO município Carrasco Bonito/TO possui densidade demográfica de 19.11475069969939 hab/km².\nO município Caseara/TO possui densidade demográfica de 2.7198940654169697 hab/km².\nO município Centenário/TO possui densidade demográfica de 1.3127334117767433 hab/km².\nO município Chapada da Natividade/TO possui densidade demográfica de 1.9903186817858813 hab/km².\nO município Chapada de Areia/TO possui densidade demográfica de 2.025028441410694 hab/km².\nO município Colinas do Tocantins/TO possui densidade demográfica de 36.54440955146057 hab/km².\nO município Colméia/TO possui densidade demográfica de 8.691658591731265 hab/km².\nO município Combinado/TO possui densidade demográfica de 22.277889111556444 hab/km².\nO município Conceição do Tocantins/TO possui densidade demográfica de 1.6723049977206748 hab/km².\nO município Couto Magalhães/TO possui densidade demográfica de 3.1586780090680358 hab/km².\nO município Cristalândia/TO possui densidade demográfica de 3.913993853612085 hab/km².\nO município Crixás do Tocantins/TO possui densidade demográfica de 1.5850976497177431 hab/km².\nO município Darcinópolis/TO possui densidade demográfica de 3.2168915786134358 hab/km².\nO município Dianópolis/TO possui densidade demográfica de 5.940366330878902 hab/km².\nO município Divinópolis do Tocantins/TO possui densidade demográfica de 2.7106239589678927 hab/km².\nO município Dois Irmãos do Tocantins/TO possui densidade demográfica de 1.9060217618124906 hab/km².\nO município Dueré/TO possui densidade demográfica de 1.3407886476780004 hab/km².\nO município Esperantina/TO possui densidade demográfica de 18.800841236458872 hab/km².\nO município Fátima/TO possui densidade demográfica de 9.937060928155441 hab/km².\nO município Figueirópolis/TO possui densidade demográfica de 2.7667390301906147 hab/km².\nO município Filadélfia/TO possui densidade demográfica de 4.277996861293309 hab/km².\nO município Formoso do Araguaia/TO possui densidade demográfica de 1.3727541051508638 hab/km².\nO município Fortaleza do Tabocão/TO possui densidade demográfica de 3.8918205804749344 hab/km².\nO município Goianorte/TO possui densidade demográfica de 2.7518351119945805 hab/km².\nO município Goiatins/TO possui densidade demográfica de 1.8824704303592048 hab/km².\nO município Guaraí/TO possui densidade demográfica de 10.228555304740407 hab/km².\nO município Gurupi/TO possui densidade demográfica de 41.803506364067125 hab/km².\nO município Ipueiras/TO possui densidade demográfica de 2.010426249616682 hab/km².\nO município Itacajá/TO possui densidade demográfica de 2.328142205442819 hab/km².\nO município Itaguatins/TO possui densidade demográfica de 8.148949111306345 hab/km².\nO município Itapiratins/TO possui densidade demográfica de 2.839319592269848 hab/km².\nO município Itaporã do Tocantins/TO possui densidade demográfica de 2.512898517955148 hab/km².\nO município Jaú do Tocantins/TO possui densidade demográfica de 1.6138607026989713 hab/km².\nO município Juarina/TO possui densidade demográfica de 4.637771541419811 hab/km².\nO município Lagoa da Confusão/TO possui densidade demográfica de 0.9664295869436405 hab/km².\nO município Lagoa do Tocantins/TO possui densidade demográfica de 3.8679307393508457 hab/km².\nO município Lajeado/TO possui densidade demográfica de 8.598716239263233 hab/km².\nO município Lavandeira/TO possui densidade demográfica de 3.088855102865611 hab/km².\nO município Lizarda/TO possui densidade demográfica de 0.650856247258978 hab/km².\nO município Luzinópolis/TO possui densidade demográfica de 9.37902418085563 hab/km².\nO município Marianópolis do Tocantins/TO possui densidade demográfica de 2.080932594423751 hab/km².\nO município Mateiros/TO possui densidade demográfica de 0.22961412844756887 hab/km².\nO município Maurilândia do Tocantins/TO possui densidade demográfica de 4.273075828806005 hab/km².\nO município Miracema do Tocantins/TO possui densidade demográfica de 7.787386722588466 hab/km².\nO município Miranorte/TO possui densidade demográfica de 12.236094686027803 hab/km².\nO município Monte do Carmo/TO possui densidade demográfica de 1.8569568138646877 hab/km².\nO município Monte Santo do Tocantins/TO possui densidade demográfica de 1.9101277999175486 hab/km².\nO município Muricilândia/TO possui densidade demográfica de 2.6562170817005857 hab/km².\nO município Natividade/TO possui densidade demográfica de 2.7771606309708954 hab/km².\nO município Nazaré/TO possui densidade demográfica de 11.078275365613397 hab/km².\nO município Nova Olinda/TO possui densidade demográfica de 6.8229705397846985 hab/km².\nO município Nova Rosalândia/TO possui densidade demográfica de 7.301814801185335 hab/km².\nO município Novo Acordo/TO possui densidade demográfica de 1.4065233971914397 hab/km².\nO município Novo Alegre/TO possui densidade demográfica de 11.424287856071965 hab/km².\nO município Novo Jardim/TO possui densidade demográfica de 1.8760451106003802 hab/km².\nO município Oliveira de Fátima/TO possui densidade demográfica de 5.037648773378674 hab/km².\nO município Palmas/TO possui densidade demográfica de 102.90138534615627 hab/km².\nO município Palmeirante/TO possui densidade demográfica de 1.8759324755189675 hab/km².\nO município Palmeiras do Tocantins/TO possui densidade demográfica de 7.674822837277711 hab/km².\nO município Palmeirópolis/TO possui densidade demográfica de 4.307076540253765 hab/km².\nO município Paraíso do Tocantins/TO possui densidade demográfica de 35.02752235698627 hab/km².\nO município Paranã/TO possui densidade demográfica de 0.9181001064811403 hab/km².\nO município Pau d`Arco/TO possui densidade demográfica de 3.3308891325023047 hab/km².\nO município Pedro Afonso/TO possui densidade demográfica de 5.738226664677508 hab/km².\nO município Peixe/TO possui densidade demográfica de 1.962500070872258 hab/km².\nO município Pequizeiro/TO possui densidade demográfica de 4.177550008265829 hab/km².\nO município Pindorama do Tocantins/TO possui densidade demográfica de 2.890147457811929 hab/km².\nO município Piraquê/TO possui densidade demográfica de 2.1351116180782537 hab/km².\nO município Pium/TO possui densidade demográfica de 0.6684781686054929 hab/km².\nO município Ponte Alta do Bom Jesus/TO possui densidade demográfica de 2.5158625577197777 hab/km².\nO município Ponte Alta do Tocantins/TO possui densidade demográfica de 1.106124819561463 hab/km².\nO município Porto Alegre do Tocantins/TO possui densidade demográfica de 5.571274857529988 hab/km².\nO município Porto Nacional/TO possui densidade demográfica de 11.044243492017834 hab/km².\nO município Praia Norte/TO possui densidade demográfica de 26.497145822522054 hab/km².\nO município Presidente Kennedy/TO possui densidade demográfica de 4.777913345967135 hab/km².\nO município Pugmil/TO possui densidade demográfica de 5.895527959584899 hab/km².\nO município Recursolândia/TO possui densidade demográfica de 1.6998547364052223 hab/km².\nO município Riachinho/TO possui densidade demográfica de 8.098863724201902 hab/km².\nO município Rio da Conceição/TO possui densidade demográfica de 2.177558694989328 hab/km².\nO município Rio dos Bois/TO possui densidade demográfica de 3.04116818725076 hab/km².\nO município Rio Sono/TO possui densidade demográfica de 0.9842045710274976 hab/km².\nO município Sampaio/TO possui densidade demográfica de 17.382698277025508 hab/km².\nO município Sandolândia/TO possui densidade demográfica de 0.942578118357885 hab/km².\nO município Santa Fé do Araguaia/TO possui densidade demográfica de 3.932447008205758 hab/km².\nO município Santa Maria do Tocantins/TO possui densidade demográfica de 2.051812883740056 hab/km².\nO município Santa Rita do Tocantins/TO possui densidade demográfica de 0.6497809126856899 hab/km².\nO município Santa Rosa do Tocantins/TO possui densidade demográfica de 2.543061694854865 hab/km².\nO município Santa Tereza do Tocantins/TO possui densidade demográfica de 4.673001055731511 hab/km².\nO município Santa Terezinha do Tocantins/TO possui densidade demográfica de 9.173835657075053 hab/km².\nO município São Bento do Tocantins/TO possui densidade demográfica de 4.166742020074148 hab/km².\nO município São Félix do Tocantins/TO possui densidade demográfica de 0.7528763333822327 hab/km².\nO município São Miguel do Tocantins/TO possui densidade demográfica de 26.280026076926934 hab/km².\nO município São Salvador do Tocantins/TO possui densidade demográfica de 2.046370329739879 hab/km².\nO município São Sebastião do Tocantins/TO possui densidade demográfica de 14.908799777220832 hab/km².\nO município São Valério/TO possui densidade demográfica de 1.7395687393583876 hab/km².\nO município Silvanópolis/TO possui densidade demográfica de 4.025960614221142 hab/km².\nO município Sítio Novo do Tocantins/TO possui densidade demográfica de 28.224985344481812 hab/km².\nO município Sucupira/TO possui densidade demográfica de 1.6986504407520087 hab/km².\nO município Taguatinga/TO possui densidade demográfica de 6.175022565028309 hab/km².\nO município Taipas do Tocantins/TO possui densidade demográfica de 1.7425192617810428 hab/km².\nO município Talismã/TO possui densidade demográfica de 1.1878158468171913 hab/km².\nO município Tocantínia/TO possui densidade demográfica de 2.589175891758918 hab/km².\nO município Tocantinópolis/TO possui densidade demográfica de 21.000492075723955 hab/km².\nO município Tupirama/TO possui densidade demográfica de 2.21002232487609 hab/km².\nO município Tupiratins/TO possui densidade demográfica de 2.34220549306944 hab/km².\nO município Wanderlândia/TO possui densidade demográfica de 7.997465514981138 hab/km².\nO município Xambioá/TO possui densidade demográfica de 9.67945854369832 hab/km².\n"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
]
] |
e7e9bbf6384933d5305e46d6aca754c8a849141a | 135,201 | ipynb | Jupyter Notebook | stock_LSTM.ipynb | purohitn/SNAS-Series | 836668a0567deff26ba34b81393b7a599d75b04e | [
"MIT"
] | null | null | null | stock_LSTM.ipynb | purohitn/SNAS-Series | 836668a0567deff26ba34b81393b7a599d75b04e | [
"MIT"
] | null | null | null | stock_LSTM.ipynb | purohitn/SNAS-Series | 836668a0567deff26ba34b81393b7a599d75b04e | [
"MIT"
] | null | null | null | 190.692525 | 57,962 | 0.870792 | [
[
[
"<a href=\"https://colab.research.google.com/github/purohitn/SNAS-Series/blob/SNAS/stock_LSTM.ipynb\" target=\"_parent\"><img src=\"https://colab.research.google.com/assets/colab-badge.svg\" alt=\"Open In Colab\"/></a>",
"_____no_output_____"
]
],
[
[
"!pip install yfinance",
"Collecting yfinance\n Downloading yfinance-0.1.64.tar.gz (26 kB)\nRequirement already satisfied: pandas>=0.24 in /usr/local/lib/python3.7/dist-packages (from yfinance) (1.1.5)\nRequirement already satisfied: numpy>=1.15 in /usr/local/lib/python3.7/dist-packages (from yfinance) (1.19.5)\nRequirement already satisfied: requests>=2.20 in /usr/local/lib/python3.7/dist-packages (from yfinance) (2.23.0)\nRequirement already satisfied: multitasking>=0.0.7 in /usr/local/lib/python3.7/dist-packages (from yfinance) (0.0.9)\nCollecting lxml>=4.5.1\n Downloading lxml-4.6.4-cp37-cp37m-manylinux_2_17_x86_64.manylinux2014_x86_64.manylinux_2_24_x86_64.whl (6.3 MB)\n\u001b[K |████████████████████████████████| 6.3 MB 6.8 MB/s \n\u001b[?25hRequirement already satisfied: python-dateutil>=2.7.3 in /usr/local/lib/python3.7/dist-packages (from pandas>=0.24->yfinance) (2.8.2)\nRequirement already satisfied: pytz>=2017.2 in /usr/local/lib/python3.7/dist-packages (from pandas>=0.24->yfinance) (2018.9)\nRequirement already satisfied: six>=1.5 in /usr/local/lib/python3.7/dist-packages (from python-dateutil>=2.7.3->pandas>=0.24->yfinance) (1.15.0)\nRequirement already satisfied: idna<3,>=2.5 in /usr/local/lib/python3.7/dist-packages (from requests>=2.20->yfinance) (2.10)\nRequirement already satisfied: chardet<4,>=3.0.2 in /usr/local/lib/python3.7/dist-packages (from requests>=2.20->yfinance) (3.0.4)\nRequirement already satisfied: urllib3!=1.25.0,!=1.25.1,<1.26,>=1.21.1 in /usr/local/lib/python3.7/dist-packages (from requests>=2.20->yfinance) (1.24.3)\nRequirement already satisfied: certifi>=2017.4.17 in /usr/local/lib/python3.7/dist-packages (from requests>=2.20->yfinance) (2021.5.30)\nBuilding wheels for collected packages: yfinance\n Building wheel for yfinance (setup.py) ... \u001b[?25l\u001b[?25hdone\n Created wheel for yfinance: filename=yfinance-0.1.64-py2.py3-none-any.whl size=24109 sha256=648aa16c04e2fc03a3837e34afd183bb6439b84cc414c7992a0969d4f3b991cf\n Stored in directory: /root/.cache/pip/wheels/86/fe/9b/a4d3d78796b699e37065e5b6c27b75cff448ddb8b24943c288\nSuccessfully built yfinance\nInstalling collected packages: lxml, yfinance\n Attempting uninstall: lxml\n Found existing installation: lxml 4.2.6\n Uninstalling lxml-4.2.6:\n Successfully uninstalled lxml-4.2.6\nSuccessfully installed lxml-4.6.4 yfinance-0.1.64\n"
],
[
"import pandas as pd\nimport numpy as np\nimport math\nimport pandas_datareader as pdd\nfrom sklearn.preprocessing import MinMaxScaler\nfrom tensorflow import keras\nfrom tensorflow.keras.models import Sequential\nfrom tensorflow.keras.layers import Dense, LSTM\nimport matplotlib.pyplot as plt",
"_____no_output_____"
],
[
"from pandas_datareader import data as pdr\nimport yfinance as yfin\nyfin.pdr_override()\n\ndf = pdr.get_data_yahoo(\"AAPL\", start=\"2013-01-01\", end=\"2019-12-30\")\nprint(df)",
"\r[*********************100%***********************] 1 of 1 completed\n Open High Low Close Adj Close Volume\nDate \n2013-01-02 19.779285 19.821428 19.343929 19.608213 16.960691 560518000\n2013-01-03 19.567142 19.631071 19.321428 19.360714 16.746611 352965200\n2013-01-04 19.177500 19.236786 18.779642 18.821428 16.280142 594333600\n2013-01-07 18.642857 18.903570 18.400000 18.710714 16.184372 484156400\n2013-01-08 18.900356 18.996071 18.616072 18.761070 16.227928 458707200\n... ... ... ... ... ... ...\n2019-12-20 70.557503 70.662498 69.639999 69.860001 68.837494 275978000\n2019-12-23 70.132500 71.062500 70.092499 71.000000 69.960800 98572000\n2019-12-24 71.172501 71.222504 70.730003 71.067497 70.027306 48478800\n2019-12-26 71.205002 72.495003 71.175003 72.477501 71.416672 93121200\n2019-12-27 72.779999 73.492500 72.029999 72.449997 71.389572 146266000\n\n[1760 rows x 6 columns]\n"
],
[
"plt.figure(figsize=(16,8))\nplt.title('close price movement')\nplt.plot(df['Close'])\nplt.xlabel('Date', fontsize=18)\nplt.ylabel('Close Price in $', fontsize=18)\nplt.show",
"_____no_output_____"
],
[
"data = df.filter(['Close'])\ndataset = data.values\nlen(dataset)",
"_____no_output_____"
],
[
"training_data_size = math.ceil(len(dataset)*.7)\ntraining_data_size",
"_____no_output_____"
],
[
"scaler = MinMaxScaler(feature_range=(0,1))\nscaled_data = scaler.fit_transform(dataset)\nscaled_data\n",
"_____no_output_____"
],
[
"train_data = scaled_data[0:training_data_size, :]\nx_train = []\ny_train = []\nfor i in range(60, len(train_data)):\n x_train.append(train_data[i-60:i, 0])\n y_train.append(train_data[i,0])\n if i <=60:\n print(x_train)\n print(y_train)",
"[array([0.09671473, 0.09248614, 0.08327231, 0.08138073, 0.08224107,\n 0.07723146, 0.08114277, 0.07918407, 0.06786508, 0.05820581,\n 0.07051327, 0.06843253, 0.06679725, 0.06970784, 0.07534597,\n 0.03659297, 0.03011276, 0.03618413, 0.0413341 , 0.04045544,\n 0.03963779, 0.03849673, 0.03160162, 0.04107174, 0.04077275,\n 0.04740548, 0.05153035, 0.05455076, 0.04721022, 0.04666715,\n 0.04641088, 0.04248737, 0.04238361, 0.03558615, 0.03388371,\n 0.03678211, 0.03189452, 0.03565937, 0.03297453, 0.03104025,\n 0.0243709 , 0.01801275, 0.02477971, 0.02143589, 0.02443801,\n 0.02513363, 0.02888628, 0.0231261 , 0.02307729, 0.02560958,\n 0.03241927, 0.03977814, 0.03902762, 0.03755704, 0.03795366,\n 0.0435552 , 0.04457421, 0.04308535, 0.03755704, 0.03180909])]\n[0.02341900173486275]\n"
],
[
"x_train, y_train = np.array(x_train), np.array(y_train)\nx_train = np.reshape(x_train, (x_train.shape[0], x_train.shape[1], 1))\nx_train.shape",
"_____no_output_____"
],
[
"model = Sequential()\nmodel.add(LSTM(50, return_sequences=True, input_shape = (x_train.shape[1], 1)))\nmodel.add(LSTM(50, return_sequences=False))\nmodel.add(Dense(25))\nmodel.add(Dense(1))",
"_____no_output_____"
],
[
"model.compile(optimizer='adam', loss='mean_squared_error')\n",
"_____no_output_____"
],
[
"model.fit(x_train, y_train, batch_size=1, epochs=10)",
"Epoch 1/10\n1172/1172 [==============================] - 30s 19ms/step - loss: 5.3807e-04\nEpoch 2/10\n1172/1172 [==============================] - 22s 19ms/step - loss: 2.9527e-04\nEpoch 3/10\n1172/1172 [==============================] - 23s 19ms/step - loss: 1.8856e-04\nEpoch 4/10\n1172/1172 [==============================] - 23s 19ms/step - loss: 1.7879e-04\nEpoch 5/10\n1172/1172 [==============================] - 23s 19ms/step - loss: 1.4583e-04\nEpoch 6/10\n1172/1172 [==============================] - 23s 19ms/step - loss: 1.1357e-04\nEpoch 7/10\n1172/1172 [==============================] - 23s 19ms/step - loss: 1.0367e-04\nEpoch 8/10\n1172/1172 [==============================] - 22s 19ms/step - loss: 1.1844e-04\nEpoch 9/10\n1172/1172 [==============================] - 22s 19ms/step - loss: 1.1450e-04\nEpoch 10/10\n1172/1172 [==============================] - 22s 19ms/step - loss: 1.0469e-04\n"
],
[
"test_data = scaled_data[training_data_size - 60: ,:]\nx_test = []\ny_test = dataset[training_data_size:, :]\nfor i in range(60, len(test_data)):\n x_test.append(test_data[i-60:i, 0])\n",
"_____no_output_____"
],
[
"x_test = np.array(x_test)\nx_test = np.reshape(x_test, (x_test.shape[0], x_test.shape[1],1))",
"_____no_output_____"
],
[
"prediction = model.predict(x_test)\nprediction = scaler.inverse_transform(prediction)",
"_____no_output_____"
],
[
"rmse = np.sqrt(np.mean(prediction- y_test)**2)\nrmse",
"_____no_output_____"
],
[
"train = data[:training_data_size]\nvalid = data[training_data_size:]\nvalid['prediction'] = prediction\nplt.figure(figsize=(16,8))\nplt.title('Model LM')\nplt.xlabel('Date', fontsize=18)\nplt.ylabel('closeprice in $', fontsize=18)\nplt.plot(train['Close'])\nplt.plot(valid[['Close', 'prediction']])\nplt.legend(['Train', 'val', 'predictions'], loc='lower right')\nplt.show",
"/usr/local/lib/python3.7/dist-packages/ipykernel_launcher.py:3: SettingWithCopyWarning: \nA value is trying to be set on a copy of a slice from a DataFrame.\nTry using .loc[row_indexer,col_indexer] = value instead\n\nSee the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy\n This is separate from the ipykernel package so we can avoid doing imports until\n"
],
[
"valid",
"_____no_output_____"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7e9d87e140d26e8f2666874707b7e2ac18ae5d1 | 619,090 | ipynb | Jupyter Notebook | Write a data science blog post.ipynb | audichandra/Heart_Disease_UCI | 6321f870f2c7ecce99883b2e37cb97dc125d223e | [
"CNRI-Python"
] | null | null | null | Write a data science blog post.ipynb | audichandra/Heart_Disease_UCI | 6321f870f2c7ecce99883b2e37cb97dc125d223e | [
"CNRI-Python"
] | null | null | null | Write a data science blog post.ipynb | audichandra/Heart_Disease_UCI | 6321f870f2c7ecce99883b2e37cb97dc125d223e | [
"CNRI-Python"
] | null | null | null | 218.066221 | 75,408 | 0.893305 | [
[
[
"# Heart Disease UCI#\n\nFor this blog project, I decided to use the the heart disease UCI dataset taken from https://www.kaggle.com/ronitf/heart-disease-uci which is already a reprocessed data from UCI machine learning repository. This dataset have been taken back from 1988 and consisted of patients with admitted heart disease and chest pain symptoms from Cleveland. \n\nThe reason that I used this dataset is because there is a family member of my friend admitted to hospital due to heart disease symptoms and I wanted to tried to test some of the common myths about heart disease to this dataset: \n1. The common gender and ages of having heart disease \n2. The relationship of blood pressure and heart rate in having heart disease \n3. The symptoms of the heart attacks is whether known or not\n4. The through heart exam result such as ECG, Fluoroscopy and Thallium \n5. The most important features in predicting either a person has heart disease or not \n",
"_____no_output_____"
],
[
"This project will follow CRISP-DM(Cross Industry Process for Data Mining) guidelines:\n\n1. Business Understanding\n2. Data Understanding\n3. Data Preparation\n4. Modeling \n5. Evaluation\n6. Deployment \n\n## 1. Business Understanding ##\n\nIn this section, we will ask the business or real-world application questions regarding the data: \n1. Does age and gender of the patients have certain trends in determining whether the patient have heart disease or not? \n2. What is the relationship between blood pressure and heart rate in having heart disease? \n3. Do we know the symptom of the heart attacks such as chest pain caused by heart disease? \n4. What is the trend of the cardiological test such as ECG, Fluoroscopy and Thallium? \n5. Out of 14 predicting features, what are the important features in predicting whether the patient have heart disease or not? (by using machine learning) ",
"_____no_output_____"
],
[
"## 2. Data Understanding##\nWe will try to get more understandings about the data in this part: ",
"_____no_output_____"
]
],
[
[
"import numpy as np\nimport pandas as pd\nimport matplotlib.pyplot as plt\nimport seaborn as sb \nfrom sklearn.ensemble import RandomForestClassifier \nfrom sklearn.tree import DecisionTreeClassifier\nfrom sklearn.tree import export_graphviz \nfrom sklearn.metrics import roc_curve, auc \nfrom sklearn.metrics import classification_report \nfrom sklearn.metrics import confusion_matrix \nfrom sklearn.model_selection import train_test_split, GridSearchCV\nfrom sklearn.metrics import accuracy_score, precision_score, recall_score, fbeta_score, f1_score\nfrom sklearn.linear_model import LogisticRegression \nfrom sklearn.linear_model import SGDClassifier\nfrom sklearn.metrics import make_scorer\nfrom sklearn.base import clone\nfrom time import time\nimport visuals as vs\nimport warnings\nwarnings.filterwarnings('ignore')\nnp.random.seed(56) ",
"_____no_output_____"
],
[
"df = pd.read_csv('heart.csv')",
"_____no_output_____"
],
[
"df.head()",
"_____no_output_____"
]
],
[
[
"Features: \n1. **age**: Patient age in years when admitted\n2. **sex**: Patient's gender (female=0, male=1)\t\n3. **cp**: Type of chest pain that patient felt\t(typical angina, atypical angina, non-angina, or asymptomatic angina)\n4. **trestbps**: Patient's resting blood pressure (mm Hg)\n5. **chol**: Patient's cholesterol level (mg/dl)\n6. **fbs**: Patient's fasting blood sugar level (< 120 mg/dl or > 120 mg/dl)\n7. **restecg**: Patient's resting electrocardiogram results (normal, ST-T wave abnormality, or left ventricular hypertrophy)\n8. **thalach**: Patient's maximum heart rate achieved during thalium stress test\n9. **exang**: Patient's exercise will induce angina or not (yes or no)\n10. **oldpeak**: ST depression (part of electrocardiogram between point S and point T) induced by exercise relative to rest\t\n11.\t**slope**:\tSlope of peak exercise ST segment (upsloping, flat, or downsloping)\t\n12. **ca**:\tNumber of major heart vessels colored by fluoroscopy \n13.\t**thal**: Thalium nuclear test result (normal, fixed defect, or reversible defect)\n14.\t**target**: Whether the patient have the hearts disease or not (disease=0, healthy=1)\n\nAfter knowing the data a little bit, we will wrangle all of the data in order to make it ready to be used in analysis.",
"_____no_output_____"
],
[
"## 3. Data Preparation ##\n\nThe process that we will do in this step are: \n1. Renaming some of the columns and their values so that it become easier to work around with \n2. Determining the impossible values as well as outlier \n3. Analyzing all of the questions that can be answered within the data without modeling",
"_____no_output_____"
]
],
[
[
"df.columns = ['age', 'gender', 'chest_pain', 'blood_pressure', 'cholesterol', 'blood_sugar', 'restecg', 'max_heart_rate',\n 'exang', 'st_depression', 'st_slope', 'major_vessels', 'thallium', 'result']",
"_____no_output_____"
],
[
"df.head()",
"_____no_output_____"
],
[
"df.describe()",
"_____no_output_____"
],
[
"df.isnull().sum()",
"_____no_output_____"
],
[
"df.info()",
"<class 'pandas.core.frame.DataFrame'>\nRangeIndex: 303 entries, 0 to 302\nData columns (total 14 columns):\nage 303 non-null int64\ngender 303 non-null int64\nchest_pain 303 non-null int64\nblood_pressure 303 non-null int64\ncholesterol 303 non-null int64\nblood_sugar 303 non-null int64\nrestecg 303 non-null int64\nmax_heart_rate 303 non-null int64\nexang 303 non-null int64\nst_depression 303 non-null float64\nst_slope 303 non-null int64\nmajor_vessels 303 non-null int64\nthallium 303 non-null int64\nresult 303 non-null int64\ndtypes: float64(1), int64(13)\nmemory usage: 33.2 KB\n"
],
[
"df['gender'].value_counts()",
"_____no_output_____"
],
[
"df['chest_pain'].value_counts()",
"_____no_output_____"
],
[
"df['blood_sugar'].value_counts()",
"_____no_output_____"
],
[
"df['restecg'].value_counts()",
"_____no_output_____"
],
[
"df['exang'].value_counts()",
"_____no_output_____"
],
[
"df['st_slope'].value_counts()",
"_____no_output_____"
],
[
"df['major_vessels'].value_counts()",
"_____no_output_____"
],
[
"df['thallium'].value_counts()",
"_____no_output_____"
],
[
"df['result'].value_counts()",
"_____no_output_____"
],
[
"df.loc[df.gender == 0, 'gender'] = 'female'\ndf.loc[df.gender == 1, 'gender'] = 'male'\n\ndf.loc[df.chest_pain == 0, 'chest_pain'] = 'asymptomatic'\ndf.loc[df.chest_pain == 1, 'chest_pain'] = 'atypical_angina'\ndf.loc[df.chest_pain == 2, 'chest_pain'] = 'non_anginal_pain'\ndf.loc[df.chest_pain == 3, 'chest_pain'] = 'typical_angina'\n\ndf.loc[df.blood_sugar == 0, 'blood_sugar'] = '<120mg/ml'\ndf.loc[df.blood_sugar == 1, 'blood_sugar'] = '>=120mg/ml'\n\ndf.loc[df.restecg == 0, 'restecg'] = 'left_ventricular_hypertrophy'\ndf.loc[df.restecg == 1, 'restecg'] = 'normal'\ndf.loc[df.restecg == 2, 'restecg'] = 'ST-T_wave_abnormality'\n\ndf.loc[df.exang == 0, 'exang'] = 'no'\ndf.loc[df.exang == 1, 'exang'] = 'yes'\n\ndf.loc[df.st_slope == 0, 'st_slope'] = 'downsloping'\ndf.loc[df.st_slope == 1, 'st_slope'] = 'flat'\ndf.loc[df.st_slope == 2, 'st_slope'] = 'upsloping'\n\ndf.loc[df.thallium == 1, 'thallium'] = 'fixed_defect'\ndf.loc[df.thallium == 2, 'thallium'] = 'normal'\ndf.loc[df.thallium == 3, 'thallium'] = 'reversible_defect'",
"_____no_output_____"
],
[
"df['gender'].value_counts()",
"_____no_output_____"
],
[
"df.describe()",
"_____no_output_____"
]
],
[
[
"There are some impossible values for the major vessels and thallium test results. For the major vessels column, it have value of 4 where it should be Nan in its original datasets. While for the thallium column, it also have a value of 0 which should have been nan in its original dataset. ",
"_____no_output_____"
]
],
[
[
"indexNames = df[ (df['major_vessels'] == 4) | (df['thallium'] == 0) ].index\ndf= df.drop(indexNames)",
"_____no_output_____"
],
[
"df['thallium'].value_counts()",
"_____no_output_____"
],
[
"indexNames",
"_____no_output_____"
],
[
"df.info()",
"<class 'pandas.core.frame.DataFrame'>\nInt64Index: 296 entries, 0 to 302\nData columns (total 14 columns):\nage 296 non-null int64\ngender 296 non-null object\nchest_pain 296 non-null object\nblood_pressure 296 non-null int64\ncholesterol 296 non-null int64\nblood_sugar 296 non-null object\nrestecg 296 non-null object\nmax_heart_rate 296 non-null int64\nexang 296 non-null object\nst_depression 296 non-null float64\nst_slope 296 non-null object\nmajor_vessels 296 non-null int64\nthallium 296 non-null object\nresult 296 non-null int64\ndtypes: float64(1), int64(6), object(7)\nmemory usage: 34.7+ KB\n"
]
],
[
[
"After dealing with wrong values, we will search for the outlier for the part with continuous value",
"_____no_output_____"
]
],
[
[
"sb.boxplot(data = df, y = 'blood_pressure') ",
"_____no_output_____"
],
[
"sb.boxplot(data = df, y = 'cholesterol') ",
"_____no_output_____"
],
[
"sb.boxplot(data = df, y = 'max_heart_rate') ",
"_____no_output_____"
],
[
"sb.boxplot(data = df, y = 'st_depression') ",
"_____no_output_____"
]
],
[
[
"We can see that there is some outliers especially for blood_pressure, cholesterol and st_depression which we decide to get rid of by knowing the threshold first ",
"_____no_output_____"
]
],
[
[
"q1 = df['blood_pressure'].quantile(0.25)\nq3 = df['blood_pressure'].quantile(0.75)\niqr = q3-q1 \nfence_low = q1-1.5*iqr\nfence_high = q3+1.5*iqr\nfence_high, fence_low",
"_____no_output_____"
],
[
"q1 = df['cholesterol'].quantile(0.25)\nq3 = df['cholesterol'].quantile(0.75)\niqr = q3-q1 \nfence_low = q1-1.5*iqr\nfence_high = q3+1.5*iqr\nfence_high, fence_low",
"_____no_output_____"
],
[
"q1 = df['st_depression'].quantile(0.25)\nq3 = df['st_depression'].quantile(0.75)\niqr = q3-q1 \nfence_low = q1-1.5*iqr\nfence_high = q3+1.5*iqr\nfence_high, fence_low",
"_____no_output_____"
]
],
[
[
"After knowing the outlier threshold, we will drop all of the value that exceed the high fence ",
"_____no_output_____"
]
],
[
[
"indexNames2 = df[ (df['blood_pressure'] >= 170) | (df['cholesterol'] >= 371.625) | (df['st_depression'] >= 4.125) ].index\ndf2= df.drop(indexNames2)",
"_____no_output_____"
],
[
"df2.info()",
"<class 'pandas.core.frame.DataFrame'>\nInt64Index: 274 entries, 0 to 302\nData columns (total 14 columns):\nage 274 non-null int64\ngender 274 non-null object\nchest_pain 274 non-null object\nblood_pressure 274 non-null int64\ncholesterol 274 non-null int64\nblood_sugar 274 non-null object\nrestecg 274 non-null object\nmax_heart_rate 274 non-null int64\nexang 274 non-null object\nst_depression 274 non-null float64\nst_slope 274 non-null object\nmajor_vessels 274 non-null int64\nthallium 274 non-null object\nresult 274 non-null int64\ndtypes: float64(1), int64(6), object(7)\nmemory usage: 32.1+ KB\n"
],
[
"sb.boxplot(data = df2, y = 'blood_pressure') ",
"_____no_output_____"
],
[
"sb.boxplot(data = df2, y = 'cholesterol') ",
"_____no_output_____"
],
[
"sb.boxplot(data = df2, y = 'st_depression') ",
"_____no_output_____"
]
],
[
[
"In this section, we will analyze the required features and the methods in answering the questions \n\n### 1. Does age and gender of the patients have certain trends in determining whether the patient have heart disease or not?### \n\nThe needed features for this question are age, gender and result columns. ",
"_____no_output_____"
]
],
[
[
"age_distribution = df2.groupby([\"age\", \"result\"]).size().reset_index()",
"_____no_output_____"
],
[
"plt.figure(figsize=(20,10))\nmy_palette = {0:'#3498db', 1:'#e74c3c'}\nax = sb.barplot(x='age', y=0, hue='result', palette=my_palette, data=age_distribution)\nplt.title('Result Distribution for each of age classes', fontsize=22, y=1.015)\nplt.xlabel('Age', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 14)\nplt.yticks(size = 14)\nleg = ax.legend(loc='center right', bbox_to_anchor=(1, 0.5), prop={'size':16})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"We can see that as the patient get older, the possibility of having heart disease is higher especially at the age of 60s ",
"_____no_output_____"
]
],
[
[
"plt.figure(figsize=(16,8))\nax = sb.boxplot(x='gender', y='age', hue='result', linewidth=11, data= df2)\nplt.title('Heart Test results distribution based on Age and Gender', fontsize=22, y=1.015)\nplt.xlabel('Gender', fontsize=16, labelpad=16)\nplt.ylabel('Age', fontsize=16, labelpad=16)\nplt.xticks(size = 14)\nplt.yticks(size = 14)\nleg = ax.legend(loc='upper right', prop={'size':13})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"As we can see that the male have wider distribution (30s-80s) among ages for the one that has heart disease compared to the female age distribution (50s-65s)",
"_____no_output_____"
],
[
"### 2. What is the relationship between blood pressure and heart rate in having heart disease?### \n\nThe features in this question will be blood pressure and max heart rate as well as result column ",
"_____no_output_____"
]
],
[
[
"df_d = df2[df2['result'] == 0]",
"_____no_output_____"
],
[
"df_nd = df2[df2['result'] == 1]",
"_____no_output_____"
],
[
"plt.figure(figsize=(16,8))\nplt.hist(df_d['max_heart_rate'], alpha=0.5, label='disease')\nplt.hist(df_nd['max_heart_rate'], alpha=0.5, label='healthy')\nplt.title('Max Heart Rate Distribution based on heart disease', fontsize=22, y=1.015)\nplt.xlabel('Max Heart Rate', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 14)\nplt.yticks(size = 14)\nplt.legend(loc='upper right', prop={'size':13})\nplt.show()",
"_____no_output_____"
]
],
[
[
"We can see that the one with healthy hearts have stronger maximum heart rate. ",
"_____no_output_____"
]
],
[
[
"plt.figure(figsize=(16,8))\nplt.hist(df_d['blood_pressure'], alpha=0.5, label='disease')\nplt.hist(df_nd['blood_pressure'], alpha=0.5, label='healthy')\nplt.title('Blood Pressure Distribution based on heart disease', fontsize=22, y=1.015)\nplt.xlabel('Blood Pressure', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 14)\nplt.yticks(size = 14)\nplt.legend(loc='upper right', prop={'size':13})\nplt.show()",
"_____no_output_____"
]
],
[
[
"there are no special trait that can be distinguished between the healthy and sick patients in their blood pressure section; however, we can see that healthy patient have lower blood pressure compared to patient with heart disease. ",
"_____no_output_____"
]
],
[
[
"plt.figure(figsize=(20,10))\nax = sb.scatterplot(x='blood_pressure', y='max_heart_rate', hue='result', data=df2)\nplt.title('The Relationship between Blood Pressure and Max Heart Rate', fontsize=22, y=1.015)\nplt.xlabel('Blood Pressure', fontsize=16, labelpad=16)\nplt.ylabel('Max Heart Rate', fontsize=16, labelpad=16)\nplt.xticks(size = 14)\nplt.yticks(size = 14)\nleg = ax.legend(loc='center right', bbox_to_anchor=(1, 0.5), prop={'size':13})\nnew_labels = [\"Result\", \"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"As the graph indicated, the lower half of the max heart rate is filled with sick patient which have lower max heart rate. we can also see that the blood pressure of healthy patient is also lower compared to sick patient which is indicated by the left side of this graph. \n\n### 3. Do we know the symptom of the heart attacks such as chest pain caused by heart disease?### \n\nFor this question, we will check remaining features that might tell us more about the symptoms of chest pain",
"_____no_output_____"
]
],
[
[
"chest_distribution = df2.groupby([\"chest_pain\", \"result\"]).size().reset_index()",
"_____no_output_____"
],
[
"plt.figure(figsize=(20,10))\nmy_palette = {0:'#3498db', 1:'#e74c3c'}\nax = sb.barplot(x='chest_pain', y=0, hue='result', palette=my_palette, data=chest_distribution)\nplt.title('Result Distribution according to Chest Pain types', fontsize=22, y=1.015)\nplt.xlabel('Chest Pain type', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 14)\nplt.yticks(size = 14)\nleg = ax.legend(loc='center right', bbox_to_anchor=(1, 0.5), prop={'size':14})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"Angina is a type of chest pain which is usually caused by the reduction of amount of blood that is pumped to heart, thus, making the heart work harder. There are four categories of angina: \n1. [asymptomatic](https://www.health.harvard.edu/heart-health/angina-and-its-silent-cousin): The type of angina that has no symptoms which can only be detected by electrocardiogram \n2. [atypical angina](https://www.timeofcare.com/typical-vs-atypical-chest-pain/): the type of angina which is categorized as probable angina (usually only consist two out of three classifications: Chest pain or discomfort symptoms, caused by exhaustion or emotional stress, can be relieved by rest or nitroglycerin) \n3. [non_anginal_pain](https://www.timeofcare.com/typical-vs-atypical-chest-pain/): the type of angina which is categorized as non-ischemic chest discomfort which only meets one or none of the classifications above\n4. [typical_angina](https://www.harringtonhospital.org/typical-and-atypical-angina-what-to-look-for/): the type of angina is categorized as definite angina which meets all three requirements above\n\nSo we can see that the asymptomatic angina is the one with the highest count among all other angina. According to [this](https://www.health.harvard.edu/heart-health/angina-and-its-silent-cousin) source, asymptomatic also one of the most dangerous type of angina since there is no symptoms. ",
"_____no_output_____"
]
],
[
[
"exang_distribution = df2.groupby([\"exang\", \"result\"]).size().reset_index()",
"_____no_output_____"
],
[
"plt.figure(figsize=(20,10))\nmy_palette = {0:'#3498db', 1:'#e74c3c'}\nax = sb.barplot(x='exang', y=0, hue='result', palette=my_palette, data=exang_distribution)\nplt.title('Result Distribution according to Exercise Induced Angina', fontsize=22, y=1.015)\nplt.xlabel('Exercise Induced Angina', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 16)\nplt.yticks(size = 16)\nleg = ax.legend(loc='left', bbox_to_anchor=(1, 0.5), prop={'size':16})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"Exercise induced angina means that when the patient exercise, they have the possibility in triggering angina which of course is in line with the logic that those with heart disease have higher possibility in triggering agina when exercise. ",
"_____no_output_____"
]
],
[
[
"plt.figure(figsize=(16,8))\nplt.hist(df_d['cholesterol'], alpha=0.5, label='disease')\nplt.hist(df_nd['cholesterol'], alpha=0.5, label='healthy')\nplt.title('Cholesterol Distribution based on heart disease', fontsize=22, y=1.015)\nplt.xlabel('Cholesterol', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 14)\nplt.yticks(size = 14)\nplt.legend(loc='upper right', prop={'size':13})\nplt.show()",
"_____no_output_____"
]
],
[
[
"We can see that there are no significant differences between the one with heart disease and the one with healthy heart as they are both have even distributions ",
"_____no_output_____"
]
],
[
[
"sugar_distribution = df2.groupby([\"blood_sugar\", \"result\"]).size().reset_index()",
"_____no_output_____"
],
[
"plt.figure(figsize=(20,10))\nmy_palette = {0:'#3498db', 1:'#e74c3c'}\nax = sb.barplot(x='blood_sugar', y=0, hue='result', palette=my_palette, data=sugar_distribution)\nplt.title('Result Distribution according to Blood Sugar', fontsize=22, y=1.015)\nplt.xlabel('Blood Sugar', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 16)\nplt.yticks(size = 16)\nleg = ax.legend(loc='center right', bbox_to_anchor=(1, 0.5), prop={'size':16})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"Blood sugar plot also indicated that there is no special trend, the same as cholesterol distribution plot ",
"_____no_output_____"
],
[
"### 4. What is the trend of the cardiological test such as ECG, Fluoroscopy and Thallium?### \nAfter knowing the symptoms, we will try to analyze the trend in the cardiological tests results",
"_____no_output_____"
]
],
[
[
"ecg_distribution = df2.groupby([\"restecg\", \"result\"]).size().reset_index()",
"_____no_output_____"
],
[
"plt.figure(figsize=(20,10))\nmy_palette = {0:'#3498db', 1:'#e74c3c'}\nax = sb.barplot(x='restecg', y=0, hue='result', palette=my_palette, data=ecg_distribution)\nplt.title('Result Distribution according to Electrocardiogram results', fontsize=22, y=1.015)\nplt.xlabel('Electrocardiogram results', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 16)\nplt.yticks(size = 16)\nleg = ax.legend(loc='left', bbox_to_anchor=(1, 0.5), prop={'size':16})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"So according to this graph of ecg results, the main findings from this are a lot of the patient with heart disease found out that they are suffering from left venticular hypertrophy (enlargement and thickening of the heart pumping chamber wall). ",
"_____no_output_____"
]
],
[
[
"plt.figure(figsize=(16,8))\nplt.hist(df_d['st_depression'], alpha=0.5, label='disease')\nplt.hist(df_nd['st_depression'], alpha=0.5, label='healthy')\nplt.title('ST Depression Distribution based on heart disease', fontsize=22, y=1.015)\nplt.xlabel('ST Depression', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 16)\nplt.yticks(size = 16)\nplt.legend(loc='upper right', prop={'size':16})\nplt.show()",
"_____no_output_____"
]
],
[
[
"[ST depression](https://litfl.com/st-segment-ecg-library/) refers to the fluctuation of ST segments in the electrocardiogram where the higher the ST depressions (mV) there is higher chance that the supply of blood to hearts is lesser, thus, higher chance of heart failing. This explanation is in line with the graph where people with heart disease have higher ST depression.",
"_____no_output_____"
]
],
[
[
"st_distribution = df2.groupby([\"st_slope\", \"result\"]).size().reset_index()",
"_____no_output_____"
],
[
"plt.figure(figsize=(20,10))\nmy_palette = {0:'#3498db', 1:'#e74c3c'}\nax = sb.barplot(x='st_slope', y=0, hue='result', palette=my_palette, data=st_distribution)\nplt.title('Result Distribution according to Slope of peak exercise ST segment', fontsize=22, y=1.015)\nplt.xlabel('ST Segments', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 16)\nplt.yticks(size = 16)\nleg = ax.legend(loc='left', bbox_to_anchor=(1, 0.5), prop={'size':16})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"In this graph, we measure the slope of the peak during ST segments. According to this [source](https://litfl.com/st-segment-ecg-library/), the flat or downsloping leads to myocardial ischaemia at certain threshold which is in line with this finding of the graph (upsloping only considered dangerous at certain threshold and combined with other tests) ",
"_____no_output_____"
]
],
[
[
"vessel_distribution = df2.groupby([\"major_vessels\", \"result\"]).size().reset_index()",
"_____no_output_____"
],
[
"plt.figure(figsize=(20,10))\nmy_palette = {0:'#3498db', 1:'#e74c3c'}\nax = sb.barplot(x='major_vessels', y=0, hue='result', palette=my_palette, data=vessel_distribution)\nplt.title('Result Distribution according to number of major vessels colored by fluoroscopy', fontsize=22, y=1.015)\nplt.xlabel('Number of major vessels', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 16)\nplt.yticks(size = 16)\nleg = ax.legend(loc='left', bbox_to_anchor=(1, 0.5), prop={'size':16})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"[Fluoroscopy](https://www.fda.gov/radiation-emitting-products/medical-x-ray-imaging/fluoroscopy) is a test which used to examine the hearts by using the material itself in order to provide continuous imaging of the examined area. The more vessels are colored by the fluoroscopy, the more troubled major vessels in the hearts of the patient. ",
"_____no_output_____"
]
],
[
[
"thallium_distribution = df2.groupby([\"thallium\", \"result\"]).size().reset_index()",
"_____no_output_____"
],
[
"plt.figure(figsize=(20,10))\nmy_palette = {0:'#3498db', 1:'#e74c3c'}\nax = sb.barplot(x='thallium', y=0, hue='result', palette=my_palette, data=thallium_distribution)\nplt.title('Result Distribution according to Thallium nuclear test', fontsize=22, y=1.015)\nplt.xlabel('Thallium Results', fontsize=16, labelpad=16)\nplt.ylabel('Count', fontsize=16, labelpad=16)\nplt.xticks(size = 16)\nplt.yticks(size = 16)\nleg = ax.legend(loc='left', bbox_to_anchor=(1, 0.5), prop={'size':16})\nnew_title = 'Result'\nleg.set_title(new_title, prop={'size':16})\nnew_labels = [\"Disease\", \"Healthy\"]\nfor t, l in zip(leg.texts, new_labels): t.set_text(l)\nax = plt.gca()",
"_____no_output_____"
]
],
[
[
"[Thallium nuclear test](https://www.jaocr.org/articles/review-of-spect-myocardial-perfusion-imaging) is the the type of test which patient is injected with thallium radioactive dye and the dye is picked up with the sensor and translated into images. There are three types of result: fixed defect (a type of perfusion defect which can be detected during stress and rest) , normal and reversible defect(a type of perfusion defect which improves at rest). a lot of patient is admitted with heart disease have reversible defect ",
"_____no_output_____"
],
[
"## 4. Modeling ## \n### 5. Out of 14 predicting features, what are the important features in predicting whether the patient have heart disease or not? (by using machine learning) ?### \n\nFor the last question, we will use machine learning algorithm in order to know which features will have the most weight in predicting whether the patient have heart disease or not",
"_____no_output_____"
]
],
[
[
"df3 = pd.get_dummies(df2, drop_first=True)",
"_____no_output_____"
],
[
"df3.head()",
"_____no_output_____"
],
[
"# Split the 'features' and 'income' data into training and testing sets\nX_train, X_test, y_train, y_test = train_test_split(df3.drop('result', 1), \n df3['result'], \n test_size = 0.3, \n random_state = 56, shuffle=True)\n\n# Show the results of the split\nprint(\"Training set has {} samples.\".format(X_train.shape[0]))\nprint(\"Testing set has {} samples.\".format(X_test.shape[0]))",
"Training set has 191 samples.\nTesting set has 83 samples.\n"
],
[
"def print_metrics(y_true, preds, model_name=None):\n '''\n INPUT:\n y_true - the y values that are actually true in the dataset (numpy array or pandas series)\n preds - the predictions for those values from some model (numpy array or pandas series)\n model_name - (str - optional) a name associated with the model if you would like to add it to the print statements \n\n OUTPUT:\n None - prints the accuracy, precision, recall, and F1 score\n '''\n if model_name == None:\n print('Accuracy score: ', format(accuracy_score(y_true, preds)))\n print('Precision score: ', format(precision_score(y_true, preds)))\n print('Recall score: ', format(recall_score(y_true, preds)))\n print('F1 score: ', format(f1_score(y_true, preds)))\n print('\\n\\n')\n \n else:\n print('Accuracy score for ' + model_name + ' :' , format(accuracy_score(y_true, preds)))\n print('Precision score ' + model_name + ' :', format(precision_score(y_true, preds)))\n print('Recall score ' + model_name + ' :', format(recall_score(y_true, preds)))\n print('F1 score ' + model_name + ' :', format(f1_score(y_true, preds)))\n print('\\n\\n')",
"_____no_output_____"
],
[
"clf_a = LogisticRegression(random_state=56)\nclf_b = RandomForestClassifier(random_state=56)\nclf_c = SGDClassifier(random_state=56)\n\n\nmodel_a = clf_a.fit(X_train, y_train)\nmodel_b = clf_b.fit(X_train, y_train)\nmodel_c = clf_c.fit(X_train, y_train)",
"_____no_output_____"
]
],
[
[
"## 5. Evaluation ## \nAfter training our model, we will predict and evaluate the metric in predicting the results",
"_____no_output_____"
]
],
[
[
"rf_preds = model_a.predict(X_test)\nprint_metrics(y_test, rf_preds, 'logistic regression')\n\nrf_preds = model_b.predict(X_test)\nprint_metrics(y_test, rf_preds, 'random forest')\n\nrf_preds = model_c.predict(X_test)\nprint_metrics(y_test, rf_preds, 'SGD classifier')",
"Accuracy score for logistic regression : 0.8554216867469879\nPrecision score logistic regression : 0.8222222222222222\nRecall score logistic regression : 0.9024390243902439\nF1 score logistic regression : 0.8604651162790697\n\n\n\nAccuracy score for random forest : 0.7951807228915663\nPrecision score random forest : 0.8\nRecall score random forest : 0.7804878048780488\nF1 score random forest : 0.7901234567901235\n\n\n\nAccuracy score for SGD classifier : 0.4939759036144578\nPrecision score SGD classifier : 0.4939759036144578\nRecall score SGD classifier : 1.0\nF1 score SGD classifier : 0.6612903225806451\n\n\n\n"
]
],
[
[
"We can see that logistic regresssion is the most suitable in predicting the result since the end result is binary and the random forest as the second most predictive ",
"_____no_output_____"
]
],
[
[
"parameters = {'C':[0.01,0.1,1,10,100,1000], 'solver':['lbfgs', 'liblinear']}\n\n# TODO: Make an fbeta_score scoring object using make_scorer()\nscorer = make_scorer(fbeta_score, beta=0.5)\n\n# TODO: Perform grid search on the classifier using 'scorer' as the scoring method using GridSearchCV()\ngrid_obj = GridSearchCV(clf_a, parameters, scoring=scorer)\n\n# TODO: Fit the grid search object to the training data and find the optimal parameters using fit()\ngrid_fit = grid_obj.fit(X_train, y_train)\n\n# Get the estimator\nbest_clf = grid_fit.best_estimator_\n\n# Make predictions using the unoptimized and model\npredictions = (clf_a.fit(X_train, y_train)).predict(X_test)\nbest_predictions = best_clf.predict(X_test)\n\n# Report the before-and-afterscores\nprint(\"Unoptimized model\\n------\")\nprint(\"Accuracy score on testing data: {:.4f}\".format(accuracy_score(y_test, predictions)))\nprint(\"F-score on testing data: {:.4f}\".format(fbeta_score(y_test, predictions, beta = 0.5)))\nprint(\"\\nOptimized Model\\n------\")\nprint(\"Final accuracy score on the testing data: {:.4f}\".format(accuracy_score(y_test, best_predictions)))\nprint(\"Final F-score on the testing data: {:.4f}\".format(fbeta_score(y_test, best_predictions, beta = 0.5)))",
"Unoptimized model\n------\nAccuracy score on testing data: 0.8554\nF-score on testing data: 0.8371\n\nOptimized Model\n------\nFinal accuracy score on the testing data: 0.8434\nFinal F-score on the testing data: 0.8373\n"
]
],
[
[
"We can see that the unoptimized version is better than optimized in accuracy; however, for the Fscore, the optimized version is better ",
"_____no_output_____"
]
],
[
[
"model = RandomForestClassifier(random_state=56).fit(X_train, y_train)\n\n# TODO: Extract the feature importances using .feature_importances_ \nimportances = model.feature_importances_\n\n# Plot\nvs.feature_plot(importances, X_train, y_train)",
"_____no_output_____"
]
],
[
[
"By using the random forest feature importances, we can get the most predictive top 5 features that can affect the end results of predicting whether patients have heart disease or not. The end results is that ST depression, thallium nuclear test results especially whether normal or reversible defect results, number of heart major vessels and the max heart rate of the patients. \n\n## 6. Deployment ## \nSo after all of the data preparation and analysis, we will conclude and take the findings which can be applied in real world\n\n**1. Does age and gender of the patients have certain trends in determining whether the patient have heart disease or not?** \n\nYes, according to the findings and the graphs, as a person become older, the possibility of having heart disease is higher especially during 50s while the most common gender in having the heart disease is male. By combining both age and gender features, we can know that male patients have wider distribution in the range of 30s-80s while female patients range in 50s-60s.\n\n**2. What is the relationship between blood pressure and heart rate in having heart disease?** \n\nAccording to the max heart rate of the patients, the healthy patient seems to have higher max heart rate compared to the sick patient while the blood pressure for sick patients seems to have higher blood pressure compared to the healthy patient. This fact is also reinforced by the combined graph where healthy patient is located at the place with higher max heart rate and lower blood pressure compared to the sick patient. \n\n**3. Do we know the symptom of the heart attacks such as chest pain caused by heart disease?** \n\nFrom the chest pain graph, we can determine that most of the sick patient with heart disease have asymptomatic or silent angina which did not show any symptoms during its attack. Other remaining features indicated that exercising can also induce the chest pain for the sick patients. If the sick patient is examined with electrocardiogram or nuclear test, we can find out that their heartbeat, heart scanning or heart examinations have abnormal results which related with reducing the amount of blood supplied to heart. \n\n**4. What is the trend of the cardiological test such as ECG, Fluoroscopy and Thallium?** \n\nECG (Electrocardiogram) test results is represented by ST depression result where higher number of the fluctuations in the ST segments, indicated that there is higher chance of contracting heart disease. The flat slope of ST depression is the most common results for the patient with heart disease. Fluoroscopy test result is indicated by major vessels column which stated that the higher the number of major vessels is colored by fluoroscopy, the higher chance that the patient have heart disease. For Thallium test, we found out that the result of the reversible defect is the most common defect that patient with heart disease have. \n\n**5. Out of 14 predicting features, what are the important features in predicting whether the patient have heart disease or not? (by using machine learning)**\n\nBy using machine learning algorithm, we can predict whether a patient have heart disease or not, given their data. After training and evaluating the results, with logistic regression we can get F-score of 86%. We also tried to predict the most influential features that determine whether patient have heart disease or not by using random forest feature importance which F-score is 79%. With feature importance, we find out top 5 features: \n\n1. ST depression \n2. Thallium test results (reversible defect) \n3. Thallium test results (normal) \n4. Major vessels of heart\n5. Maximum Heart rate \n\nall of these features which deemed as important by features importance, seemed to have some similarities with the previous question answers such as that most of the factors can only be determined by thorough tests and cannot be determined by appeared symptoms.\n\n## Conclusion:##\n1. The most common trait of having heart disease seemed attach to older male with high blood pressure and lower maximum heart rate. Exercise might also induce chest pain or discomfort. \n2. Chest pain or discomfort might not be an important predictor in determining heart disease or not since the most common chest pain type is the asymptomatic or the silent chest pain with no chest pain or discomfort shown. \n3. The most important predictor in predicting the heart disease or not result is the through tests such as ECG, Fluoroscopy and Thallium test which is why it is recommended to get our heart examined by cardiologist through these tests so that we can prevent the worst from happening ",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
]
] |
e7e9f06c7dd23c50f2782c2031595d8e09755f78 | 64,969 | ipynb | Jupyter Notebook | Singaporean Food in Indonesia/Phrase III (Sumatra, Kalimantan, Sulawesi, Papua).ipynb | miradelimanr/update-code | 68270534eab0166a05bc5ed6bd18a1aa0ca85b7f | [
"CC0-1.0"
] | null | null | null | Singaporean Food in Indonesia/Phrase III (Sumatra, Kalimantan, Sulawesi, Papua).ipynb | miradelimanr/update-code | 68270534eab0166a05bc5ed6bd18a1aa0ca85b7f | [
"CC0-1.0"
] | 8 | 2022-03-31T02:49:54.000Z | 2022-03-31T06:31:07.000Z | Singaporean Food in Indonesia/Phrase III (Sumatra, Kalimantan, Sulawesi, Papua).ipynb | miradelimanr/update-code | 68270534eab0166a05bc5ed6bd18a1aa0ca85b7f | [
"CC0-1.0"
] | null | null | null | 32,484.5 | 64,968 | 0.653435 | [
[
[
"# Singaporean Food in Indonesia Phrase III : Sumatra, Kalimantan, Sulawesi, and Papua Area\n\nThis is the last phrase of Singaporean food in Indonesia series. In this notebook I'll show you list of Singaporean restaurant in Sumatra (Medan, Padang, Palembang), Kalimantan (Pontianak, Palangkaraya, Balikpapan), Sulawesi (Manado), and Papua (Jayapura). So here is the list curated from Google Maps and various platform website. This time I'll be using Tripadvisor, Traveloka Eats, and Pergi Kuliner as the source.\n\nOriginally there are 101 restaurants selected all around Indonesia,which 23 of them are located in this area. Note that the list here is not always the authentic one. There are coffee shop named Kopitiam and Chinese restaurant which has Singaporean food but never claimed themselves as Singaporean restaurant. At least they got kaya toast, hainanse rice, laksa, chili crab and etc, will explain more on menu.\n\n# Content\n* Overview\n* Menu\n* Facility\n* Rating",
"_____no_output_____"
]
],
[
[
"from pandasql import sqldf\nimport pandas as pd\nsingapore = pd.read_csv(\"../input/singapore-food/singapore_id.csv\", sep = \";\")",
"_____no_output_____"
]
],
[
[
"# Overview\n\nHere is all the list of restaurants sorted by region and city, there are also their unique menu, price, and ratings. Most of prices ranged from 20k until 65k and ratings are quite rave. Medan and Palembang is the most available restaurant.",
"_____no_output_____"
]
],
[
[
"singapore = sqldf(\"\"\"SELECT no, restaurant_name, region, city, unique_menu, price, \nltrim(google_rating) as google_rating, ltrim(platform_rating) as platform_rating \nFROM singapore \nWHERE region LIKE '%Sumatra' OR region LIKE '%Kalimantan' OR region LIKE '%Sulawesi' OR region LIKE '%Papua'\"\"\")\nsingapore.style.hide_index()",
"_____no_output_____"
]
],
[
[
"# Menu\n\nLike the previous part, I'll divide those menu on three category, which are Rice & Noodle Menu, Poultry Menu, and Toast & Snack Menu.\n\n**Rice & Noodle**\n\nHere is their Rice and Noodle Menu. For the rice they mostly have hainanse rice and various porridge. For noodle they have noodle (mostly mee goreng) and kway teow. Also there are laksa and bakmi ayam as well. All shown here are representative for each city, so then check issue for complete csv available.\n\nThere are some restaurant doesn't serving noodle which are **Singapore Chicken Rice (Pontianak), One Chick (Pontianak), Bubur Singapore (Makassar), Aman Hainanse Chicken Rice, and Liu Kee (Medan)**, only hainanse rice available. **Rajawali Kopitiam (Palembang)** does have bakmi ayam only. \n\nFor disambiguation, mee is mie/noodle, fried rice is nasi goreng, kway teow is kwetiaw, beehoon is bihun, and hainanse rice is nasi hainam. Bakmi ayam is quite similar with wonton noodle but it's different after all.",
"_____no_output_____"
]
],
[
[
"singapore = sqldf(\"\"\"SELECT restaurant_name, city, rice_menu, noodle, noodle_specific\nFROM singapore\nWHERE region LIKE '%Sumatra' OR region LIKE '%Kalimantan' OR region LIKE '%Sulawesi' OR region LIKE '%Papua'\nGROUP BY city\"\"\")\nsingapore.style.hide_index()",
"_____no_output_____"
]
],
[
[
"**Poultry Menu**\n\nBased on the list here, the poultry menu are mostly dominated by chicken but only as a topping (signed by 'yes'), but some places have chicken speciality like kungpao and blackpepper. The seafood is quite rare but some places mostly has prawn as majority. Restaurant serving seafood are **New Star Kopitiam Resto (Balikpapan)**--prawn only, **Bubur Singapore (Makassar), M Kopitiam (Palembang), and New Town Kopitiam Bukit Golf (Palembang)**. There are three places serving pork, which are **Singapore Chicken Rice (Pontianak), Aman Hainanse Chicken Rice (Medan), and SING Bakuteh (Manado)**. Mostly the other poultry dominated by vegetables.",
"_____no_output_____"
]
],
[
[
"singapore = sqldf(\"\"\"SELECT restaurant_name, city, chicken, seafood, poultry_other\nFROM singapore\nWHERE region LIKE '%Sumatra' OR region LIKE '%Kalimantan' OR region LIKE '%Sulawesi' OR region LIKE '%Papua'\nGROUP BY city\"\"\")\nsingapore.style.hide_index()",
"_____no_output_____"
]
],
[
[
"**Toast & Snack Menu**\n\nSimilar with the previous part, kaya toast is also popular in this area. Which means kaya toast is the most popular Singaporean toast for entire Indonesia. As for snack the fries are the most popular, but there is a place named **Aman Hainanse Chicken Rice (Medan)** which has unique snack which no other place have, like chasio, siobak, and pekamche. In this section, toast and snack are listed separately.",
"_____no_output_____"
]
],
[
[
"singapore = sqldf(\"\"\"SELECT restaurant_name, city, toast\nFROM singapore\nWHERE (region LIKE '%Sumatra' OR region LIKE '%Kalimantan' OR region LIKE '%Sulawesi' OR region LIKE '%Papua') AND (toast NOT LIKE '%no')\"\"\")\nsingapore.style.hide_index()",
"_____no_output_____"
],
[
"singapore = sqldf(\"\"\"SELECT restaurant_name, city, snack_dish\nFROM singapore\nWHERE (region LIKE '%Sumatra' OR region LIKE '%Kalimantan' OR region LIKE '%Sulawesi' OR region LIKE '%Papua') AND (snack_dish NOT LIKE '%no')\"\"\")\nsingapore.style.hide_index()",
"_____no_output_____"
]
],
[
[
"# Facility\n\nAll of them have takeaway options and majority of them has delivery options except some places. None of them has outdoor seat and smoking area, except Bangi Kopitiam, M Kopitiam, New Town Kopitiam, Pace Tan Pu Kopitiam, and Lins Kopitiam for the outdoor, and SING Bakuteh Manado and Rajawali Kopitiam for the smoking place. Note that sometimes outdoor area is also a smoking area. \n\nOnly three places serving alcohol, which are **Bangi Kopitiam (Palembang), New Town Kopitiam (Palembang),** and **M Kopitiam (Palembang)**. Lastly, wifi is available in small number, mostly in Palembang and Papua area.\n\nGrouped by city.",
"_____no_output_____"
]
],
[
[
"singapore = sqldf(\"\"\"SELECT restaurant_name, city, takeaway, delivery, outdoor_seat, smoking_area, alcohol_served, wifi\nFROM singapore\nWHERE region LIKE '%Sumatra' OR region LIKE '%Kalimantan' OR region LIKE '%Sulawesi' OR region LIKE '%Papua'\nGROUP BY city\"\"\")\nsingapore.style.hide_index()",
"_____no_output_____"
]
],
[
[
"# Rating\n\nThese ratings are ordered by highest total rating, should they have more than 3.5 then the restaurant is worth to try. All the facility and menu are representated by all both google and platform rating and count, so in the meantime only ratings are counted here. Based on the results, **Liu Kee Hainanse Chicken Rice (Medan), M Kopitiam (Palembang)**, and **Aman Hainanse Chicken Rice (Medan)** are the top 3 among the list.\n\nBased on the area, for Sumatra they got **Liu Kee Hainanse Chicken Rice** which is the best from Medan (scored 4,9). Then from Palembang they have **M Kopitiam** which is the most proper (scored 4,85). **Pondok Kopitiam** is the only one Singaporean related kopitiam remaining in Padang (scored 4,5).\n\nFor Kalimantan area, **Onechick (Pontianak)** has the highest rating among their region (scored 4,5). **Abun Kopitiam** is better than New Star Kopitiam Resto in Balikpapan (scored both 4,35 and 3,8). **Palangka Kopitiam** is the only one Singaporean related cuisine in Palangkaraya (scored 3,95).\n\nIn Sulawesi, **SING Bakuteh Manado** is the best on the region (scored 4,5) and **Bubur Singapore** is the most popular on Makassar (scored 4,45).\n\nLastly, **Bangi Kopitiam Jayapura** is the best from Jayapura among the three (scored 4,55).",
"_____no_output_____"
]
],
[
[
"singapore = sqldf(\"\"\"SELECT restaurant_name, city, \nltrim(google_rating) as google_rating, \nltrim(platform_rating) as platform_rating, \nltrim((google_rating + platform_rating)/2) as total_rating,\nCASE\n WHEN (google_rating + platform_rating)/2 >= 3.5 THEN \"Recommended\"\n ELSE \"Reconsider\"\nEND AS \"Recommendation\"\nFROM singapore WHERE region LIKE '%Sumatra' OR region LIKE '%Kalimantan' OR region LIKE '%Sulawesi' OR region LIKE '%Papua'\nORDER BY total_rating DESC\"\"\")\nsingapore.style.hide_index()",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7e9f70f3474c635fce350ee21e40385a7cbc0d0 | 92,961 | ipynb | Jupyter Notebook | notebooks/altair_notebooks/07-LayeredCharts.ipynb | WSU-DataScience/ICOTS10_Data_Visualization | f71b48fb1d9cf5919281e2a55db02589f541d92e | [
"BSD-3-Clause"
] | 1 | 2019-12-28T15:08:15.000Z | 2019-12-28T15:08:15.000Z | notebooks/altair_notebooks/07-LayeredCharts.ipynb | WSU-DataScience/ICOTS10_Data_Visualization | f71b48fb1d9cf5919281e2a55db02589f541d92e | [
"BSD-3-Clause"
] | null | null | null | notebooks/altair_notebooks/07-LayeredCharts.ipynb | WSU-DataScience/ICOTS10_Data_Visualization | f71b48fb1d9cf5919281e2a55db02589f541d92e | [
"BSD-3-Clause"
] | null | null | null | 205.665929 | 26,313 | 0.88422 | [
[
[
"# Layered Charts",
"_____no_output_____"
],
[
"A `LayeredChart` allows you to stack multiple individual charts on top of each other as layers. For example, this could be used to create a chart with both lines and points.",
"_____no_output_____"
],
[
"## Imports",
"_____no_output_____"
]
],
[
[
"import altair as alt\nimport pandas as pd\nimport numpy as np",
"_____no_output_____"
],
[
"# Uncomment/run this line to enable Altair in the classic notebook (not JupyterLab)\nalt.renderers.enable('notebook')",
"_____no_output_____"
]
],
[
[
"## Data",
"_____no_output_____"
]
],
[
[
"np.random.seed(181)\ndata = pd.DataFrame({'x': np.arange(10),\n 'y':np.random.rand(10)})",
"_____no_output_____"
],
[
"data.head()",
"_____no_output_____"
]
],
[
[
"## Layered charts",
"_____no_output_____"
],
[
"Suppose you have defined two charts, and you would like to plot them on the same set of axes.\nThis comes up often when creating a compound chart with points and lines marking the same data.\nFor example:",
"_____no_output_____"
]
],
[
[
"layer1 = alt.Chart(data).mark_point().encode(\n x='x:Q',\n y='y:Q'\n)\n\nlayer2 = alt.Chart(data).mark_line().encode(\n x='x:Q',\n y='y:Q'\n)",
"_____no_output_____"
]
],
[
[
"The most succinct way to layer two charts is to use the ``+`` operator:",
"_____no_output_____"
]
],
[
[
"layer1 + layer2",
"_____no_output_____"
]
],
[
[
"One problem with this is that you end up specifying the encodings and data multiple times; you can instead build both layers from the same base chart:",
"_____no_output_____"
]
],
[
[
"base = alt.Chart(data).encode(\n x='x:Q',\n y='y:Q'\n)\n\nbase.mark_line() + base.mark_point()",
"_____no_output_____"
]
],
[
[
"To be a bit more explicit, you can use the ``alt.layer`` function, which produces the same thing:",
"_____no_output_____"
]
],
[
[
"alt.layer(\n base.mark_line(),\n base.mark_point()\n)",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7e9ff547fa69d6484bdf1b28dfa7cc3be88004a | 32,844 | ipynb | Jupyter Notebook | Muskingum_routing.ipynb | hcwinsemius/hydrology_computing | e4b7eb8bf217f2ae293e8c57de0312545aee754b | [
"MIT"
] | null | null | null | Muskingum_routing.ipynb | hcwinsemius/hydrology_computing | e4b7eb8bf217f2ae293e8c57de0312545aee754b | [
"MIT"
] | null | null | null | Muskingum_routing.ipynb | hcwinsemius/hydrology_computing | e4b7eb8bf217f2ae293e8c57de0312545aee754b | [
"MIT"
] | null | null | null | 132.435484 | 24,330 | 0.850201 | [
[
[
"%matplotlib inline\nimport matplotlib.pyplot as plt\nimport numpy as np\nimport matplotlib.pyplot as plt\nfrom ipywidgets import interact\n",
"_____no_output_____"
]
],
[
[
"In this notebook, we solve a storage flow relationship, assuming the so-called Muskingum equation. An important law we use (as always) is the continuity equation (i.e. how much flow goes into and out of a given river section, how much does this change the storage?)\n\n$$\n\\frac{\\partial{A}}{\\partial{t}} + \\frac{\\partial{Q}}{\\partial{x}} = 0\n$$\n\nHere $A$ [L$^2$] is the wetted cross-section, $t$ is the time [T], $Q$ [L$^3$ T$^{-1}$] is the flow and $x$ is the longitudinal position [L], usually we use meters and seconds for the magnitudes L and T.\n\nWe can integrate this equation over a certain length $x$ and simply look at the storage over a given channel section $S$, assuming that at any moment in time $S=Ax$, yielding:\n\n$$\n\\frac{\\Delta A\\ x}{\\Delta{t}} + \\Delta{Q} = 0\n$$\nor in simpler terms:\n$$\n\\frac{\\Delta S}{\\Delta t} = -\\Delta Q\n$$\nWhen integrating over a certain time span $t_1$ until $t_2$ we yield:\n$$\n\\frac{S_2-S_1}{\\Delta t}=0.5\\left(I_1+I_2\\right)-0.5\\left(O_1+O_2\\right)\n$$\nwhere $I$ and $O$ represent $Q$ at the inflow and outflow boundaries of the channel stretch $x$ respectively. \n\nOk, so far we have only simplified the continuity equation. Now we need a storage-flow relationship. Muskingum assumes that the storage can be described with a linear relationship of the weighted average of inflows and outflows in a given channel section. ADD EXPLANATION, WHAT DOES THIS MEAN IN PRACTICE?\n\n$$\nS=k\\left[fI+\\left(1-f\\right)O\\right]\n$$\nIt is pretty easy to see that this relationship approaches a linear reservoir when $f$ approaches 0. We can also subsitute this in the earlier found discretized continuity equation:\n$$\n\\frac{k}{\\Delta t}\\left[f\\left(I_2-I_1\\right)+\\left(1-f\\right)\\left(O_2-O_1\\right)\\right]=0.5\\left(I_1+I_2\\right)-0.5\\left(O_1+O_2\\right)\n$$\nThis allows us to relate the outflow from a storage section at a future time step to the inflows over the time step and outflow over the previous time step. Reordering then provides:\n$$\nS_2 = c_0I_2+c_1I_1 + c_2O_1\n$$\nwith:\n$$\nc_0 = \\frac{-2f+\\Delta t/k}{2\\left(1-f\\right)+\\Delta t/k}, \nc_1 = \\frac{2f+\\Delta t/k}{2\\left(1-f\\right)+\\Delta t/k}, \nc_2 = \\frac{2\\left(1-f\\right)-\\Delta t/k}{2\\left(1-f\\right)+\\Delta t/k}\n$$\nOk, this is extremely simple! Let's put this in a little model!",
"_____no_output_____"
]
],
[
[
"\nclass simple_runoff(object):\n \"\"\"\n A very very simple rainfall runoff response model, computing specific runoff as follows:\n q = max(P-I, 0)*rc\n where P is precipitation in mm per day, I is interception in mm/day, rc is runoff coefficient [-]\n Consequently, it will compute flows at the outlet as follows:\n Q(t) = q(t)A * f(t)\n It will convert flows to m3/s (instead of day) before returning\n where f(t) is a unit hydrograph convolution function, computed assuming a triangular shape,\n with a time of concentration given in days\n Input:\n I: interception threshold [mm/day]\n rc: runoff coefficient [-]\n A: catchment surface area [km2]\n t_c: time of concentration [day]\n \"\"\"\n def __init__(self, I, rc, A, t_c):\n self.I = I\n self.rc = rc\n self.A = A\n self.t_c = t_c\n self.triangle_UH()\n \n def triangle_UH(self):\n \"\"\"\n Make a unit hydrograph assuming a time of concentration in time steps\n \"\"\"\n uh_right = np.append(np.linspace(0, 1, self.t_c), np.linspace(1, 0, self.t_c)[1:])\n uh_left = np.zeros(len(uh_right))\n self.conv = np.append(uh_left, uh_right)/uh_right.sum()\n\n def compute_flows(self, P):\n q = np.maximum(P-self.I, 0)*self.rc\n _Q = np.convolve(q*A*1e3/86400, self.conv, 'same')\n return pd.Series(_Q, index=pd.DatetimeIndex(start=P.index[0], periods=len(_Q), freq=P.index.freqstr))\n \nclass Muskingum(object):\n def __init__(self, k, f, dt):\n self.k = k # residence time\n self.f = f # weighting factor\n self.dt = dt\n self.c0 = (-2*f+dt/k)/(2*(1-f)+dt/k)\n self.c1 = (2*f+dt/k)/(2*(1-f)+dt/k)\n self.c2 = (2*(1-f)-dt/k)/(2*(1-f)+dt/k)\n \n def compute_O_2(self, I_1, I_2, O_1):\n # check for NaNs in O_1\n if np.isnan(O_1):\n O_1 = 0\n return self.c0*I_2+self.c1*I_1+self.c2*O_1\n\n def compute_outflows(self, s_inflows):\n s_outflows = pd.Series(index=s_inflows.index, )\n for _i in range(1, len(s_inflows)):\n s_outflows.iloc[_i] = compute_O_2(s_inflows.iloc[_i-1], s_inflows.iloc[_i], s_outflows.iloc[_i-1])\n return s_outflows\n",
"_____no_output_____"
],
[
"import pandas as pd\n\ns = pd.Series(index=pd.DatetimeIndex(start='2001-01-01', end='2001-01-30', freq='D'))\ns2 = pd.Series()\nP.index[0]",
"_____no_output_____"
],
[
"# prepare some flows from a unit hydrograph principe\np = np.array([0., 5., 20., 18., 15., 12., 8., 10., 2., 0., 0.])\nP = pd.Series(p, index=pd.DatetimeIndex(start='2001-01-01', periods=len(p), freq='D')) # mm/day\n\n# make a model\nI = 3. # interception threshold [mm/day]\nrc = 0.5# event runoff coefficient [-]\nA = 150000 # surface of catchment [km2]\nt_c = 5\nrunoff_model = simple_runoff(I, rc, A, t_c)\nQ = runoff_model.compute_flows(P)\n",
"_____no_output_____"
],
[
"ax = plt.axes()\n\ndf = pd.DataFrame({'P': P, 'Q': Q})\ndf['P'].plot(ax=ax, secondary_y=True, label='rainfall')\ndf['Q'].plot(ax=ax, label='Discharge')",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code"
] | [
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
]
] |
e7ea0265841be6411bc6f623f5c492cfa7dac50f | 6,987 | ipynb | Jupyter Notebook | docs/source/MIP/solved/Plastic Injection Production Planning.ipynb | ffraile/operations-research | 3e143ac4ec7031f008505cc2695095ab8dfa2f28 | [
"MIT"
] | null | null | null | docs/source/MIP/solved/Plastic Injection Production Planning.ipynb | ffraile/operations-research | 3e143ac4ec7031f008505cc2695095ab8dfa2f28 | [
"MIT"
] | null | null | null | docs/source/MIP/solved/Plastic Injection Production Planning.ipynb | ffraile/operations-research | 3e143ac4ec7031f008505cc2695095ab8dfa2f28 | [
"MIT"
] | null | null | null | 43.397516 | 339 | 0.611994 | [
[
[
"# Plastic Injection Production Planning\n## Problem definition\n\nPatrick \"Eel\" O'Brian's (PEOB) Ltd. is a company that manufactures plastic components for the automotive sector. \nAs an intern at PEOB Ltd, your first assignment is to write an Integer Programming Model to calculate the optimal production plan for the plastic injection machine. \nThe injection machine heats plastic and injects it into a mould to get a specific shape. Each mould can thus only be used to manufacture specific component types. Your first version will take into account the storage costs and the delayed orders cost which are defined below. \n\n**Storage costs**\nThe storage or inventory costs represent the cost of storing the inventory levels of every component type at every planning period and are modeled as a fixed cost per unit and planning period. The storage costs are different for every component type.\n\n**Delayed orders costs**\nAt every planning period, you may delay part of the demand for next planning periods. Customers will apply a fixed penalti for every delayed unit at every period. The delayed costs are also different for every component type.\n\nYour model needs to take into account the following additional data: \n\n**Initial Inventory Levels (units)**: There is an initial inventory level for every component type available at the first planning period that needs to be taken into account.\n\n**Minimum and Maximum Inventory Levels (units)**: There is a maximum and a minimum inventory level for every component type.\n\n**Machine capacity (units)**: The machine capacity represents the number of units of a given component type that the machine can produce using a given mould. If a mould cannot be used to manufacture a component type, the machine capacity is zero for that combination of component type and mould.\n\n**Demand (Units)**: The company has several confirmed orders for the following periods and therefore, our model needs to take into account the demand for every product at every planning period. \n\n**a.** Write down the indexes and decision variables\n**Indices**\n\n- t = periods to produce $t \\in [0,..,T]$\n\n- m = moulds $m \\in [0,..,M]$\n\n- p = products $p \\in [0,..,P]$\n\n**Decision Variables**\n\n- $Y_{pt}$ = units to store for produt p on period t (Integer)\n\n- $D_{pt}$ = delayed production for produt p on period t (Integer)\n\n- $X_{pt}$ = production for produt p on period t (Integer)\n\n- $S_{mt}$ = (Binary) {1 if mould m is used on period t, 0 otherwise}\n\n**b.** Write down the objective function\nThe objective function is to minimise storage costs and delayed order costs (eur):\n\n$\\min z = \\sum_{t=0}^{T}{\\sum_{p=0}^{P}{C_d·D_{pt}}} + \\sum_{t=0}^{T}{\\sum_{p=0}^{P}{C_s·Y_{pt}}}$ (eur)\n\nWhere: \n\n- $C_s$ represents the storage costs per unit and period (eur)\n\n- $C_d$ represents the delayed order costs per unit and period (eur)\n\n**c.** Write down the constraints\n\n**Problem Data**\n- $M_{mp}$ Machine Capacity using mould m to produce product p(units)\n\n- $R_{pt}$ Demand for product p in period t\n\n- $Y_{p0}$ Initial inventory level of product p\n\n- $Y_{pmax}$ Maximum inventory level of product p\n\n- $Y_{pmin}$ Minimum inventory level of product p\n\n\n**Machine Capacity Constraint**\n\n$X_{pt} = \\sum_{m=0}^{M}{M_{mp}·S_{mt}} \\forall p,t$\n\n$\\sum_{m=0}^{M}{S_{mt}} = 1 \\forall t$\n\nMeaning that the number of units manufactured in period t of product p is equal to the machine capacity of the machine using the mould m. The second constraint ensures that the model is consistent and only one mould is used in one period. \n\n**Demand**\n\n$Y_{pt-1} + X_{pt} - Y_{pt} = R_{pt} + D_{pt-1} - D_{pt} \\forall p, t \\in [1,T]$\n\n(Initial Inventory)\n\n$X_{p0} - Y_{p0} = R_{p0} - D_{p0} \\forall p, t=0$\n\n**Inventory levels**\n\n$Y_{pt} \\geq Y_{minp} \\forall p,t$\n\n$Y_{pt} \\leq Y_{maxp} \\forall p,t$\n\nYou have successfully validated your model and it is already providing valuable information for the company. Now the company would like to extend the model to take into account as well the set up costs: \n\n**Set up costs**\nAs explained above, the injection machine uses different moulds to manufacture different parts. Each time that a mould is changed to make a different component, the operators need to setup the machine. The company estimates that this setup cost only depends on the mould that is used and is only applied when a product is changed.\n\n**d.** Modify the model to take into account the set up costs.\n\nWe introduce a new decision variable to model the sequence of mould changes and simplify the model: \n\n- $Su_{mt}$ Mould setup sequence (Binary) 1 if mould m is changed in period t, 0 otherwise.\n- $C_m$ Setup costs of mould m (Eur). \n\nNow, the objective function becomes:\n\n\n$\\min z =\\sum_{t=0}^{T}{\\sum_{m=0}^{M}{C_m·Su_{mt}}} + \\sum_{t=0}^{T}{\\sum_{p=0}^{P}{C_s·D_{pt}}} + \\sum_{t=0}^{T}{\\sum_{p=0}^{P}{C_d·Y_{pt}}}$ (Eur)\n\nWe also need to introduce the following constraints for the new decision variable:\n\n$Su_{m0} = S_{m0} \\forall m$\n\nMeaning that we take into account the costs of the initial setup of the machine.\n\n$Su_{mt} \\geq S_{mt} - S_{mt-1}$\n\nMeaning that we only take into account in the costs mould changes (that is, when the mould used is different for two consecutive periods).\n\n\n",
"_____no_output_____"
]
]
] | [
"markdown"
] | [
[
"markdown"
]
] |
e7ea0e2d05e6452b478470440560a0e694184f68 | 34,681 | ipynb | Jupyter Notebook | notebooks/sagemaker_databrew_ml_workflow_blog.ipynb | aws-samples/aws-databrew-ml-stepfunction-workflow | f188f5ac5d77b4b14f4c7842a530927b54746ceb | [
"MIT-0"
] | 2 | 2021-09-13T17:13:47.000Z | 2022-01-18T12:17:41.000Z | notebooks/sagemaker_databrew_ml_workflow_blog.ipynb | aws-samples/aws-databrew-ml-stepfunction-workflow | f188f5ac5d77b4b14f4c7842a530927b54746ceb | [
"MIT-0"
] | null | null | null | notebooks/sagemaker_databrew_ml_workflow_blog.ipynb | aws-samples/aws-databrew-ml-stepfunction-workflow | f188f5ac5d77b4b14f4c7842a530927b54746ceb | [
"MIT-0"
] | 1 | 2021-09-13T17:05:04.000Z | 2021-09-13T17:05:04.000Z | 39.410227 | 566 | 0.636487 | [
[
[
"# Sagemaker Model training workflow with AWS Glue Databrew reciepe and AWS Step Functions.\n\n1. [Introduction](#Introduction)\n1. [Setup](#Setup)\n1. [Create Resources](#Create-Resources)\n1. [Build a Machine Learning Workflow](#Build-a-Machine-Learning-Workflow)\n1. [Run the Workflow](#Run-the-Workflow)\n1. [Clean Up](#Clean-Up)",
"_____no_output_____"
],
[
"## Introduction\n\nAs with growing data and large scale adoption of machine learning solutions, cleansing and visualizing data for model training has become key task in the ML workflow. As data scientist and engineers look for more insights from data they also want to reduce time to derive new insights and look for data profiling and transformation capabilities from visual preparation tool.\nAs part of this blog post, we walk through a solution where we create ML workflow using AWS step functions within Sagemaker notebook and use DataBrew for visual data preparation step and run DataBrew recipe jobs as part of ML workflow.\n\nThis notebook is part SageMaker model training with DataBrew recipe job blog post.\n\n## Use case overview\n\nFor our use case, we use direct marketing campaigns public dataset. The marketing campaigns were based on phone calls. This dataset has few biographic and economic status about campaign contacts and their final decision to access product.\nThe classification goal is to predict if the client will subscribe a term deposit (variable y).\nThe dataset we use is publicly available and it is attributed by S. Moro, P. Cortez and P. Rita to the University Of California Irvine Repository Of Machine Learning Datasets.\n\nhttps://archive.ics.uci.edu/ml/datasets/Bank+Marketing\n\nFor our use case, this campaign CSV file is maintained by your organization’s Marketing team, which uploads CSV file to Amazon Simple Storage Service (Amazon S3). We then create series of data preparation steps using AWS DataBrew and create product subscription model using ML workflow using Sagemaker & AWS step functions SDK.\n\n* [AWS Glue Databrew](https://aws.amazon.com/glue/features/databrew/)\n* [AWS Glue Databrew Deverloper Guide](https://docs.aws.amazon.com/databrew/latest/dg/what-is.html)\n* [AWS Step Functions](https://aws.amazon.com/step-functions/)\n* [AWS Step Functions Developer Guide](https://docs.aws.amazon.com/step-functions/latest/dg/welcome.html)\n* [AWS Step Functions Data Science SDK](https://aws-step-functions-data-science-sdk.readthedocs.io)\n",
"_____no_output_____"
],
[
"## Visual Workflow \n\n",
"_____no_output_____"
],
[
"## DataBrew Plugin Setup (Optional)\n\nFirst, we'll install AWS Glue databrew plugin with Sagemaker and load all the required modules, adding this plugin will help you to work with familiar AWS Glue Databrew console within Sagemaker. \nThen we'll create fine-grained IAM roles for the AWS Glue databrew,Lambda and Step Functions resources that we will create. The IAM roles grant the services permissions within your AWS environment.",
"_____no_output_____"
],
[
"## AWS Glue Databrew Plugin Installation\n\nMake sure to add the correct permissions to the role your SageMaker Studio runs with. A good place to start is by:\n\n1. Adding the *AwsGlueDataBrewFullAccessPolicy* managed policy to your SageMaker execution role, and;\n\n2. Creating *AwsGlueDataBrewSpecificS3BucketPolicy* customer managed policy with s3:GetObject and s3:PutObject permissions for the bucket/s in which you would like to operate and adding this policy to your SageMaker execution role. Refer to IAM policy for S3 documentation for more details.\n\nDetailed steps for plugin installation is documented in below page, please use sagemaker system terminal and follow below steps.\n\n* [SageMaker Plugin installation](https://github.com/aws/aws-glue-databrew-jupyter-extension/blob/main/SageMaker-Installation-Instructions.md#installing-the-plugin)",
"_____no_output_____"
],
[
"",
"_____no_output_____"
],
[
"### Adding Databrew receipe steps\n\nAfter creating new DataBrew project and importing dataset, please proceed with below transformations.",
"_____no_output_____"
],
[
"### Categorical data mapping\n\nOrdinal categorical values are ordered or hierarchical like Education level or economic status. These can be labeled as 1, 2 and 3 numerical format which represent lowest to highest ordering. DataBrew has an easier way to handle such variables by using Categorical mapping. It can be accessed from the toolbar under Mapping.\nCategorical mapping also used for custom one to one mapping like “Yes” / “No” to 1/0 values. Here is an example mapping for the output variable y which gets converted to numeric values 1/0 using categorical mapping function.\n\n",
"_____no_output_____"
],
[
"### One hot encoding\n\nFor all non-ordinal categorical values like marital status or education, One-hot encoding is the most common way to convert them to numerical format. It can be accessed from column actions next to the column name or from the DataBrew project toolbar under Encoding. \n\n",
"_____no_output_____"
],
[
"### Binning\n\nBinning is a data pre-processing technique used to reduce the effects of minor observation errors and the binning transformation allows you to group numbers of more or less continuous values into a smaller number of \"bins\". For example, in the current data set we have ages of people which we can group into a smaller number of age intervals.\n\nYou can access the binning from scale menu on the toolbar.\n\n\n",
"_____no_output_____"
],
[
"### Binarization\n\nBinarization is the process of dividing data into two groups and assigning one out of two values to all the members of the same group. You can use the Binarize transformation by defining a threshold t and assigning the value 0 to all the data points below the threshold and 1 to those above it. \nIn our marketing dataset the duration column specifies the last contact duration, in seconds (numeric). This metric is highly useful to predict outcome as usually the longer the conversation the customer is interested towards the offering. Adding a binarized long call metric based on call duration will be helpful in our prediction model.\nAdding 5 min (300 sec) threshold to add new metric long call.\n\n\n",
"_____no_output_____"
],
[
"### Add a Flag column\n\nAnother useful recipe step is to add a column with transformation applied on a column, we will add a flag column for those who has house but no loan to give better weightage.\n\n",
"_____no_output_____"
],
[
"### Delete unused columns\n\nFinally drop unused columns before writing final output file for model creation. In the next step we will use Sagemaker inbuilt model XGBoost algorithm for model training which require the output variable to be the first column in your dataset.\n\n",
"_____no_output_____"
],
[
"After saving above recipe please create new DataBrew recipe job and use it in ETL step below.",
"_____no_output_____"
],
[
"## Sagemaker Permission Setup\n\nBefore beginning this tutorial, make sure you have the required permissions to create the resources required as part of the solution.\nYou have to setup below roles / permission to implement this ML workflow.\n\nAdd following permission policy to your Sagemaker Studio execution role.\n1.\tAWSStepFunctionsFullAccess\n2.\tAWS Lambda function (Write for Access level)\n3.\tAWSGlueDataBrewFullAccess\n\nCreate new role for Step function execution with related permission for orchestrating DataBrew Jobs, Sagemaker training & Lambda function. \t\n1.\tAmazonSageMaker-StepFunctionsWorkflowExecutionRole\n2.\tAdd Iam:PassRole permission to Sagemaker service in policy definition.\n\nAWS Glue DataBrew role to access data stored in S3 and create DataBrew recipe jobs.\n\nAWS Lambda role to query Sagemaker training status.\n",
"_____no_output_____"
],
[
"To make it easier for you to get started, we created an AWS CloudFormation template that helps configure Sagemaker notebook role with the required policies for Step function Orchestration and new role for DataBrew job creation and AWS Lambda execution. The source code for the CloudFormation template are available in the GitHub repo.\n\n\nYou have to pass below parameters to the template to pre-configure the roles to allow fine grained resource access.\n\nSageMakerExecutionRole \t- Copy the role name from Sagemaker studio\n\nDatabrewGlueJobName\t - Preferred name for your DataBrew recipe job\n\nS3Bucket\t\t\t - Source data S3 bucket name.\n",
"_____no_output_____"
],
[
"### Import the Required Modules",
"_____no_output_____"
]
],
[
[
"# verify latest version of stepfunctions.\nimport sys\n\n# verify step function version\n!pip show stepfunctions\n\n# clone the repo and install SDK version > 2.2.0 required for databrew integration \n# https://github.com/aws/aws-step-functions-data-science-sdk-python/pull/151\n!git clone https://github.com/aws/aws-step-functions-data-science-sdk-python.git /tmp/aws-step-functions-data-science-sdk-python\n!pip install /tmp/aws-step-functions-data-science-sdk-python/.",
"_____no_output_____"
],
[
"import uuid\nimport logging\nimport stepfunctions\nimport boto3\nimport sagemaker\n\nfrom sagemaker.amazon.amazon_estimator import image_uris\nfrom sagemaker.inputs import TrainingInput\nfrom sagemaker.s3 import S3Uploader\nfrom stepfunctions import steps\nfrom stepfunctions.steps import TrainingStep, ModelStep\nfrom stepfunctions.inputs import ExecutionInput\nfrom stepfunctions.workflow import Workflow\n\nsession = sagemaker.Session()\nstepfunctions.set_stream_logger(level=logging.INFO)\n\nregion = boto3.Session().region_name\nbucket = session.default_bucket()\nid = uuid.uuid4().hex\n\n\n#Create a unique name for the AWS Glue DataBrew recipe & Glue etl job to be created. If you change the \n#default name, you may need to change the Step Functions execution role.\nrecipe_job_name = 'job-marketing-research-recipe'\netl_job_name = 'job-marketing-research-etl-{}'.format(id)\n\n#Create a unique name for the AWS Lambda function to be created. If you change\n#the default name, you may need to change the Step Functions execution role.\nfunction_name = 'sage_training_query_status-{}'.format(id)",
"_____no_output_____"
]
],
[
[
"### Configure Execution Roles",
"_____no_output_____"
]
],
[
[
"# paste the AmazonSageMaker-StepFunctionsWorkflowExecutionRole ARN (please refer permission setup section)\nworkflow_execution_role = ''\n\n# SageMaker Execution Role\n# You can use sagemaker.get_execution_role() if running inside sagemaker's notebook instance\nsagemaker_execution_role = sagemaker.get_execution_role() #Replace with ARN if not in an AWS SageMaker notebook",
"_____no_output_____"
],
[
"sagemaker_execution_role",
"_____no_output_____"
],
[
"# paste the query_training_status_role role ARN (refer prerequisites section)\nlambda_role = ''\n\n# paste the glue etl role this will help glue job to write to S3.\nglue_role = ''",
"_____no_output_____"
],
[
"session = sagemaker.Session()\nbucket = session.default_bucket()\nprint(bucket)\nproject_name = 'databrew_blog'",
"_____no_output_____"
]
],
[
[
"### Source DataSet Location\n\nCopy the train dataset to S3 location for DataBrew transformation and to train the processed data.",
"_____no_output_____"
]
],
[
[
"data_source = S3Uploader.upload(local_path='./data/bank-additional.csv',\n desired_s3_uri='s3://{}/{}'.format(bucket, project_name),\n sagemaker_session=session)\nrecipe_prefix = 'recipe'\ntrain_prefix = 'train'\nval_prefix = 'validation'\n\nrecipe_data = 's3://{}/{}/{}/'.format(bucket, project_name, recipe_prefix)\ntrain_data = 's3://{}/{}/{}/'.format(bucket, project_name, train_prefix)\nvalidation_data = 's3://{}/{}/{}/'.format(bucket, project_name, val_prefix)",
"_____no_output_____"
]
],
[
[
"### Create the AWS Glue Job",
"_____no_output_____"
]
],
[
[
"glue_script_location = S3Uploader.upload(local_path='./code/glue_etl.py',\n desired_s3_uri='s3://{}/{}'.format(bucket, project_name),\n sagemaker_session=session)\nglue_client = boto3.client('glue')\n\nresponse = glue_client.create_job(\n Name=etl_job_name,\n Description='PySpark job to split data in to training and validation data sets and remove header',\n Role=glue_role, # you can pass your existing AWS Glue role here if you have used Glue before\n ExecutionProperty={\n 'MaxConcurrentRuns': 2\n },\n Command={\n 'Name': 'glueetl',\n 'ScriptLocation': glue_script_location,\n 'PythonVersion': '3'\n },\n DefaultArguments={\n '--job-language': 'python'\n },\n GlueVersion='2.0',\n WorkerType='Standard',\n NumberOfWorkers=2,\n Timeout=60\n)",
"_____no_output_____"
]
],
[
[
"### Create the AWS Lambda Function",
"_____no_output_____"
]
],
[
[
"import zipfile\nzip_name = 'lambda_training_job_status.zip'\nlambda_source_code = './code/lambda_training_job_status.py'\n\nzf = zipfile.ZipFile(zip_name, mode='w')\nzf.write(lambda_source_code, arcname=lambda_source_code.split('/')[-1])\nzf.close()\n\n\nS3Uploader.upload(local_path=zip_name, \n desired_s3_uri='s3://{}/{}'.format(bucket, project_name),\n sagemaker_session=session)",
"_____no_output_____"
],
[
"lambda_client = boto3.client('lambda')\n\nresponse = lambda_client.create_function(\n FunctionName=function_name,\n Runtime='python3.7',\n Role=lambda_role,\n Handler='lambda_training_job_status.lambda_handler',\n Code={\n 'S3Bucket': bucket,\n 'S3Key': '{}/{}'.format(project_name, zip_name)\n },\n Description='Queries a SageMaker training job and return the results.',\n Timeout=15,\n MemorySize=128\n)",
"_____no_output_____"
]
],
[
[
"### Configure the AWS SageMaker Estimator",
"_____no_output_____"
]
],
[
[
"container = sagemaker.image_uris.retrieve('xgboost', region, '1.2-1')\n\nxgb = sagemaker.estimator.Estimator(container,\n sagemaker_execution_role, \n train_instance_count=1, \n train_instance_type='ml.m4.xlarge',\n output_path='s3://{}/{}/output'.format(bucket, project_name))\n\nxgb.set_hyperparameters(max_depth=5,\n eta=0.2,\n gamma=4,\n min_child_weight=6,\n subsample=0.8,\n objective='binary:logistic',\n eval_metric='error',\n num_round=100)",
"_____no_output_____"
]
],
[
[
"\n## Build a Machine Learning Workflow",
"_____no_output_____"
],
[
"You can use a state machine workflow to create a model training pipeline. The AWS Stepfunctions Data Science SDK provides several AWS SageMaker workflow steps that you can use to construct an ML pipeline. In this tutorial you will create the following steps:\n\n* [**ETLStep**](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/services.html) - Starts an AWS Glue Databrew job to extract the latest data from our source database and prepare our data.\n* [**TrainingStep**](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.sagemaker.TrainingStep) - Creates the training step and passes the defined estimator.\n* [**ModelStep**](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.sagemaker.ModelStep) - Creates a model in SageMaker using the artifacts created during the TrainingStep.\n* [**LambdaStep**](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/compute.html#stepfunctions.steps.compute.LambdaStep) - Creates the task state step within our workflow that calls a Lambda function.\n* [**ChoiceStateStep**](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/states.html#stepfunctions.steps.states.Choice) - Creates the choice state step within our workflow.\n* [**EndpointConfigStep**](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.sagemaker.EndpointConfigStep) - Creates the endpoint config step to define the new configuration for our endpoint.\n* [**EndpointStep**](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.sagemaker.EndpointStep) - Creates the endpoint step to update our model endpoint.\n* [**FailStateStep**](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/states.html#stepfunctions.steps.states.Fail) - Creates fail state step within our workflow.",
"_____no_output_____"
]
],
[
[
"# SageMaker expects unique names for each job, model and endpoint. \n# If these names are not unique the execution will fail.\nexecution_input = ExecutionInput(schema={\n 'TrainingJobName': str,\n 'DatabrewJobName': str,\n 'GlueETLJobName': str,\n 'ModelName': str,\n 'EndpointName': str,\n 'LambdaFunctionName': str\n})",
"_____no_output_____"
]
],
[
[
"### Create a step with AWS GlueDataBrew recipe Job\nIn the following cell, we create a DataBrew step that runs an AWS Glue DataBrew recipe job. The Glue job extracts the latest data from our source location, removes unnecessary columns, and perform few data cleansing operations. AWS Glue DataBrew is performing this extraction, transformation, and load (ETL) in a serverless fashion, so there are no compute resources to configure and manage.",
"_____no_output_____"
]
],
[
[
"recipe_step = steps.GlueDataBrewStartJobRunStep(\n 'Extract, Transform, Load',\n parameters={\"Name\": execution_input['DatabrewJobName']}\n)",
"_____no_output_____"
]
],
[
[
"### Create an ETL step with AWS Glue Job\nIn the following cell, we create a Glue step thats runs an AWS Glue job. The Glue job splits the data in to training and validation sets, and saves the data to CSV format in S3. Glue is performing this extraction, transformation, and load (ETL) in a serverless fashion, so there are no compute resources to configure and manage. See the [GlueStartJobRunStep](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/compute.html#stepfunctions.steps.compute.GlueStartJobRunStep) Compute step in the AWS Step Functions Data Science SDK documentation.",
"_____no_output_____"
]
],
[
[
"etl_step = steps.GlueStartJobRunStep('Split Train & Test DataSet',\n parameters={\"JobName\": execution_input['GlueETLJobName'],\n \"Arguments\":{\n '--S3_SOURCE': recipe_data,\n '--S3_DEST': 's3a://{}/{}/'.format(bucket, project_name),\n '--TRAIN_KEY': train_prefix + '/',\n '--VAL_KEY': val_prefix +'/'}\n }\n)",
"_____no_output_____"
]
],
[
[
"### Create a SageMaker Training Step \n\nIn the following cell, we create the training step and pass the estimator we defined above. See [TrainingStep](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.sagemaker.TrainingStep) in the AWS Step Functions Data Science SDK documentation to learn more.",
"_____no_output_____"
]
],
[
[
"training_step = steps.TrainingStep(\n 'Model Training', \n estimator=xgb,\n data={\n 'train': TrainingInput(train_data, content_type='text/csv'),\n 'validation': TrainingInput(validation_data, content_type='text/csv')\n },\n job_name=execution_input['TrainingJobName'],\n wait_for_completion=True\n)",
"_____no_output_____"
]
],
[
[
"### Create a Model Step \n\nIn the following cell, we define a model step that will create a model in Amazon SageMaker using the artifacts created during the TrainingStep. See [ModelStep](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.sagemaker.ModelStep) in the AWS Step Functions Data Science SDK documentation to learn more.\n\nThe model creation step typically follows the training step. The Step Functions SDK provides the [get_expected_model](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.sagemaker.TrainingStep.get_expected_model) method in the TrainingStep class to provide a reference for the trained model artifacts. Please note that this method is only useful when the ModelStep directly follows the TrainingStep.",
"_____no_output_____"
]
],
[
[
"model_step = steps.ModelStep(\n 'Save Model',\n model=training_step.get_expected_model(),\n model_name=execution_input['ModelName'],\n result_path='$.ModelStepResults'\n)",
"_____no_output_____"
]
],
[
[
"### Create a Lambda Step\nIn the following cell, we define a lambda step that will invoke the previously created lambda function as part of our Step Function workflow. See [LambdaStep](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/compute.html#stepfunctions.steps.compute.LambdaStep) in the AWS Step Functions Data Science SDK documentation to learn more.",
"_____no_output_____"
]
],
[
[
"lambda_step = steps.compute.LambdaStep(\n 'Query Training Results',\n parameters={ \n \"FunctionName\": execution_input['LambdaFunctionName'],\n 'Payload':{\n \"TrainingJobName.$\": '$.TrainingJobName'\n }\n }\n)",
"_____no_output_____"
]
],
[
[
"### Create a Choice State Step \nIn the following cell, we create a choice step in order to build a dynamic workflow. This choice step branches based off of the results of our SageMaker training step: did the training job fail or should the model be saved and the endpoint be updated? We will add specific rules to this choice step later in this notebook.",
"_____no_output_____"
]
],
[
[
"check_accuracy_step = steps.states.Choice(\n 'Accuracy > 90%'\n)",
"_____no_output_____"
]
],
[
[
"### Create an Endpoint Configuration Step\nIn the following cell we create an endpoint configuration step. See [EndpointConfigStep](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.sagemaker.EndpointConfigStep) in the AWS Step Functions Data Science SDK documentation to learn more.",
"_____no_output_____"
]
],
[
[
"endpoint_config_step = steps.EndpointConfigStep(\n \"Create Model Endpoint Config\",\n endpoint_config_name=execution_input['ModelName'],\n model_name=execution_input['ModelName'],\n initial_instance_count=1,\n instance_type='ml.m4.xlarge'\n)",
"_____no_output_____"
]
],
[
[
"### Update the Model Endpoint Step\nIn the following cell, we create the Endpoint step to deploy the new model as a managed API endpoint, updating an existing SageMaker endpoint if our choice state is successful.",
"_____no_output_____"
]
],
[
[
"endpoint_step = steps.EndpointStep(\n 'Update Model Endpoint',\n endpoint_name=execution_input['EndpointName'],\n endpoint_config_name=execution_input['ModelName'],\n update=False\n)",
"_____no_output_____"
]
],
[
[
"### Create the Fail State Step\nIn addition, we create a Fail step which proceeds from our choice state if the validation accuracy of our model is lower than the threshold we define. See [FailStateStep](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/states.html#stepfunctions.steps.states.Fail) in the AWS Step Functions Data Science SDK documentation to learn more.",
"_____no_output_____"
]
],
[
[
"fail_step = steps.states.Fail(\n 'Model Accuracy Too Low',\n comment='Validation accuracy lower than threshold'\n)",
"_____no_output_____"
]
],
[
[
"### Add Rules to Choice State\nIn the cells below, we add a threshold rule to our choice state. Therefore, if the validation accuracy of our model is below 0.90, we move to the Fail State. If the validation accuracy of our model is above 0.90, we move to the save model step with proceeding endpoint update. See [here](https://github.com/dmlc/xgboost/blob/master/doc/parameter.rst) for more information on how XGBoost calculates classification error.\n\nFor binary classification problems the XGBoost algorithm defines the model error as: \n\n\\begin{equation*}\n\\frac{incorret\\:predictions}{total\\:number\\:of\\:predictions}\n\\end{equation*}\n\nTo achieve an accuracy of 90%, we need error <.10.",
"_____no_output_____"
]
],
[
[
"threshold_rule = steps.choice_rule.ChoiceRule.NumericLessThan(variable=lambda_step.output()['Payload']['trainingMetrics'][0]['Value'], value=.1)\n\ncheck_accuracy_step.add_choice(rule=threshold_rule, next_step=endpoint_config_step)\ncheck_accuracy_step.default_choice(next_step=fail_step)",
"_____no_output_____"
]
],
[
[
"### Link all the Steps Together\nFinally, create your workflow definition by chaining all of the steps together that we've created. See [Chain](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/sagemaker.html#stepfunctions.steps.states.Chain) in the AWS Step Functions Data Science SDK documentation to learn more.",
"_____no_output_____"
]
],
[
[
"endpoint_config_step.next(endpoint_step)",
"_____no_output_____"
],
[
"workflow_definition = steps.Chain([\n recipe_step,\n etl_step,\n training_step,\n model_step,\n lambda_step,\n check_accuracy_step\n])",
"_____no_output_____"
]
],
[
[
"## Run the Workflow\nCreate your workflow using the workflow definition above, and render the graph with [render_graph](https://aws-step-functions-data-science-sdk.readthedocs.io/en/latest/workflow.html#stepfunctions.workflow.Workflow.render_graph):",
"_____no_output_____"
]
],
[
[
"workflow = Workflow(\n name='MarketingCampaignInference_{}'.format(id),\n definition=workflow_definition,\n role=workflow_execution_role,\n execution_input=execution_input\n)\n\n# render workflow graph\nworkflow.render_graph()\n\n# create workflow\nworkflow.create()\n\n",
"_____no_output_____"
],
[
"# run the workflow\nexecution = workflow.execute(\n inputs={\n 'TrainingJobName': 'regression-{}'.format(id), # Each Sagemaker Job requires a unique name,\n 'DatabrewJobName': recipe_job_name,\n 'GlueETLJobName': etl_job_name,\n 'ModelName': 'MarketingCampaignPrediction-{}'.format(id), # Each Model requires a unique name,\n 'EndpointName': 'MarketingCampaign', # Each Endpoint requires a unique name\n 'LambdaFunctionName': function_name\n }\n)",
"_____no_output_____"
]
],
[
[
"## Step function output\n\n",
"_____no_output_____"
],
[
"## Clean Up\nWhen you are done, make sure to clean up your AWS account by deleting resources you won't be reusing.",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
]
] |
e7ea0e2d2a03e0fd3f261d60e58abe72506ae118 | 99,490 | ipynb | Jupyter Notebook | Delivery_time.ipynb | Ashish-Dhage7/Simple_Linear_Regression | a9a89320b21cb551f12b32fb0b690b64f340b73d | [
"MIT"
] | null | null | null | Delivery_time.ipynb | Ashish-Dhage7/Simple_Linear_Regression | a9a89320b21cb551f12b32fb0b690b64f340b73d | [
"MIT"
] | null | null | null | Delivery_time.ipynb | Ashish-Dhage7/Simple_Linear_Regression | a9a89320b21cb551f12b32fb0b690b64f340b73d | [
"MIT"
] | null | null | null | 86.43788 | 16,084 | 0.735561 | [
[
[
"import pandas as pd\nimport numpy as np\nimport pandas_profiling as pp\nimport sweetviz as sv\nimport seaborn as sns\nimport statsmodels.formula.api as smf\nfrom ml_metrics import rmse\nfrom matplotlib import pyplot as plt",
"_____no_output_____"
],
[
"data=pd.read_csv(\"delivery_time.csv\")\n#data.head()\nDelivery_data=data.rename(columns={'Delivery Time':'Delivery_Time','Sorting Time':'Sorting_Time'})\nDelivery_data.head()",
"_____no_output_____"
],
[
"#Structure of data\ntype(Delivery_data)\nDelivery_data.shape",
"_____no_output_____"
],
[
"#data types\nDelivery_data.dtypes",
"_____no_output_____"
],
[
"#Checking Null values\nDelivery_data.isnull().sum()\n\n#NO Null Values",
"_____no_output_____"
],
[
"Delivery_data.info()",
"<class 'pandas.core.frame.DataFrame'>\nRangeIndex: 21 entries, 0 to 20\nData columns (total 2 columns):\n # Column Non-Null Count Dtype \n--- ------ -------------- ----- \n 0 Delivery_Time 21 non-null float64\n 1 Sorting_Time 21 non-null int64 \ndtypes: float64(1), int64(1)\nmemory usage: 464.0 bytes\n"
],
[
"#Print the duplicated rows\nDelivery_data[Delivery_data.duplicated()]\n\n#NO Duplicate values in any rows ",
"_____no_output_____"
],
[
"#Box plot\nDelivery_data.boxplot(column=['Delivery_Time'])\n#No outliers in Delivery_Time column",
"_____no_output_____"
],
[
"#Box plot\nDelivery_data.boxplot(column=['Sorting_Time'])\n#No outliers in Sorting_Time column",
"_____no_output_____"
],
[
"#Descriptive stat of Delivery_Time\nDelivery_data['Delivery_Time'].describe()",
"_____no_output_____"
],
[
"#Descriptive stat of Sorting_Time\nDelivery_data['Sorting_Time'].describe()",
"_____no_output_____"
],
[
"# Create the default pairplot\nsns.pairplot(Delivery_data)",
"_____no_output_____"
],
[
"Delivery_data.corr()",
"_____no_output_____"
],
[
"#Building our first model\nmodel1=smf.ols(\"Delivery_Time~Sorting_Time\",data=Delivery_data).fit()\nmodel1.summary()",
"_____no_output_____"
],
[
"#Building our second model(log transformation)\nmodel2=smf.ols(\"Delivery_Time~np.log(Sorting_Time)\",data=Delivery_data).fit()\nmodel2.summary()",
"_____no_output_____"
],
[
"#Building our third model(Exponential transformation)\nmodel3=smf.ols(\"np.log(Delivery_Time)~Sorting_Time\",data=Delivery_data).fit()\nmodel3.summary()",
"_____no_output_____"
],
[
"#Building our fourth model(Quadratic model)\nDelivery_data[\"Sorting_Time_sq\"] = Delivery_data.Sorting_Time*Delivery_data.Sorting_Time\nmodel4=smf.ols(\"Delivery_Time~Sorting_Time+Sorting_Time_sq\",data=Delivery_data).fit()\nmodel4.summary()",
"_____no_output_____"
],
[
"#Storing R^2 values in a data frame\nrsqr_model1=model1.rsquared\nrsqr_model2=model2.rsquared\nrsqr_model3=model3.rsquared\nrsqr_model4=model4.rsquared\n\nd1={'Model':['model1','model2','model3','model4'],'R^2 values':[rsqr_model1,rsqr_model2,rsqr_model3,rsqr_model4]}\nrsqr_frame=pd.DataFrame(d1)\nrsqr_frame\n#After Analysis of all 4 models we conclude model3 has highest R^2 value so we choose model3 as our final model ",
"_____no_output_____"
],
[
"#After Analysis of all 4 models we conclude model3 has highest R^2 value so we choose model3 as our final model\npred_Model3_log=model3.predict(Delivery_data.Sorting_Time)\npred_Model3=np.exp(pred_Model3_log) # as we have used log(AT) in preparing model so we need to convert it back\nrmse(pred_Model3,Delivery_data.Delivery_Time)",
"_____no_output_____"
],
[
"#Predicted delivery time using sorting time \npred_Model3.head()",
"_____no_output_____"
],
[
"# Visualization of regresion line over the scatter plot YearsofExperience and Salary\nplt.scatter(x=Delivery_data['Sorting_Time'],y=Delivery_data['Delivery_Time'],color='green');plt.plot(Delivery_data['Sorting_Time'],pred_Model3,color='blue');plt.xlabel('Sorting_Time');plt.ylabel('Delivery_Time')",
"_____no_output_____"
],
[
"# so we will conclude the model3 has highest R^2 value \n# getting residuals of the entire data set\nresid = model3.resid_pearson \nresid\nplt.plot(resid,'o');plt.axhline(y=0,color='green');plt.xlabel(\"Observation Number\");plt.ylabel(\"Standardized Residual\")",
"_____no_output_____"
],
[
"# Predicted vs actual values\nplt.scatter(x=pred_Model3,y=Delivery_data.Delivery_Time);plt.xlabel(\"Predicted Delivery_Time\");plt.ylabel(\"Actual Delivery_Time\")",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ea19095089c3e8b09e7deebd954d27801b54b3 | 40,516 | ipynb | Jupyter Notebook | chapter_6_exercises.ipynb | JuliaNeumann/nltk_book_exercises | 2021832e3c9ff3a1a911de9b86094efb0e696f00 | [
"MIT"
] | 44 | 2017-11-11T01:49:12.000Z | 2022-03-25T08:09:04.000Z | chapter_6_exercises.ipynb | ramashwink/nltk_book_exercises | 2021832e3c9ff3a1a911de9b86094efb0e696f00 | [
"MIT"
] | null | null | null | chapter_6_exercises.ipynb | ramashwink/nltk_book_exercises | 2021832e3c9ff3a1a911de9b86094efb0e696f00 | [
"MIT"
] | 29 | 2017-10-05T02:59:42.000Z | 2022-03-25T08:08:38.000Z | 48.580336 | 354 | 0.450884 | [
[
[
"__Exercise 1__",
"_____no_output_____"
]
],
[
[
"# MACHINE TRANSLATION:\n# see e.g. http://www.aclweb.org/anthology/R11-1077, https://nlp.stanford.edu/courses/cs224n/2010/reports/bipins.pdf\n# data: parallel corpora, aligned at sentence level (automatically or manually)\n# size: usually assumed the larger the better, 2nd paper: 100,00 documents\n# reasons for large amount: probability that a word or combination of words has been seen during training increases",
"_____no_output_____"
]
],
[
[
"__Exercise 2__",
"_____no_output_____"
]
],
[
[
"import nltk\nfrom nltk.corpus import names\nimport random\nnames = ([(name, 'male') for name in names.words('male.txt')] + [(name, 'female') for name in names.words('female.txt')])\nrandom.shuffle(names)\ntest, devtest, training = names[:500], names[500:1000], names[1000:]\n\ndef gender_features1(name):\n features = {}\n features[\"firstletter\"] = name[0].lower()\n features[\"lastletter\"] = name[-1].lower()\n for letter in 'abcdefghijklmnopqrstuvwxyz':\n features[\"count(%s)\" % letter] = name.lower().count(letter)\n features[\"has(%s)\" % letter] = (letter in name.lower())\n features[\"suffix2\"] = name[-2:].lower()\n return features\n\ntrain_set = [(gender_features1(n), g) for (n,g) in training]\ndevtest_set = [(gender_features1(n), g) for (n,g) in devtest]\nclassifier = nltk.NaiveBayesClassifier.train(train_set)\nprint nltk.classify.accuracy(classifier, devtest_set)",
"0.786\n"
],
[
"def error_analysis(gender_features):\n errors = []\n for (name, tag) in devtest:\n guess = classifier.classify(gender_features(name))\n if guess != tag:\n errors.append((tag, guess, name))\n print 'no. of errors: ', len(errors) \n \n for (tag, guess, name) in sorted(errors): # doctest: +ELLIPSIS +NORMALIZE_WHITESPACE\n print 'correct=%-8s guess=%-8s name=%-30s' % (tag, guess, name) \n \nerror_analysis(gender_features1) ",
"no. of errors: 107\ncorrect=female guess=male name=Alison \ncorrect=female guess=male name=Aubry \ncorrect=female guess=male name=Beilul \ncorrect=female guess=male name=Bell \ncorrect=female guess=male name=Beret \ncorrect=female guess=male name=Bess \ncorrect=female guess=male name=Betsey \ncorrect=female guess=male name=Bidget \ncorrect=female guess=male name=Brear \ncorrect=female guess=male name=Cherry \ncorrect=female guess=male name=Christen \ncorrect=female guess=male name=Christian \ncorrect=female guess=male name=Cody \ncorrect=female guess=male name=Con \ncorrect=female guess=male name=Deerdre \ncorrect=female guess=male name=Demetris \ncorrect=female guess=male name=Dorry \ncorrect=female guess=male name=Elsbeth \ncorrect=female guess=male name=Em \ncorrect=female guess=male name=Emmy \ncorrect=female guess=male name=Esme \ncorrect=female guess=male name=Estell \ncorrect=female guess=male name=Ethyl \ncorrect=female guess=male name=Florence \ncorrect=female guess=male name=Friederike \ncorrect=female guess=male name=Gabriell \ncorrect=female guess=male name=Gunvor \ncorrect=female guess=male name=Gwennie \ncorrect=female guess=male name=Gwyneth \ncorrect=female guess=male name=Harmony \ncorrect=female guess=male name=Hester \ncorrect=female guess=male name=Hope \ncorrect=female guess=male name=Janifer \ncorrect=female guess=male name=Janot \ncorrect=female guess=male name=Jody \ncorrect=female guess=male name=Judith \ncorrect=female guess=male name=Kim \ncorrect=female guess=male name=Kore \ncorrect=female guess=male name=Margery \ncorrect=female guess=male name=Margret \ncorrect=female guess=male name=Marion \ncorrect=female guess=male name=Marry \ncorrect=female guess=male name=Meg \ncorrect=female guess=male name=Meggan \ncorrect=female guess=male name=Mercy \ncorrect=female guess=male name=Olympe \ncorrect=female guess=male name=Patsy \ncorrect=female guess=male name=Philis \ncorrect=female guess=male name=Philly \ncorrect=female guess=male name=Pier \ncorrect=female guess=male name=Raquel \ncorrect=female guess=male name=Roseann \ncorrect=female guess=male name=Sapphire \ncorrect=female guess=male name=Scarlet \ncorrect=female guess=male name=Shannon \ncorrect=female guess=male name=Sharyl \ncorrect=female guess=male name=Solange \ncorrect=female guess=male name=Sophey \ncorrect=female guess=male name=Van \ncorrect=female guess=male name=Wendy \ncorrect=female guess=male name=Wilone \ncorrect=female guess=male name=Winnah \ncorrect=male guess=female name=Abby \ncorrect=male guess=female name=Allan \ncorrect=male guess=female name=Arnie \ncorrect=male guess=female name=Bailey \ncorrect=male guess=female name=Baily \ncorrect=male guess=female name=Benjy \ncorrect=male guess=female name=Bubba \ncorrect=male guess=female name=Charlie \ncorrect=male guess=female name=Dwane \ncorrect=male guess=female name=Elisha \ncorrect=male guess=female name=Emanuel \ncorrect=male guess=female name=Fonsie \ncorrect=male guess=female name=Franklyn \ncorrect=male guess=female name=Hannibal \ncorrect=male guess=female name=Jeramie \ncorrect=male guess=female name=Jere \ncorrect=male guess=female name=Jermayne \ncorrect=male guess=female name=Jerrie \ncorrect=male guess=female name=Jimmie \ncorrect=male guess=female name=Jude \ncorrect=male guess=female name=Kevin \ncorrect=male guess=female name=Kyle \ncorrect=male guess=female name=Lawrence \ncorrect=male guess=female name=Lazare \ncorrect=male guess=female name=Lin \ncorrect=male guess=female name=Lonnie \ncorrect=male guess=female name=Michele \ncorrect=male guess=female name=Micky \ncorrect=male guess=female name=Moishe \ncorrect=male guess=female name=Neil \ncorrect=male guess=female name=Noble \ncorrect=male guess=female name=Odie \ncorrect=male guess=female name=Prentice \ncorrect=male guess=female name=Reza \ncorrect=male guess=female name=Ricki \ncorrect=male guess=female name=Ronnie \ncorrect=male guess=female name=Samuele \ncorrect=male guess=female name=Sly \ncorrect=male guess=female name=Sydney \ncorrect=male guess=female name=Tann \ncorrect=male guess=female name=Terence \ncorrect=male guess=female name=Uli \ncorrect=male guess=female name=Vail \ncorrect=male guess=female name=Val \ncorrect=male guess=female name=Vasili \n"
],
[
"def gender_features2(name):\n features = {}\n features[\"firstletter\"] = name[0].lower()\n features[\"lastletter\"] = name[-1].lower()\n for letter in 'abcdefghijklmnopqrstuvwxyz':\n features[\"count(%s)\" % letter] = name.lower().count(letter)\n features[\"has(%s)\" % letter] = (letter in name.lower())\n features[\"suffix2\"] = name[-2:].lower()\n features[\"suffix3\"] = name[-3:].lower()\n return features\n\ntrain_set = [(gender_features2(n), g) for (n,g) in training]\ndevtest_set = [(gender_features2(n), g) for (n,g) in devtest]\nclassifier = nltk.NaiveBayesClassifier.train(train_set)\nprint nltk.classify.accuracy(classifier, devtest_set)",
"0.792\n"
],
[
"error_analysis(gender_features2)",
"no. of errors: 104\ncorrect=female guess=male name=Alison \ncorrect=female guess=male name=Aubry \ncorrect=female guess=male name=Beilul \ncorrect=female guess=male name=Bell \ncorrect=female guess=male name=Beret \ncorrect=female guess=male name=Bess \ncorrect=female guess=male name=Betsey \ncorrect=female guess=male name=Bidget \ncorrect=female guess=male name=Brear \ncorrect=female guess=male name=Cal \ncorrect=female guess=male name=Cherry \ncorrect=female guess=male name=Christen \ncorrect=female guess=male name=Christian \ncorrect=female guess=male name=Cody \ncorrect=female guess=male name=Con \ncorrect=female guess=male name=Demetris \ncorrect=female guess=male name=Dorry \ncorrect=female guess=male name=Ealasaid \ncorrect=female guess=male name=Elsbeth \ncorrect=female guess=male name=Em \ncorrect=female guess=male name=Emmy \ncorrect=female guess=male name=Esme \ncorrect=female guess=male name=Estell \ncorrect=female guess=male name=Ethyl \ncorrect=female guess=male name=Florence \ncorrect=female guess=male name=France \ncorrect=female guess=male name=Friederike \ncorrect=female guess=male name=Gabriell \ncorrect=female guess=male name=Gillan \ncorrect=female guess=male name=Gunvor \ncorrect=female guess=male name=Gwyneth \ncorrect=female guess=male name=Harmony \ncorrect=female guess=male name=Hester \ncorrect=female guess=male name=Hope \ncorrect=female guess=male name=Janifer \ncorrect=female guess=male name=Janot \ncorrect=female guess=male name=Jody \ncorrect=female guess=male name=Judith \ncorrect=female guess=male name=Kim \ncorrect=female guess=male name=Margery \ncorrect=female guess=male name=Margret \ncorrect=female guess=male name=Marion \ncorrect=female guess=male name=Marry \ncorrect=female guess=male name=Meg \ncorrect=female guess=male name=Meggan \ncorrect=female guess=male name=Mercy \ncorrect=female guess=male name=Murial \ncorrect=female guess=male name=Olympe \ncorrect=female guess=male name=Patsy \ncorrect=female guess=male name=Paule \ncorrect=female guess=male name=Philis \ncorrect=female guess=male name=Pier \ncorrect=female guess=male name=Raquel \ncorrect=female guess=male name=Sal \ncorrect=female guess=male name=Scarlet \ncorrect=female guess=male name=Shannon \ncorrect=female guess=male name=Sharyl \ncorrect=female guess=male name=Sophey \ncorrect=female guess=male name=Van \ncorrect=female guess=male name=Wendy \ncorrect=female guess=male name=Wilone \ncorrect=female guess=male name=Winnah \ncorrect=male guess=female name=Abby \ncorrect=male guess=female name=Allan \ncorrect=male guess=female name=Arnie \ncorrect=male guess=female name=Bailey \ncorrect=male guess=female name=Baily \ncorrect=male guess=female name=Benjy \ncorrect=male guess=female name=Bubba \ncorrect=male guess=female name=Carroll \ncorrect=male guess=female name=Charlie \ncorrect=male guess=female name=Dwane \ncorrect=male guess=female name=Elisha \ncorrect=male guess=female name=Fonsie \ncorrect=male guess=female name=Franklyn \ncorrect=male guess=female name=Hannibal \ncorrect=male guess=female name=Herrmann \ncorrect=male guess=female name=Jeramie \ncorrect=male guess=female name=Jere \ncorrect=male guess=female name=Jermayne \ncorrect=male guess=female name=Jerrie \ncorrect=male guess=female name=Jimmie \ncorrect=male guess=female name=Jude \ncorrect=male guess=female name=Kyle \ncorrect=male guess=female name=Lazare \ncorrect=male guess=female name=Lin \ncorrect=male guess=female name=Lonnie \ncorrect=male guess=female name=Michele \ncorrect=male guess=female name=Micky \ncorrect=male guess=female name=Neil \ncorrect=male guess=female name=Noble \ncorrect=male guess=female name=Odie \ncorrect=male guess=female name=Penny \ncorrect=male guess=female name=Prentice \ncorrect=male guess=female name=Reza \ncorrect=male guess=female name=Ricki \ncorrect=male guess=female name=Ronnie \ncorrect=male guess=female name=Samuele \ncorrect=male guess=female name=Sydney \ncorrect=male guess=female name=Tann \ncorrect=male guess=female name=Tobie \ncorrect=male guess=female name=Uli \ncorrect=male guess=female name=Vail \ncorrect=male guess=female name=Vasili \n"
],
[
"def gender_features3(name):\n features = {}\n features[\"firstletter\"] = name[0].lower()\n features[\"lastletter\"] = name[-1].lower()\n for letter in 'abcdefghijklmnopqrstuvwxyz':\n features[\"count(%s)\" % letter] = name.lower().count(letter)\n features[\"has(%s)\" % letter] = (letter in name.lower())\n features[\"suffix2\"] = name[-2:].lower()\n features[\"suffix3\"] = name[-3:].lower()\n features[\"prefix3\"] = name[:3].lower()\n return features\n\ntrain_set = [(gender_features3(n), g) for (n,g) in training]\ndevtest_set = [(gender_features3(n), g) for (n,g) in devtest]\nclassifier = nltk.NaiveBayesClassifier.train(train_set)\nprint nltk.classify.accuracy(classifier, devtest_set)",
"0.8\n"
],
[
"error_analysis(gender_features3)",
"no. of errors: 100\ncorrect=female guess=male name=Alison \ncorrect=female guess=male name=Aubry \ncorrect=female guess=male name=Beilul \ncorrect=female guess=male name=Bell \ncorrect=female guess=male name=Beret \ncorrect=female guess=male name=Bess \ncorrect=female guess=male name=Bidget \ncorrect=female guess=male name=Brear \ncorrect=female guess=male name=Caitrin \ncorrect=female guess=male name=Cal \ncorrect=female guess=male name=Cherry \ncorrect=female guess=male name=Christen \ncorrect=female guess=male name=Christian \ncorrect=female guess=male name=Cody \ncorrect=female guess=male name=Con \ncorrect=female guess=male name=Demetris \ncorrect=female guess=male name=Dorry \ncorrect=female guess=male name=Ealasaid \ncorrect=female guess=male name=Em \ncorrect=female guess=male name=Emmy \ncorrect=female guess=male name=Esme \ncorrect=female guess=male name=Ethyl \ncorrect=female guess=male name=France \ncorrect=female guess=male name=Friederike \ncorrect=female guess=male name=Gabriell \ncorrect=female guess=male name=Gillan \ncorrect=female guess=male name=Gunvor \ncorrect=female guess=male name=Gwyneth \ncorrect=female guess=male name=Harmony \ncorrect=female guess=male name=Hester \ncorrect=female guess=male name=Hope \ncorrect=female guess=male name=Janifer \ncorrect=female guess=male name=Janot \ncorrect=female guess=male name=Jody \ncorrect=female guess=male name=Judith \ncorrect=female guess=male name=Kim \ncorrect=female guess=male name=Margery \ncorrect=female guess=male name=Margret \ncorrect=female guess=male name=Marion \ncorrect=female guess=male name=Meg \ncorrect=female guess=male name=Mercy \ncorrect=female guess=male name=Murial \ncorrect=female guess=male name=Nicol \ncorrect=female guess=male name=Olympe \ncorrect=female guess=male name=Patsy \ncorrect=female guess=male name=Paule \ncorrect=female guess=male name=Philis \ncorrect=female guess=male name=Philly \ncorrect=female guess=male name=Pier \ncorrect=female guess=male name=Raquel \ncorrect=female guess=male name=Sal \ncorrect=female guess=male name=Scarlet \ncorrect=female guess=male name=Shannon \ncorrect=female guess=male name=Solange \ncorrect=female guess=male name=Sophey \ncorrect=female guess=male name=Van \ncorrect=female guess=male name=Wendy \ncorrect=female guess=male name=Wilone \ncorrect=female guess=male name=Winnah \ncorrect=male guess=female name=Abby \ncorrect=male guess=female name=Allan \ncorrect=male guess=female name=Arnie \ncorrect=male guess=female name=Baily \ncorrect=male guess=female name=Bubba \ncorrect=male guess=female name=Carroll \ncorrect=male guess=female name=Charlie \ncorrect=male guess=female name=Dory \ncorrect=male guess=female name=Elisha \ncorrect=male guess=female name=Esteban \ncorrect=male guess=female name=Florian \ncorrect=male guess=female name=Fonsie \ncorrect=male guess=female name=Franklyn \ncorrect=male guess=female name=Hannibal \ncorrect=male guess=female name=Herrmann \ncorrect=male guess=female name=Jeramie \ncorrect=male guess=female name=Jere \ncorrect=male guess=female name=Jermayne \ncorrect=male guess=female name=Jerrie \ncorrect=male guess=female name=Jimmie \ncorrect=male guess=female name=Jo \ncorrect=male guess=female name=Jude \ncorrect=male guess=female name=Kyle \ncorrect=male guess=female name=Lin \ncorrect=male guess=female name=Lonnie \ncorrect=male guess=female name=Michele \ncorrect=male guess=female name=Micky \ncorrect=male guess=female name=Neil \ncorrect=male guess=female name=Noble \ncorrect=male guess=female name=Noe \ncorrect=male guess=female name=Odie \ncorrect=male guess=female name=Prince \ncorrect=male guess=female name=Reza \ncorrect=male guess=female name=Ricki \ncorrect=male guess=female name=Ronnie \ncorrect=male guess=female name=Samuele \ncorrect=male guess=female name=Sydney \ncorrect=male guess=female name=Tann \ncorrect=male guess=female name=Uli \ncorrect=male guess=female name=Val \ncorrect=male guess=female name=Vasili \n"
],
[
"def gender_features4(name):\n features = {}\n features[\"firstletter\"] = name[0].lower()\n features[\"lastletter\"] = name[-1].lower()\n for letter in 'abcdefghijklmnopqrstuvwxyz':\n features[\"count(%s)\" % letter] = name.lower().count(letter)\n features[\"has(%s)\" % letter] = (letter in name.lower())\n features[\"suffix2\"] = name[-2:].lower()\n features[\"suffix3\"] = name[-3:].lower()\n features[\"prefix3\"] = name[:3].lower()\n features[\"num_vowels\"] = len([letter for letter in name if letter in 'aeiouy'])\n return features\n\ntrain_set = [(gender_features4(n), g) for (n,g) in training]\ndevtest_set = [(gender_features4(n), g) for (n,g) in devtest]\nclassifier = nltk.NaiveBayesClassifier.train(train_set)\nprint nltk.classify.accuracy(classifier, devtest_set)",
"0.802\n"
],
[
"# final performance test:\ntest_set = [(gender_features4(n), g) for (n,g) in test]\nprint nltk.classify.accuracy(classifier, test_set)",
"0.82\n"
],
[
"# performance slightly worse than in dev-test -> features reflect some idiosyncracies of dev-test",
"_____no_output_____"
]
],
[
[
"__Exercise 3)__",
"_____no_output_____"
]
],
[
[
"from nltk.corpus import senseval\ninstances = senseval.instances('serve.pos')\nsize = int(len(instances) * 0.1)\ntraining, test = instances[size:], instances[:size]",
"_____no_output_____"
],
[
"training[0]",
"_____no_output_____"
],
[
"def sense_features(instance):\n features = {}\n features[\"word-type\"] = instance.word\n features[\"word-tag\"] = instance.context[instance.position][1] \n features[\"prev-word\"] = instance.context[instance.position-1][0]\n features[\"prev-word-tag\"] = instance.context[instance.position-1][1]\n features[\"next-word\"] = instance.context[instance.position+1][0]\n features[\"next-word-tag\"] = instance.context[instance.position+1][1]\n return features\n\ntrain_set = [(sense_features(instance), instance.senses) for instance in training]\ntest_set = [(sense_features(instance), instance.senses) for instance in test]\nclassifier = nltk.NaiveBayesClassifier.train(train_set)\nprint nltk.classify.accuracy(classifier, test_set)",
"0.807780320366\n"
]
],
[
[
"__Exercise 4)__",
"_____no_output_____"
]
],
[
[
"from nltk.corpus import movie_reviews\ndocuments = [(list(movie_reviews.words(fileid)), category) for category in movie_reviews.categories() for fileid in movie_reviews.fileids(category)]\nrandom.shuffle(documents)\n\nall_words = nltk.FreqDist(w.lower() for w in movie_reviews.words())\nword_features = all_words.keys()[:2000]\ndef document_features(document):\n document_words = set(document)\n features = {}\n for word in word_features:\n features['contains(%s)' % word] = (word in document_words)\n return features\n\nfeaturesets = [(document_features(d), c) for (d,c) in documents]\ntrain_set, test_set = featuresets[100:], featuresets[:100]\nclassifier = nltk.NaiveBayesClassifier.train(train_set)\nprint nltk.classify.accuracy(classifier, test_set)\nclassifier.show_most_informative_features(30)",
"0.77\nMost Informative Features\n contains(doubts) = True pos : neg = 9.8 : 1.0\n contains(sans) = True neg : pos = 8.8 : 1.0\n contains(hugo) = True pos : neg = 7.8 : 1.0\n contains(effortlessly) = True pos : neg = 7.5 : 1.0\n contains(dismissed) = True pos : neg = 7.1 : 1.0\n contains(mediocrity) = True neg : pos = 6.9 : 1.0\n contains(overwhelmed) = True pos : neg = 6.4 : 1.0\n contains(fabric) = True pos : neg = 6.4 : 1.0\n contains(wires) = True neg : pos = 6.2 : 1.0\n contains(wits) = True pos : neg = 5.8 : 1.0\n contains(topping) = True pos : neg = 5.8 : 1.0\n contains(ugh) = True neg : pos = 5.7 : 1.0\n contains(uplifting) = True pos : neg = 5.7 : 1.0\n contains(bruckheimer) = True neg : pos = 5.6 : 1.0\n contains(bounce) = True neg : pos = 5.6 : 1.0\n contains(lang) = True pos : neg = 5.1 : 1.0\n contains(understands) = True pos : neg = 4.5 : 1.0\n contains(matheson) = True pos : neg = 4.4 : 1.0\n contains(controversy) = True pos : neg = 4.3 : 1.0\n contains(quicker) = True neg : pos = 4.3 : 1.0\n contains(maxwell) = True neg : pos = 4.3 : 1.0\n contains(locks) = True neg : pos = 4.3 : 1.0\n contains(tsui) = True neg : pos = 4.3 : 1.0\n contains(admired) = True pos : neg = 4.2 : 1.0\n contains(cronenberg) = True pos : neg = 3.9 : 1.0\n contains(derivative) = True neg : pos = 3.8 : 1.0\n contains(attorney) = True pos : neg = 3.8 : 1.0\n contains(existential) = True pos : neg = 3.7 : 1.0\n contains(bandits) = True pos : neg = 3.7 : 1.0\n contains(restoring) = True pos : neg = 3.7 : 1.0\n"
],
[
"# most of them already indicate some judgment in themselves ('ugh', 'mediocrity') or belong to typical phrases that\n# indicate one direction of judgement ('understands' -> '... understands how to create atmosphere' or something like that)\n# some seem to be names of actors etc. which tend to be judged one direction or the other\n# surprising -> '33', 'wires'",
"_____no_output_____"
]
],
[
[
"__Exercise 5)__",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
]
] |
e7ea2d369c7475da69eef03e8d77f4ebde1f3106 | 1,868 | ipynb | Jupyter Notebook | Lab7_1.ipynb | hocthucv/bt | 7ceb36259c358cb67e77bb0a1148d75e21fd7f97 | [
"Unlicense"
] | null | null | null | Lab7_1.ipynb | hocthucv/bt | 7ceb36259c358cb67e77bb0a1148d75e21fd7f97 | [
"Unlicense"
] | null | null | null | Lab7_1.ipynb | hocthucv/bt | 7ceb36259c358cb67e77bb0a1148d75e21fd7f97 | [
"Unlicense"
] | null | null | null | 26.685714 | 214 | 0.415953 | [
[
[
"<a href=\"https://colab.research.google.com/github/hocthucv/bt/blob/main/Lab7_1.ipynb\" target=\"_parent\"><img src=\"https://colab.research.google.com/assets/colab-badge.svg\" alt=\"Open In Colab\"/></a>",
"_____no_output_____"
]
],
[
[
"def isBalanced(S):\n stack = []\n brac = {\"{\": \"}\", \"[\": \"]\", \"(\" : \")\"}\n for i in S:\n if i in brac.keys():\n stack.append(i)\n elif i in brac.values():\n if i == brac.get(stack[-1]):\n stack.pop()\n else:\n stack.append(i)\n return \"Success\" if not stack else S.index(stack.pop()) + 1\n\nS = input()\nprint(isBalanced(S))",
"{[}\n3\n"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
]
] |
e7ea35a0da710c9ae4c6b100c7a655687686f4ca | 651,675 | ipynb | Jupyter Notebook | data/Generate Plots.ipynb | robertvsiii/kaic-analysis | f80f85bbaeb1190cba2bb18ed732af915374ff9c | [
"MIT"
] | null | null | null | data/Generate Plots.ipynb | robertvsiii/kaic-analysis | f80f85bbaeb1190cba2bb18ed732af915374ff9c | [
"MIT"
] | null | null | null | data/Generate Plots.ipynb | robertvsiii/kaic-analysis | f80f85bbaeb1190cba2bb18ed732af915374ff9c | [
"MIT"
] | null | null | null | 498.603673 | 55,044 | 0.940343 | [
[
[
"<h1>Table of Contents<span class=\"tocSkip\"></span></h1>\n<div class=\"toc\"><ul class=\"toc-item\"><li><span><a href=\"#Figure-1:-Introduction\" data-toc-modified-id=\"Figure-1:-Introduction-1\"><span class=\"toc-item-num\">1 </span>Figure 1: Introduction</a></span></li><li><span><a href=\"#Figure-2:-Model-Performance\" data-toc-modified-id=\"Figure-2:-Model-Performance-2\"><span class=\"toc-item-num\">2 </span>Figure 2: Model Performance</a></span><ul class=\"toc-item\"><li><span><a href=\"#Load-KaiABC-model\" data-toc-modified-id=\"Load-KaiABC-model-2.1\"><span class=\"toc-item-num\">2.1 </span>Load KaiABC model</a></span><ul class=\"toc-item\"><li><span><a href=\"#Estimate-Errors\" data-toc-modified-id=\"Estimate-Errors-2.1.1\"><span class=\"toc-item-num\">2.1.1 </span>Estimate Errors</a></span></li></ul></li><li><span><a href=\"#Plot-KaiABC-model-with-$N_{\\rm-eff}$\" data-toc-modified-id=\"Plot-KaiABC-model-with-$N_{\\rm-eff}$-2.2\"><span class=\"toc-item-num\">2.2 </span>Plot KaiABC model with $N_{\\rm eff}$</a></span></li></ul></li><li><span><a href=\"#Figure-3:-Plot-all-data-together\" data-toc-modified-id=\"Figure-3:-Plot-all-data-together-3\"><span class=\"toc-item-num\">3 </span>Figure 3: Plot all data together</a></span></li><li><span><a href=\"#Figure-4:-New-Model\" data-toc-modified-id=\"Figure-4:-New-Model-4\"><span class=\"toc-item-num\">4 </span>Figure 4: New Model</a></span></li><li><span><a href=\"#Supplemental-Plots\" data-toc-modified-id=\"Supplemental-Plots-5\"><span class=\"toc-item-num\">5 </span>Supplemental Plots</a></span></li></ul></div>",
"_____no_output_____"
]
],
[
[
"import numpy as np\nimport matplotlib.pyplot as plt\nfrom decimal import Decimal\nimport pandas as pd\nimport pickle\nfrom matplotlib.backends import backend_pdf as bpdf\nfrom kaic_analysis.scripts import FirstPassage, RunModel, Current, StateData, FindParam, LoadExperiment, PlotExperiment, EntropyRate\nfrom kaic_analysis.toymodel import SimulateClockKinetic\nimport os\nfrom sklearn.decomposition import PCA\nimport seaborn as sns\nimport scipy.interpolate as interpolate\nimport bootstrapped.bootstrap as bs\nimport bootstrapped.stats_functions as bs_stats\nimport scipy.optimize as opt\n%matplotlib inline\n\ndef var(values, axis=1):\n '''Returns the variance of each row of a matrix'''\n return np.var(np.asmatrix(values), axis=axis).A1\n\ndef compute_maxeig(A,C,N):\n K0 = np.asarray([[-C*N*(np.sin(2*np.pi*(i-1-j)/N)-np.sin(2*np.pi*(i-j)/N))*(1-np.exp(-A/N)) for j in range(N)] for i in range (N)])\n K1 = np.diag(np.ones(N-1),k=1)*N - np.eye(N)*N*(1+np.exp(-A/N)) + np.diag(np.ones(N-1),k=-1)*N*np.exp(-A/N)\n K = K0 + K1\n Keig = np.linalg.eig(K)[0]\n max_ind = np.argmax(np.real(Keig))\n return np.real(Keig[max_ind])\n\ndef compute_maxeig_imag(A,C,N):\n K0 = np.asarray([[-C*N*(np.sin(2*np.pi*(i-1-j)/N)-np.sin(2*np.pi*(i-j)/N))*(1-np.exp(-A/N)) for j in range(N)] for i in range (N)])\n K1 = np.diag(np.ones(N-1),k=1)*N - np.eye(N)*N*(1+np.exp(-A/N)) + np.diag(np.ones(N-1),k=-1)*N*np.exp(-A/N)\n K = K0 + K1\n Keig = np.linalg.eig(K)[0]\n max_ind = np.argmax(np.real(Keig))\n return np.imag(Keig[max_ind])",
"_____no_output_____"
]
],
[
[
"# Figure 1: Introduction",
"_____no_output_____"
]
],
[
[
"fig,ax = plt.subplots(figsize=(3.25,1.75))\nfig.subplots_adjust(left=0.17,bottom=0.25,right=0.95)\n\ncode_folder = '../KMC_KaiC_rev2'\ndata_low=RunModel(folder=code_folder,paramdict={'volume':1,'sample_cnt':1e4,'tequ':50,'rnd_seed':np.random.randint(1e6),'ATPfrac':0.45},name='data_low')\ndata_WT=RunModel(folder=code_folder,paramdict={'volume':1,'sample_cnt':3e5,'tequ':50,'rnd_seed':np.random.randint(1e6)},name='data_WT')\nos.chdir('../data')\n\ndata_WT.index = data_WT.index-data_WT.index[0]\n(data_WT['pT']/6).plot(ax=ax,color='k',legend=False)\n\nax.set_xlim((0,200))\nax.spines['top'].set_visible(False)\nax.spines['right'].set_visible(False)\n\nax.set_xlabel('Time (hours)')\nax.set_ylabel(r'$f_T$')\npdf = bpdf.PdfPages('Plots/timeseries_kaic.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
],
[
"nT = data_WT['pT'].values*360 #volume = 1 corresponds to 360 hexamers\nnS = data_WT['pS'].values*360\nsamp = np.arange(100000,300000,20,dtype=int)\n\nfig,ax = plt.subplots(figsize=(3,3))\nfig.subplots_adjust(left=0.17,bottom=0.25,right=0.95)\nax.plot(nT[samp],nS[samp])\nax.set_aspect('equal', 'box')\nax.set_xlabel('Number of phosphorylated threonines')\nax.set_ylabel('Number of phosphorylated serines')\nax.spines['top'].set_visible(False)\nax.spines['right'].set_visible(False)\npdf = bpdf.PdfPages('Plots/limit.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()\n\nfig,ax = plt.subplots(figsize=(3,3))\nfig.subplots_adjust(left=0.17,bottom=0.25,right=0.9)\nax.plot(nT[100000:],nS[100000:])\nax.set_xlim((500,525))\nax.set_ylim((400,425))\nax.set_aspect('equal', 'box')\nax.set_xlabel('Number of phosphorylated threonines')\nax.set_ylabel('Number of phosphorylated serines')\nax.grid(True)\npdf = bpdf.PdfPages('Plots/zoom.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
],
[
"param_name = 'ATPfrac'\nrun_nums = list(range(17,22))\nfig,ax = plt.subplots(figsize=(3.25,1.75))\nfig.subplots_adjust(left=0.17,bottom=0.25,right=0.95)\n\ndata = LoadExperiment(param_name,run_nums,date='2018-08-24',folder='kaic_data')\n\nbins = np.linspace(0,150,150)\n\nname = 'ATPfrac = 0.99999998477'\nfor Ncyc in range(1,6):\n ax.hist(FirstPassage(data[2][name],Ncyc=Ncyc),bins=bins,density=True,alpha=0.5)\nax.set_xlim((0,150))\nax.set_xlabel('Time (hours)')\nax.set_ylabel('Fraction of runs')\npdf = bpdf.PdfPages('Plots/hist.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
],
[
"Ncyclist = np.arange(1,30)\n\nfig,ax = plt.subplots(figsize=(3.25,1.75))\nfig.subplots_adjust(left=0.17,bottom=0.25,right=0.95)\n\nax.set_xlabel(r'$n$')\nax.set_ylabel(r'${\\rm var}(\\tau_n)$')\n\n\n\nvartau = []\nmeantau = []\nvarw=[]\nmeanw=[]\nfor N in Ncyclist:\n taus = np.asarray(FirstPassage(data[2][name],Ncyc=N))\n bs_mean = bs.bootstrap(taus,stat_func=bs_stats.mean)\n bs_var = bs.bootstrap(taus,stat_func=var)\n vartau.append(bs_var.value)\n meantau.append(bs_mean.value)\n varw.append(2./(bs_var.upper_bound-bs_var.lower_bound))\n meanw.append(2./(bs_mean.upper_bound-bs_mean.lower_bound))\n \nvarw = np.asarray(varw)\nmeanw=np.asarray(meanw)\nvartau = np.asarray(vartau)\nmeantau = np.asarray(meantau)\n \n[slope, intercept], cov = np.polyfit(Ncyclist,vartau,1,w=varw,cov=True)\nax.errorbar(Ncyclist,np.asarray(vartau),yerr=1/np.asarray(varw),color='k',alpha=0.5)\nax.plot(Ncyclist,intercept+slope*Ncyclist,color='k')\n\npdf = bpdf.PdfPages('Plots/D.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
]
],
[
[
"# Figure 2: Model Performance",
"_____no_output_____"
],
[
"## Load KaiABC model",
"_____no_output_____"
]
],
[
[
"param_name = 'ATPfrac'\nrun_nums = list(range(1,13))\ndata = LoadExperiment(param_name,run_nums,date='2018-08-23',folder='kaic_data')[2]\nrun_nums = list(range(13,22))\ndata.update(LoadExperiment(param_name,run_nums,date='2018-08-24',folder='kaic_data')[2])\n\nkeylist = list(data.keys())\nATPfracs = [Decimal(keylist[j].split('=')[1]) for j in range(len(keylist))]\nATPfracs.sort()\nnamelist = [param_name+' = '+str(ATPfracs[j]) for j in range(len(ATPfracs))]\n\nNcyclist = np.arange(1,30)\nD0 = []\nD0_err = []\nD0_2 = []\nD0_2_err = []\nT = []\nvary = []\ncolors = sns.color_palette(\"RdBu_r\",21)\n\nfig,axs = plt.subplots(5,4,figsize=(6.5,9),sharex=True,sharey=True)\nfor k in range(4):\n axs[4,k].set_xlabel('Number of Cycles')\naxs[2,0].set_ylabel('Variance of Completion Time')\n\naxs = axs.reshape(-1)\nfig.subplots_adjust(hspace=0.4)\n\nfig2,ax2 = plt.subplots(5,4,figsize=(6.5,9),sharex=True,sharey=True)\nfor k in range(4):\n ax2[4,k].set_xlabel('Number of Cycles')\nax2[2,0].set_ylabel('Variance of Variance of Completion Time')\nax2 = ax2.reshape(-1)\nfig2.subplots_adjust(hspace=0.4)\n\nk = 0\n\nfor name in namelist[1:]:\n\n vartau = []\n meantau = []\n varw=[]\n meanw=[]\n for N in Ncyclist:\n taus = np.asarray(FirstPassage(data[name],Ncyc=N))\n bs_mean = bs.bootstrap(taus,stat_func=bs_stats.mean,alpha=0.36)\n bs_var = bs.bootstrap(taus,stat_func=var,alpha=0.36)\n vartau.append(bs_var.value)\n meantau.append(bs_mean.value)\n varw.append(2./(bs_var.upper_bound-bs_var.lower_bound))\n meanw.append(2./(bs_mean.upper_bound-bs_mean.lower_bound))\n \n varw = np.asarray(varw)\n meanw=np.asarray(meanw)\n vartau = np.asarray(vartau)\n meantau = np.asarray(meantau)\n \n ax2[k].plot(Ncyclist,1./varw,color = colors[0])\n slope, intercept = np.polyfit(Ncyclist,1./varw,1)\n ax2[k].plot(Ncyclist,intercept+slope*Ncyclist,color=colors[-1])\n vary.append(slope**2)\n \n slope, intercept = np.polyfit(Ncyclist,meantau,1,w=meanw)\n T.append(slope)\n \n [slope, intercept], cov = np.polyfit(Ncyclist,vartau,1,w=varw,cov=True)\n D0.append(slope)\n D0_2.append(vartau[-1]/Ncyclist[-1])\n cov = np.linalg.inv(np.asarray([[2*np.sum(varw**2*Ncyclist**2),2*np.sum(varw**2*Ncyclist)],\n [2*np.sum(varw**2*Ncyclist),2*np.sum(varw**2)]]))\n D0_err.append(np.sqrt(cov[0,0]))\n D0_2_err.append(np.sqrt(1./varw[-1]))\n axs[k].errorbar(Ncyclist,np.asarray(vartau),yerr=1/np.asarray(varw),color=colors[0])\n axs[k].plot(Ncyclist,intercept+slope*Ncyclist,color=colors[-1])\n axs[k].set_title(name[:14])\n k+=1\n \npdf = bpdf.PdfPages('Plots/KaiC_fits.pdf')\npdf.savefig(fig)\npdf.close()\n\npdf = bpdf.PdfPages('Plots/KaiC_fits_var.pdf')\npdf.savefig(fig2)\npdf.close()\nplt.show()\n\nT = np.asarray(T)\nD = np.asarray(D0)/T\nD_err = np.asarray(D0_err)/T\nD2 = np.asarray(D0_2)/T\nD2_err = np.asarray(D0_2_err)/T\nD3_err = np.sqrt(np.asarray(vary))/T\n\n\nrun_nums = list(range(1,13))\ndata = LoadExperiment(param_name,run_nums,date='2018-08-23',folder='kaic_data')[1]\nrun_nums = list(range(13,22))\ndata = data.join(LoadExperiment(param_name,run_nums,date='2018-08-24',folder='kaic_data')[1])\n\nScyc = data[namelist].values[0][1:]*T\n\nwith open('ModelData.dat', 'wb') as f:\n pickle.dump([T, D, D_err, Scyc],f)",
"_____no_output_____"
]
],
[
[
"### Estimate Errors",
"_____no_output_____"
],
[
"The graphs of the bootstrap error versus number of cycles indicate that the standard deviation of the completion time increases linearly with the number of cycles. This is what we would expect to happen if each run of the experiment (making the same number of trajectories and estimating the variance for each number of cycles) produces a slope $D_0+y$, where $y$ has mean 0 and variance $\\sigma_y^2$, and is fixed for each iteration of the simulation.\n\nSpecifically, we have\n\\begin{align}\n{\\rm var}(\\tau_n) &= (D_0 + y)N_{\\rm cyc}\\\\\n\\sqrt{{\\rm var}({\\rm var}(\\tau_n))} &= \\sigma_y N_{\\rm cyc}.\n\\end{align}\n\nUnder this noise model, the uncertainty in the slope is simply $\\sigma_y$.",
"_____no_output_____"
],
[
"## Plot KaiABC model with $N_{\\rm eff}$",
"_____no_output_____"
]
],
[
[
"with open('ModelData.dat','rb') as f:\n [T,D,D_err,Scyc] = pickle.load(f)\n \nDelWmin = 2000\nDelWmax = 3100\nDelWvec = np.exp(np.linspace(np.log(DelWmin),np.log(DelWmax),5000))\n\nM = 180*2\nNeff = 1.1\nNcoh = T/D\nNcoh_err = (T/D**2)*D3_err\nk=0\n\nScyc = Scyc[:-1]\nNcoh = Ncoh[:-1]\nNcoh_err = Ncoh_err[:-1]\n\nfig,ax=plt.subplots(figsize=(3.5,3))\nfig.subplots_adjust(left=0.2,bottom=0.2)\nax.plot(Scyc/M,Ncoh/M,label='KaiABC Model')\nax.errorbar(Scyc/M,Ncoh/M,yerr=Ncoh_err/M,linestyle='',capsize=2)\nax.plot([DelWmin,DelWmax],[Neff,Neff],'k--',label = r'$N_{\\rm eff}/M = $'+str(Neff))\nplt.legend(loc=4)\n\nax.set_xlabel(r'Entropy Production per Cycle $\\Delta S/M$')\nax.set_ylabel(r'Number of Coherent Cycles $\\mathcal{N}/M$')\n\npdf = bpdf.PdfPages('Plots/Figure2.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
]
],
[
[
"# Figure 3: Plot all data together",
"_____no_output_____"
],
[
"Empirical curve:\n\\begin{align}\n\\frac{V}{\\mathcal{N}} - C &= W_0 (W-W_c)^\\alpha\\\\\n\\frac{\\mathcal{N}}{V} &= \\frac{1}{C+ W_0(W-W_c)^\\alpha}\n\\end{align}",
"_____no_output_____"
]
],
[
[
"with open('ModelData.dat','rb') as f:\n [T,D,D_err,Scyc] = pickle.load(f)\n\ndef CaoN(DelW,params = {}):\n N = (params['C']+params['W0']*(DelW-params['Wc'])**params['alpha'])**(-1)\n N[np.where(DelW<params['Wc'])[0]] = np.nan\n return N\n\n#For comparing experiment data:\nVKai = 3e13 #Convert volume to single hexamers (assuming 3.4 uM concentration of monomers, and 100 uL wells)\nal = 1/(2*np.pi**2)\n\n#Parameters from Cao2015\nparamlist = {'Activator-Inhibitor':\n {'C':0.6,\n 'W0':380,\n 'Wc':360,\n 'alpha':-0.99},\n 'AI Envelope':\n {'C':0.36,\n 'W0':194,\n 'Wc':400,\n 'alpha':-1},\n 'Repressilator':\n {'Wc':1.75,\n 'W0':25.9,\n 'alpha':-1.1,\n 'C':0.4},\n 'Brusselator':\n {'Wc':100.4,\n 'W0':846,\n 'alpha':-1.0,\n 'C':0.5},\n 'Glycolysis':\n {'Wc':80.5,\n 'W0':151.4,\n 'alpha':-1.1,\n 'C':0.5},\n 'KaiABC Experiment':\n {'Wc':10.6*16*6,\n 'W0':0.28*16*6*VKai*al,\n 'alpha':-1.0,\n 'C':0.04*VKai*al}}\n\nfig,ax=plt.subplots(figsize=(3.25,3))\nfig.subplots_adjust(bottom=0.15,left=0.2,right=0.95,top=0.95)\ndel paramlist['AI Envelope']\ndel paramlist['KaiABC Experiment']\nDelWmin = 1\nDelWmax = 0.6*10000\nDelWvec = np.exp(np.linspace(np.log(DelWmin),np.log(DelWmax),5000))\nax.plot(DelWvec,DelWvec/2,'k--',label='Thermodynamic Bound')\nfor item in ['Repressilator','Glycolysis','Activator-Inhibitor','Brusselator']:\n ax.plot(DelWvec,CaoN(DelWvec,params=paramlist[item]),label=item)\n\nN = 1\nM = 180*2\nNeff = 1.2*N*M\nNcoh = T/D\nNcoh_err = (T/D**2)*D_err\nk=0\n\nax.plot(Scyc[:-1]/M,Ncoh[:-1]/M,label='KaiABC Model')\n\nplt.legend(loc=1,fontsize=8)\nax.set_ylim((4e-1,2e1))\nax.set_xlim((10,3100))\nax.set_yscale('log')\nax.set_xscale('log')\n\nax.set_xlabel(r'$\\Delta S/M$')\nax.set_ylabel(r'$\\mathcal{N}/M$')\n\npdf = bpdf.PdfPages('Plots/Figure3.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()\n\n",
"/anaconda3/lib/python3.6/site-packages/ipykernel_launcher.py:5: RuntimeWarning: invalid value encountered in power\n \"\"\"\n"
]
],
[
[
"# Figure 4: New Model",
"_____no_output_____"
]
],
[
[
"date = '2018-08-23'\ndate2 = '2018-08-24'\ndate3 = '2018-08-25'\nD0 = []\nD0_err = []\nT = []\nScyc = []\nsigy =[]\ncolors = sns.color_palette(\"RdBu_r\",20)\nk = 0\nfor expt_number in range(3):\n low = 18*expt_number+1\n high = 18*(expt_number+1)\n if expt_number == 2: #Skip simulations that failed\n low = 48\n for run_number in range(low,high):\n try:\n t = pd.read_csv('toy_data/t_'+date+'_'+str(run_number)+'.csv',header=None)\n t = t - t.loc[0]\n data = pd.read_csv('toy_data/data_'+date+'_'+str(run_number)+'.csv',index_col=0)\n except:\n try:\n t = pd.read_csv('toy_data/t_'+date2+'_'+str(run_number)+'.csv',header=None)\n t = t - t.loc[0]\n data = pd.read_csv('toy_data/data_'+date2+'_'+str(run_number)+'.csv',index_col=0)\n except:\n t = pd.read_csv('toy_data/t_'+date3+'_'+str(run_number)+'.csv',header=None)\n t = t - t.loc[0]\n data = pd.read_csv('toy_data/data_'+date3+'_'+str(run_number)+'.csv',index_col=0)\n\n\n fig,axs = plt.subplots(2,figsize=(4,8))\n fig2,axs2 = plt.subplots(2,figsize=(4,8))\n vartau = []\n meantau = []\n varw=[]\n meanw=[]\n sigy=[]\n \n for N in t.index:\n taus = t.loc[N].values\n bs_mean = bs.bootstrap(taus,stat_func=bs_stats.mean)\n bs_var = bs.bootstrap(taus,stat_func=var)\n vartau.append(bs_var.value)\n meantau.append(bs_mean.value)\n varw.append(2./(bs_var.upper_bound-bs_var.lower_bound))\n meanw.append(2./(bs_mean.upper_bound-bs_mean.lower_bound))\n \n varw = np.asarray(varw)\n meanw=np.asarray(meanw)\n vartau = np.asarray(vartau)\n meantau = np.asarray(meantau)\n \n usable = np.where(~np.isnan(meantau))[0]\n usable = usable[1:]\n \n try:\n slope, intercept = np.polyfit(t.index.values[usable],meantau[usable],1,w=meanw[usable])\n T.append(slope)\n axs[0].set_title('A = '+str(data['A'].loc[0]))\n axs[0].errorbar(t.index,meantau,yerr=1/meanw,color=colors[k])\n axs[0].plot(t.index,intercept+slope*t.index,color=colors[k])\n axs[0].set_xlabel('Number of Cycles')\n axs[0].set_ylabel('Mean completion time')\n \n axs2[0].plot(t.index,1./varw)\n [slope, intercept], cov = np.polyfit(t.index.values[usable],1./varw[usable],1,cov=True)\n sigy.append(slope)\n \n [slope, intercept], cov = np.polyfit(t.index.values[usable],vartau[usable],1,w=varw[usable],cov=True)\n D0.append(slope)\n cov = np.linalg.inv(np.asarray([[np.nansum(varw[usable]**2*t.index.values[usable]**2),np.nansum(varw[usable]**2*t.index.values[usable])],\n [np.nansum(varw[usable]**2*t.index.values[usable]),np.nansum(varw[usable]**2)]]))\n\n D0_err.append(np.sqrt(cov[0,0]))\n axs[1].errorbar(t.index,np.asarray(vartau),yerr=1/np.asarray(varw),color=colors[k])\n axs[1].plot(t.index,intercept+slope*t.index,color=colors[k])\n axs[1].set_xlabel('Number of Cycles')\n axs[1].set_ylabel('Variance of completion time')\n Scyc.append(data['Sdot'].mean()*T[-1])\n k+=1\n except:\n print(str(run_number)+' failed!')\n try:\n del T[k]\n except:\n e = 1\n try:\n del D0[k]\n except:\n e = 1\n \n plt.show()\n \n\n T = np.asarray(T)\n D = np.asarray(D0)/T\n D_err = np.asarray(sigy)/T\n\n Scyc = np.asarray(Scyc)\n with open('ToyData_'+str(expt_number)+'_2.dat', 'wb') as f:\n pickle.dump([T, D, D_err, Scyc],f)",
"/anaconda3/lib/python3.6/site-packages/ipykernel_launcher.py:46: RuntimeWarning: divide by zero encountered in double_scalars\n/anaconda3/lib/python3.6/site-packages/ipykernel_launcher.py:47: RuntimeWarning: divide by zero encountered in double_scalars\n"
],
[
"fig,ax=plt.subplots(figsize=(3.25,3))\nfig.subplots_adjust(bottom=0.15,left=0.21,right=0.95,top=0.95)\ncolors = sns.color_palette()\n\nxvec = np.exp(np.linspace(-2,np.log(30),120))\nNlist = [3,10,50]\nM = 100\ncolors = sns.color_palette()\nk=0\nfor n in [2,1,0]:\n with open('ToyData_'+str(n)+'_2.dat', 'rb') as f:\n [T, D, D_err, Scyc] = pickle.load(f)\n N = Nlist[n]\n Neff = 1.3*N*M\n Ncoh = T/D\n Ncoh_err = 2*(T/D**2)*D_err\n\n ax.errorbar(Scyc/M,Ncoh/M,yerr=Ncoh_err/M,color=colors[k],label='N = '+str(N)) \n k+=1\nax.plot(xvec,xvec/2,'--',color='k',label='Thermodynamic Bound')\nplt.legend(loc=2,fontsize=10)\nax.set_xlim((0,30))\nax.set_ylim((0,20))\n\n\nax.set_xlabel(r'$\\Delta S/M$',fontsize=14)\nax.set_ylabel(r'$\\mathcal{N}/M$',fontsize=14)\n\npdf = bpdf.PdfPages('Plots/toymodel.pdf')\npdf.savefig(fig)\npdf.close()\n\nplt.show()",
"_____no_output_____"
]
],
[
[
"# Supplemental Plots",
"_____no_output_____"
]
],
[
[
"data = []\nN = 6\nM = 100\nC = 5\nkwargs = {'tmax':4,'nsteps':1,'N':N,'M':M,'A':4.4,'C':C}\nout1 = SimulateClockKinetic(**kwargs)\nkwargs = {'tmax':4,'nsteps':1,'N':N,'M':M,'A':9.529,'C':C}\nout2 = SimulateClockKinetic(**kwargs)",
"_____no_output_____"
],
[
"fig,ax=plt.subplots()\nax.plot(out1['t'],out1['f'][:,0], label=r'$\\dot{S} = 160\\,k_B/{\\rm hr}$')\nax.plot(out2['t'],out2['f'][:,0], label=r'$\\dot{S} = 880\\,k_B/{\\rm hr}$')\nplt.legend()\nax.set_xlabel('Time (hours)')\nax.set_ylabel('Fraction in state 1')\npdf = bpdf.PdfPages('Plots/timeseries_new.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
],
[
"fig,ax=plt.subplots()\n\nN = 6\nM = 100\nC = 5\n\nskip = 1\nmodel = PCA(n_components=2).fit(out1['f'])\nf1 = model.transform(out1['f'])\nax.plot(f1[np.arange(0,len(f1),skip),0],f1[np.arange(0,len(f1),skip),1],label=r'$\\dot{S} = 160\\,k_B/{\\rm hr}$')\n\nskip = 1\nmodel = PCA(n_components=2).fit(out2['f'])\nf2 = model.transform(out2['f'])\nax.plot(f2[np.arange(0,len(f2),skip),0],f2[np.arange(0,len(f2),skip),1],label=r'$\\dot{S} = 880\\,k_B/{\\rm hr}$')\n\nax.plot([0],[0],'ko',markersize=8)\nax.plot([0,0],[0,0.6],'k',linewidth=2)\nax.set_ylim((-0.5,0.55))\n\nplt.legend(loc=1)\nax.set_xlabel('PCA 1')\nax.set_ylabel('PCA 2')\npdf = bpdf.PdfPages('Plots/phase_new.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
],
[
"Cvec = np.arange(2,7)\nAcvec = {}\nperiod = {}\nNvec = np.arange(3,50)\n\n\nfor C in Cvec:\n Acvec.update({'C = '+str(C): []})\n period.update({'C = '+str(C): []})\n for N in Nvec:\n try:\n Acvec['C = '+str(C)].append(opt.brentq(compute_maxeig,0,2,args=(C,N)))\n period['C = '+str(C)].append(2*np.pi/compute_maxeig_imag(Acvec['C = '+str(C)][-1],C,N))\n except:\n Acvec['C = '+str(C)].append(np.nan)\n period['C = '+str(C)].append(np.nan)\n \nwith open('Ac2.dat','wb') as f:\n pickle.dump([Nvec,Acvec,period],f)",
"_____no_output_____"
],
[
"with open('Ac2.dat','rb') as f:\n Nvec,Acvec,period = pickle.load(f)\n \nfig,ax=plt.subplots(figsize=(2.5,2.5))\nfig.subplots_adjust(left=0.22,bottom=0.22)\nfor item in Acvec.keys():\n ax.plot(Nvec,Acvec[item],label=item[-1])\nplt.legend(title = r'$C$')\nax.set_xlabel(r'$N$')\nax.set_ylabel(r'$A_c$')\nax.set_ylim((0,1))\n\npdf = bpdf.PdfPages('Plots/Figure6b.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
],
[
"fig,ax=plt.subplots(figsize=(3.5,3))\nfor item in Acvec.keys():\n ax.plot(Nvec,period[item],label=item[-1])\nplt.legend(title = r'$C$')\nax.set_xlabel(r'$N$')\nax.set_ylabel(r'Period at critical point')\npdf = bpdf.PdfPages('Plots/Figure5c.pdf')\npdf.savefig(fig)\npdf.close()\nplt.show()",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ea46fa4178d835be7d2100a298c8baae08f7f2 | 47,293 | ipynb | Jupyter Notebook | PyCitySchools/Resources/PyCitySchools_starter_unsolved.ipynb | fkokro/Pandas---Challenge | 9695736dcb44c10033ab8fdd0b801e54bebebcc8 | [
"ADSL"
] | null | null | null | PyCitySchools/Resources/PyCitySchools_starter_unsolved.ipynb | fkokro/Pandas---Challenge | 9695736dcb44c10033ab8fdd0b801e54bebebcc8 | [
"ADSL"
] | null | null | null | PyCitySchools/Resources/PyCitySchools_starter_unsolved.ipynb | fkokro/Pandas---Challenge | 9695736dcb44c10033ab8fdd0b801e54bebebcc8 | [
"ADSL"
] | null | null | null | 32.481456 | 202 | 0.401835 | [
[
[
"### Note\n* Instructions have been included for each segment. You do not have to follow them exactly, but they are included to help you think through the steps.",
"_____no_output_____"
]
],
[
[
"# Dependencies and Setup\nimport pandas as pd\n\n# File to Load (Remember to Change These)\nschool_data_to_load = \"Resources/schools_complete.csv\"\nstudent_data_to_load = \"Resources/students_complete.csv\"\n\n# Read School and Student Data File and store into Pandas Data Frames\nschool_data = pd.read_csv(school_data_to_load)\nstudent_data = pd.read_csv(student_data_to_load)\n\n# Combine the data into a single dataset\nschool_data_complete = pd.merge(student_data, school_data, how=\"left\", on=[\"school_name\", \"school_name\"])\nschool_data_complete ",
"_____no_output_____"
]
],
[
[
"## District Summary\n\n* Calculate the total number of schools\n\n* Calculate the total number of students\n\n* Calculate the total budget\n\n* Calculate the average math score \n\n* Calculate the average reading score\n\n* Calculate the overall passing rate (overall average score), i.e. (avg. math score + avg. reading score)/2\n\n* Calculate the percentage of students with a passing math score (70 or greater)\n\n* Calculate the percentage of students with a passing reading score (70 or greater)\n\n* Create a dataframe to hold the above results\n\n* Optional: give the displayed data cleaner formatting",
"_____no_output_____"
]
],
[
[
"#Calculates the total number of schools\narr_of_schools = school_data_complete[\"school_name\"].unique()\ntotal_schools = len(arr_of_schools)",
"_____no_output_____"
],
[
"#Calulates the number of students\narr_of_students = school_data_complete[\"student_name\"]\ntotal_students = len(arr_of_students)",
"_____no_output_____"
],
[
"#Calulates the total budget for all schools\narr_of_budgets = school_data_complete[\"budget\"].unique()\ntotal_budget = arr_of_budgets.sum()",
"_____no_output_____"
],
[
"#Calulates the average math score for all students\navg_math = school_data_complete[\"math_score\"].mean()",
"_____no_output_____"
],
[
"#Calulates the average math score for all students\navg_read = school_data_complete[\"reading_score\"].mean()",
"_____no_output_____"
],
[
"#Calculates the overall passing score\navg_overall = \"{:.4f}\".format((avg_read + avg_math)/2)",
"_____no_output_____"
],
[
"#Calculates the percentage of students with a passing math score (70 or greater)\npercentage_math = \"{:.6%}\".format((len(school_data_complete.loc[(school_data_complete[\"math_score\"]>= 70)])/total_students))",
"_____no_output_____"
],
[
"#Calculates the percentage of students with a passing reading score (70 or greater)\npercentage_read = \"{:.6%}\".format((len(school_data_complete.loc[(school_data_complete[\"reading_score\"]>= 70)])/total_students))",
"_____no_output_____"
],
[
"#Creates a dataframe to hold the above results\n\nframe_df = pd.DataFrame({\n \"Total Schools\":[total_schools],\n \"Total Students\":[\"{:,}\".format(total_students)],\n \"Total Budget\":[\"${:,}\".format(total_budget)],\n \"Average Math Score\":[avg_math],\n \"Average Reading Score\":[avg_read],\n \"% Passing Math\":[percentage_math],\n \"% Passing Reading\":[percentage_read],\n \"% Overall Passing Rate\":[avg_overall]\n})",
"_____no_output_____"
],
[
"frame_df",
"_____no_output_____"
]
],
[
[
"## School Summary",
"_____no_output_____"
],
[
"* Create an overview table that summarizes key metrics about each school, including:\n * School Name\n * School Type\n * Total Students\n * Total School Budget\n * Per Student Budget\n * Average Math Score\n * Average Reading Score\n * % Passing Math\n * % Passing Reading\n * Overall Passing Rate (Average of the above two)\n \n* Create a dataframe to hold the above results",
"_____no_output_____"
],
[
"## Top Performing Schools (By Passing Rate)",
"_____no_output_____"
],
[
"* Sort and display the top five schools in overall passing rate",
"_____no_output_____"
]
],
[
[
"#Sets index to school name\nschool_data_complete_name_index = school_data_complete.set_index(\"school_name\")",
"_____no_output_____"
]
],
[
[
"## Bottom Performing Schools (By Passing Rate)",
"_____no_output_____"
],
[
"* Sort and display the five worst-performing schools",
"_____no_output_____"
]
],
[
[
" ",
"_____no_output_____"
]
],
[
[
"## Math Scores by Grade",
"_____no_output_____"
],
[
"* Create a table that lists the average Reading Score for students of each grade level (9th, 10th, 11th, 12th) at each school.\n\n * Create a pandas series for each grade. Hint: use a conditional statement.\n \n * Group each series by school\n \n * Combine the series into a dataframe\n \n * Optional: give the displayed data cleaner formatting",
"_____no_output_____"
],
[
"## Reading Score by Grade ",
"_____no_output_____"
],
[
"* Perform the same operations as above for reading scores",
"_____no_output_____"
],
[
"## Scores by School Spending",
"_____no_output_____"
],
[
"* Create a table that breaks down school performances based on average Spending Ranges (Per Student). Use 4 reasonable bins to group school spending. Include in the table each of the following:\n * Average Math Score\n * Average Reading Score\n * % Passing Math\n * % Passing Reading\n * Overall Passing Rate (Average of the above two)",
"_____no_output_____"
]
],
[
[
"# Sample bins. Feel free to create your own bins.\nspending_bins = [0, 585, 615, 645, 675]\ngroup_names = [\"<$585\", \"$585-615\", \"$615-645\", \"$645-675\"]",
"_____no_output_____"
]
],
[
[
"## Scores by School Size",
"_____no_output_____"
],
[
"* Perform the same operations as above, based on school size.",
"_____no_output_____"
]
],
[
[
"# Sample bins. Feel free to create your own bins.\nsize_bins = [0, 1000, 2000, 5000]\ngroup_names = [\"Small (<1000)\", \"Medium (1000-2000)\", \"Large (2000-5000)\"]",
"_____no_output_____"
]
],
[
[
"## Scores by School Type",
"_____no_output_____"
],
[
"* Perform the same operations as above, based on school type.",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
]
] |
e7ea4ebfca478a22a89a6531d52f90a11d363d49 | 19,142 | ipynb | Jupyter Notebook | examples/retina_train_5class.ipynb | YuBeomGon/pytorch_retina | a1713ecbf99e3cf2f8f5edce3329b808b4f9dee8 | [
"Apache-2.0"
] | null | null | null | examples/retina_train_5class.ipynb | YuBeomGon/pytorch_retina | a1713ecbf99e3cf2f8f5edce3329b808b4f9dee8 | [
"Apache-2.0"
] | null | null | null | examples/retina_train_5class.ipynb | YuBeomGon/pytorch_retina | a1713ecbf99e3cf2f8f5edce3329b808b4f9dee8 | [
"Apache-2.0"
] | null | null | null | 32.890034 | 136 | 0.528837 | [
[
[
"import numpy as np \nimport pandas as pd\nimport os\nimport matplotlib.pyplot as plt\nfrom PIL import Image, ImageDraw, ImageEnhance\nimport albumentations as A\nimport albumentations.pytorch\nfrom tqdm.notebook import tqdm\n\nimport cv2\nimport re\nimport time\n\nimport sys\nsys.path.append('../')\nfrom retinanet import coco_eval\nfrom retinanet import csv_eval\nfrom retinanet import model\n# from retinanet import retina\nfrom retinanet.dataloader import *\nfrom retinanet.anchors import Anchors\nfrom retinanet.losses import *\nfrom retinanet.scheduler import *\nfrom retinanet.parallel import DataParallelModel, DataParallelCriterion\n\n# from sklearn.model_selection import train_test_split\n# from sklearn.preprocessing import LabelEncoder\n\n#Torch\nimport torch\nimport torch.nn as nn\nfrom torch.utils.data import Dataset,DataLoader\nfrom torch.utils.data.sampler import SequentialSampler, RandomSampler\nfrom torch.optim import Adam, lr_scheduler\nimport torch.optim as optim\n",
"_____no_output_____"
],
[
"# device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')\n# print ('Available devices ', torch.cuda.device_count())\n\n# print ('Current cuda device ', torch.cuda.current_device())\n# print(torch.cuda.get_device_name(device))\n\n# GPU 할당 변경하기\nGPU_NUM = 0 # 원하는 GPU 번호 입력\ndevice = torch.device(f'cuda:{GPU_NUM}' if torch.cuda.is_available() else 'cpu')\ntorch.cuda.set_device(device) # change allocation of current GPU\nprint(device)\nprint ('Current cuda device ', torch.cuda.current_device()) # check\ndevice_ids = [0,1,2,4]",
"cuda:0\nCurrent cuda device 0\n"
],
[
"%time\nPATH_TO_WEIGHTS = '../coco_resnet_50_map_0_335_state_dict.pt'\npretrained_retinanet = model.resnet50(num_classes=80, device=device)\npretrained_retinanet.load_state_dict(torch.load(PATH_TO_WEIGHTS))\n\n\nretinanet = model.resnet50(num_classes=5, device=device)\nfor param, state in zip(pretrained_retinanet.parameters(), pretrained_retinanet.state_dict()) :\n #print(state)\n if 'classificationModel' not in state :\n retinanet.state_dict()[state] = param\n else :\n print(state)\n \nfor param, state in zip(pretrained_retinanet.fpn.parameters(), pretrained_retinanet.fpn.state_dict()) :\n #print(state)\n retinanet.fpn.state_dict()[state] = param\n\nfor param, state in zip(pretrained_retinanet.regressionModel.parameters(), pretrained_retinanet.regressionModel.state_dict()) :\n #print(state)\n retinanet.regressionModel.state_dict()[state] = param ",
"CPU times: user 4 µs, sys: 7 µs, total: 11 µs\nWall time: 21.9 µs\n"
],
[
"\n# retinanet.to(device)\nretinanet = torch.nn.DataParallel(retinanet, device_ids = [0,1,2,4], output_device=0).to(device)\n# retinanet = DataParallelModel(retinanet, device_ids = device_ids)\nretinanet.to(device)\n# retinanet.cuda()\nretinanet.module.freeze_bn()",
"_____no_output_____"
],
[
"# train_transforms\n",
"_____no_output_____"
],
[
"# train_info = np.load('../data/train.npy', allow_pickle=True, encoding='latin1').item()\n# train_info\n\nbatch_size = 32\ndataset_train = PapsDataset('../data/', set_name='train',\n transform=train_transforms)\n\ntrain_data_loader = DataLoader(\n dataset_train,\n batch_size=batch_size,\n shuffle=True,\n num_workers=16,\n collate_fn=collate_fn\n)",
"loading annotations into memory...\nDone (t=0.82s)\ncreating index...\nindex created!\n"
],
[
"criterion = FocalLoss(device)\ncriterion = criterion.to(device)\nretinanet.training = True\n\n# https://gaussian37.github.io/dl-pytorch-lr_scheduler/\noptimizer = optim.Adam(retinanet.parameters(), lr = 1e-7)\nscheduler = CosineAnnealingWarmUpRestarts(optimizer, T_0=20, T_mult=1, eta_max=0.0001, T_up=5, gamma=0.5)\n# CosineAnnealingWarmRestarts",
"_____no_output_____"
],
[
"# for data in tqdm(train_data_loader) :\n# pass\n# # print(data)\n# # sdfsf",
"_____no_output_____"
],
[
"#for i, data in enumerate(tqdm(train_data_loader)) :\nEPOCH_NUM = 60\nloss_per_epoch = 0.5\nepoch_time_list = []\nfor epoch in range(EPOCH_NUM) :\n epoch_loss = []\n total_loss = 0\n tk0 = tqdm(train_data_loader, total=len(train_data_loader), leave=False)\n EPOCH_LEARING_RATE = optimizer.param_groups[0][\"lr\"]\n start_time = time.time()\n print(\"*****{}th epoch, learning rate {}\".format(epoch, EPOCH_LEARING_RATE))\n\n for step, data in enumerate(tk0) :\n images, box, label, targets = data\n batch_size = len(images)\n\n # images = list(image.to(device) for image in images)\n c, h, w = images[0].shape\n images = torch.cat(images).view(-1, c, h, w).to(device)\n# print(images.shape)\n# targets = [{k: v.to(device) for k, v in t.items()} for t in targets]\n targets = [ t.to(device) for t in targets]\n\n# classification_loss, regression_loss = retinanet([images, targets])\n outputs = retinanet([images, targets])\n classification, regression, anchors, annotations = (outputs)\n classification_loss, regression_loss = criterion(classification, regression, anchors, annotations)\n\n# output = retinanet(images)\n# features, regression, classification = output\n# classification_loss, regression_loss = criterion(classification, regression, modified_anchors, targets) \n classification_loss = classification_loss.mean()\n regression_loss = regression_loss.mean()\n loss = classification_loss + regression_loss \n total_loss += loss.item()\n \n epoch_loss.append((loss.item()))\n tk0.set_postfix(lr=optimizer.param_groups[0][\"lr\"], batch_loss=loss.item(), cls_loss=classification_loss.item(), \n reg_loss=regression_loss.item(), avg_loss=total_loss/(step+1))\n\n optimizer.zero_grad()\n loss.backward()\n torch.nn.utils.clip_grad_norm_(retinanet.parameters(), 0.1)\n optimizer.step() \n\n print('{}th epochs loss is {}'.format(epoch, np.mean(epoch_loss)))\n if loss_per_epoch > np.mean(epoch_loss):\n print('best model is saved')\n torch.save(retinanet.state_dict(), 'best_model.pt')\n loss_per_epoch = np.mean(epoch_loss)\n# scheduler.step(np.mean(epoch_loss))\n scheduler.step()\n epoch_time_list.append(time.time() - start_time)\n\ntorch.save(retinanet.state_dict(), '../trained_models/model.pt')\n",
"_____no_output_____"
],
[
"# label_list",
"_____no_output_____"
],
[
"dataset_val = PapsDataset('../data/', set_name='val',\n transform=val_transforms)\n\nval_data_loader = DataLoader(\n dataset_val,\n batch_size=1,\n shuffle=True,\n num_workers=2,\n collate_fn=collate_fn\n)",
"loading annotations into memory...\nDone (t=0.43s)\ncreating index...\nindex created!\n"
],
[
"retinanet.load_state_dict(torch.load('../trained_models/model.pt'))",
"_____no_output_____"
],
[
"from pycocotools.cocoeval import COCOeval\nimport json\nimport torch",
"_____no_output_____"
],
[
"retinanet.eval()\nstart_time = time.time()\nthreshold = 0.1\nresults = []\nGT_results = []\nimage_ids = []\ncnt = 0\nscores_list = []\n\nfor index, data in enumerate(tqdm(val_data_loader)) :\n if cnt > 300 :\n break\n cnt += 1\n with torch.no_grad(): \n images, tbox, tlabel, targets = data\n batch_size = len(images)\n# print(tbox)\n# print(len(tbox[0]))\n\n c, h, w = images[0].shape\n images = torch.cat(images).view(-1, c, h, w).to(device)\n\n outputs = retinanet(images)\n scores, labels, boxes = (outputs)\n \n scores = scores.cpu()\n labels = labels.cpu()\n boxes = boxes.cpu() \n \n scores_list.append(scores)\n\n if boxes.shape[0] > 0:\n # change to (x, y, w, h) (MS COCO standard)\n boxes[:, 2] -= boxes[:, 0]\n boxes[:, 3] -= boxes[:, 1]\n# print(boxes)\n\n # compute predicted labels and scores\n #for box, score, label in zip(boxes[0], scores[0], labels[0]):\n for box_id in range(boxes.shape[0]):\n score = float(scores[box_id])\n label = int(labels[box_id])\n box = boxes[box_id, :]\n\n # scores are sorted, so we can break\n if score < threshold:\n break\n\n # append detection for each positively labeled class\n image_result = {\n 'image_id' : dataset_val.image_ids[index],\n 'category_id' : dataset_val.label_to_coco_label(label),\n 'score' : float(score),\n 'bbox' : box.tolist(),\n }\n\n # append detection to results\n results.append(image_result)\n \n if len(tbox[0]) > 0: \n\n # compute predicted labels and scores\n #for box, score, label in zip(boxes[0], scores[0], labels[0]):\n for box_id in range(len(tbox[0])):\n score = float(0.99)\n label = (tlabel[0][box_id])\n box = list(tbox[0][box_id])\n box[2] -= box[0]\n box[3] -= box[1] \n\n # append detection for each positively labeled class\n image_result = {\n 'image_id' : dataset_val.image_ids[index],\n 'category_id' : dataset_val.label_to_coco_label(label),\n 'score' : float(score),\n 'bbox' : list(box),\n }\n\n # append detection to results\n GT_results.append(image_result) \n\n # append image to list of processed images\n image_ids.append(dataset_val.image_ids[index])\n\n # print progress\n print('{}/{}'.format(index, len(dataset_val)), end='\\r') \n\nif not len(results):\n print('No object detected')\nprint('GT_results', len(GT_results)) \nprint('pred_results', len(results)) \n\n# write output\njson.dump(results, open('{}_bbox_results.json'.format(dataset_val.set_name), 'w'), indent=4)\n# write GT\njson.dump(GT_results, open('{}_GTbbox_results.json'.format(dataset_val.set_name), 'w'), indent=4) \n\nprint('validation time :', time.time() - start_time)\n\n",
"_____no_output_____"
],
[
"# load results in COCO evaluation tool\ncoco_true = dataset_val.coco\ncoco_pred = coco_true.loadRes('{}_bbox_results.json'.format(dataset_val.set_name))\ncoco_gt = coco_true.loadRes('{}_GTbbox_results.json'.format(dataset_val.set_name))\n\n# run COCO evaluation\n# coco_eval = COCOeval(coco_true, coco_pred, 'bbox')\ncoco_eval = COCOeval(coco_gt, coco_pred, 'bbox')\ncoco_eval.params.imgIds = image_ids\n# coco_eval.params.catIds = [0]\ncoco_eval.evaluate()\ncoco_eval.accumulate()\ncoco_eval.summarize() ",
"Loading and preparing results...\nDONE (t=0.02s)\ncreating index...\nindex created!\nLoading and preparing results...\nDONE (t=0.02s)\ncreating index...\nindex created!\nRunning per image evaluation...\nEvaluate annotation type *bbox*\nDONE (t=0.10s).\nAccumulating evaluation results...\nDONE (t=0.04s).\n Average Precision (AP) @[ IoU=0.50:0.95 | area= all | maxDets=100 ] = 0.366\n Average Precision (AP) @[ IoU=0.50 | area= all | maxDets=100 ] = 0.564\n Average Precision (AP) @[ IoU=0.75 | area= all | maxDets=100 ] = 0.443\n Average Precision (AP) @[ IoU=0.50:0.95 | area= small | maxDets=100 ] = -1.000\n Average Precision (AP) @[ IoU=0.50:0.95 | area=medium | maxDets=100 ] = 0.366\n Average Precision (AP) @[ IoU=0.50:0.95 | area= large | maxDets=100 ] = -1.000\n Average Recall (AR) @[ IoU=0.50:0.95 | area= all | maxDets= 1 ] = 0.290\n Average Recall (AR) @[ IoU=0.50:0.95 | area= all | maxDets= 10 ] = 0.565\n Average Recall (AR) @[ IoU=0.50:0.95 | area= all | maxDets=100 ] = 0.582\n Average Recall (AR) @[ IoU=0.50:0.95 | area= small | maxDets=100 ] = -1.000\n Average Recall (AR) @[ IoU=0.50:0.95 | area=medium | maxDets=100 ] = 0.582\n Average Recall (AR) @[ IoU=0.50:0.95 | area= large | maxDets=100 ] = -1.000\n"
],
[
"coco_eval.params.catIds",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ea6d998581b77d54764e9fe3d2dc93b24e30d1 | 2,274 | ipynb | Jupyter Notebook | misc/notes/Authentication/Intro to Authentication.ipynb | alittlebirdie00/coma | 4670e4be2f687f48b9c75118788aa7f90b72b5d4 | [
"MIT"
] | 2 | 2018-09-16T21:39:24.000Z | 2019-05-03T06:24:18.000Z | misc/notes/Authentication/Intro to Authentication.ipynb | alittlebirdie00/coma | 4670e4be2f687f48b9c75118788aa7f90b72b5d4 | [
"MIT"
] | 46 | 2018-08-28T13:38:04.000Z | 2019-10-02T18:54:51.000Z | misc/notes/Authentication/Intro to Authentication.ipynb | alittlebirdie00/coma | 4670e4be2f687f48b9c75118788aa7f90b72b5d4 | [
"MIT"
] | 1 | 2018-08-30T11:08:31.000Z | 2018-08-30T11:08:31.000Z | 33.441176 | 134 | 0.582234 | [
[
[
"empty"
]
]
] | [
"empty"
] | [
[
"empty"
]
] |
e7ea7b0a364439a30bc60dba0af08b0d1af368fb | 7,690 | ipynb | Jupyter Notebook | Notebooks/DiabetesNotebook.ipynb | codeshruti/HealthCheck | 3e1579991ba9574eae0441f9597e06cd434c83c5 | [
"MIT"
] | 1 | 2021-03-15T10:43:42.000Z | 2021-03-15T10:43:42.000Z | Notebooks/DiabetesNotebook.ipynb | codeshruti/HealthCheck | 3e1579991ba9574eae0441f9597e06cd434c83c5 | [
"MIT"
] | null | null | null | Notebooks/DiabetesNotebook.ipynb | codeshruti/HealthCheck | 3e1579991ba9574eae0441f9597e06cd434c83c5 | [
"MIT"
] | null | null | null | 29.239544 | 163 | 0.418466 | [
[
[
"# Importing essential libraries\nimport numpy as np\nimport pandas as pd\nimport pickle",
"_____no_output_____"
],
[
"# Loading the dataset\ndf = pd.read_csv('./diabetes.csv')",
"_____no_output_____"
],
[
"df.head(5)",
"_____no_output_____"
],
[
"# Renaming DiabetesPedigreeFunction as DPF\ndf = df.rename(columns={'DiabetesPedigreeFunction':'DPF'})",
"_____no_output_____"
],
[
"# Replacing the 0 values from ['Glucose','BloodPressure','SkinThickness','Insulin','BMI'] by NaN\ndf_copy = df.copy(deep=True)\ndf_copy[['Glucose','BloodPressure','SkinThickness','Insulin','BMI']] = df_copy[['Glucose','BloodPressure','SkinThickness','Insulin','BMI']].replace(0,np.NaN)",
"_____no_output_____"
],
[
"# Replacing NaN value by mean, median depending upon distribution\ndf_copy['Glucose'].fillna(df_copy['Glucose'].mean(), inplace=True)\ndf_copy['BloodPressure'].fillna(df_copy['BloodPressure'].mean(), inplace=True)\ndf_copy['SkinThickness'].fillna(df_copy['SkinThickness'].median(), inplace=True)\ndf_copy['Insulin'].fillna(df_copy['Insulin'].median(), inplace=True)\ndf_copy['BMI'].fillna(df_copy['BMI'].median(), inplace=True)",
"_____no_output_____"
],
[
"# Model Building\nfrom sklearn.model_selection import train_test_split\nX = df.drop(columns='Outcome')\ny = df['Outcome']\nX_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.20, random_state=0)",
"_____no_output_____"
],
[
"# Creating Random Forest Model\nfrom sklearn.ensemble import RandomForestClassifier\nclassifier = RandomForestClassifier(n_estimators=20)\nclassifier.fit(X_train, y_train)",
"_____no_output_____"
],
[
"# Creating a pickle file for the classifier\nfilename = 'diabetes-model.pkl'\npickle.dump(classifier, open(filename, 'wb'))",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ea7f7e18956fe9b07067367b723346486d6f42 | 15,704 | ipynb | Jupyter Notebook | site/en/guide/graph_optimization.ipynb | atharva1503/docs | d810ab4728a4810615c83e9b17861ed6d9d76f1f | [
"Apache-2.0"
] | 2 | 2020-06-20T14:10:44.000Z | 2020-10-12T07:10:34.000Z | site/en/guide/graph_optimization.ipynb | atharva1503/docs | d810ab4728a4810615c83e9b17861ed6d9d76f1f | [
"Apache-2.0"
] | null | null | null | site/en/guide/graph_optimization.ipynb | atharva1503/docs | d810ab4728a4810615c83e9b17861ed6d9d76f1f | [
"Apache-2.0"
] | null | null | null | 36.950588 | 521 | 0.573994 | [
[
[
"##### Copyright 2020 The TensorFlow Authors.",
"_____no_output_____"
]
],
[
[
"#@title Licensed under the Apache License, Version 2.0 (the \"License\");\n# you may not use this file except in compliance with the License.\n# You may obtain a copy of the License at\n#\n# https://www.apache.org/licenses/LICENSE-2.0\n#\n# Unless required by applicable law or agreed to in writing, software\n# distributed under the License is distributed on an \"AS IS\" BASIS,\n# WITHOUT WARRANTIES OR CONDITIONS OF ANY KIND, either express or implied.\n# See the License for the specific language governing permissions and\n# limitations under the License.",
"_____no_output_____"
]
],
[
[
"# TensorFlow graph optimization with Grappler",
"_____no_output_____"
],
[
"<table class=\"tfo-notebook-buttons\" align=\"left\">\n <td>\n <a target=\"_blank\" href=\"https://www.tensorflow.org/guide/graph_optimization\"><img src=\"https://www.tensorflow.org/images/tf_logo_32px.png\" />View on TensorFlow.org</a>\n </td>\n <td>\n <a target=\"_blank\" href=\"https://colab.research.google.com/github/tensorflow/docs/blob/master/site/en/guide/graph_optimization.ipynb\"><img src=\"https://www.tensorflow.org/images/colab_logo_32px.png\" />Run in Google Colab</a>\n </td>\n <td>\n <a target=\"_blank\" href=\"https://github.com/tensorflow/docs/blob/master/site/en/guide/graph_optimization.ipynb\"><img src=\"https://www.tensorflow.org/images/GitHub-Mark-32px.png\" />View source on GitHub</a>\n </td>\n <td>\n <a href=\"https://storage.googleapis.com/tensorflow_docs/docs/site/en/guide/graph_optimization.ipynb\"><img src=\"https://www.tensorflow.org/images/download_logo_32px.png\" />Download notebook</a>\n </td>\n</table>",
"_____no_output_____"
],
[
"## Overview\n\nTensorFlow uses both graph and eager executions to execute computations. A `tf.Graph` contains a set of `tf.Operation` objects (ops) which represent units of computation and `tf.Tensor` objects which represent the units of data that flow between ops.\n\nGrappler is the default graph optimization system in the TensorFlow runtime. Grappler applies optimizations in graph mode (within `tf.function`) to improve the perormance of your TensorFlow computations through graph simplifications and other high-level optimizations such as inlining function bodies to enable inter-procedural optimizations. Optimizing the `tf.Graph` also reduces the device peak memory usage and improves hardware utilization by optimizing the mapping of graph nodes to compute resources. \n\nUse `tf.config.optimizer.set_experimental_options()` for finer control over your `tf.Graph` optimizations.\n\n",
"_____no_output_____"
],
[
"## Available graph optimizers\n\nGrappler performs graph optimizations through a top-level driver called the `MetaOptimizer`. The following graph optimizers are available with TensorFlow: \n\n* *Constant folding optimizer -* Statically infers the value of tensors when possible by folding constant nodes in the graph and materializes the result using constants.\n* *Arithmetic optimizer -* Simplifies arithmetic operations by eliminating common subexpressions and simplifying arithmetic statements. \n* *Layout optimizer -* Optimizes tensor layouts to execute data format dependent operations such as convolutions more efficiently.\n* *Remapper optimizer -* Remaps subgraphs onto more efficient implementations by replacing commonly occuring subgraphs with optimized fused monolithic kernels.\n* *Memory optimizer -* Analyzes the graph to inspect the peak memory usage for each operation and inserts CPU-GPU memory copy operations for swapping GPU memory to CPU to reduce the peak memory usage.\n* *Dependency optimizer -* Removes or rearranges control dependencies to shorten the critical path for a model step or enables other\noptimizations. Also removes nodes that are effectively no-ops such as Identity.\n* *Pruning optimizer -* Prunes nodes that have no effect on the output from the graph. It is usually run first to reduce the size of the graph and speed up processing in other Grappler passes.\n* *Function optimizer -* Optimizes the function library of a TensorFlow program and inlines function bodies to enable other inter-procedural optimizations.\n* *Shape optimizer -* Optimizes subgraphs that operate on shape and shape related information.\n* *Autoparallel optimizer -* Automatically parallelizes graphs by splitting along the batch dimension. This optimizer is turned OFF by default.\n* *Loop optimizer -* Optimizes the graph control flow by hoisting loop-invariant subgraphs out of loops and by removing redundant stack operations in loops. Also optimizes loops with statically known trip counts and removes statically known dead branches in conditionals.\n* *Scoped allocator optimizer -* Introduces scoped allocators to reduce data movement and to consolidate some operations.\n* *Pin to host optimizer -* Swaps small operations onto the CPU. This optimizer is turned OFF by default. \n* *Auto mixed precision optimizer -* Converts data types to float16 where applicable to improve performance. Currently applies only to GPUs.\n* *Debug stripper -* Strips nodes related to debugging operations such as `tf.debugging.Assert`, `tf.debugging.check_numerics`, and `tf.print` from the graph. This optimizer is turned OFF by default.",
"_____no_output_____"
],
[
"## Setup\n",
"_____no_output_____"
]
],
[
[
"from __future__ import absolute_import, division, print_function, unicode_literals\n\nimport numpy as np\nimport timeit\nimport traceback\nimport contextlib\n\ntry:\n %tensorflow_version 2.x\nexcept Exception:\n pass\n\nimport tensorflow as tf",
"_____no_output_____"
]
],
[
[
"Create a context manager to easily toggle optimizer states.",
"_____no_output_____"
]
],
[
[
"@contextlib.contextmanager\ndef options(options):\n old_opts = tf.config.optimizer.get_experimental_options()\n tf.config.optimizer.set_experimental_options(options)\n try:\n yield\n finally:\n tf.config.optimizer.set_experimental_options(old_opts)",
"_____no_output_____"
]
],
[
[
"## Compare execution performance with and without Grappler\n\nTensorFlow 2 and beyond executes [eagerly](../eager.md) by default. Use `tf.function` to switch the default execution to Graph mode. Grappler runs automatically in the background to apply the graph optimizations above and improve execution performance. \n\n",
"_____no_output_____"
],
[
"### Constant folding optimizer\n\nAs a preliminary example, consider a function which performs operations on constants and returns an output.",
"_____no_output_____"
]
],
[
[
"def test_function_1():\n @tf.function\n def simple_function(input_arg):\n print('Tracing!')\n a = tf.constant(np.random.randn(2000,2000), dtype = tf.float32)\n c = a\n for n in range(50):\n c = c@a\n return tf.reduce_mean(c+input_arg)\n\n return simple_function",
"_____no_output_____"
]
],
[
[
"Turn off the constant folding optimizer and execute the function:",
"_____no_output_____"
]
],
[
[
"with options({'constant_folding': False}):\n print(tf.config.optimizer.get_experimental_options())\n simple_function = test_function_1()\n # Trace once\n x = tf.constant(2.2)\n simple_function(x)\n print(\"Vanilla execution:\", timeit.timeit(lambda: simple_function(x), number = 1), \"s\")",
"_____no_output_____"
]
],
[
[
"Enable the constant folding optimizer and execute the function again to observe a speed-up in function execution.",
"_____no_output_____"
]
],
[
[
"with options({'constant_folding': True}):\n print(tf.config.optimizer.get_experimental_options())\n simple_function = test_function_1()\n # Trace once\n x = tf.constant(2.2)\n simple_function(x)\n print(\"Constant folded execution:\", timeit.timeit(lambda: simple_function(x), number = 1), \"s\")",
"_____no_output_____"
]
],
[
[
"### Debug stripper optimizer\n\nConsider a simple function that checks the numeric value of its input argument and returns it. ",
"_____no_output_____"
]
],
[
[
"def test_function_2():\n @tf.function\n def simple_func(input_arg):\n output = input_arg\n tf.debugging.check_numerics(output, \"Bad!\")\n return output\n return simple_func",
"_____no_output_____"
]
],
[
[
"First, execute the function with the debug stripper optimizer turned off. ",
"_____no_output_____"
]
],
[
[
"test_func = test_function_2()\np1 = tf.constant(float('inf'))\ntry:\n test_func(p1)\nexcept tf.errors.InvalidArgumentError as e:\n traceback.print_exc(limit=2)",
"_____no_output_____"
]
],
[
[
"`tf.debugging.check_numerics` raises an invalid argument error because of the `Inf` argument to `test_func`.",
"_____no_output_____"
],
[
"Enable the debug stripper optimizer and execute the function again. ",
"_____no_output_____"
]
],
[
[
"with options({'debug_stripper': True}):\n test_func2 = test_function_2()\n p1 = tf.constant(float('inf'))\n try:\n test_func2(p1)\n except tf.errors.InvalidArgumentError as e:\n traceback.print_exc(limit=2)",
"_____no_output_____"
]
],
[
[
"The debug stripper optimizer strips the `tf.debug.check_numerics` node from the graph and executes the function without raising any errors. ",
"_____no_output_____"
],
[
"## Summary\n\nThe TensorFlow runtime uses Grappler to optimize graphs automatically before execution. Use `tf.config.optimizer.set_experimental_options` to enable or disable the various graph optimizers. \n\nFor more information on Grappler, see <a href=\"http://web.stanford.edu/class/cs245/slides/TFGraphOptimizationsStanford.pdf\" class=\"external\">TensorFlow Graph Optimizations</a>.",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
]
] |
e7ea886acdca67eb337162284122fc89934841ab | 17,017 | ipynb | Jupyter Notebook | sparkify_cassandra_project.ipynb | iamfeniak/sparkify-etl-pipeline-cassandra | 655617fd5fa1352a6f3e4acafc7d4f731fc67945 | [
"MIT"
] | null | null | null | sparkify_cassandra_project.ipynb | iamfeniak/sparkify-etl-pipeline-cassandra | 655617fd5fa1352a6f3e4acafc7d4f731fc67945 | [
"MIT"
] | null | null | null | sparkify_cassandra_project.ipynb | iamfeniak/sparkify-etl-pipeline-cassandra | 655617fd5fa1352a6f3e4acafc7d4f731fc67945 | [
"MIT"
] | null | null | null | 27.140351 | 190 | 0.531997 | [
[
[
"# Part I. ETL Pipeline for Pre-Processing the Files",
"_____no_output_____"
],
[
"#### Import Python packages ",
"_____no_output_____"
]
],
[
[
"# Import Python packages \nimport pandas as pd\nimport cassandra\nimport re\nimport os\nimport glob\nimport numpy as np\nimport json\nimport csv",
"_____no_output_____"
]
],
[
[
"#### Creating list of filepaths to process original event csv data files",
"_____no_output_____"
]
],
[
[
"# checking your current working directory\nprint(os.getcwd())\n\n# Get your current folder and subfolder event data\nfilepath = os.getcwd() + '/event_data'\n\n# Create a for loop to create a list of files and collect each filepath\nfor root, dirs, files in os.walk(filepath):\n \n# join the file path and roots with the subdirectories using glob\n file_path_list = glob.glob(os.path.join(root,'*'))",
"/home/workspace\n"
]
],
[
[
"#### Processing the files to create the data file csv that will be used for Apache Casssandra tables",
"_____no_output_____"
]
],
[
[
"# initiating an empty list of rows that will be generated from each file\nfull_data_rows_list = [] \n \n# for every filepath in the file path list \nfor f in file_path_list:\n\n # reading csv file \n with open(f, 'r', encoding = 'utf8', newline='') as csvfile: \n # creating a csv reader object \n csvreader = csv.reader(csvfile) \n next(csvreader)\n \n # extracting each data row one by one and append it \n for line in csvreader:\n #print(line)\n full_data_rows_list.append(line) \n\n# creating a smaller event data csv file called event_datafile_full csv that will be used to insert data into the \\\n# Apache Cassandra tables\ncsv.register_dialect('myDialect', quoting=csv.QUOTE_ALL, skipinitialspace=True)\n\nwith open('event_datafile_new.csv', 'w', encoding = 'utf8', newline='') as f:\n writer = csv.writer(f, dialect='myDialect')\n writer.writerow(['artist','firstName','gender','itemInSession','lastName','length',\\\n 'level','location','sessionId','song','userId'])\n for row in full_data_rows_list:\n if (row[0] == ''):\n continue\n writer.writerow((row[0], row[2], row[3], row[4], row[5], row[6], row[7], row[8], row[12], row[13], row[16]))",
"_____no_output_____"
],
[
"# check the number of rows in your csv file\nwith open('event_datafile_new.csv', 'r', encoding = 'utf8') as f:\n print(sum(1 for line in f))",
"6821\n"
]
],
[
[
"### The event_datafile_new.csv now contains the following columns: \n- artist \n- firstName of user\n- gender of user\n- item number in session\n- last name of user\n- length of the song\n- level (paid or free song)\n- location of the user\n- sessionId\n- song title\n- userId\n\nThe image below is a screenshot of what the denormalized data should appear like in the <font color=red>**event_datafile_new.csv**</font> after the code above is run:<br>\n\n<img src=\"images/image_event_datafile_new.jpg\">",
"_____no_output_____"
],
[
"#### Creating a Cluster",
"_____no_output_____"
]
],
[
[
"from cassandra.cluster import Cluster\n\ntry: \n # This should make a connection to a Cassandra instance your local machine \n # (127.0.0.1)\n cluster = Cluster(['127.0.0.1'])\n # To establish connection and begin executing queries, need a session\n session = cluster.connect()\nexcept Exception as e:\n print(e)",
"_____no_output_____"
]
],
[
[
"#### Create Keyspace",
"_____no_output_____"
]
],
[
[
"try:\n session.execute(\"\"\"\n CREATE KEYSPACE IF NOT EXISTS sparkify \n WITH REPLICATION = \n { 'class' : 'SimpleStrategy', 'replication_factor' : 1 }\"\"\")\nexcept Exception as e:\n print(e)",
"_____no_output_____"
]
],
[
[
"#### Set Keyspace",
"_____no_output_____"
]
],
[
[
"try:\n session.set_keyspace('sparkify')\nexcept Exception as e:\n print(e)",
"_____no_output_____"
]
],
[
[
"# Part II. Time to answer important query questions",
"_____no_output_____"
],
[
"### Query 1. Give me the artist, song title and song's length in the music app history that was heard during sessionId = 338, and itemInSession = 4\n\nThis query focuses on session history, so the table will be named accordingly.\n\nFiltering needs to be done by session_id AND item_in_session. \n\nLooking at the data, it's safe to assume that both aforementioned columns would identify row uniquely, so both columns will be included for primary key. ",
"_____no_output_____"
]
],
[
[
"session_history_create = \"\"\"CREATE TABLE IF NOT EXISTS session_history(\n session_id int, \n item_in_session int, \n artist_name text,\n song_title text, \n song_length float, \n PRIMARY KEY(session_id, item_in_session));\"\"\"\n\ntry:\n session.execute(session_history_create)\nexcept Exception as e:\n print(e) ",
"_____no_output_____"
],
[
"file = 'event_datafile_new.csv'\n\nwith open(file, encoding = 'utf8') as f:\n csvreader = csv.reader(f)\n next(csvreader) # skip header\n for line in csvreader:\n query = \"INSERT INTO session_history(session_id, item_in_session, artist_name, song_title, song_length)\"\n query = query + \"VALUES(%s, %s, %s, %s, %s);\"\n session.execute(query, (int(line[8]), int(line[3]), line[0], line[9], float(line[5])))",
"_____no_output_____"
]
],
[
[
"### Let's test query 1 with an example ",
"_____no_output_____"
]
],
[
[
"query = \"\"\"SELECT artist_name, song_title, song_length \n FROM session_history \n WHERE session_id=338 AND item_in_session=4;\"\"\"\ntry:\n result = pd.DataFrame(list(session.execute(query)))\n print(result.to_string())\nexcept Exception as e:\n print(e) ",
" artist_name song_title song_length\n0 Faithless Music Matters (Mark Knight Dub) 495.307312\n"
]
],
[
[
"### Query 2. Give me only the following: name of artist, song (sorted by itemInSession) and user (first and last name) for userid = 10, sessionid = 182\n\nThis query focuses on specific user session history. \n\nWe will be retrieving specific listening details by user_id and session_id. \n\nTo support query, user_id, session_id and item_in_session will be used for primary key (where item_in_session is needed to give sorted output. ",
"_____no_output_____"
]
],
[
[
"user_session_history_create = \"\"\"\n CREATE TABLE IF NOT EXISTS user_session_history(\n user_id int,\n session_id int, \n item_in_session int, \n artist_name text,\n song_title text, \n user_first_name text,\n user_last_name text,\n PRIMARY KEY(user_id, session_id, item_in_session));\"\"\"\n\ntry:\n session.execute(user_session_history_create)\nexcept Exception as e:\n print(e) ",
"_____no_output_____"
],
[
"file = 'event_datafile_new.csv'\n\nwith open(file, encoding = 'utf8') as f:\n csvreader = csv.reader(f)\n next(csvreader) # skip header\n for line in csvreader:\n query = \"INSERT INTO user_session_history(user_id, session_id, item_in_session, artist_name, song_title, user_first_name, user_last_name)\"\n query = query + \"VALUES(%s, %s, %s, %s, %s, %s, %s);\"\n session.execute(query, (int(line[10]), int(line[8]), int(line[3]), line[0], line[9], line[1], line[4]))",
"_____no_output_____"
]
],
[
[
"### Let's test query 2 with an example ",
"_____no_output_____"
]
],
[
[
"query = \"\"\"SELECT artist_name, song_title, user_first_name, user_last_name\n FROM user_session_history \n WHERE user_id=10 AND session_id=182;\"\"\"\n\ntry:\n result = pd.DataFrame(list(session.execute(query)))\n print(result.to_string())\nexcept Exception as e:\n print(e) ",
" artist_name song_title user_first_name user_last_name\n0 Down To The Bone Keep On Keepin' On Sylvie Cruz\n1 Three Drives Greece 2000 Sylvie Cruz\n2 Sebastien Tellier Kilometer Sylvie Cruz\n3 Lonnie Gordon Catch You Baby (Steve Pitron & Max Sanna Radio... Sylvie Cruz\n"
]
],
[
[
"### Query 3. Give me every user name (first and last) in my music app history who listened to the song 'All Hands Against His Own'\n\nThis query focuses on song history, highlighting which songs are being listened to by which users. \n\nWe will be retrieving specific listening details by song_title. \n\nTo support query, song_title and user_id will be used for primary key. \n\nNote: Song names might not be unique (e.g. different artists might have same song names). To remove ambiguity, in future it might makes sense to specify more details about this query. ",
"_____no_output_____"
]
],
[
[
"song_user_history_create = \"\"\"\n CREATE TABLE IF NOT EXISTS song_user_history(\n user_id int,\n song_title text, \n user_first_name text,\n user_last_name text,\n PRIMARY KEY(song_title, user_id));\"\"\"\n\ntry:\n session.execute(song_user_history_create)\nexcept Exception as e:\n print(e) ",
"_____no_output_____"
],
[
"file = 'event_datafile_new.csv'\n\nwith open(file, encoding = 'utf8') as f:\n csvreader = csv.reader(f)\n next(csvreader) # skip header\n for line in csvreader:\n query = \"INSERT INTO song_user_history(user_id, song_title, user_first_name, user_last_name)\"\n query = query + \"VALUES(%s, %s, %s, %s);\"\n session.execute(query, (int(line[10]), line[9], line[1], line[4]))",
"_____no_output_____"
]
],
[
[
"### Let's test query 3 with an example ",
"_____no_output_____"
]
],
[
[
"query = \"\"\"SELECT user_first_name, user_last_name\n FROM song_user_history \n WHERE song_title='All Hands Against His Own';\"\"\"\n\ntry:\n result = pd.DataFrame(list(session.execute(query)))\n print(result.to_string())\nexcept Exception as e:\n print(e) ",
" user_first_name user_last_name\n0 Jacqueline Lynch\n1 Tegan Levine\n2 Sara Johnson\n"
]
],
[
[
"### Dropping the tables before closing out the sessions",
"_____no_output_____"
]
],
[
[
"drop_session_history = \"DROP TABLE IF EXISTS session_history;\"\nsession.execute(drop_session_history)\ndrop_user_session_history = \"DROP TABLE IF EXISTS user_session_history;\"\nsession.execute(drop_user_session_history)\ndrop_song_user_history = \"DROP TABLE IF EXISTS song_user_history;\"\nsession.execute(drop_song_user_history)",
"_____no_output_____"
]
],
[
[
"# Don't forget to close the session and cluster connection¶",
"_____no_output_____"
]
],
[
[
"session.shutdown()\ncluster.shutdown()",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7ea89ac400666dc833315c21cb6e3d1638407fe | 32,640 | ipynb | Jupyter Notebook | FinanceProjectMain.ipynb | AmaaniGoose/FinanceProject | c8acbe141ee0d930e129356a1a414085ba927659 | [
"MIT"
] | null | null | null | FinanceProjectMain.ipynb | AmaaniGoose/FinanceProject | c8acbe141ee0d930e129356a1a414085ba927659 | [
"MIT"
] | null | null | null | FinanceProjectMain.ipynb | AmaaniGoose/FinanceProject | c8acbe141ee0d930e129356a1a414085ba927659 | [
"MIT"
] | 1 | 2021-01-03T15:46:32.000Z | 2021-01-03T15:46:32.000Z | 85.894737 | 18,924 | 0.7856 | [
[
[
"Welcome!",
"_____no_output_____"
],
[
"In finance momentum refers to the phenomenon of cross-sectional predictability of returns by past price data. A standard example would be the well documented tendency of stocks that have had high returns over the past one to twelve months to continue outperform stocks that have performed poorly over the same period. Positive returns from buying past winners and selling past losers is a long-standing market anomaly in financial research documented for basically every asset class and literally for hundreds of years. Note that since the stocks are compared to their peers we talk about cross-sectional predictability, in contrast to the time-series momentum, or trend following, where decision to buy or sell a stock depends on its own past performance only. Over the past quarter of a century the finance literature has proposed numerous ways to measure the momentum, e.g. in terms of lookback horizon, and identified a host of confounding variables, like market volatility, predicting its performance as an investment strategy. The emerging field of financial machine learning further finds past price data to be among the strongest predictors of future returns, dominating fundamental variables like book-to-market ratio.",
"_____no_output_____"
]
],
[
[
"import csv\nimport random\nimport matplotlib.pyplot as plt\nimport numpy as np\nimport scipy.stats as statfunc\nimport pandas as pd\nimport plotly.graph_objs as go\nimport mplfinance as mpf\nfrom sklearn import mixture as mix\nimport seaborn as sns \nfrom pandas_datareader import data as web",
"_____no_output_____"
],
[
"df= web.get_data_yahoo('RELIANCE.NS',start= '2018-04-08', end='2020-12-12')\ndf=df[['Open','High','Low','Close','Volume']]\ndf['open']=df['Open'].shift(1)\ndf['high']=df['High'].shift(1)\ndf['low']=df['Low'].shift(1)\ndf['close']=df['Close'].shift(1)\ndf['volume']=df['Volume'].shift(1)\ndf=df[['open','high','low','close','volume']]\n\n",
"_____no_output_____"
],
[
"df=df.dropna()",
"_____no_output_____"
],
[
"unsup = mix.GaussianMixture(n_components=4, covariance_type=\"spherical\", n_init=100, random_state=42)",
"_____no_output_____"
],
[
"unsup.fit(np.reshape(df,(-1,df.shape[1])))\nregime = unsup.predict(np.reshape(df,(-1,df.shape[1])))",
"_____no_output_____"
],
[
"df['Return']= np.log(df['close']/df['close'].shift(1))",
"_____no_output_____"
],
[
"Regimes=pd.DataFrame(regime,columns=['Regime'],index=df.index)\\\n .join(df, how='inner')\\\n .assign(market_cu_return=df.Return.cumsum())\\\n .reset_index(drop=False)\\\n .rename(columns={'index':'Date'})",
"_____no_output_____"
],
[
"order=[0,1,2,3]\nfig = sns.FacetGrid(data=Regimes,hue='Regime',hue_order=order,aspect=2,size= 4)\nfig.map(plt.scatter,'Date','market_cu_return', s=4).add_legend()\nplt.show()",
"C:\\ProgramData\\Anaconda3\\lib\\site-packages\\seaborn\\axisgrid.py:230: UserWarning: The `size` paramter has been renamed to `height`; please update your code.\n warnings.warn(msg, UserWarning)\n"
],
[
"for i in order:\n print('Mean for regime %i: '%i,unsup.means_[i][0])\n print('Co-Variancefor regime %i: '%i,(unsup.covariances_[i]))",
"Mean for regime 0: 10784.766638714711\nCo-Variancefor regime 0: 36141.444765725864\nMean for regime 1: 12047.572769668644\nCo-Variancefor regime 1: 23143.320584827794\nMean for regime 2: 9447.270093875897\nCo-Variancefor regime 2: 453141.17885501473\nMean for regime 3: 11509.5640340791\nCo-Variancefor regime 3: 30112.327401162816\n"
]
],
[
[
"# Version 2: Using technical indicators",
"_____no_output_____"
]
],
[
[
"print(df)",
" open high low close volume\nDate \n2018-04-09 NaN NaN NaN NaN NaN\n2018-04-10 912.549988 919.000000 912.549988 916.000000 3572251.0\n2018-04-11 918.400024 923.900024 914.150024 917.049988 3865402.0\n2018-04-12 921.799988 932.750000 916.049988 930.849976 6007539.0\n2018-04-13 929.849976 936.650024 924.200012 928.700012 4947725.0\n... ... ... ... ... ...\n2020-12-07 1969.000000 1969.000000 1940.000000 1946.750000 8521388.0\n2020-12-08 1940.599976 1965.000000 1940.599976 1958.199951 8418767.0\n2020-12-09 1961.150024 2014.250000 1950.000000 1993.750000 20030506.0\n2020-12-10 2009.949951 2033.800049 1999.250000 2026.949951 13464375.0\n2020-12-11 2021.599976 2028.500000 2001.000000 2007.000000 7414229.0\n\n[660 rows x 5 columns]\n"
],
[
"def RSI(df, base=\"Close\", period=21):\n \"\"\"\n Function to compute Relative Strength Index (RSI)\n \n Args :\n df : Pandas DataFrame which contains ['date', 'open', 'high', 'low', 'close', 'volume'] columns\n base : String indicating the column name from which the MACD needs to be computed from (Default Close)\n period : Integer indicates the period of computation in terms of number of candles\n \n Returns :\n df : Pandas DataFrame with new columns added for \n Relative Strength Index (RSI_$period)\n \"\"\"\n \n delta = df[base].diff()\n up, down = delta.copy(), delta.copy()\n\n up[up < 0] = 0\n down[down > 0] = 0\n \n rUp = up.ewm(com=period - 1, adjust=False).mean()\n rDown = down.ewm(com=period - 1, adjust=False).mean().abs()\n\n df['RSI_' + str(period)] = 100 - 100 / (1 + rUp / rDown)\n df['RSI_' + str(period)].fillna(0, inplace=True)\n\n return df",
"_____no_output_____"
],
[
"def BBand(df, base='Close', period=20, multiplier=2):\n \"\"\"\n Function to compute Bollinger Band (BBand)\n \n Args :\n df : Pandas DataFrame which contains ['date', 'open', 'high', 'low', 'close', 'volume'] columns\n base : String indicating the column name from which the MACD needs to be computed from (Default Close)\n period : Integer indicates the period of computation in terms of number of candles\n multiplier : Integer indicates value to multiply the SD\n \n Returns :\n df : Pandas DataFrame with new columns added for \n Upper Band (UpperBB_$period_$multiplier)\n Lower Band (LowerBB_$period_$multiplier)\n \"\"\"\n \n upper = 'UpperBB_' + str(period) + '_' + str(multiplier)\n lower = 'LowerBB_' + str(period) + '_' + str(multiplier)\n \n sma = df[base].rolling(window=period, min_periods=period - 1).mean()\n sd = df[base].rolling(window=period).std()\n df[upper] = sma + (multiplier * sd)\n df[lower] = sma - (multiplier * sd)\n \n df[upper].fillna(0, inplace=True)\n df[lower].fillna(0, inplace=True)\n \n return df",
"_____no_output_____"
],
[
"def MACD(df, fastEMA=12, slowEMA=26, signal=9, base='Close'):\n \"\"\"\n Function to compute Moving Average Convergence Divergence (MACD)\n \n Args :\n df : Pandas DataFrame which contains ['date', 'open', 'high', 'low', 'close', 'volume'] columns\n fastEMA : Integer indicates faster EMA\n slowEMA : Integer indicates slower EMA\n signal : Integer indicates the signal generator for MACD\n base : String indicating the column name from which the MACD needs to be computed from (Default Close)\n \n Returns :\n df : Pandas DataFrame with new columns added for \n Fast EMA (ema_$fastEMA)\n Slow EMA (ema_$slowEMA)\n MACD (macd_$fastEMA_$slowEMA_$signal)\n MACD Signal (signal_$fastEMA_$slowEMA_$signal)\n MACD Histogram (MACD (hist_$fastEMA_$slowEMA_$signal)) \n \"\"\"\n\n fE = \"ema_\" + str(fastEMA)\n sE = \"ema_\" + str(slowEMA)\n macd = \"macd_\" + str(fastEMA) + \"_\" + str(slowEMA) + \"_\" + str(signal)\n sig = \"signal_\" + str(fastEMA) + \"_\" + str(slowEMA) + \"_\" + str(signal)\n hist = \"hist_\" + str(fastEMA) + \"_\" + str(slowEMA) + \"_\" + str(signal)\n\n # Compute fast and slow EMA \n EMA(df, base, fE, fastEMA)\n EMA(df, base, sE, slowEMA)\n \n # Compute MACD\n df[macd] = np.where(np.logical_and(np.logical_not(df[fE] == 0), np.logical_not(df[sE] == 0)), df[fE] - df[sE], 0)\n \n # Compute MACD Signal\n EMA(df, macd, sig, signal)\n \n # Compute MACD Histogram\n df[hist] = np.where(np.logical_and(np.logical_not(df[macd] == 0), np.logical_not(df[sig] == 0)), df[macd] - df[sig], 0)\n \n return df",
"_____no_output_____"
],
[
"def ATR(df, period, ohlc=['Open', 'High', 'Low', 'Close']):\n \"\"\"\n Function to compute Average True Range (ATR)\n \n Args :\n df : Pandas DataFrame which contains ['date', 'open', 'high', 'low', 'close', 'volume'] columns\n period : Integer indicates the period of computation in terms of number of candles\n ohlc: List defining OHLC Column names (default ['Open', 'High', 'Low', 'Close'])\n \n Returns :\n df : Pandas DataFrame with new columns added for \n True Range (TR)\n ATR (ATR_$period)\n \"\"\"\n atr = 'ATR_' + str(period)\n\n # Compute true range only if it is not computed and stored earlier in the df\n if not 'TR' in df.columns:\n df['h-l'] = df[ohlc[1]] - df[ohlc[2]]\n df['h-yc'] = abs(df[ohlc[1]] - df[ohlc[3]].shift())\n df['l-yc'] = abs(df[ohlc[2]] - df[ohlc[3]].shift())\n \n df['TR'] = df[['h-l', 'h-yc', 'l-yc']].max(axis=1)\n \n df.drop(['h-l', 'h-yc', 'l-yc'], inplace=True, axis=1)\n\n # Compute EMA of true range using ATR formula after ignoring first row\n EMA(df, 'TR', atr, period, alpha=True)\n \n return df",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7ea930c40f26efa52736fe3b12c35c87c8f684e | 21,459 | ipynb | Jupyter Notebook | Kaggle-Competitions/Predict-Bio-Response/Predict-Bio-Response-Model-Building.ipynb | gopala-kr/ds-notebooks | bc35430ecdd851f2ceab8f2437eec4d77cb59423 | [
"MIT"
] | 1 | 2019-05-10T09:16:23.000Z | 2019-05-10T09:16:23.000Z | Kaggle-Competitions/Predict-Bio-Response/Predict-Bio-Response-Model-Building.ipynb | gopala-kr/ds-notebooks | bc35430ecdd851f2ceab8f2437eec4d77cb59423 | [
"MIT"
] | null | null | null | Kaggle-Competitions/Predict-Bio-Response/Predict-Bio-Response-Model-Building.ipynb | gopala-kr/ds-notebooks | bc35430ecdd851f2ceab8f2437eec4d77cb59423 | [
"MIT"
] | 1 | 2019-05-10T09:17:28.000Z | 2019-05-10T09:17:28.000Z | 93.3 | 8,582 | 0.85456 | [
[
[
"import pandas as pd\nimport numpy as np\n\nimport matplotlib.pyplot as plt\nimport seaborn as sns\n\nimport warnings\nwarnings.filterwarnings('ignore')\n\nfrom sklearn.cross_validation import train_test_split, StratifiedKFold, cross_val_score\nfrom sklearn.metrics import log_loss\nfrom sklearn.linear_model import LogisticRegression\nfrom sklearn.ensemble import RandomForestClassifier\nfrom sklearn.manifold import TSNE\n\nnp.random.seed(1234)\n\n%matplotlib inline",
"_____no_output_____"
],
[
"train = pd.read_csv('./data/train.csv')\ntest = pd.read_csv('./data/test.csv')",
"_____no_output_____"
]
],
[
[
"## Objectives\n\n* Consider only two features of the dataset obtained after t-sne\n* Find out best performing value of perplexity for t-sne using cross-validation\n* Use linear model with no calibration vs non linear model to see how well these perform",
"_____no_output_____"
]
],
[
[
"def get_training_set(train):\n X = train[train.columns.drop('Activity')]\n y = train.Activity\n \n return X, y\n\nX, y = get_training_set(train)\nXtest = test",
"_____no_output_____"
],
[
"X_train, X_test, y_train, y_test = train_test_split(X, y, stratify=y, test_size=0.3, random_state=44)",
"_____no_output_____"
]
],
[
[
"## Cross validation",
"_____no_output_____"
]
],
[
[
"def get_cross_val_scores_by_perplexity(X, y, clf):\n skf = StratifiedKFold(y, n_folds=3, shuffle=True, random_state=43)\n perplexity = [50, 75, 100]\n errors = []\n \n for p in perplexity:\n tsne = TSNE(n_components=2, perplexity=p)\n Xhat = tsne.fit_transform(X)\n \n cv_scores = cross_val_score(clf, Xhat, y, scoring='log_loss', cv=skf, n_jobs=-1)\n errors.append(cv_scores.mean())\n \n perplexity = np.array(perplexity)\n errors = np.array(errors)\n \n return perplexity, errors",
"_____no_output_____"
],
[
"perplexity, errors = get_cross_val_scores_by_perplexity(X, y, LogisticRegression(C=1.))",
"_____no_output_____"
],
[
"errors = map(abs, errors)",
"_____no_output_____"
],
[
"plt.scatter(perplexity, errors, color='b', alpha=0.5)\nplt.xlabel('Perplexity')\nplt.ylabel('Log Loss');",
"_____no_output_____"
],
[
"perplexity, errors = get_cross_val_scores_by_perplexity(X, y, RandomForestClassifier(n_jobs=-1))\nerrors = map(abs, errors)",
"_____no_output_____"
],
[
"plt.scatter(perplexity, errors, color='b', alpha=0.5)\nplt.xlabel('Perplexity')\nplt.ylabel('Log Loss');",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ea93a13ffc39995880f6dbe9df775c53cb9b15 | 416,370 | ipynb | Jupyter Notebook | bool-pvalues.ipynb | sobuch/polynomial-distinguishers | 5a007abd222d00cbf99f1083c3b537343d2fff56 | [
"MIT"
] | 5 | 2017-03-03T13:53:51.000Z | 2019-05-09T09:47:28.000Z | bool-pvalues.ipynb | sobuch/polynomial-distinguishers | 5a007abd222d00cbf99f1083c3b537343d2fff56 | [
"MIT"
] | 5 | 2017-10-07T11:15:09.000Z | 2021-01-25T17:03:59.000Z | bool-pvalues.ipynb | sobuch/polynomial-distinguishers | 5a007abd222d00cbf99f1083c3b537343d2fff56 | [
"MIT"
] | 6 | 2017-03-26T17:06:20.000Z | 2021-11-15T22:22:33.000Z | 235.770102 | 75,116 | 0.897334 | [
[
[
"import os\nimport json\nimport collections\nimport itertools\nimport copy\nimport pandas as pd\nimport seaborn as sns\nfrom matplotlib import pyplot\nimport matplotlib as mpl\nimport matplotlib.pyplot as plt\nimport matplotlib.ticker as ticker\nimport numpy as np\nimport matplotlib\nfrom scipy import stats\nfrom scipy.misc import derivative\nimport coloredlogs\nimport logging\n\n# Pval generation related functions, previously in the notebook\nfrom booltest.pvals import *\n\ncoloredlogs.CHROOT_FILES = []\ncoloredlogs.install(level=logging.INFO, use_chroot=False)",
"_____no_output_____"
],
[
"def pvalue_comp(fnc, extremes, dx, bin_tup, by_bins=True):\n \"\"\"Extremes = [(val, direction +1\\-1)] \"\"\"\n nints = len(extremes)\n areas = [0] * nints\n nbounds = [x[0] for x in extremes]\n nbins = [binize(x[0], bin_tup) for x in extremes]\n bmin = min(nbounds)\n bmax = max(nbounds)\n cp = 0\n iterc = 0\n results = []\n print('OK: ', nints, nbins, ' size: ', bin_tup[4])\n \n while cp <= 1.0: # integration step\n iterc += 1\n if iterc > 10000:\n raise ValueError('exc') # Hard-termination to avoid infinite cycle.\n \n # Integration by increasing pvalue and tabulating.\n # Each area grows at the same pace. pvalue is a sum of areas.\n # Termination - bounds are crossing / touching.\n \n # Integrate each area with one step but in such a way the area is the same. \n max_area = max(areas)\n min_area = min(areas)\n sum_area = sum(areas)\n err = max([abs(x) for x in all_diffs(areas)])\n areas_str = ['%.7f' % x for x in areas]\n #print('Main iter: %s, cp: %.7f, mina: %.7f, maxa: %.7f, suma: %.7f, err: %.7f, a: [%s], n: %s' \n # % (iterc, cp, min_area, max_area, sum_area, err, ', '.join(areas_str), nbins))\n \n subit = 0\n while any([x <= min_area for x in areas]):\n subit += 1\n #print('.. subit: %s' % subit)\n \n for ix in range(nints):\n if areas[ix] > min_area :\n continue\n if by_bins:\n areas[ix] += get_bin_val(nbins[ix], bin_tup)\n nbounds[ix] = get_bin_start(nbins[ix], bin_tup)\n nbins[ix] = move_bound(nbins[ix], 1, extremes[ix][1])\n else:\n areas[ix] += fnc(nbounds[ix])\n nbounds[ix] = move_bound(nbounds[ix], dx, extremes[ix][1]) \n cp = sum(areas)\n \n crit_int = [None]*nints\n for i in range(nints):\n crit_int[i] = (extremes[i][0], nbounds[i]) if extremes[i][1] > 0 else (nbounds[i], extremes[i][0])\n \n results.append((cp, crit_int, copy.deepcopy(areas), err))\n \n #print('Main iter: %s, cp: %s, mina: %s, maxa: %s, suma: %s, a: %s' \n # % (iterc, cp, min(areas), max(areas), sum(areas), areas))\n #print('Total: %s' % (sum([get_bin_val(ix, bin_tup) for ix in range(len(bin_tup[0]))])))\n #print(json.dumps(results, indent=2))\n return results\n\n\ndef tabulate_pvals(val, nbins=200, abs_val=False, target_pvals=[0.0, 0.00001, 0.0001, 0.0005, 0.001, 0.005, 0.01]):\n inp_iter = val['zscores']\n if abs_val:\n inp_iter = [abs(x) for x in inp_iter]\n \n bin_tup = get_bins(inp_iter, nbins=nbins, full=True)\n bb = get_distrib_fbins(inp_iter, bin_tup)\n \n bin_size = bin_tup[1]\n minv, maxv = bin_tup[2], bin_tup[3] \n bins = np.array([x[0] for x in bb])\n \n # Tabulate pvalues\n # build_integrator(bin_tup)\n extremes = [\n [minv, 1],\n [0, -1],\n [0, +1],\n [maxv, -1]\n ] if not abs_val else [\n [minv, 1],\n [maxv, -1]\n ]\n \n print('%s-%s-%s-%s-%s' % (val['method'], val['block'], val['deg'], val['comb_deg'], val['data_size']))\n pvals = pvalue_comp(lambda x: binned_pmf(x, bin_tup), extremes, \n dx=1./(nbins/10.), bin_tup=bin_tup, by_bins=True)\n\n res_pdata = []\n for target in target_pvals:\n chosen = 0\n for i in range(len(pvals)):\n chosen = i\n if pvals[i][0] >= target:\n chosen = i - 1 if i > 0 else 0\n break\n \n cdata = pvals[chosen]\n res_pdata.append(collections.OrderedDict([\n ('pval_target', target),\n ('pval', cdata[0]),\n ('crit', cdata[1]),\n ('areas', cdata[2]),\n ('err', cdata[3]),\n ]))\n\n return collections.OrderedDict([\n ('method', val['method']), \n ('block', val['block']),\n ('deg', val['deg']),\n ('comb_deg', val['comb_deg']),\n ('data_size', val['data_size']),\n ('nsamples', len(inp_iter)),\n ('nbins', nbins),\n ('abs_val', abs_val),\n ('binsize', bin_size),\n ('minv', minv),\n ('maxv', maxv),\n ('extremes', extremes),\n ('pvals', res_pdata)\n ])\n",
"_____no_output_____"
],
[
"js1 = json.load(open('/tmp/results_bat_1588463858-booltest1-ref3-40k.json'))",
"_____no_output_____"
],
[
"js2 = json.load(open('/tmp/results_bat_1588274711-booltest1-ref-60k.json'))",
"_____no_output_____"
],
[
"js = js1 + js2",
"_____no_output_____"
],
[
"if js:\n rs = extract_zscores_from_bat(js)",
"2020-05-03 15:09:13 phx.local booltest.pvals[14921] DEBUG Input candidates: 3600000\n2020-05-03 15:09:29 phx.local booltest.pvals[14921] DEBUG Skipped same seed: 0\n2020-05-03 15:09:30 phx.local booltest.pvals[14921] DEBUG Skipped same hash: 0\n2020-05-03 15:09:30 phx.local booltest.pvals[14921] DEBUG Skipped same dfile: 0\n"
],
[
"len(rs[0]['zscores'])",
"_____no_output_____"
],
[
"pval_db = []\ndata = data_filt = rs\nfor dix, val in enumerate(data_filt):\n res = tabulate_pvals(val, abs_val=True)\n pval_db.append(res)\n print('Dump %s' % dix)\njson.dump(pval_db, open('pval_db.json', 'w+'), indent=2)",
"AES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-1-1-10000000\nOK: 2 [0, 163] size: 100000\nDump 0\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-1-2-10000000\nOK: 2 [0, 149] size: 100000\nDump 1\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-1-3-10000000\nOK: 2 [0, 146] size: 100000\nDump 2\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-2-1-10000000\nOK: 2 [0, 155] size: 100000\nDump 3\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-2-2-10000000\nOK: 2 [0, 149] size: 100000\nDump 4\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-2-3-10000000\nOK: 2 [0, 149] size: 100000\nDump 5\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-3-1-10000000\nOK: 2 [0, 149] size: 100000\nDump 6\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-3-2-10000000\nOK: 2 [0, 145] size: 100000\nDump 7\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-128-3-3-10000000\nOK: 2 [0, 147] size: 100000\nDump 8\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-1-1-10000000\nOK: 2 [0, 153] size: 100000\nDump 9\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-1-2-10000000\nOK: 2 [0, 151] size: 100000\nDump 10\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-1-3-10000000\nOK: 2 [0, 149] size: 100000\nDump 11\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-2-1-10000000\nOK: 2 [0, 153] size: 100000\nDump 12\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-2-2-10000000\nOK: 2 [0, 147] size: 100000\nDump 13\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-2-3-10000000\nOK: 2 [0, 151] size: 100000\nDump 14\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-3-1-10000000\nOK: 2 [0, 144] size: 100000\nDump 15\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-3-2-10000000\nOK: 2 [0, 144] size: 100000\nDump 16\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-256-3-3-10000000\nOK: 2 [0, 141] size: 100000\nDump 17\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-1-1-10000000\nOK: 2 [0, 151] size: 100000\nDump 18\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-1-2-10000000\nOK: 2 [0, 148] size: 100000\nDump 19\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-1-3-10000000\nOK: 2 [0, 148] size: 100000\nDump 20\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-2-1-10000000\nOK: 2 [0, 147] size: 100000\nDump 21\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-2-2-10000000\nOK: 2 [0, 144] size: 100000\nDump 22\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-2-3-10000000\nOK: 2 [0, 146] size: 100000\nDump 23\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-3-1-10000000\nOK: 2 [0, 145] size: 100000\nDump 24\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-3-2-10000000\nOK: 2 [0, 138] size: 100000\nDump 25\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-384-3-3-10000000\nOK: 2 [0, 143] size: 100000\nDump 26\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-1-1-10000000\nOK: 2 [0, 153] size: 100000\nDump 27\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-1-2-10000000\nOK: 2 [0, 147] size: 100000\nDump 28\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-1-3-10000000\nOK: 2 [0, 145] size: 100000\nDump 29\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-2-1-10000000\nOK: 2 [0, 146] size: 100000\nDump 30\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-2-2-10000000\nOK: 2 [0, 145] size: 100000\nDump 31\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-2-3-10000000\nOK: 2 [0, 148] size: 100000\nDump 32\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-3-1-10000000\nOK: 2 [0, 140] size: 100000\nDump 33\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-3-2-10000000\nOK: 2 [0, 141] size: 100000\nDump 34\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7-512-3-3-10000000\nOK: 2 [0, 141] size: 100000\nDump 35\n"
]
],
[
[
"## Old way - csv",
"_____no_output_____"
]
],
[
[
"#js = json.load(open('ref_1554219251.json'))\n#csv = open('ref_1554219251.csv').read()\ncsv = open('ref_1558361146.csv').read()\n\ncsv_data = []\nfor rec in [x.strip() for x in csv.split(\"\\n\")]:\n p = rec.split(';')\n if len(p) < 6:\n continue\n cur = collections.OrderedDict([\n ('method', p[0]), \n ('block', int(p[1])),\n ('deg', int(p[2])),\n ('comb_deg', int(p[3])),\n ('data_size', int(p[4])),\n ('zscores', [float(x.replace(',','.')) for x in p[6:]])\n ])\n csv_data.append(cur)\nprint(json.dumps(csv_data[0])) ",
"{\"method\": \"in0-kctr-ri1\", \"block\": 128, \"deg\": 1, \"comb_deg\": 2, \"data_size\": 10, \"zscores\": [-3.77991]}\n"
],
[
"data = csv_data\ndata_filt = [x for x in data if x and len(x['zscores']) > 19000]\ndata_filt.sort(key=lambda x: (x['method'], x['block'], x['deg'], x['comb_deg'], x['data_size']))\nnp.random.seed(87655677)\nprint('#of inputs with data count over threshold: ', len(data_filt))",
"#of inputs with data count over threshold: 144\n"
],
[
"pval_db = []\nfor dix, val in enumerate(data_filt):\n res = tabulate_pvals(val, abs_val=True)\n pval_db.append(res)\n print('Dump %s' % dix)\njson.dump(pval_db, open('pval_db.json', 'w+'), indent=2)",
"inctr-krnd-ri0-128-1-1-10\nOK: 2 [0, 153] size: 20000\nDump 0\ninctr-krnd-ri0-128-1-2-10\nOK: 2 [0, 146] size: 19999\nDump 1\ninctr-krnd-ri0-128-1-3-10\nOK: 2 [0, 138] size: 20000\nDump 2\ninctr-krnd-ri0-128-2-1-10\nOK: 2 [0, 149] size: 20000\nDump 3\ninctr-krnd-ri0-128-2-2-10\nOK: 2 [0, 147] size: 20000\nDump 4\ninctr-krnd-ri0-128-2-3-10\nOK: 2 [0, 143] size: 20001\nDump 5\ninctr-krnd-ri0-128-3-1-10\nOK: 2 [0, 144] size: 20000\nDump 6\ninctr-krnd-ri0-128-3-2-10\nOK: 2 [0, 139] size: 20001\nDump 7\ninctr-krnd-ri0-128-3-3-10\nOK: 2 [0, 145] size: 20001\nDump 8\ninctr-krnd-ri0-256-1-1-10\nOK: 2 [0, 148] size: 20000\nDump 9\ninctr-krnd-ri0-256-1-2-10\nOK: 2 [0, 149] size: 19999\nDump 10\ninctr-krnd-ri0-256-1-3-10\nOK: 2 [0, 149] size: 20000\nDump 11\ninctr-krnd-ri0-256-2-1-10\nOK: 2 [0, 144] size: 20001\nDump 12\ninctr-krnd-ri0-256-2-2-10\nOK: 2 [0, 142] size: 20000\nDump 13\ninctr-krnd-ri0-256-2-3-10\nOK: 2 [0, 144] size: 20000\nDump 14\ninctr-krnd-ri0-256-3-1-10\nOK: 2 [0, 141] size: 20001\nDump 15\ninctr-krnd-ri0-256-3-2-10\nOK: 2 [0, 132] size: 20001\nDump 16\ninctr-krnd-ri0-256-3-3-10\nOK: 2 [0, 138] size: 20001\nDump 17\ninctr-krnd-ri0-384-1-1-10\nOK: 2 [0, 149] size: 20001\nDump 18\ninctr-krnd-ri0-384-1-2-10\nOK: 2 [0, 139] size: 20000\nDump 19\ninctr-krnd-ri0-384-1-3-10\nOK: 2 [0, 135] size: 20001\nDump 20\ninctr-krnd-ri0-384-2-1-10\nOK: 2 [0, 144] size: 20001\nDump 21\ninctr-krnd-ri0-384-2-2-10\nOK: 2 [0, 143] size: 20001\nDump 22\ninctr-krnd-ri0-384-2-3-10\nOK: 2 [0, 144] size: 20001\nDump 23\ninctr-krnd-ri0-384-3-1-10\nOK: 2 [0, 140] size: 20000\nDump 24\ninctr-krnd-ri0-384-3-2-10\nOK: 2 [0, 135] size: 20000\nDump 25\ninctr-krnd-ri0-384-3-3-10\nOK: 2 [0, 136] size: 20001\nDump 26\ninctr-krnd-ri0-512-1-1-10\nOK: 2 [0, 150] size: 20000\nDump 27\ninctr-krnd-ri0-512-1-2-10\nOK: 2 [0, 145] size: 20000\nDump 28\ninctr-krnd-ri0-512-1-3-10\nOK: 2 [0, 142] size: 20001\nDump 29\ninctr-krnd-ri0-512-2-1-10\nOK: 2 [0, 141] size: 20000\nDump 30\ninctr-krnd-ri0-512-2-2-10\nOK: 2 [0, 139] size: 20001\nDump 31\ninctr-krnd-ri0-512-2-3-10\nOK: 2 [0, 150] size: 20001\nDump 32\ninctr-krnd-ri0-512-3-1-10\nOK: 2 [0, 134] size: 20001\nDump 33\ninctr-krnd-ri0-512-3-2-10\nOK: 2 [0, 130] size: 20000\nDump 34\ninctr-krnd-ri0-512-3-3-10\nOK: 2 [0, 137] size: 20000\nDump 35\ninhw-krnd-ri0-128-1-1-10\nOK: 2 [0, 155] size: 40006\nDump 36\ninhw-krnd-ri0-128-1-2-10\nOK: 2 [0, 145] size: 40006\nDump 37\ninhw-krnd-ri0-128-1-3-10\nOK: 2 [0, 146] size: 40007\nDump 38\ninhw-krnd-ri0-128-2-1-10\nOK: 2 [0, 148] size: 40007\nDump 39\ninhw-krnd-ri0-128-2-2-10\nOK: 2 [0, 148] size: 40007\nDump 40\ninhw-krnd-ri0-128-2-3-10\nOK: 2 [0, 146] size: 40007\nDump 41\ninhw-krnd-ri0-128-3-1-10\nOK: 2 [0, 155] size: 40005\nDump 42\ninhw-krnd-ri0-128-3-2-10\nOK: 2 [0, 146] size: 40003\nDump 43\ninhw-krnd-ri0-128-3-3-10\nOK: 2 [0, 142] size: 40008\nDump 44\ninhw-krnd-ri0-256-1-1-10\nOK: 2 [0, 154] size: 40007\nDump 45\ninhw-krnd-ri0-256-1-2-10\nOK: 2 [0, 151] size: 40007\nDump 46\ninhw-krnd-ri0-256-1-3-10\nOK: 2 [0, 138] size: 40007\nDump 47\ninhw-krnd-ri0-256-2-1-10\nOK: 2 [0, 153] size: 40004\nDump 48\ninhw-krnd-ri0-256-2-2-10\nOK: 2 [0, 149] size: 40005\nDump 49\ninhw-krnd-ri0-256-2-3-10\nOK: 2 [0, 142] size: 40008\nDump 50\ninhw-krnd-ri0-256-3-1-10\nOK: 2 [0, 138] size: 40006\nDump 51\ninhw-krnd-ri0-256-3-2-10\nOK: 2 [0, 140] size: 40007\nDump 52\ninhw-krnd-ri0-256-3-3-10\nOK: 2 [0, 138] size: 40009\nDump 53\ninhw-krnd-ri0-384-1-1-10\nOK: 2 [0, 153] size: 40006\nDump 54\ninhw-krnd-ri0-384-1-2-10\nOK: 2 [0, 144] size: 40008\nDump 55\ninhw-krnd-ri0-384-1-3-10\nOK: 2 [0, 138] size: 40007\nDump 56\ninhw-krnd-ri0-384-2-1-10\nOK: 2 [0, 147] size: 40006\nDump 57\ninhw-krnd-ri0-384-2-2-10\nOK: 2 [0, 146] size: 40007\nDump 58\ninhw-krnd-ri0-384-2-3-10\nOK: 2 [0, 148] size: 40006\nDump 59\ninhw-krnd-ri0-384-3-1-10\nOK: 2 [0, 138] size: 40008\nDump 60\ninhw-krnd-ri0-384-3-2-10\nOK: 2 [0, 136] size: 40006\nDump 61\ninhw-krnd-ri0-384-3-3-10\nOK: 2 [0, 140] size: 40006\nDump 62\ninhw-krnd-ri0-512-1-1-10\nOK: 2 [0, 151] size: 40007\nDump 63\ninhw-krnd-ri0-512-1-2-10\nOK: 2 [0, 154] size: 40005\nDump 64\ninhw-krnd-ri0-512-1-3-10\nOK: 2 [0, 141] size: 40004\nDump 65\ninhw-krnd-ri0-512-2-1-10\nOK: 2 [0, 142] size: 40008\nDump 66\ninhw-krnd-ri0-512-2-2-10\nOK: 2 [0, 152] size: 40005\nDump 67\ninhw-krnd-ri0-512-2-3-10\nOK: 2 [0, 150] size: 40008\nDump 68\ninhw-krnd-ri0-512-3-1-10\nOK: 2 [0, 136] size: 40005\nDump 69\ninhw-krnd-ri0-512-3-2-10\nOK: 2 [0, 138] size: 40007\nDump 70\ninhw-krnd-ri0-512-3-3-10\nOK: 2 [0, 145] size: 40007\nDump 71\ninhw4-krnd-ri0-128-1-1-10\nOK: 2 [0, 151] size: 20000\nDump 72\ninhw4-krnd-ri0-128-1-2-10\nOK: 2 [0, 139] size: 20001\nDump 73\ninhw4-krnd-ri0-128-1-3-10\nOK: 2 [0, 145] size: 20000\nDump 74\ninhw4-krnd-ri0-128-2-1-10\nOK: 2 [0, 147] size: 20000\nDump 75\ninhw4-krnd-ri0-128-2-2-10\nOK: 2 [0, 148] size: 20000\nDump 76\ninhw4-krnd-ri0-128-2-3-10\nOK: 2 [0, 144] size: 20000\nDump 77\ninhw4-krnd-ri0-128-3-1-10\nOK: 2 [0, 155] size: 20000\nDump 78\ninhw4-krnd-ri0-128-3-2-10\nOK: 2 [0, 146] size: 20000\nDump 79\ninhw4-krnd-ri0-128-3-3-10\nOK: 2 [0, 142] size: 20001\nDump 80\ninhw4-krnd-ri0-256-1-1-10\nOK: 2 [0, 154] size: 20000\nDump 81\ninhw4-krnd-ri0-256-1-2-10\nOK: 2 [0, 150] size: 20000\nDump 82\ninhw4-krnd-ri0-256-1-3-10\nOK: 2 [0, 136] size: 20000\nDump 83\ninhw4-krnd-ri0-256-2-1-10\nOK: 2 [0, 153] size: 20000\nDump 84\ninhw4-krnd-ri0-256-2-2-10\nOK: 2 [0, 141] size: 20000\nDump 85\ninhw4-krnd-ri0-256-2-3-10\nOK: 2 [0, 139] size: 20002\nDump 86\ninhw4-krnd-ri0-256-3-1-10\nOK: 2 [0, 136] size: 20000\nDump 87\ninhw4-krnd-ri0-256-3-2-10\nOK: 2 [0, 139] size: 20000\nDump 88\ninhw4-krnd-ri0-256-3-3-10\nOK: 2 [0, 133] size: 20002\nDump 89\ninhw4-krnd-ri0-384-1-1-10\nOK: 2 [0, 146] size: 19999\nDump 90\ninhw4-krnd-ri0-384-1-2-10\nOK: 2 [0, 143] size: 20001\nDump 91\ninhw4-krnd-ri0-384-1-3-10\nOK: 2 [0, 136] size: 20000\nDump 92\ninhw4-krnd-ri0-384-2-1-10\nOK: 2 [0, 145] size: 20000\nDump 93\ninhw4-krnd-ri0-384-2-2-10\nOK: 2 [0, 141] size: 20001\nDump 94\ninhw4-krnd-ri0-384-2-3-10\nOK: 2 [0, 147] size: 20000\nDump 95\ninhw4-krnd-ri0-384-3-1-10\nOK: 2 [0, 138] size: 20001\nDump 96\ninhw4-krnd-ri0-384-3-2-10\nOK: 2 [0, 131] size: 20000\nDump 97\ninhw4-krnd-ri0-384-3-3-10\nOK: 2 [0, 133] size: 20000\nDump 98\ninhw4-krnd-ri0-512-1-1-10\nOK: 2 [0, 147] size: 20000\nDump 99\ninhw4-krnd-ri0-512-1-2-10\nOK: 2 [0, 143] size: 19999\nDump 100\ninhw4-krnd-ri0-512-1-3-10\nOK: 2 [0, 135] size: 19997\nDump 101\ninhw4-krnd-ri0-512-2-1-10\nOK: 2 [0, 142] size: 20001\nDump 102\ninhw4-krnd-ri0-512-2-2-10\nOK: 2 [0, 147] size: 19999\nDump 103\ninhw4-krnd-ri0-512-2-3-10\nOK: 2 [0, 150] size: 20001\nDump 104\ninhw4-krnd-ri0-512-3-1-10\nOK: 2 [0, 135] size: 20000\nDump 105\ninhw4-krnd-ri0-512-3-2-10\nOK: 2 [0, 138] size: 20000\nDump 106\ninhw4-krnd-ri0-512-3-3-10\nOK: 2 [0, 145] size: 20001\nDump 107\ninhw6-krnd-ri0-128-1-1-10\nOK: 2 [0, 155] size: 20000\nDump 108\ninhw6-krnd-ri0-128-1-2-10\nOK: 2 [0, 145] size: 20000\nDump 109\ninhw6-krnd-ri0-128-1-3-10\nOK: 2 [0, 140] size: 20000\nDump 110\ninhw6-krnd-ri0-128-2-1-10\nOK: 2 [0, 147] size: 20000\nDump 111\ninhw6-krnd-ri0-128-2-2-10\nOK: 2 [0, 141] size: 20000\nDump 112\ninhw6-krnd-ri0-128-2-3-10\nOK: 2 [0, 146] size: 20000\nDump 113\ninhw6-krnd-ri0-128-3-1-10\nOK: 2 [0, 138] size: 20000\nDump 114\ninhw6-krnd-ri0-128-3-2-10\nOK: 2 [0, 135] size: 20000\nDump 115\ninhw6-krnd-ri0-128-3-3-10\nOK: 2 [0, 140] size: 20000\nDump 116\ninhw6-krnd-ri0-256-1-1-10\nOK: 2 [0, 150] size: 20000\nDump 117\ninhw6-krnd-ri0-256-1-2-10\nOK: 2 [0, 142] size: 20000\nDump 118\ninhw6-krnd-ri0-256-1-3-10\nOK: 2 [0, 138] size: 20000\nDump 119\ninhw6-krnd-ri0-256-2-1-10\nOK: 2 [0, 146] size: 20000\nDump 120\ninhw6-krnd-ri0-256-2-2-10\nOK: 2 [0, 149] size: 20000\nDump 121\ninhw6-krnd-ri0-256-2-3-10\nOK: 2 [0, 141] size: 20000\nDump 122\ninhw6-krnd-ri0-256-3-1-10\nOK: 2 [0, 138] size: 20000\nDump 123\ninhw6-krnd-ri0-256-3-2-10\nOK: 2 [0, 135] size: 20000\nDump 124\ninhw6-krnd-ri0-256-3-3-10\nOK: 2 [0, 138] size: 20000\nDump 125\ninhw6-krnd-ri0-384-1-1-10\nOK: 2 [0, 152] size: 20000\nDump 126\ninhw6-krnd-ri0-384-1-2-10\nOK: 2 [0, 143] size: 20000\nDump 127\ninhw6-krnd-ri0-384-1-3-10\nOK: 2 [0, 137] size: 20000\nDump 128\ninhw6-krnd-ri0-384-2-1-10\nOK: 2 [0, 147] size: 20000\nDump 129\ninhw6-krnd-ri0-384-2-2-10\nOK: 2 [0, 146] size: 20000\nDump 130\ninhw6-krnd-ri0-384-2-3-10\nOK: 2 [0, 145] size: 20000\n"
],
[
"nbins = 200\nabs_val = True\n \nfor dix, val in enumerate(data_filt):\n inp_iter = (val['zscores'])\n if abs_val:\n inp_iter = [abs(x) for x in inp_iter]\n \n print('%s[%s:%s:%s:%s]: %s %s' \n % (val['method'], val['block'], val['deg'], val['comb_deg'], \n val['data_size'], len(val['zscores']),\n '',#dst.ppf([1-0.0001, 1-0.001, 1-0.01, 1-0.05, 1-0.10, 1-0.5, 0, 1, 0.0001, 0.001, 0.1, 0.9])\n #dst.stats(moments='mvsk')\n ))\n \n bin_tup = get_bins(inp_iter, nbins=nbins, full=True)\n bb = get_distrib_fbins(inp_iter, bin_tup)\n \n bin_size = bin_tup[1]\n minv, maxv = bin_tup[2], bin_tup[3] \n bins = np.array([x[0] for x in bb])\n dst = stats.rv_discrete(values=([x[0] for x in bb], [x[1] for x in bb]))\n print(stats.rv_discrete)\n \n x=np.array([bins[0], bins[1], bins[6]])\n print(dst.pmf(x))\n print(dst._pmf(x))\n \n # Tabulate pvalues\n build_integrator(bin_tup)\n extremes = [\n [minv, 1],\n [0, -1],\n [0, +1],\n [maxv, -1]\n ] if not abs_val else [\n [minv, 1],\n [maxv, -1]\n ]\n \n pvals = pvalue_comp(lambda x: binned_pmf(x, bin_tup), extremes, \n dx=1./(nbins/10.), bin_tup=bin_tup, by_bins=True)\n \n n_sample = 100\n rvs = dst.rvs(size=n_sample)\n f, l = np.histogram(rvs, bins=bins)\n f = np.append(f, [0])\n probs = np.array([x[1] for x in bb])\n #print(bins, len(bins))\n #print(probs, len(probs))\n #print(f, len(f))\n #sfreq = np.vstack([np.array([x[0] for x in bb]), f, probs*n_sample]).T\n #print(sfreq)\n \n print('%s[%s:%s:%s:%s]: %s %s' \n % (val['method'], val['block'], val['deg'], val['comb_deg'], \n val['data_size'], len(val['zscores']),\n dst.ppf([1-0.0001, 1-0.001, 1-0.01, 1-0.05, 1-0.10, 1-0.5, 0, 1, 0.0001, 0.001, 0.1, 0.9])\n #dst.stats(moments='mvsk')\n ))\n \n x = np.linspace(min(bins),max(bins),1000)\n plt.plot(x, dst.cdf(x))\n plt.show()\n \n cdf_dev = derivative(dst.cdf, x, dx=0.5)\n plt.plot(x,cdf_dev)\n \n sec_x = pvals[40] # 49\n print('Plotting area under: ', sec_x)\n for ix in range(len(sec_x[1])):\n section = np.arange(sec_x[1][ix][0], sec_x[1][ix][1], 1/20.)\n plt.fill_between(section, derivative(dst.cdf, section, dx=0.5))\n plt.show()\n \n #for pv in pvals:\n # sec_x = pv\n # for ix in range(len(sec_x[1])):\n # section = np.arange(sec_x[1][ix][0], sec_x[1][ix][1], 1/20.)\n # plt.fill_between(section, derivative(dst.cdf, section, dx=0.5))\n # plt.show()\n \n x = np.linspace(0,100,10000)\n plt.plot(x,dst.ppf(x))\n plt.show()\n \n x = np.linspace(minv,maxv,10000)\n plt.plot(bins, dst._pmf(bins))\n plt.show()\n \n x = np.linspace(minv,maxv,10000)\n plt.plot(x, [binned_pmf(y, bin_tup) for y in x])\n for ix in range(len(sec_x[1])):\n section = np.linspace(sec_x[1][ix][0], sec_x[1][ix][1], 10000) #np.arange(sec_x[1][ix][0], sec_x[1][ix][1], 1/20.)\n plt.fill_between(section, [binned_pmf(y, bin_tup)+0.0005 for y in section])\n plt.show()\n \n # Idea: pvalue function = pms of the distribution. \n # If test returns z-score with p=0 then we reject the hypothesis as we didnt get such zscore\n # If test returns with p=0.3 we dont reject as we have our alpha set somehow...\n # Problem: number of bins. If too many, we have small probabilities -> some alphas not reachable.\n #if dix > 3:\n break\n ",
"AES-r10-e-cfg80a9374f52f32147903b0b2240c7[128:1:1:10000000]: 100000 \n<class 'scipy.stats._distn_infrastructure.rv_discrete'>\n[0. 0. 0.]\n[1.0e-05 3.0e-05 1.6e-04]\nOK: 2 [0, 163] size: 100000\nAES-r10-e-cfg80a9374f52f32147903b0b2240c7[128:1:1:10000000]: 100000 [5.00498027 4.48483105 3.93730555 3.52666142 3.3350275 2.76012572\n 0.692451 6.15478382 1.77457982 1.93883747 2.3494816 3.3350275 ]\n"
],
[
"np.arange(7)\n#np.zeros(np.shape(0.5),'d')\n#print(dst.ppf([1-0.01, 1-0.05, 1-0.10, 0.5, 0.6]))",
"_____no_output_____"
],
[
"import itertools\ndef crit_regions(data, add_zscores=False):\n iter_block = [128, 256, 384, 512]\n iter_deg = [1, 2, 3]\n iter_cdeg = [1, 2, 3]\n iter = itertools.product(iter_block, iter_deg, iter_cdeg)\n for ix, crec in enumerate(iter):\n c = tuple(crec)\n reldata = [x for x in data if (x['block'], x['deg'], x['comb_deg']) == c and len(x['zscores']) > 9999]\n reldata.sort(key=lambda x: -len(x['zscores']))\n relrecs = []\n for crel in reldata:\n zsz = [abs(x) for x in crel['zscores']]\n relrecs.append((crel['method'], len(zsz), min(zsz), max(zsz), crel['zscores'] if add_zscores else None))\n \n yield (crec, relrecs)",
"_____no_output_____"
],
[
"np.mean(list(range(10)))",
"_____no_output_____"
],
[
"# Prints markdown critical region table\ndef crit_regions_table(data):\n dt = list(crit_regions(data, True))\n max_num = 9\n hdr = ['block', 'deg', 'comb-deg', 'samples', 'alpha', 'min', 'max', 'mean', 'stddev']\n max_hdr = max([len(x) for x in hdr])\n max_field = max(max_hdr, max_num) + 1\n \n hdr_spaced = [x.rjust(max_field) for x in hdr]\n print('|%s |' % ' |'.join(hdr_spaced))\n print('|%s|' % '|'.join([''.rjust(max_field + 1, '-') for _ in hdr]))\n for r in dt:\n cr, cd = r[0], r[1][0]\n zsz = [abs(x) for x in cd[4]]\n elems = [*cr] + [cd[1], '%.1e' % (1./cd[1]), \n '%9.6f' % cd[2], '%9.6f' % cd[3], \n '%9.6f' % np.mean(zsz), '%9.6f' % np.std(zsz)]\n elems = [str(x).rjust(max_field) for x in elems]\n print('|%s |' % ' |'.join(elems))\n \n\ncrit_regions_table(data)",
"| block | deg | comb-deg | samples | alpha | min | max | mean | stddev |\n|-----------|-----------|-----------|-----------|-----------|-----------|-----------|-----------|-----------|\n| 128 | 1 | 1 | 100000 | 1.0e-05 | 1.692451 | 6.167706 | 2.827212 | 0.392073 |\n| 128 | 1 | 2 | 100000 | 1.0e-05 | 3.205285 | 6.203124 | 3.968438 | 0.296394 |\n| 128 | 1 | 3 | 100000 | 1.0e-05 | 4.075543 | 6.790043 | 4.786912 | 0.252671 |\n| 128 | 2 | 1 | 100000 | 1.0e-05 | 2.935793 | 6.447060 | 3.888612 | 0.336659 |\n| 128 | 2 | 2 | 100000 | 1.0e-05 | 4.358132 | 7.336712 | 5.284895 | 0.303586 |\n| 128 | 2 | 3 | 100000 | 1.0e-05 | 4.873983 | 7.796648 | 5.770370 | 0.295979 |\n| 128 | 3 | 1 | 100000 | 1.0e-05 | 3.866804 | 6.823321 | 4.708614 | 0.290063 |\n| 128 | 3 | 2 | 100000 | 1.0e-05 | 5.698085 | 8.376927 | 6.560998 | 0.265912 |\n| 128 | 3 | 3 | 100000 | 1.0e-05 | 6.749871 | 9.570848 | 7.616965 | 0.282312 |\n| 256 | 1 | 1 | 100000 | 1.0e-05 | 1.999939 | 5.302164 | 3.044472 | 0.368626 |\n| 256 | 1 | 2 | 100000 | 1.0e-05 | 3.191316 | 6.303923 | 3.969717 | 0.297037 |\n| 256 | 1 | 3 | 100000 | 1.0e-05 | 4.096477 | 7.026620 | 4.786630 | 0.252239 |\n| 256 | 2 | 1 | 100000 | 1.0e-05 | 3.304946 | 6.601629 | 4.221227 | 0.310189 |\n| 256 | 2 | 2 | 100000 | 1.0e-05 | 4.775841 | 7.609938 | 5.659143 | 0.293182 |\n| 256 | 2 | 3 | 100000 | 1.0e-05 | 5.188120 | 8.305500 | 6.051743 | 0.289642 |\n| 256 | 3 | 1 | 100000 | 1.0e-05 | 4.389393 | 7.001934 | 5.130026 | 0.263766 |\n| 256 | 3 | 2 | 100000 | 1.0e-05 | 6.402618 | 9.003259 | 7.139777 | 0.245306 |\n| 256 | 3 | 3 | 100000 | 1.0e-05 | 7.408662 | 9.833698 | 8.247723 | 0.263262 |\n| 384 | 1 | 1 | 100000 | 1.0e-05 | 2.175556 | 5.326058 | 3.165671 | 0.359287 |\n| 384 | 1 | 2 | 100000 | 1.0e-05 | 3.214039 | 6.110398 | 3.968937 | 0.296144 |\n| 384 | 1 | 3 | 100000 | 1.0e-05 | 4.138595 | 7.017427 | 4.787164 | 0.252258 |\n| 384 | 2 | 1 | 100000 | 1.0e-05 | 3.563257 | 6.406780 | 4.405090 | 0.297651 |\n| 384 | 2 | 2 | 100000 | 1.0e-05 | 4.972945 | 7.606778 | 5.859061 | 0.288056 |\n| 384 | 2 | 3 | 100000 | 1.0e-05 | 5.355741 | 8.065769 | 6.201906 | 0.284984 |\n| 384 | 3 | 1 | 100000 | 1.0e-05 | 4.621511 | 7.317737 | 5.363479 | 0.251736 |\n| 384 | 3 | 2 | 100000 | 1.0e-05 | 6.729801 | 8.957334 | 7.454564 | 0.235683 |\n| 384 | 3 | 3 | 100000 | 1.0e-05 | 7.666722 | 10.225207 | 8.585460 | 0.254527 |\n| 512 | 1 | 1 | 100000 | 1.0e-05 | 2.292019 | 5.580788 | 3.250047 | 0.351051 |\n| 512 | 1 | 2 | 100000 | 1.0e-05 | 3.177457 | 5.980500 | 3.968829 | 0.295422 |\n| 512 | 1 | 3 | 100000 | 1.0e-05 | 4.143849 | 6.820400 | 4.786089 | 0.251366 |\n| 512 | 2 | 1 | 100000 | 1.0e-05 | 3.736198 | 6.505483 | 4.530410 | 0.289203 |\n| 512 | 2 | 2 | 100000 | 1.0e-05 | 5.156339 | 7.839674 | 5.997271 | 0.286677 |\n| 512 | 2 | 3 | 100000 | 1.0e-05 | 5.459804 | 8.324528 | 6.304574 | 0.283540 |\n| 512 | 3 | 1 | 100000 | 1.0e-05 | 4.840198 | 7.196232 | 5.520881 | 0.242361 |\n| 512 | 3 | 2 | 100000 | 1.0e-05 | 6.931561 | 9.324313 | 7.667548 | 0.228464 |\n| 512 | 3 | 3 | 100000 | 1.0e-05 | 7.886802 | 10.312977 | 8.815687 | 0.249666 |\n"
],
[
"cregions = list(crit_regions(data, True))",
"_____no_output_____"
],
[
"colors = [\n *sns.color_palette(\"ch:1.0,-.1,light=.7,dark=.4\", 9),\n *sns.color_palette(\"ch:2.0,-.1,light=.7,dark=.4\", 9),\n *sns.color_palette(\"ch:2.9,-.1,light=.7,dark=.4\", 9),\n *sns.color_palette(\"ch:0.5,-.1,light=.7,dark=.4\", 9),\n]\n\nsns.palplot(colors)",
"_____no_output_____"
],
[
"# z-score dist plot\na4_dims = (10.7, 6.27) #None #(2*11.7, 8.27)\nylim = (0.0, 2.2740367425079864)\nxlim = (1.1900278616886606, 11.30353802905374)\n\n\ndef distplots(cregions, colors, desc, fname, xlim=None, ylim=None):\n fig, ax = pyplot.subplots(figsize=a4_dims)\n \n for i in range(len(cregions)):\n zs = [abs(x) for x in cregions[i][1][0][4]]\n lbl = '-'.join([str(x) for x in cregions[i][0]])\n \n r = sns.distplot(a=zs, ax=ax, hist=True, norm_hist=False, bins='auto', label=lbl, kde=True, color=colors[i])\n lx = r.lines[-1].get_xdata()\n ly = r.lines[-1].get_ydata()\n maxi = np.argmax(ly)\n \n ax.annotate(lbl, (lx[maxi], ly[maxi] + 0.1), xytext=(lx[maxi], 2.1),\n rotation=90, ha='center', va='center', \n arrowprops={'arrowstyle':'->', 'connectionstyle':'arc3',\n \"linestyle\":\"--\", \"linewidth\":0.8, \"color\":'0.6'})\n print(desc, i, len(zs), ly[maxi])\n \n plt.legend(loc='upper right')\n plt.title(desc if desc else 'z-score distributions')\n ax.set_xlabel(\"z-score\") \n ax.set_ylabel(\"Hist\")\n if xlim: ax.set_xlim(xlim)\n if ylim: ax.set_ylim(ylim)\n \n plt.savefig(fname, bbox_inches='tight', dpi=400)\n #plt.savefig(os.path.splitext(fname)[0] + '.pdf', bbox_inches='tight', dpi=400)\n return fig, ax\n\n# fig, ax = distplots(cregions, colors, 'z-score distributions', 'zscore-dists.png', xlim, ylim)\n# print('ylim', ax.get_ylim())\n# print('xlim', ax.get_xlim())\n\ndistplots(cregions[0*9:1*9], colors[0*9:1*9], 'z-score distributions, m=128', 'zscore-dists-128.png', xlim, ylim)\ndistplots(cregions[1*9:2*9], colors[1*9:2*9], 'z-score distributions, m=256', 'zscore-dists-256.png', xlim, ylim)\ndistplots(cregions[2*9:3*9], colors[2*9:3*9], 'z-score distributions, m=384', 'zscore-dists-384.png', xlim, ylim)\ndistplots(cregions[3*9:4*9], colors[3*9:4*9], 'z-score distributions, m=512', 'zscore-dists-512.png', xlim, ylim)",
"z-score distributions, m=128 0 100000 1.0860145915038812\nz-score distributions, m=128 1 100000 1.4898752914235662\nz-score distributions, m=128 2 100000 1.775244407949746\nz-score distributions, m=128 3 100000 1.2619474040479957\nz-score distributions, m=128 4 100000 1.3988954645627572\nz-score distributions, m=128 5 100000 1.4293099840415195\nz-score distributions, m=128 6 100000 1.457388007034366\nz-score distributions, m=128 7 100000 1.5833428682501467\nz-score distributions, m=128 8 100000 1.470101465047176\nz-score distributions, m=256 0 100000 1.155550064087031\nz-score distributions, m=256 1 100000 1.4922073675233691\nz-score distributions, m=256 2 100000 1.7754761820092513\nz-score distributions, m=256 3 100000 1.390848892630593\nz-score distributions, m=256 4 100000 1.4526803121198308\nz-score distributions, m=256 5 100000 1.4782328530024624\nz-score distributions, m=256 6 100000 1.6457794888760504\nz-score distributions, m=256 7 100000 1.720981496703932\nz-score distributions, m=256 8 100000 1.5743692739576993\nz-score distributions, m=384 0 100000 1.1973841102488112\nz-score distributions, m=384 1 100000 1.49988259393262\nz-score distributions, m=384 2 100000 1.7893995977758541\nz-score distributions, m=384 3 100000 1.4387241480371875\nz-score distributions, m=384 4 100000 1.474419247403731\nz-score distributions, m=384 5 100000 1.484486413165005\nz-score distributions, m=384 6 100000 1.732010655541338\nz-score distributions, m=384 7 100000 1.8058103891297945\nz-score distributions, m=384 8 100000 1.6482672294324072\nz-score distributions, m=512 0 100000 1.2331322903255877\nz-score distributions, m=512 1 100000 1.4913518557787002\nz-score distributions, m=512 2 100000 1.7637543383491219\nz-score distributions, m=512 3 100000 1.5132793986675874\nz-score distributions, m=512 4 100000 1.4921456241920505\nz-score distributions, m=512 5 100000 1.5039691830383564\nz-score distributions, m=512 6 100000 1.7899256094378988\nz-score distributions, m=512 7 100000 1.8570929715831315\nz-score distributions, m=512 8 100000 1.6844789082574791\n"
],
[
"a4_dims = (2*11.7, 8.27)\nfig, ax = pyplot.subplots(figsize=a4_dims)\nzs = data_filt[1]['zscores']\n\nfor i in range(5):\n zs = [(x) for x in data_filt[i]['zscores']]\n print(len(zs))\n sns.distplot(a=zs, ax=ax, hist=True, norm_hist=False, bins='auto')",
"_____no_output_____"
]
],
[
[
"# pvalues\n - pvalue = probability (in the null hypothesis distribution) to be observed as a value equal to or more extreme than the value observed\n \n## computation \n - Derive CDF -> find 0 regions = extremes\n - Integrate from 0 regions towards region of increasing integral value. \n - Once sum of all integrations is alpha, stop. Integrated area is a critical region\n - Computation for x: integrate until the first integral boundary hits x. pvalue = sum of integrals\n - Tabulation: for each desired pvalue compute boundaries (4 values) where critical region starts. \n - pvalue(x): need to do the integration OR function table (\\forall zscores: P(zscore) > 0).\n - In our case 4 extremes, integrate: \n - -\\inf towards 0\n - +\\inf towards 0\n - 0 towards +\\inf\n - 0 towards -\\inf\n - 10000 samples, pvalue = 0 -> 1/10000. \n - absolutize -> we have a new distribution -> 2x more datapoints, 2 tails. \n ",
"_____no_output_____"
]
],
[
[
"np.arange(-1, 1, 1/20.)",
"_____no_output_____"
],
[
"counter = [0] * 8\nMAXV = 2\n\ndef inc(counter):\n global MAXV\n ln = len(counter) - 1\n while ln >= 0:\n counter[ln] = (counter[ln] + 1) % MAXV\n if (counter[ln] != 0):\n return(counter)\n ln-=1\n raise ValueError('Overflow')\n\n \ndef dec(counter):\n global MAXV\n ln = len(counter) - 1\n while ln >= 0:\n counter[ln] = (counter[ln] - 1) % MAXV\n if (counter[ln] != MAXV-1):\n return counter\n ln-=1\n raise ValueError('Underflow')\n \n \nfor i in range(20):\n print(inc(counter))\nprint('-'*80)\nfor i in range(20):\n print(dec(counter))",
"[0, 0, 0, 0, 0, 0, 0, 1]\n[0, 0, 0, 0, 0, 0, 1, 0]\n[0, 0, 0, 0, 0, 0, 1, 1]\n[0, 0, 0, 0, 0, 1, 0, 0]\n[0, 0, 0, 0, 0, 1, 0, 1]\n[0, 0, 0, 0, 0, 1, 1, 0]\n[0, 0, 0, 0, 0, 1, 1, 1]\n[0, 0, 0, 0, 1, 0, 0, 0]\n[0, 0, 0, 0, 1, 0, 0, 1]\n[0, 0, 0, 0, 1, 0, 1, 0]\n[0, 0, 0, 0, 1, 0, 1, 1]\n[0, 0, 0, 0, 1, 1, 0, 0]\n[0, 0, 0, 0, 1, 1, 0, 1]\n[0, 0, 0, 0, 1, 1, 1, 0]\n[0, 0, 0, 0, 1, 1, 1, 1]\n[0, 0, 0, 1, 0, 0, 0, 0]\n[0, 0, 0, 1, 0, 0, 0, 1]\n[0, 0, 0, 1, 0, 0, 1, 0]\n[0, 0, 0, 1, 0, 0, 1, 1]\n[0, 0, 0, 1, 0, 1, 0, 0]\n--------------------------------------------------------------------------------\n[0, 0, 0, 1, 0, 0, 1, 1]\n[0, 0, 0, 1, 0, 0, 1, 0]\n[0, 0, 0, 1, 0, 0, 0, 1]\n[0, 0, 0, 1, 0, 0, 0, 0]\n[0, 0, 0, 0, 1, 1, 1, 1]\n[0, 0, 0, 0, 1, 1, 1, 0]\n[0, 0, 0, 0, 1, 1, 0, 1]\n[0, 0, 0, 0, 1, 1, 0, 0]\n[0, 0, 0, 0, 1, 0, 1, 1]\n[0, 0, 0, 0, 1, 0, 1, 0]\n[0, 0, 0, 0, 1, 0, 0, 1]\n[0, 0, 0, 0, 1, 0, 0, 0]\n[0, 0, 0, 0, 0, 1, 1, 1]\n[0, 0, 0, 0, 0, 1, 1, 0]\n[0, 0, 0, 0, 0, 1, 0, 1]\n[0, 0, 0, 0, 0, 1, 0, 0]\n[0, 0, 0, 0, 0, 0, 1, 1]\n[0, 0, 0, 0, 0, 0, 1, 0]\n[0, 0, 0, 0, 0, 0, 0, 1]\n[0, 0, 0, 0, 0, 0, 0, 0]\n"
],
[
"from booltest import common\n",
"_____no_output_____"
],
[
"common.generate_seed(2)",
"_____no_output_____"
],
[
"import os\nimport time\ntmp_files = os.scandir('/tmp')\nfor i in tmp_files:\n print(i)\n time.sleep(10)",
"<DirEntry 'com.apple.launchd.RxJEu0LeJ5'>\n<DirEntry 'dumps'>\n<DirEntry 'powerlog'>\n<DirEntry 'com.apple.launchd.nOl6PZPwzO'>\n<DirEntry 'com.adobe.reader.rna.0.1f5.DC'>\n<DirEntry 'com.apple.launchd.w3Z9dzVsCy'>\n<DirEntry 'com.adobe.reader.rna.225.1f5'>\n"
],
[
"3*3*3*10000*3\n",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7eaa8b291ce43ae1553320bc41e7d7c8f7e1cf9 | 502 | ipynb | Jupyter Notebook | chapter1 install/Untitled.ipynb | wishwisdom/fn_tutorial | 26cc6be0e98d8f7a38e8d2dec30a1a9e7a6b54e7 | [
"MIT"
] | null | null | null | chapter1 install/Untitled.ipynb | wishwisdom/fn_tutorial | 26cc6be0e98d8f7a38e8d2dec30a1a9e7a6b54e7 | [
"MIT"
] | null | null | null | chapter1 install/Untitled.ipynb | wishwisdom/fn_tutorial | 26cc6be0e98d8f7a38e8d2dec30a1a9e7a6b54e7 | [
"MIT"
] | null | null | null | 15.6875 | 28 | 0.505976 | [] | [] | [] |
e7eabe61164d0a386819cc6289dd7bce253d3893 | 251,307 | ipynb | Jupyter Notebook | PaddleCare/Deep_learning/Grad_CAM_Pytorch-1.01-Stephenfang51-patch-1/Grad_CAM_Pytorch_1_tutorialv2.ipynb | momozi1996/Medical_AI_analysis | e1736a9e0d4c1b0996badbbeb870703fef119ab2 | [
"OML"
] | 2 | 2022-01-26T13:54:28.000Z | 2022-03-01T05:54:14.000Z | PaddleCare/Deep_learning/Grad_CAM_Pytorch-1.01-Stephenfang51-patch-1/Grad_CAM_Pytorch_1_tutorialv2.ipynb | momozi1996/Medical_AI_analysis | e1736a9e0d4c1b0996badbbeb870703fef119ab2 | [
"OML"
] | null | null | null | PaddleCare/Deep_learning/Grad_CAM_Pytorch-1.01-Stephenfang51-patch-1/Grad_CAM_Pytorch_1_tutorialv2.ipynb | momozi1996/Medical_AI_analysis | e1736a9e0d4c1b0996badbbeb870703fef119ab2 | [
"OML"
] | null | null | null | 654.445313 | 239,076 | 0.945405 | [
[
[
"### GradCAM (Gradient class activation mappinng)\n\n这个版本主要用来了解制作Grad_Cam的过程,并没有特意封装\n\n每行代码几乎都有注解\n\n---\n\n\nCNN卷积一直都是一个神秘的过程\n\n过去一直被以黑盒来形容\n\n能够窥探CNN是一件很有趣的事情, 特别是还能够帮助我们在进行一些任务的时候\n\n了解模型判别目标物的过程是不是出了什么问题\n\n而Grad-CAM就是一个很好的可视化选择\n\n因为能够生成高分辨率的图并叠加在原图上, 让我们在研究一些模型判别错误的时候, 有个更直观的了解\n\n那么具体是如何生成Grad CAM的呢?\n老规矩先上图, 说明流程一定要配图\n\n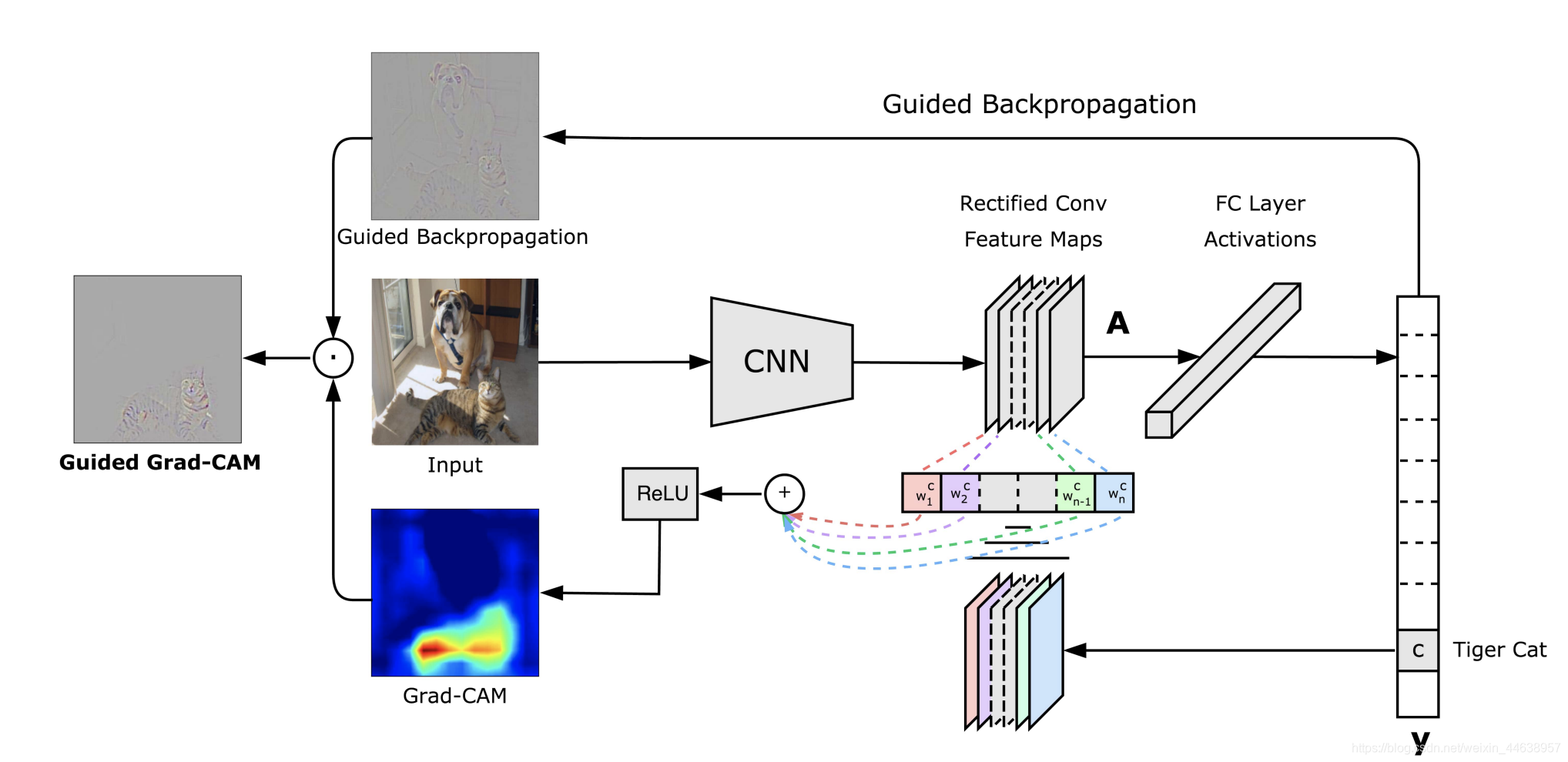\n借用一下论文作者的图\n\n我们能看到Input的地方就是我们要输入进网络进行判别的\n假设这里输入的input 类别是 Tiger cat \n\n一如往常的进入CNN提取特征\n\n现在看到图中央的Rectified Conv Feature Maps 这个层就是我们要的目标层, 而这个层通常会是整个网路特征提取的最后一层, 为什么呢? 因为通常越深层的网络越能提取到越关键的特征, 这个是不变的\n\n如果你这边可以理解了就继续往下看呗\n\n我们能看到目标层下面有一排的 $w_1^c w_2^c....$等等的\n**这里的w就是权重, 也就是我们需要的目标层512维 每一维对类别Tiger cat的重要程度**\n>这里的权重不要理解为前向传播的 每个节点的权重, 两者是不同的\n\n<br>\n而这些权重就是能够让模型判别为Tiger cat的最重要的依据\n我们要这些权重与目标层的feature map进行linear combination并提取出来\n<br>\n<br>\n那么该怎么求出这些权重呢?\n\n该是上公式的时候了, 公式由论文提出\n\n$L_{Grad-CAM}^c = ReLU(\\sum_k\\alpha^c_kA^k)$\n\n$\\alpha_k^c$ 就是我们上面说的权重\n\n怎么求?摆出论文公式\n\n$\\alpha_k^c = \\frac{1}{Z} \\sum_i \\sum_j$ $\\frac{\\partial y^c}{\\partial A^k_{ij}}$\n\n$\\frac{1}{Z} \\sum_i \\sum_j$ 表示进行全局池化,也就是求feature map的平均值\n\n$\\frac{\\partial y^c}{\\partial A^k_{ij}}$ 表示最终类别对我们要的目标层求梯度\n\n所有我们第一步先求出$A^k$ 的梯度之后, 在进行全局池化求得每一个feature map的平均梯度值就会是我们要的$\\alpha_k^c$ , 整个池化过程可以是下图红框处\n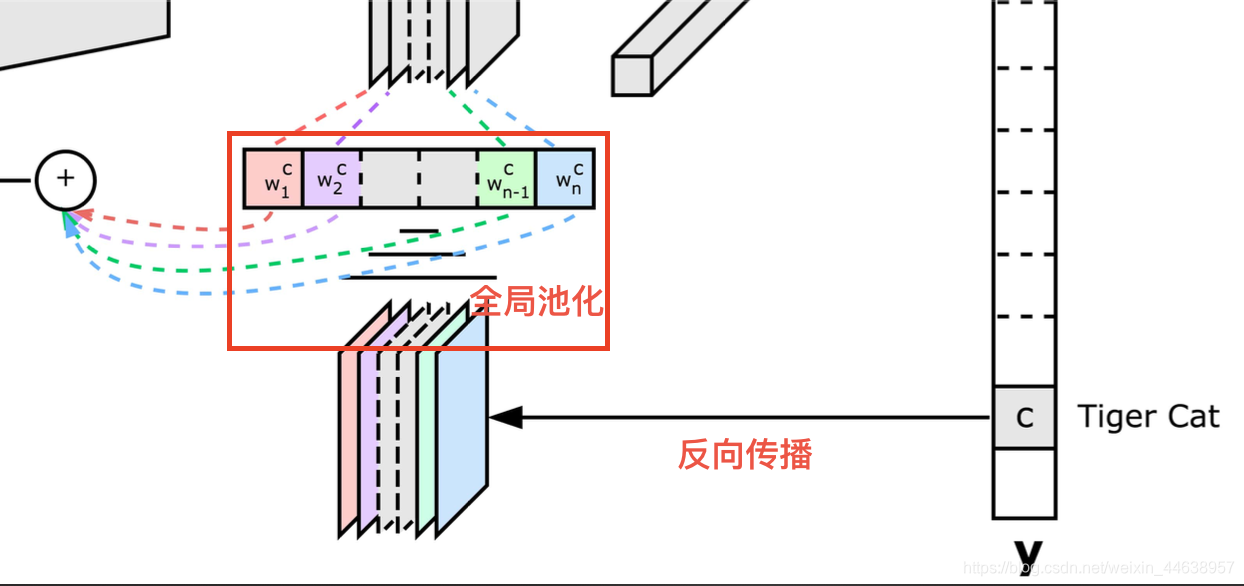\n\n这个$\\alpha_k^c$ 代表的就是 经过全局池化求出梯度平均值$w_1^c w_2^c....$\n也就是我们前面所说的**目标层的512维 每一维对类别Tiger cat的重要程度**\n\n>这边很好理解, 就是把512层feature map分别取平均值, 就取出512个均值, 这512个均值就能分别表示每层的重要程度, 有的值高就显得重要, 有的低就不重要\n\n\n\n\n\n好的 回到$L_{Grad-CAM}^c = ReLU(\\sum_k\\alpha^c_kA^k)$\n\n现在$\\alpha_k^c$ 我们有了\n\n$A^k$ 表示feature map A 然后深度是k, 如果网络是vgg16, 那么k就是512\n\n把我们求得的$\\alpha_k^c$ 与 $A^k$两者进行相乘(这里做的就是线性相乘), k如果是512, 那么将512张feature map都与权重进行相乘然后加总$\\sum_k$\n\n好,最终在经过Relu进行过滤\n我们回想一下Relu的作用是什么?\n\n是不是就是让大于0的参数原值输出,小于0的参数就直接等于0 相当于舍弃\n\n\n\n\n<img src=\"https://img-blog.csdnimg.cn/2019080623401089.png?x-oss-process=image/watermark,type_ZmFuZ3poZW5naGVpdGk,shadow_10,text_aHR0cHM6Ly9ibG9nLmNzZG4ubmV0L3dlaXhpbl80NDYzODk1Nw==,size_16,color_FFFFFF,t_70\" width = 400>\n\n其实Relu在这里扮演很重要的角色, 因为我们真正感兴趣的就是哪些能够代表Tiger Cat这个类别的特征, 而那些小于0被舍弃的就是属于其他类别的, 在这里用不上, 所有经过relu的参数能有更好的表达能力\n\n于是 我们就提取出来这个Tiger Cat类别的CAM图啦\n\n那么要注意一下, 这个提取出来的CAM 大小以vgg16来说是14*14像素\n因为经过了很多层的卷积\n\n我们要将这个图进行放大到与原输入的尺寸一样大在进行叠加才能展现GradCAM容易分析的优势\n\n当然中间有很多实现的细节包含利用openCV将色彩空间转换\n\n\n就如下图是我做的一个范例\n\n那么是不是就很容易理解网络参数对于黑猩猩面部的权重被激活的比较多,白话的说就是网络是靠猩猩脸部来判别 哦 原来这是黑猩猩啊 !\n\n当然这样的效果是在预训练网络上(vgg16 imagenet 1000类 包含黑猩猩类)才会有的, 预训练好的参数早就可以轻易的判别黑猩猩了\n如果只是单纯的丢到不是预训练的网络会是下面这样子\n\n\n<img src=\"https://img-blog.csdnimg.cn/20190806235450213.png?x-oss-process=image/watermark,type_ZmFuZ3poZW5naGVpdGk,shadow_10,text_aHR0cHM6Ly9ibG9nLmNzZG4ubmV0L3dlaXhpbl80NDYzODk1Nw==,size_16,color_FFFFFF,t_70\" width=400>\n\n\n所以网络需要进行训练, 随着训练, 权重会越能突显这个类别的特征\n最后透过某些特定的纹路就能进行判别\n\n\n<br>\n<br>\n<br>\n<br>\n<br>\n<mark>注意一下,代码中说的目标层就是我们锁定的VGG16中的第29层relu层</mark>\n\n\n\n论文连接: https://arxiv.org/pdf/1610.02391v1.pdf",
"_____no_output_____"
]
],
[
[
"import torch as t\nfrom torchvision import models\nimport cv2\nimport sys\nimport numpy as np\nimport matplotlib.pyplot as plt\n",
"_____no_output_____"
],
[
"class FeatureExtractor():\n \"\"\"\n 1. 提取目标层特征\n 2. register 目标层梯度\n \"\"\"\n def __init__(self, model, target_layers):\n self.model = model\n self.model_features = model.features\n self.target_layers = target_layers\n self.gradients = list()\n def save_gradient(self, grad):\n self.gradients.append(grad)\n def get_gradients(self):\n return self.gradients\n def __call__(self, x):\n target_activations = list()\n self.gradients = list()\n for name, module in self.model_features._modules.items(): #遍历的方式遍历网络的每一层\n x = module(x) #input 会经过遍历的每一层\n if name in self.target_layers: #设个条件,如果到了你指定的层, 则继续\n x.register_hook(self.save_gradient) #利用hook来记录目标层的梯度\n target_activations += [x] #这里只取得目标层的features\n x = x.view(x.size(0), -1) #reshape成 全连接进入分类器\n x = self.model.classifier(x)#进入分类器\n return target_activations, x,\n\n \ndef preprocess_image(img):\n \"\"\"\n 预处理层\n 将图像进行标准化处理\n \"\"\"\n mean = [0.485, 0.456, 0.406] \n stds = [0.229, 0.224, 0.225]\n preprocessed_img = img.copy()[:, :, ::-1] # BGR > RGB\n \n #标准化处理, 将bgr三层都处理\n for i in range(3):\n\n preprocessed_img[:, :, i] = preprocessed_img[:, :, i] - mean[i]\n preprocessed_img[:, :, i] = preprocessed_img[:, :, i] / stds[i]\n \n preprocessed_img = \\\n np.ascontiguousarray(np.transpose(preprocessed_img, (2, 0, 1))) #transpose HWC > CHW\n preprocessed_img = t.from_numpy(preprocessed_img) #totensor\n preprocessed_img.unsqueeze_(0)\n input = t.tensor(preprocessed_img, requires_grad=True)\n \n return input\n\n",
"_____no_output_____"
],
[
"def show_cam_on_image(img, mask):\n heatmap = cv2.applyColorMap(np.uint8(255*mask), cv2.COLORMAP_JET) #利用色彩空间转换将heatmap凸显\n heatmap = np.float32(heatmap)/255 #归一化\n cam = heatmap + np.float32(img) #将heatmap 叠加到原图\n cam = cam / np.max(cam)\n cv2.imwrite('GradCam_test.jpg', np.uint8(255 * cam))#生成图像\n \n cam = cam[:, :, ::-1] #BGR > RGB\n plt.figure(figsize=(10, 10))\n plt.imshow(np.uint8(255*cam))",
"_____no_output_____"
],
[
"class GradCam():\n \"\"\"\n GradCam主要执行\n 1.提取特征(调用FeatureExtractor)\n 2.反向传播求目标层梯度\n 3.实现目标层的CAM图\n \"\"\"\n def __init__(self, model, target_layer_names):\n self.model = model\n\n\n self.extractor = FeatureExtractor(self.model, target_layer_names)\n def forward(self, input):\n return self.model(input)\n def __call__(self, input):\n features, output = self.extractor(input) #这里的feature 对应的就是目标层的输出, output是图像经过分类网络的输出\n output.data\n one_hot = output.max() #取1000个类中最大的值\n \n self.model.features.zero_grad() #梯度清零\n self.model.classifier.zero_grad() #梯度清零\n one_hot.backward(retain_graph=True) #反向传播之后,为了取得目标层梯度\n \n grad_val = self.extractor.get_gradients()[-1].data.numpy()\n #调用函数get_gradients(), 得到目标层求得的梯\n \n target = features[-1] \n #features 目前是list 要把里面relu层的输出取出来, 也就是我们要的目标层 shape(1, 512, 14, 14)\n target = target.data.numpy()[0, :] #(1, 512, 14, 14) > (512, 14, 14) \n \n \n weights = np.mean(grad_val, axis = (2, 3))[0, :] #array shape (512, ) 求出relu梯度的 512层 每层权重\n \n cam = np.zeros(target.shape[1:]) #做一个空白map,待会将值填上\n #(14, 14) shape(512, 14, 14)tuple 索引[1:] 也就是从14开始开始\n \n #for loop的方式将平均后的权重乘上目标层的每个feature map, 并且加到刚刚生成的空白map上\n for i, w in enumerate(weights): \n cam += w * target[i, :, :] \n #w * target[i, :, :]\n #target[i, :, :] = array:shape(14, 14)\n #w = 512个的权重均值 shape(512, )\n #每个均值分别乘上target的feature map\n #在放到空白的14*14上(cam)\n #最终 14*14的空白map 会被填满\n \n cam = cv2.resize(cam, (224, 224)) #将14*14的featuremap 放大回224*224\n cam = cam - np.min(cam)\n cam = cam / np.max(cam)\n return cam",
"_____no_output_____"
],
[
"grad_cam = GradCam(model = models.vgg16(pretrained=True), \\\n target_layer_names = [\"29\"])\n\n#使用预训练vgg16\n#我们的目标层取第29层relu, relu层只保留有用的结果, 所以取其层最能突显出特征\n",
"_____no_output_____"
],
[
"\nimg = cv2.imread('./images/gorilla.jpg') #读取图像\nimg = np.float32(cv2.resize(img, (224, 224))) / 255 #为了丢到vgg16要求的224*224 先进行缩放并且归一化\ninput = preprocess_image(img)\n\nmask = grad_cam(input)\nshow_cam_on_image(img, mask)\n",
"/Library/Frameworks/Python.framework/Versions/3.7/lib/python3.7/site-packages/ipykernel_launcher.py:47: UserWarning: To copy construct from a tensor, it is recommended to use sourceTensor.clone().detach() or sourceTensor.clone().detach().requires_grad_(True), rather than torch.tensor(sourceTensor).\n"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7eac68e1ee12a5f72b5fd9912fec75971fe4743 | 7,971 | ipynb | Jupyter Notebook | nixie/nixie.ipynb | tanico-rikudo/raspi4 | ead6064ee5eaf0fb3459487047903aeb031189b7 | [
"MIT"
] | null | null | null | nixie/nixie.ipynb | tanico-rikudo/raspi4 | ead6064ee5eaf0fb3459487047903aeb031189b7 | [
"MIT"
] | null | null | null | nixie/nixie.ipynb | tanico-rikudo/raspi4 | ead6064ee5eaf0fb3459487047903aeb031189b7 | [
"MIT"
] | null | null | null | 24.754658 | 112 | 0.508217 | [
[
[
"# -*- coding: utf-8 -*-\n\nimport time\nimport RPi.GPIO as GPIO\nfrom datetime import datetime as dt\nfrom dateutil.relativedelta import relativedelta\n\nGPIO.setmode(GPIO.BCM)\n# GPIO.setmode(GPIO.BOARD)",
"_____no_output_____"
],
[
"bin_hl={'0': GPIO.LOW, '1':GPIO.HIGH}\n\nfrom enum import Enum\nclass Pin(Enum):\n A=4\n B=18\n C=24\n D=19\n d1=13\n d2=6\n\ndef gpio_setup():\n GPIO.setup(Pin.A.value, GPIO.OUT,initial=GPIO.LOW)\n GPIO.setup(Pin.B.value, GPIO.OUT,initial=GPIO.LOW)\n GPIO.setup(Pin.C.value, GPIO.OUT,initial=GPIO.LOW)\n GPIO.setup(Pin.D.value, GPIO.OUT,initial=GPIO.LOW)\n GPIO.setup(Pin.d1.value, GPIO.OUT,initial=GPIO.LOW)\n GPIO.setup(Pin.d2.value, GPIO.OUT,initial=GPIO.LOW)\n \ndef show_num_on_in14(dec_num, pin):\n assert dec_num < 10 , 'num must be <10' \n assert dec_num >= 0 , 'num must be >0'\n GPIO.output(Pin.A.value, bin_hl[bin(~dec_num>>0)[-1]])\n GPIO.output(Pin.B.value, bin_hl[bin(~dec_num>>1)[-1]])\n GPIO.output(Pin.C.value, bin_hl[bin(~dec_num>>2)[-1]])\n GPIO.output(Pin.D.value, bin_hl[bin(~dec_num>>3)[-1]])\n \ndef on(pin):\n GPIO.output(pin.value, GPIO.HIGH)\n\ndef off(pin):\n GPIO.output(pin.value, GPIO.LOW)\n\n\ndef show_pins(pin_assign, seconds=5, milliseconds=0):\n st = dt.now()\n expect_ed = st + relativedelta(seconds=seconds, microseconds=milliseconds*1000)\n while expect_ed > dt.now():\n for assgin_data in pin_assign:\n on(assgin_data[\"pin\"])\n show_num_on_in14(assgin_data[\"val\"],Pin)\n time.sleep(0.008)\n off(assgin_data[\"pin\"])\n time.sleep(0.002)\n \ndef check_all_decimal_num(Pin):\n for j in range(10000):\n for i in range(10):\n show_num_on_in14(i, Pin)\n time.sleep(0.1)\n \ndef clock():\n while True:\n sec = str(dt.now().second).zfill(2)\n pin_assign = [\n {\"pin\" : Pin.d1, \"val\" : int(sec[1])},\n { \"pin\" : Pin.d2, \"val\" :int(sec[0])}]\n show_pins(pin_assign, seconds=0, milliseconds=500)\n",
"_____no_output_____"
],
[
"gpio_setup()",
"_____no_output_____"
],
[
"on(Pin.d1)\non(Pin.d2)",
"_____no_output_____"
],
[
"show_num_on_in14(2, Pin)",
"_____no_output_____"
],
[
"pin_assign = [\n {\"pin\" : Pin.d1, \"val\" : 1},\n { \"pin\" : Pin.d2, \"val\" : 5}]\n",
"_____no_output_____"
],
[
"check_all_decimal_num(Pin)",
"_____no_output_____"
],
[
"show_pins(pin_assign, seconds=5, milliseconds=0)",
"_____no_output_____"
]
],
[
[
"### #1 ~ #9 (NO transistor)",
"_____no_output_____"
]
],
[
[
"GPIO.output(Pin.A.value, GPIO.LOW)\nGPIO.output(Pin.B.value, GPIO.LOW)\nGPIO.output(Pin.C.value,GPIO.LOW)\nGPIO.output(Pin.D.value, GPIO.LOW)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.HIGH)\nGPIO.output(Pin.B.value,GPIO.LOW)\nGPIO.output(Pin.C.value,GPIO.LOW)\nGPIO.output(Pin.D.value, GPIO.LOW)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.LOW)\nGPIO.output(Pin.B.value,GPIO.HIGH)\nGPIO.output(Pin.C.value,GPIO.LOW)\nGPIO.output(Pin.D.value, GPIO.LOW)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.HIGH)\nGPIO.output(Pin.B.value,GPIO.HIGH)\nGPIO.output(Pin.C.value,GPIO.LOW)\nGPIO.output(Pin.D.value, GPIO.LOW)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.LOW)\nGPIO.output(Pin.B.value,GPIO.LOW)\nGPIO.output(Pin.C.value,GPIO.HIGH)\nGPIO.output(Pin.D.value, GPIO.LOW)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.HIGH)\nGPIO.output(Pin.B.value,GPIO.LOW)\nGPIO.output(Pin.C.value,GPIO.HIGH)\nGPIO.output(Pin.D.value, GPIO.LOW)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.LOW)\nGPIO.output(Pin.B.value,GPIO.HIGH)\nGPIO.output(Pin.C.value,GPIO.HIGH)\nGPIO.output(Pin.D.value, GPIO.LOW)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.HIGH)\nGPIO.output(Pin.B.value,GPIO.HIGH)\nGPIO.output(Pin.C.value,GPIO.HIGH)\nGPIO.output(Pin.D.value, GPIO.LOW)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.LOW)\nGPIO.output(Pin.B.value,GPIO.LOW)\nGPIO.output(Pin.C.value,GPIO.LOW)\nGPIO.output(Pin.D.value, GPIO.HIGH)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.HIGH)\nGPIO.output(Pin.B.value,GPIO.LOW)\nGPIO.output(Pin.C.value,GPIO.LOW)\nGPIO.output(Pin.D.value, GPIO.HIGH)",
"_____no_output_____"
],
[
"GPIO.output(Pin.A.value,GPIO.HIGH)\nGPIO.output(Pin.B.value,GPIO.HIGH)\nGPIO.output(Pin.C.value,GPIO.HIGH)\nGPIO.output(Pin.D.value, GPIO.HIGH)",
"_____no_output_____"
],
[
" \nGPIO.cleanup()",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ead1fa57f788871a7bb57c458f826082c485c3 | 49,726 | ipynb | Jupyter Notebook | employee_bd.ipynb | jchamberlain2020/SQL_Challenge | 726bcba3b92e6dc68870eb6fb4e2839b64466569 | [
"ADSL"
] | null | null | null | employee_bd.ipynb | jchamberlain2020/SQL_Challenge | 726bcba3b92e6dc68870eb6fb4e2839b64466569 | [
"ADSL"
] | null | null | null | employee_bd.ipynb | jchamberlain2020/SQL_Challenge | 726bcba3b92e6dc68870eb6fb4e2839b64466569 | [
"ADSL"
] | null | null | null | 41.892165 | 15,120 | 0.550919 | [
[
[
"#Import dependencies\nimport pandas as pd\nimport matplotlib.pyplot as plt\n%matplotlib inline\nimport numpy as np\nimport matplotlib.mlab as mlab",
"_____no_output_____"
],
[
"from sqlalchemy import create_engine\nengine = create_engine (f'postgresql://postgres:postgres@localhost:5432/employees')\nconnection = engine.connect()",
"_____no_output_____"
],
[
"#Import the Employee Table\nemployees = pd.read_sql('select * from employees', connection, parse_dates=['birth_date', 'hire_date'])\nemployees.head()",
"_____no_output_____"
],
[
"#Import department table\ndepartment = pd.read_sql('select * from departments', connection)\ndepartment.head()",
"_____no_output_____"
],
[
"#Import salaries table\nsalaries = pd.read_sql('select * from salaries', connection, parse_dates=['from_date', 'to_date'])\nsalaries.head()",
"_____no_output_____"
],
[
"#Import department manager table\ndept_manager = pd.read_sql('select * from dept_manager', connection, parse_dates=['from_date', 'to_date'])\ndept_manager",
"_____no_output_____"
],
[
"#Replace null dates\ndept_manager.to_date = dept_manager['to_date'].fillna(pd.to_datetime('2050-12-31'))\ndept_manager.head()",
"_____no_output_____"
],
[
"#Import titles table\ntitles = pd.read_sql('select * from titles', connection, parse_dates=['from_date', 'to_date'])\ntitles.head()",
"_____no_output_____"
],
[
"#Replace null values\ntitles.to_date = titles['to_date'].fillna(pd.to_datetime('2050-12-31'))\ntitles.head()",
"_____no_output_____"
],
[
"#Count by title\ntitles.title.value_counts()",
"_____no_output_____"
],
[
"#Merge dataframe of titles and salaries\nemployee_salaries = titles.merge(salaries, on=\"emp_no\")\nemployee_salaries.head()",
"_____no_output_____"
],
[
"#Show columns employee number, title, and salary - drop other columns\nemployee_salaries_df = employee_salaries[['emp_no', 'title', 'salary']]\nemployee_salaries_df",
"_____no_output_____"
],
[
"#Average of salary per title\nemployee_salaries_df.groupby('title')['salary'].mean().round(2)",
"_____no_output_____"
],
[
"employees_grouped_by_title = employee_salaries_df.groupby(['title'])['salary'].mean()\nemployees_grouped_by_title.plot.bar()\nplt.xlabel(\"Job Title\")\nplt.ylabel(\"Average Salary\")",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7eae0de9e8e677aa659809ade25ab756c4e82c0 | 18,276 | ipynb | Jupyter Notebook | docs/_downloads/f11c58c36c9b8a5daf09d3f9a792ef84/memory_format_tutorial.ipynb | creduo/PyTorch-tutorials-kr | 75c20a4af6af77267c19fb2107728b4bed93311a | [
"BSD-3-Clause"
] | 1 | 2021-07-08T12:37:20.000Z | 2021-07-08T12:37:20.000Z | docs/_downloads/f11c58c36c9b8a5daf09d3f9a792ef84/memory_format_tutorial.ipynb | creduo/PyTorch-tutorials-kr | 75c20a4af6af77267c19fb2107728b4bed93311a | [
"BSD-3-Clause"
] | null | null | null | docs/_downloads/f11c58c36c9b8a5daf09d3f9a792ef84/memory_format_tutorial.ipynb | creduo/PyTorch-tutorials-kr | 75c20a4af6af77267c19fb2107728b4bed93311a | [
"BSD-3-Clause"
] | null | null | null | 50.908078 | 2,321 | 0.571405 | [
[
[
"%matplotlib inline",
"_____no_output_____"
]
],
[
[
"\n(experimental) Channels Last Memory Format in PyTorch\n*******************************************************\n**Author**: `Vitaly Fedyunin <https://github.com/VitalyFedyunin>`_\n\nWhat is Channels Last\n---------------------\n\nChannels Last memory format is an alternative way of ordering NCHW tensors in memory preserving dimensions ordering. Channels Last tensors ordered in such a way that channels become the densest dimension (aka storing images pixel-per-pixel).\n\nFor example, classic (contiguous) storage of NCHW tensor (in our case it is two 2x2 images with 3 color channels) look like this:\n\n.. figure:: /_static/img/classic_memory_format.png \n :alt: classic_memory_format\n\nChannels Last memory format orders data differently:\n\n.. figure:: /_static/img/channels_last_memory_format.png \n :alt: channels_last_memory_format\n\nPytorch supports memory formats (and provides back compatibility with existing models including eager, JIT, and TorchScript) by utilizing existing strides structure.\nFor example, 10x3x16x16 batch in Channels Last format will have strides equal to (768, 1, 48, 3).\n\n",
"_____no_output_____"
],
[
"Channels Last memory format is implemented for 4D NCWH Tensors only.\n\n\n",
"_____no_output_____"
]
],
[
[
"import torch\nN, C, H, W = 10, 3, 32, 32",
"_____no_output_____"
]
],
[
[
"Memory Format API\n-----------------------\n\nHere is how to convert tensors between contiguous and channels \nlast memory formats.\n\n",
"_____no_output_____"
],
[
"Classic PyTorch contiguous tensor\n\n",
"_____no_output_____"
]
],
[
[
"x = torch.empty(N, C, H, W)\nprint(x.stride()) # Ouputs: (3072, 1024, 32, 1)",
"_____no_output_____"
]
],
[
[
"Conversion operator\n\n",
"_____no_output_____"
]
],
[
[
"x = x.contiguous(memory_format=torch.channels_last)\nprint(x.shape) # Outputs: (10, 3, 32, 32) as dimensions order preserved\nprint(x.stride()) # Outputs: (3072, 1, 96, 3)",
"_____no_output_____"
]
],
[
[
"Back to contiguous\n\n",
"_____no_output_____"
]
],
[
[
"x = x.contiguous(memory_format=torch.contiguous_format)\nprint(x.stride()) # Outputs: (3072, 1024, 32, 1)",
"_____no_output_____"
]
],
[
[
"Alternative option\n\n",
"_____no_output_____"
]
],
[
[
"x = x.to(memory_format=torch.channels_last)\nprint(x.stride()) # Ouputs: (3072, 1, 96, 3)",
"_____no_output_____"
]
],
[
[
"Format checks\n\n",
"_____no_output_____"
]
],
[
[
"print(x.is_contiguous(memory_format=torch.channels_last)) # Ouputs: True",
"_____no_output_____"
]
],
[
[
"Create as Channels Last\n\n",
"_____no_output_____"
]
],
[
[
"x = torch.empty(N, C, H, W, memory_format=torch.channels_last)\nprint(x.stride()) # Ouputs: (3072, 1, 96, 3)",
"_____no_output_____"
]
],
[
[
"``clone`` preserves memory format\n\n",
"_____no_output_____"
]
],
[
[
"y = x.clone()\nprint(y.stride()) # Ouputs: (3072, 1, 96, 3)",
"_____no_output_____"
]
],
[
[
"``to``, ``cuda``, ``float`` ... preserves memory format\n\n",
"_____no_output_____"
]
],
[
[
"if torch.cuda.is_available():\n y = x.cuda()\n print(y.stride()) # Ouputs: (3072, 1, 96, 3)",
"_____no_output_____"
]
],
[
[
"``empty_like``, ``*_like`` operators preserves memory format\n\n",
"_____no_output_____"
]
],
[
[
"y = torch.empty_like(x)\nprint(y.stride()) # Ouputs: (3072, 1, 96, 3)",
"_____no_output_____"
]
],
[
[
"Pointwise operators preserves memory format\n\n",
"_____no_output_____"
]
],
[
[
"z = x + y\nprint(z.stride()) # Ouputs: (3072, 1, 96, 3)",
"_____no_output_____"
]
],
[
[
"Conv, Batchnorm modules support Channels Last\n(only works for CudNN >= 7.6)\n\n",
"_____no_output_____"
]
],
[
[
"if torch.backends.cudnn.version() >= 7603:\n input = torch.randint(1, 10, (2, 8, 4, 4), dtype=torch.float32, device=\"cuda\", requires_grad=True)\n model = torch.nn.Conv2d(8, 4, 3).cuda().float()\n\n input = input.contiguous(memory_format=torch.channels_last)\n model = model.to(memory_format=torch.channels_last) # Module parameters need to be Channels Last\n\n out = model(input)\n print(out.is_contiguous(memory_format=torch.channels_last)) # Ouputs: True",
"_____no_output_____"
]
],
[
[
"Performance Gains\n-------------------------------------------------------------------------------------------\nThe most significant performance gains are observed on NVidia's hardware with \nTensor Cores support. We were able to archive over 22% perf gains while running '\nAMP (Automated Mixed Precision) training scripts supplied by NVidia https://github.com/NVIDIA/apex.\n\n``python main_amp.py -a resnet50 --b 200 --workers 16 --opt-level O2 ./data``\n\n",
"_____no_output_____"
]
],
[
[
"# opt_level = O2\n# keep_batchnorm_fp32 = None <class 'NoneType'>\n# loss_scale = None <class 'NoneType'>\n# CUDNN VERSION: 7603\n# => creating model 'resnet50'\n# Selected optimization level O2: FP16 training with FP32 batchnorm and FP32 master weights.\n# Defaults for this optimization level are:\n# enabled : True\n# opt_level : O2\n# cast_model_type : torch.float16\n# patch_torch_functions : False\n# keep_batchnorm_fp32 : True\n# master_weights : True\n# loss_scale : dynamic\n# Processing user overrides (additional kwargs that are not None)...\n# After processing overrides, optimization options are:\n# enabled : True\n# opt_level : O2\n# cast_model_type : torch.float16\n# patch_torch_functions : False\n# keep_batchnorm_fp32 : True\n# master_weights : True\n# loss_scale : dynamic\n# Epoch: [0][10/125] Time 0.866 (0.866) Speed 230.949 (230.949) Loss 0.6735125184 (0.6735) Prec@1 61.000 (61.000) Prec@5 100.000 (100.000)\n# Epoch: [0][20/125] Time 0.259 (0.562) Speed 773.481 (355.693) Loss 0.6968704462 (0.6852) Prec@1 55.000 (58.000) Prec@5 100.000 (100.000)\n# Epoch: [0][30/125] Time 0.258 (0.461) Speed 775.089 (433.965) Loss 0.7877287269 (0.7194) Prec@1 51.500 (55.833) Prec@5 100.000 (100.000)\n# Epoch: [0][40/125] Time 0.259 (0.410) Speed 771.710 (487.281) Loss 0.8285319805 (0.7467) Prec@1 48.500 (54.000) Prec@5 100.000 (100.000)\n# Epoch: [0][50/125] Time 0.260 (0.380) Speed 770.090 (525.908) Loss 0.7370464802 (0.7447) Prec@1 56.500 (54.500) Prec@5 100.000 (100.000)\n# Epoch: [0][60/125] Time 0.258 (0.360) Speed 775.623 (555.728) Loss 0.7592862844 (0.7472) Prec@1 51.000 (53.917) Prec@5 100.000 (100.000)\n# Epoch: [0][70/125] Time 0.258 (0.345) Speed 774.746 (579.115) Loss 1.9698858261 (0.9218) Prec@1 49.500 (53.286) Prec@5 100.000 (100.000)\n# Epoch: [0][80/125] Time 0.260 (0.335) Speed 770.324 (597.659) Loss 2.2505953312 (1.0879) Prec@1 50.500 (52.938) Prec@5 100.000 (100.000)",
"_____no_output_____"
]
],
[
[
"Passing ``--channels-last true`` allows running a model in Channels Last format with observed 22% perf gain.\n\n``python main_amp.py -a resnet50 --b 200 --workers 16 --opt-level O2 --channels-last true ./data``\n\n",
"_____no_output_____"
]
],
[
[
"# opt_level = O2\n# keep_batchnorm_fp32 = None <class 'NoneType'>\n# loss_scale = None <class 'NoneType'>\n#\n# CUDNN VERSION: 7603\n#\n# => creating model 'resnet50'\n# Selected optimization level O2: FP16 training with FP32 batchnorm and FP32 master weights.\n#\n# Defaults for this optimization level are:\n# enabled : True\n# opt_level : O2\n# cast_model_type : torch.float16\n# patch_torch_functions : False\n# keep_batchnorm_fp32 : True\n# master_weights : True\n# loss_scale : dynamic\n# Processing user overrides (additional kwargs that are not None)...\n# After processing overrides, optimization options are:\n# enabled : True\n# opt_level : O2\n# cast_model_type : torch.float16\n# patch_torch_functions : False\n# keep_batchnorm_fp32 : True\n# master_weights : True\n# loss_scale : dynamic\n#\n# Epoch: [0][10/125] Time 0.767 (0.767) Speed 260.785 (260.785) Loss 0.7579724789 (0.7580) Prec@1 53.500 (53.500) Prec@5 100.000 (100.000)\n# Epoch: [0][20/125] Time 0.198 (0.482) Speed 1012.135 (414.716) Loss 0.7007197738 (0.7293) Prec@1 49.000 (51.250) Prec@5 100.000 (100.000)\n# Epoch: [0][30/125] Time 0.198 (0.387) Speed 1010.977 (516.198) Loss 0.7113101482 (0.7233) Prec@1 55.500 (52.667) Prec@5 100.000 (100.000)\n# Epoch: [0][40/125] Time 0.197 (0.340) Speed 1013.023 (588.333) Loss 0.8943189979 (0.7661) Prec@1 54.000 (53.000) Prec@5 100.000 (100.000)\n# Epoch: [0][50/125] Time 0.198 (0.312) Speed 1010.541 (641.977) Loss 1.7113249302 (0.9551) Prec@1 51.000 (52.600) Prec@5 100.000 (100.000)\n# Epoch: [0][60/125] Time 0.198 (0.293) Speed 1011.163 (683.574) Loss 5.8537774086 (1.7716) Prec@1 50.500 (52.250) Prec@5 100.000 (100.000)\n# Epoch: [0][70/125] Time 0.198 (0.279) Speed 1011.453 (716.767) Loss 5.7595844269 (2.3413) Prec@1 46.500 (51.429) Prec@5 100.000 (100.000)\n# Epoch: [0][80/125] Time 0.198 (0.269) Speed 1011.827 (743.883) Loss 2.8196096420 (2.4011) Prec@1 47.500 (50.938) Prec@5 100.000 (100.000)",
"_____no_output_____"
]
],
[
[
"The following list of models has the full support of Channels Last and showing 8%-35% perf gains on Volta devices:\n``alexnet``, ``mnasnet0_5``, ``mnasnet0_75``, ``mnasnet1_0``, ``mnasnet1_3``, ``mobilenet_v2``, ``resnet101``, ``resnet152``, ``resnet18``, ``resnet34``, ``resnet50``, ``resnext50_32x4d``, ``shufflenet_v2_x0_5``, ``shufflenet_v2_x1_0``, ``shufflenet_v2_x1_5``, ``shufflenet_v2_x2_0``, ``squeezenet1_0``, ``squeezenet1_1``, ``vgg11``, ``vgg11_bn``, ``vgg13``, ``vgg13_bn``, ``vgg16``, ``vgg16_bn``, ``vgg19``, ``vgg19_bn``, ``wide_resnet101_2``, ``wide_resnet50_2``\n\n\n",
"_____no_output_____"
],
[
"Converting existing models\n--------------------------\n\nChannels Last support not limited by existing models, as any model can be converted to Channels Last and propagate format through the graph as soon as input formatted correctly. \n\n\n",
"_____no_output_____"
]
],
[
[
"# Need to be done once, after model initialization (or load)\nmodel = model.to(memory_format=torch.channels_last) # Replace with your model\n\n# Need to be done for every input\ninput = input.to(memory_format=torch.channels_last) # Replace with your input\noutput = model(input)",
"_____no_output_____"
]
],
[
[
"However, not all operators fully converted to support Channels Last (usually returning \ncontiguous output instead). That means you need to verify the list of used operators \nagainst supported operators list https://github.com/pytorch/pytorch/wiki/Operators-with-Channels-Last-support, \nor introduce memory format checks into eager execution mode and run your model.\n\nAfter running the code below, operators will raise an exception if the output of the \noperator doesn't match the memory format of the input.\n\n\n\n",
"_____no_output_____"
]
],
[
[
"def contains_cl(args):\n for t in args:\n if isinstance(t, torch.Tensor):\n if t.is_contiguous(memory_format=torch.channels_last) and not t.is_contiguous():\n return True\n elif isinstance(t, list) or isinstance(t, tuple):\n if contains_cl(list(t)):\n return True\n return False\n\n\ndef print_inputs(args, indent=''):\n for t in args:\n if isinstance(t, torch.Tensor):\n print(indent, t.stride(), t.shape, t.device, t.dtype)\n elif isinstance(t, list) or isinstance(t, tuple):\n print(indent, type(t))\n print_inputs(list(t), indent=indent + ' ')\n else:\n print(indent, t)\n\n\ndef check_wrapper(fn):\n name = fn.__name__\n\n def check_cl(*args, **kwargs):\n was_cl = contains_cl(args)\n try:\n result = fn(*args, **kwargs)\n except Exception as e:\n print(\"`{}` inputs are:\".format(name))\n print_inputs(args)\n print('-------------------')\n raise e\n failed = False\n if was_cl:\n if isinstance(result, torch.Tensor):\n if result.dim() == 4 and not result.is_contiguous(memory_format=torch.channels_last):\n print(\"`{}` got channels_last input, but output is not channels_last:\".format(name),\n result.shape, result.stride(), result.device, result.dtype)\n failed = True\n if failed and True:\n print(\"`{}` inputs are:\".format(name))\n print_inputs(args)\n raise Exception(\n 'Operator `{}` lost channels_last property'.format(name))\n return result\n return check_cl\n\n\ndef attribute(m):\n for i in dir(m):\n e = getattr(m, i)\n exclude_functions = ['is_cuda', 'has_names', 'numel',\n 'stride', 'Tensor', 'is_contiguous', '__class__']\n if i not in exclude_functions and not i.startswith('_') and '__call__' in dir(e):\n try:\n setattr(m, i, check_wrapper(e))\n except Exception as e:\n print(i)\n print(e)\n\n\nattribute(torch.Tensor)\nattribute(torch.nn.functional)\nattribute(torch)",
"_____no_output_____"
]
],
[
[
"If you found an operator that doesn't support Channels Last tensors\nand you want to contribute, feel free to use following developers \nguide https://github.com/pytorch/pytorch/wiki/Writing-memory-format-aware-operators.\n\n\n",
"_____no_output_____"
],
[
"Work to do\n----------\nThere are still many things to do, such as:\n\n- Resolving ambiguity of N1HW and NC11 Tensors;\n- Testing of Distributed Training support;\n- Improving operators coverage.\n\nIf you have feedback and/or suggestions for improvement, please let us\nknow by creating `an issue <https://github.com/pytorch/pytorch/issues>`_.\n\n",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
]
] |
e7eafe2b26168062c37288e396d6c5c24187505c | 3,762 | ipynb | Jupyter Notebook | tutorials/W0D0_NeuroVideoSeries/student/W0D0_Tutorial4.ipynb | janeite/course-content | 2a3ba168c5bfb5fd5e8305fe3ae79465b0add52c | [
"CC-BY-4.0",
"BSD-3-Clause"
] | 2,294 | 2020-05-11T12:05:35.000Z | 2022-03-28T21:23:34.000Z | tutorials/W0D0_NeuroVideoSeries/student/W0D0_Tutorial4.ipynb | janeite/course-content | 2a3ba168c5bfb5fd5e8305fe3ae79465b0add52c | [
"CC-BY-4.0",
"BSD-3-Clause"
] | 629 | 2020-05-11T15:42:26.000Z | 2022-03-29T12:23:35.000Z | tutorials/W0D0_NeuroVideoSeries/student/W0D0_Tutorial4.ipynb | janeite/course-content | 2a3ba168c5bfb5fd5e8305fe3ae79465b0add52c | [
"CC-BY-4.0",
"BSD-3-Clause"
] | 917 | 2020-05-11T12:47:53.000Z | 2022-03-31T12:14:41.000Z | 27.661765 | 281 | 0.565657 | [
[
[
"<a href=\"https://colab.research.google.com/github/NeuromatchAcademy/course-content/blob/master/tutorials/W0D0_NeuroVideoSeries/student/W0D0_Tutorial4.ipynb\" target=\"_parent\"><img src=\"https://colab.research.google.com/assets/colab-badge.svg\" alt=\"Open In Colab\"/></a>",
"_____no_output_____"
],
[
"# Live in Lab\n**Neuro Video Series**\n\n**By Neuromatch Academy**\n\n**Content creators**: Anne Churchland, Chaoqun Yin, Alex Kostiuk, Lukas Oesch,Michael Ryan, Ashley Chen, Joao Couto\n\n**Content reviewers**: Tara van Viegen,\tOluwatomisin Faniyan, Pooya Pakarian, Sirisha Sripada\n\n**Video editors, captioners, and translators**: Jeremy Forest, Tara van Viegen, Carolina Shimabuku, Shuze Liu",
"_____no_output_____"
],
[
"**Our 2021 Sponsors, including Presenting Sponsor Facebook Reality Labs**\n\n<p align='center'><img src='https://github.com/NeuromatchAcademy/widgets/blob/master/sponsors.png?raw=True'/></p>",
"_____no_output_____"
],
[
"## Video",
"_____no_output_____"
]
],
[
[
"# @markdown\nfrom ipywidgets import widgets\n\nout2 = widgets.Output()\nwith out2:\n from IPython.display import IFrame\n class BiliVideo(IFrame):\n def __init__(self, id, page=1, width=400, height=300, **kwargs):\n self.id=id\n src = \"https://player.bilibili.com/player.html?bvid={0}&page={1}\".format(id, page)\n super(BiliVideo, self).__init__(src, width, height, **kwargs)\n\n video = BiliVideo(id=\"BV1r5411T7pP\", width=854, height=480, fs=1)\n print(\"Video available at https://www.bilibili.com/video/{0}\".format(video.id))\n display(video)\n\nout1 = widgets.Output()\nwith out1:\n from IPython.display import YouTubeVideo\n video = YouTubeVideo(id=\"HCx-sOp9R7M\", width=854, height=480, fs=1, rel=0)\n print(\"Video available at https://youtube.com/watch?v=\" + video.id)\n display(video)\n\nout = widgets.Tab([out1, out2])\nout.set_title(0, 'Youtube')\nout.set_title(1, 'Bilibili')\n\ndisplay(out)",
"_____no_output_____"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code"
]
] |
e7eb0381e18b5b38e6e73f0847aed6778e026a25 | 462,451 | ipynb | Jupyter Notebook | 02_end_to_end_machine_learning_project.ipynb | Ruqyai/handson-ml2 | a38def95c88bfa884f5e7a370b363ae8ce93b37e | [
"Apache-2.0"
] | 1 | 2020-10-21T09:55:15.000Z | 2020-10-21T09:55:15.000Z | 02_end_to_end_machine_learning_project.ipynb | Ruqyai/handson-ml2 | a38def95c88bfa884f5e7a370b363ae8ce93b37e | [
"Apache-2.0"
] | null | null | null | 02_end_to_end_machine_learning_project.ipynb | Ruqyai/handson-ml2 | a38def95c88bfa884f5e7a370b363ae8ce93b37e | [
"Apache-2.0"
] | null | null | null | 119.65097 | 155,712 | 0.828014 | [
[
[
"**Chapter 2 – End-to-end Machine Learning project**\n\n*Welcome to Machine Learning Housing Corp.! Your task is to predict median house values in Californian districts, given a number of features from these districts.*\n\n*This notebook contains all the sample code and solutions to the exercices in chapter 2.*",
"_____no_output_____"
],
[
"# Setup",
"_____no_output_____"
],
[
"First, let's import a few common modules, ensure MatplotLib plots figures inline and prepare a function to save the figures. We also check that Python 3.5 or later is installed (although Python 2.x may work, it is deprecated so we strongly recommend you use Python 3 instead), as well as Scikit-Learn ≥0.20.",
"_____no_output_____"
]
],
[
[
"# Python ≥3.5 is required\nimport sys\nassert sys.version_info >= (3, 5)\n\n# Scikit-Learn ≥0.20 is required\nimport sklearn\nassert sklearn.__version__ >= \"0.20\"\n\n# Common imports\nimport numpy as np\nimport os\n\n# To plot pretty figures\n%matplotlib inline\nimport matplotlib as mpl\nimport matplotlib.pyplot as plt\nmpl.rc('axes', labelsize=14)\nmpl.rc('xtick', labelsize=12)\nmpl.rc('ytick', labelsize=12)\n\n# Where to save the figures\nPROJECT_ROOT_DIR = \".\"\nCHAPTER_ID = \"end_to_end_project\"\nIMAGES_PATH = os.path.join(PROJECT_ROOT_DIR, \"images\", CHAPTER_ID)\nos.makedirs(IMAGES_PATH, exist_ok=True)\n\ndef save_fig(fig_id, tight_layout=True, fig_extension=\"png\", resolution=300):\n path = os.path.join(IMAGES_PATH, fig_id + \".\" + fig_extension)\n print(\"Saving figure\", fig_id)\n if tight_layout:\n plt.tight_layout()\n plt.savefig(path, format=fig_extension, dpi=resolution)\n\n# Ignore useless warnings (see SciPy issue #5998)\nimport warnings\nwarnings.filterwarnings(action=\"ignore\", message=\"^internal gelsd\")",
"_____no_output_____"
]
],
[
[
"# Get the data",
"_____no_output_____"
]
],
[
[
"import os\nimport tarfile\nimport urllib\n\nDOWNLOAD_ROOT = \"https://raw.githubusercontent.com/ageron/handson-ml2/master/\"\nHOUSING_PATH = os.path.join(\"datasets\", \"housing\")\nHOUSING_URL = DOWNLOAD_ROOT + \"datasets/housing/housing.tgz\"\n\ndef fetch_housing_data(housing_url=HOUSING_URL, housing_path=HOUSING_PATH):\n if not os.path.isdir(housing_path):\n os.makedirs(housing_path)\n tgz_path = os.path.join(housing_path, \"housing.tgz\")\n urllib.request.urlretrieve(housing_url, tgz_path)\n housing_tgz = tarfile.open(tgz_path)\n housing_tgz.extractall(path=housing_path)\n housing_tgz.close()",
"_____no_output_____"
],
[
"fetch_housing_data()",
"_____no_output_____"
],
[
"import pandas as pd\n\ndef load_housing_data(housing_path=HOUSING_PATH):\n csv_path = os.path.join(housing_path, \"housing.csv\")\n return pd.read_csv(csv_path)",
"_____no_output_____"
],
[
"housing = load_housing_data()\nhousing.head()",
"_____no_output_____"
],
[
"housing.info()",
"<class 'pandas.core.frame.DataFrame'>\nRangeIndex: 20640 entries, 0 to 20639\nData columns (total 10 columns):\nlongitude 20640 non-null float64\nlatitude 20640 non-null float64\nhousing_median_age 20640 non-null float64\ntotal_rooms 20640 non-null float64\ntotal_bedrooms 20433 non-null float64\npopulation 20640 non-null float64\nhouseholds 20640 non-null float64\nmedian_income 20640 non-null float64\nmedian_house_value 20640 non-null float64\nocean_proximity 20640 non-null object\ndtypes: float64(9), object(1)\nmemory usage: 1.6+ MB\n"
],
[
"housing[\"ocean_proximity\"].value_counts()",
"_____no_output_____"
],
[
"housing.describe()",
"_____no_output_____"
],
[
"%matplotlib inline\nimport matplotlib.pyplot as plt\nhousing.hist(bins=50, figsize=(20,15))\nsave_fig(\"attribute_histogram_plots\")\nplt.show()",
"Saving figure attribute_histogram_plots\n"
],
[
"# to make this notebook's output identical at every run\nnp.random.seed(42)",
"_____no_output_____"
],
[
"import numpy as np\n\n# For illustration only. Sklearn has train_test_split()\ndef split_train_test(data, test_ratio):\n shuffled_indices = np.random.permutation(len(data))\n test_set_size = int(len(data) * test_ratio)\n test_indices = shuffled_indices[:test_set_size]\n train_indices = shuffled_indices[test_set_size:]\n return data.iloc[train_indices], data.iloc[test_indices]",
"_____no_output_____"
],
[
"train_set, test_set = split_train_test(housing, 0.2)\nlen(train_set)",
"_____no_output_____"
],
[
"len(test_set)",
"_____no_output_____"
],
[
"from zlib import crc32\n\ndef test_set_check(identifier, test_ratio):\n return crc32(np.int64(identifier)) & 0xffffffff < test_ratio * 2**32\n\ndef split_train_test_by_id(data, test_ratio, id_column):\n ids = data[id_column]\n in_test_set = ids.apply(lambda id_: test_set_check(id_, test_ratio))\n return data.loc[~in_test_set], data.loc[in_test_set]",
"_____no_output_____"
]
],
[
[
"The implementation of `test_set_check()` above works fine in both Python 2 and Python 3. In earlier releases, the following implementation was proposed, which supported any hash function, but was much slower and did not support Python 2:",
"_____no_output_____"
]
],
[
[
"import hashlib\n\ndef test_set_check(identifier, test_ratio, hash=hashlib.md5):\n return hash(np.int64(identifier)).digest()[-1] < 256 * test_ratio",
"_____no_output_____"
]
],
[
[
"If you want an implementation that supports any hash function and is compatible with both Python 2 and Python 3, here is one:",
"_____no_output_____"
]
],
[
[
"def test_set_check(identifier, test_ratio, hash=hashlib.md5):\n return bytearray(hash(np.int64(identifier)).digest())[-1] < 256 * test_ratio",
"_____no_output_____"
],
[
"housing_with_id = housing.reset_index() # adds an `index` column\ntrain_set, test_set = split_train_test_by_id(housing_with_id, 0.2, \"index\")",
"_____no_output_____"
],
[
"housing_with_id[\"id\"] = housing[\"longitude\"] * 1000 + housing[\"latitude\"]\ntrain_set, test_set = split_train_test_by_id(housing_with_id, 0.2, \"id\")",
"_____no_output_____"
],
[
"test_set.head()",
"_____no_output_____"
],
[
"from sklearn.model_selection import train_test_split\n\ntrain_set, test_set = train_test_split(housing, test_size=0.2, random_state=42)",
"_____no_output_____"
],
[
"test_set.head()",
"_____no_output_____"
],
[
"housing[\"median_income\"].hist()",
"_____no_output_____"
],
[
"housing[\"income_cat\"] = pd.cut(housing[\"median_income\"],\n bins=[0., 1.5, 3.0, 4.5, 6., np.inf],\n labels=[1, 2, 3, 4, 5])",
"_____no_output_____"
],
[
"housing[\"income_cat\"].value_counts()",
"_____no_output_____"
],
[
"housing[\"income_cat\"].hist()",
"_____no_output_____"
],
[
"from sklearn.model_selection import StratifiedShuffleSplit\n\nsplit = StratifiedShuffleSplit(n_splits=1, test_size=0.2, random_state=42)\nfor train_index, test_index in split.split(housing, housing[\"income_cat\"]):\n strat_train_set = housing.loc[train_index]\n strat_test_set = housing.loc[test_index]",
"_____no_output_____"
],
[
"strat_test_set[\"income_cat\"].value_counts() / len(strat_test_set)",
"_____no_output_____"
],
[
"housing[\"income_cat\"].value_counts() / len(housing)",
"_____no_output_____"
],
[
"def income_cat_proportions(data):\n return data[\"income_cat\"].value_counts() / len(data)\n\ntrain_set, test_set = train_test_split(housing, test_size=0.2, random_state=42)\n\ncompare_props = pd.DataFrame({\n \"Overall\": income_cat_proportions(housing),\n \"Stratified\": income_cat_proportions(strat_test_set),\n \"Random\": income_cat_proportions(test_set),\n}).sort_index()\ncompare_props[\"Rand. %error\"] = 100 * compare_props[\"Random\"] / compare_props[\"Overall\"] - 100\ncompare_props[\"Strat. %error\"] = 100 * compare_props[\"Stratified\"] / compare_props[\"Overall\"] - 100",
"_____no_output_____"
],
[
"compare_props",
"_____no_output_____"
],
[
"for set_ in (strat_train_set, strat_test_set):\n set_.drop(\"income_cat\", axis=1, inplace=True)",
"_____no_output_____"
]
],
[
[
"# Discover and visualize the data to gain insights",
"_____no_output_____"
]
],
[
[
"housing = strat_train_set.copy()",
"_____no_output_____"
],
[
"housing.plot(kind=\"scatter\", x=\"longitude\", y=\"latitude\")\nsave_fig(\"bad_visualization_plot\")",
"Saving figure bad_visualization_plot\n"
],
[
"housing.plot(kind=\"scatter\", x=\"longitude\", y=\"latitude\", alpha=0.1)\nsave_fig(\"better_visualization_plot\")",
"Saving figure better_visualization_plot\n"
]
],
[
[
"The argument `sharex=False` fixes a display bug (the x-axis values and legend were not displayed). This is a temporary fix (see: https://github.com/pandas-dev/pandas/issues/10611 ). Thanks to Wilmer Arellano for pointing it out.",
"_____no_output_____"
]
],
[
[
"housing.plot(kind=\"scatter\", x=\"longitude\", y=\"latitude\", alpha=0.4,\n s=housing[\"population\"]/100, label=\"population\", figsize=(10,7),\n c=\"median_house_value\", cmap=plt.get_cmap(\"jet\"), colorbar=True,\n sharex=False)\nplt.legend()\nsave_fig(\"housing_prices_scatterplot\")",
"Saving figure housing_prices_scatterplot\n"
],
[
"import matplotlib.image as mpimg\ncalifornia_img=mpimg.imread(PROJECT_ROOT_DIR + '/images/end_to_end_project/california.png')\nax = housing.plot(kind=\"scatter\", x=\"longitude\", y=\"latitude\", figsize=(10,7),\n s=housing['population']/100, label=\"Population\",\n c=\"median_house_value\", cmap=plt.get_cmap(\"jet\"),\n colorbar=False, alpha=0.4,\n )\nplt.imshow(california_img, extent=[-124.55, -113.80, 32.45, 42.05], alpha=0.5,\n cmap=plt.get_cmap(\"jet\"))\nplt.ylabel(\"Latitude\", fontsize=14)\nplt.xlabel(\"Longitude\", fontsize=14)\n\nprices = housing[\"median_house_value\"]\ntick_values = np.linspace(prices.min(), prices.max(), 11)\ncbar = plt.colorbar()\ncbar.ax.set_yticklabels([\"$%dk\"%(round(v/1000)) for v in tick_values], fontsize=14)\ncbar.set_label('Median House Value', fontsize=16)\n\nplt.legend(fontsize=16)\nsave_fig(\"california_housing_prices_plot\")\nplt.show()",
"_____no_output_____"
],
[
"corr_matrix = housing.corr()",
"_____no_output_____"
],
[
"corr_matrix[\"median_house_value\"].sort_values(ascending=False)",
"_____no_output_____"
],
[
"# from pandas.tools.plotting import scatter_matrix # For older versions of Pandas\nfrom pandas.plotting import scatter_matrix\n\nattributes = [\"median_house_value\", \"median_income\", \"total_rooms\",\n \"housing_median_age\"]\nscatter_matrix(housing[attributes], figsize=(12, 8))\nsave_fig(\"scatter_matrix_plot\")",
"_____no_output_____"
],
[
"housing.plot(kind=\"scatter\", x=\"median_income\", y=\"median_house_value\",\n alpha=0.1)\nplt.axis([0, 16, 0, 550000])\nsave_fig(\"income_vs_house_value_scatterplot\")",
"_____no_output_____"
],
[
"housing[\"rooms_per_household\"] = housing[\"total_rooms\"]/housing[\"households\"]\nhousing[\"bedrooms_per_room\"] = housing[\"total_bedrooms\"]/housing[\"total_rooms\"]\nhousing[\"population_per_household\"]=housing[\"population\"]/housing[\"households\"]",
"_____no_output_____"
],
[
"corr_matrix = housing.corr()\ncorr_matrix[\"median_house_value\"].sort_values(ascending=False)",
"_____no_output_____"
],
[
"housing.plot(kind=\"scatter\", x=\"rooms_per_household\", y=\"median_house_value\",\n alpha=0.2)\nplt.axis([0, 5, 0, 520000])\nplt.show()",
"_____no_output_____"
],
[
"housing.describe()",
"_____no_output_____"
]
],
[
[
"# Prepare the data for Machine Learning algorithms",
"_____no_output_____"
]
],
[
[
"housing = strat_train_set.drop(\"median_house_value\", axis=1) # drop labels for training set\nhousing_labels = strat_train_set[\"median_house_value\"].copy()",
"_____no_output_____"
],
[
"sample_incomplete_rows = housing[housing.isnull().any(axis=1)].head()\nsample_incomplete_rows",
"_____no_output_____"
],
[
"sample_incomplete_rows.dropna(subset=[\"total_bedrooms\"]) # option 1",
"_____no_output_____"
],
[
"sample_incomplete_rows.drop(\"total_bedrooms\", axis=1) # option 2",
"_____no_output_____"
],
[
"median = housing[\"total_bedrooms\"].median()\nsample_incomplete_rows[\"total_bedrooms\"].fillna(median, inplace=True) # option 3",
"_____no_output_____"
],
[
"sample_incomplete_rows",
"_____no_output_____"
],
[
"from sklearn.impute import SimpleImputer\nimputer = SimpleImputer(strategy=\"median\")",
"_____no_output_____"
]
],
[
[
"Remove the text attribute because median can only be calculated on numerical attributes:",
"_____no_output_____"
]
],
[
[
"housing_num = housing.drop(\"ocean_proximity\", axis=1)\n# alternatively: housing_num = housing.select_dtypes(include=[np.number])",
"_____no_output_____"
],
[
"imputer.fit(housing_num)",
"_____no_output_____"
],
[
"imputer.statistics_",
"_____no_output_____"
]
],
[
[
"Check that this is the same as manually computing the median of each attribute:",
"_____no_output_____"
]
],
[
[
"housing_num.median().values",
"_____no_output_____"
]
],
[
[
"Transform the training set:",
"_____no_output_____"
]
],
[
[
"X = imputer.transform(housing_num)",
"_____no_output_____"
],
[
"housing_tr = pd.DataFrame(X, columns=housing_num.columns,\n index=housing.index)",
"_____no_output_____"
],
[
"housing_tr.loc[sample_incomplete_rows.index.values]",
"_____no_output_____"
],
[
"imputer.strategy",
"_____no_output_____"
],
[
"housing_tr = pd.DataFrame(X, columns=housing_num.columns,\n index=housing_num.index)",
"_____no_output_____"
],
[
"housing_tr.head()",
"_____no_output_____"
]
],
[
[
"Now let's preprocess the categorical input feature, `ocean_proximity`:",
"_____no_output_____"
]
],
[
[
"housing_cat = housing[[\"ocean_proximity\"]]\nhousing_cat.head(10)",
"_____no_output_____"
],
[
"from sklearn.preprocessing import OrdinalEncoder\n\nordinal_encoder = OrdinalEncoder()\nhousing_cat_encoded = ordinal_encoder.fit_transform(housing_cat)\nhousing_cat_encoded[:10]",
"_____no_output_____"
],
[
"ordinal_encoder.categories_",
"_____no_output_____"
],
[
"from sklearn.preprocessing import OneHotEncoder\n\ncat_encoder = OneHotEncoder()\nhousing_cat_1hot = cat_encoder.fit_transform(housing_cat)\nhousing_cat_1hot",
"_____no_output_____"
]
],
[
[
"By default, the `OneHotEncoder` class returns a sparse array, but we can convert it to a dense array if needed by calling the `toarray()` method:",
"_____no_output_____"
]
],
[
[
"housing_cat_1hot.toarray()",
"_____no_output_____"
]
],
[
[
"Alternatively, you can set `sparse=False` when creating the `OneHotEncoder`:",
"_____no_output_____"
]
],
[
[
"cat_encoder = OneHotEncoder(sparse=False)\nhousing_cat_1hot = cat_encoder.fit_transform(housing_cat)\nhousing_cat_1hot",
"_____no_output_____"
],
[
"cat_encoder.categories_",
"_____no_output_____"
]
],
[
[
"Let's create a custom transformer to add extra attributes:",
"_____no_output_____"
]
],
[
[
"from sklearn.base import BaseEstimator, TransformerMixin\n\n# column index\nrooms_ix, bedrooms_ix, population_ix, households_ix = 3, 4, 5, 6\n\nclass CombinedAttributesAdder(BaseEstimator, TransformerMixin):\n def __init__(self, add_bedrooms_per_room = True): # no *args or **kargs\n self.add_bedrooms_per_room = add_bedrooms_per_room\n def fit(self, X, y=None):\n return self # nothing else to do\n def transform(self, X):\n rooms_per_household = X[:, rooms_ix] / X[:, households_ix]\n population_per_household = X[:, population_ix] / X[:, households_ix]\n if self.add_bedrooms_per_room:\n bedrooms_per_room = X[:, bedrooms_ix] / X[:, rooms_ix]\n return np.c_[X, rooms_per_household, population_per_household,\n bedrooms_per_room]\n else:\n return np.c_[X, rooms_per_household, population_per_household]\n\nattr_adder = CombinedAttributesAdder(add_bedrooms_per_room=False)\nhousing_extra_attribs = attr_adder.transform(housing.values)",
"_____no_output_____"
],
[
"housing_extra_attribs = pd.DataFrame(\n housing_extra_attribs,\n columns=list(housing.columns)+[\"rooms_per_household\", \"population_per_household\"],\n index=housing.index)\nhousing_extra_attribs.head()",
"_____no_output_____"
]
],
[
[
"Now let's build a pipeline for preprocessing the numerical attributes:",
"_____no_output_____"
]
],
[
[
"from sklearn.pipeline import Pipeline\nfrom sklearn.preprocessing import StandardScaler\n\nnum_pipeline = Pipeline([\n ('imputer', SimpleImputer(strategy=\"median\")),\n ('attribs_adder', CombinedAttributesAdder()),\n ('std_scaler', StandardScaler()),\n ])\n\nhousing_num_tr = num_pipeline.fit_transform(housing_num)",
"_____no_output_____"
],
[
"housing_num_tr",
"_____no_output_____"
],
[
"from sklearn.compose import ColumnTransformer\n\nnum_attribs = list(housing_num)\ncat_attribs = [\"ocean_proximity\"]\n\nfull_pipeline = ColumnTransformer([\n (\"num\", num_pipeline, num_attribs),\n (\"cat\", OneHotEncoder(), cat_attribs),\n ])\n\nhousing_prepared = full_pipeline.fit_transform(housing)",
"_____no_output_____"
],
[
"housing_prepared",
"_____no_output_____"
],
[
"housing_prepared.shape",
"_____no_output_____"
]
],
[
[
"For reference, here is the old solution based on a `DataFrameSelector` transformer (to just select a subset of the Pandas `DataFrame` columns), and a `FeatureUnion`:",
"_____no_output_____"
]
],
[
[
"from sklearn.base import BaseEstimator, TransformerMixin\n\n# Create a class to select numerical or categorical columns \nclass OldDataFrameSelector(BaseEstimator, TransformerMixin):\n def __init__(self, attribute_names):\n self.attribute_names = attribute_names\n def fit(self, X, y=None):\n return self\n def transform(self, X):\n return X[self.attribute_names].values",
"_____no_output_____"
]
],
[
[
"Now let's join all these components into a big pipeline that will preprocess both the numerical and the categorical features:",
"_____no_output_____"
]
],
[
[
"num_attribs = list(housing_num)\ncat_attribs = [\"ocean_proximity\"]\n\nold_num_pipeline = Pipeline([\n ('selector', OldDataFrameSelector(num_attribs)),\n ('imputer', SimpleImputer(strategy=\"median\")),\n ('attribs_adder', CombinedAttributesAdder()),\n ('std_scaler', StandardScaler()),\n ])\n\nold_cat_pipeline = Pipeline([\n ('selector', OldDataFrameSelector(cat_attribs)),\n ('cat_encoder', OneHotEncoder(sparse=False)),\n ])",
"_____no_output_____"
],
[
"from sklearn.pipeline import FeatureUnion\n\nold_full_pipeline = FeatureUnion(transformer_list=[\n (\"num_pipeline\", old_num_pipeline),\n (\"cat_pipeline\", old_cat_pipeline),\n ])",
"_____no_output_____"
],
[
"old_housing_prepared = old_full_pipeline.fit_transform(housing)\nold_housing_prepared",
"_____no_output_____"
]
],
[
[
"The result is the same as with the `ColumnTransformer`:",
"_____no_output_____"
]
],
[
[
"np.allclose(housing_prepared, old_housing_prepared)",
"_____no_output_____"
]
],
[
[
"# Select and train a model ",
"_____no_output_____"
]
],
[
[
"from sklearn.linear_model import LinearRegression\n\nlin_reg = LinearRegression()\nlin_reg.fit(housing_prepared, housing_labels)",
"_____no_output_____"
],
[
"# let's try the full preprocessing pipeline on a few training instances\nsome_data = housing.iloc[:5]\nsome_labels = housing_labels.iloc[:5]\nsome_data_prepared = full_pipeline.transform(some_data)\n\nprint(\"Predictions:\", lin_reg.predict(some_data_prepared))",
"_____no_output_____"
]
],
[
[
"Compare against the actual values:",
"_____no_output_____"
]
],
[
[
"print(\"Labels:\", list(some_labels))",
"_____no_output_____"
],
[
"some_data_prepared",
"_____no_output_____"
],
[
"from sklearn.metrics import mean_squared_error\n\nhousing_predictions = lin_reg.predict(housing_prepared)\nlin_mse = mean_squared_error(housing_labels, housing_predictions)\nlin_rmse = np.sqrt(lin_mse)\nlin_rmse",
"_____no_output_____"
],
[
"from sklearn.metrics import mean_absolute_error\n\nlin_mae = mean_absolute_error(housing_labels, housing_predictions)\nlin_mae",
"_____no_output_____"
],
[
"from sklearn.tree import DecisionTreeRegressor\n\ntree_reg = DecisionTreeRegressor(random_state=42)\ntree_reg.fit(housing_prepared, housing_labels)",
"_____no_output_____"
],
[
"housing_predictions = tree_reg.predict(housing_prepared)\ntree_mse = mean_squared_error(housing_labels, housing_predictions)\ntree_rmse = np.sqrt(tree_mse)\ntree_rmse",
"_____no_output_____"
]
],
[
[
"# Fine-tune your model",
"_____no_output_____"
]
],
[
[
"from sklearn.model_selection import cross_val_score\n\nscores = cross_val_score(tree_reg, housing_prepared, housing_labels,\n scoring=\"neg_mean_squared_error\", cv=10)\ntree_rmse_scores = np.sqrt(-scores)",
"_____no_output_____"
],
[
"def display_scores(scores):\n print(\"Scores:\", scores)\n print(\"Mean:\", scores.mean())\n print(\"Standard deviation:\", scores.std())\n\ndisplay_scores(tree_rmse_scores)",
"_____no_output_____"
],
[
"lin_scores = cross_val_score(lin_reg, housing_prepared, housing_labels,\n scoring=\"neg_mean_squared_error\", cv=10)\nlin_rmse_scores = np.sqrt(-lin_scores)\ndisplay_scores(lin_rmse_scores)",
"_____no_output_____"
]
],
[
[
"**Note**: we specify `n_estimators=100` to be future-proof since the default value is going to change to 100 in Scikit-Learn 0.22 (for simplicity, this is not shown in the book).",
"_____no_output_____"
]
],
[
[
"from sklearn.ensemble import RandomForestRegressor\n\nforest_reg = RandomForestRegressor(n_estimators=100, random_state=42)\nforest_reg.fit(housing_prepared, housing_labels)",
"_____no_output_____"
],
[
"housing_predictions = forest_reg.predict(housing_prepared)\nforest_mse = mean_squared_error(housing_labels, housing_predictions)\nforest_rmse = np.sqrt(forest_mse)\nforest_rmse",
"_____no_output_____"
],
[
"from sklearn.model_selection import cross_val_score\n\nforest_scores = cross_val_score(forest_reg, housing_prepared, housing_labels,\n scoring=\"neg_mean_squared_error\", cv=10)\nforest_rmse_scores = np.sqrt(-forest_scores)\ndisplay_scores(forest_rmse_scores)",
"_____no_output_____"
],
[
"scores = cross_val_score(lin_reg, housing_prepared, housing_labels, scoring=\"neg_mean_squared_error\", cv=10)\npd.Series(np.sqrt(-scores)).describe()",
"_____no_output_____"
],
[
"from sklearn.svm import SVR\n\nsvm_reg = SVR(kernel=\"linear\")\nsvm_reg.fit(housing_prepared, housing_labels)\nhousing_predictions = svm_reg.predict(housing_prepared)\nsvm_mse = mean_squared_error(housing_labels, housing_predictions)\nsvm_rmse = np.sqrt(svm_mse)\nsvm_rmse",
"_____no_output_____"
],
[
"from sklearn.model_selection import GridSearchCV\n\nparam_grid = [\n # try 12 (3×4) combinations of hyperparameters\n {'n_estimators': [3, 10, 30], 'max_features': [2, 4, 6, 8]},\n # then try 6 (2×3) combinations with bootstrap set as False\n {'bootstrap': [False], 'n_estimators': [3, 10], 'max_features': [2, 3, 4]},\n ]\n\nforest_reg = RandomForestRegressor(random_state=42)\n# train across 5 folds, that's a total of (12+6)*5=90 rounds of training \ngrid_search = GridSearchCV(forest_reg, param_grid, cv=5,\n scoring='neg_mean_squared_error',\n return_train_score=True)\ngrid_search.fit(housing_prepared, housing_labels)",
"_____no_output_____"
]
],
[
[
"The best hyperparameter combination found:",
"_____no_output_____"
]
],
[
[
"grid_search.best_params_",
"_____no_output_____"
],
[
"grid_search.best_estimator_",
"_____no_output_____"
]
],
[
[
"Let's look at the score of each hyperparameter combination tested during the grid search:",
"_____no_output_____"
]
],
[
[
"cvres = grid_search.cv_results_\nfor mean_score, params in zip(cvres[\"mean_test_score\"], cvres[\"params\"]):\n print(np.sqrt(-mean_score), params)",
"_____no_output_____"
],
[
"pd.DataFrame(grid_search.cv_results_)",
"_____no_output_____"
],
[
"from sklearn.model_selection import RandomizedSearchCV\nfrom scipy.stats import randint\n\nparam_distribs = {\n 'n_estimators': randint(low=1, high=200),\n 'max_features': randint(low=1, high=8),\n }\n\nforest_reg = RandomForestRegressor(random_state=42)\nrnd_search = RandomizedSearchCV(forest_reg, param_distributions=param_distribs,\n n_iter=10, cv=5, scoring='neg_mean_squared_error', random_state=42)\nrnd_search.fit(housing_prepared, housing_labels)",
"_____no_output_____"
],
[
"cvres = rnd_search.cv_results_\nfor mean_score, params in zip(cvres[\"mean_test_score\"], cvres[\"params\"]):\n print(np.sqrt(-mean_score), params)",
"_____no_output_____"
],
[
"feature_importances = grid_search.best_estimator_.feature_importances_\nfeature_importances",
"_____no_output_____"
],
[
"extra_attribs = [\"rooms_per_hhold\", \"pop_per_hhold\", \"bedrooms_per_room\"]\n#cat_encoder = cat_pipeline.named_steps[\"cat_encoder\"] # old solution\ncat_encoder = full_pipeline.named_transformers_[\"cat\"]\ncat_one_hot_attribs = list(cat_encoder.categories_[0])\nattributes = num_attribs + extra_attribs + cat_one_hot_attribs\nsorted(zip(feature_importances, attributes), reverse=True)",
"_____no_output_____"
],
[
"final_model = grid_search.best_estimator_\n\nX_test = strat_test_set.drop(\"median_house_value\", axis=1)\ny_test = strat_test_set[\"median_house_value\"].copy()\n\nX_test_prepared = full_pipeline.transform(X_test)\nfinal_predictions = final_model.predict(X_test_prepared)\n\nfinal_mse = mean_squared_error(y_test, final_predictions)\nfinal_rmse = np.sqrt(final_mse)",
"_____no_output_____"
],
[
"final_rmse",
"_____no_output_____"
]
],
[
[
"We can compute a 95% confidence interval for the test RMSE:",
"_____no_output_____"
]
],
[
[
"from scipy import stats\n\nconfidence = 0.95\nsquared_errors = (final_predictions - y_test) ** 2\nnp.sqrt(stats.t.interval(confidence, len(squared_errors) - 1,\n loc=squared_errors.mean(),\n scale=stats.sem(squared_errors)))",
"_____no_output_____"
]
],
[
[
"We could compute the interval manually like this:",
"_____no_output_____"
]
],
[
[
"m = len(squared_errors)\nmean = squared_errors.mean()\ntscore = stats.t.ppf((1 + confidence) / 2, df=m - 1)\ntmargin = tscore * squared_errors.std(ddof=1) / np.sqrt(m)\nnp.sqrt(mean - tmargin), np.sqrt(mean + tmargin)",
"_____no_output_____"
]
],
[
[
"Alternatively, we could use a z-scores rather than t-scores:",
"_____no_output_____"
]
],
[
[
"zscore = stats.norm.ppf((1 + confidence) / 2)\nzmargin = zscore * squared_errors.std(ddof=1) / np.sqrt(m)\nnp.sqrt(mean - zmargin), np.sqrt(mean + zmargin)",
"_____no_output_____"
]
],
[
[
"# Extra material",
"_____no_output_____"
],
[
"## A full pipeline with both preparation and prediction",
"_____no_output_____"
]
],
[
[
"full_pipeline_with_predictor = Pipeline([\n (\"preparation\", full_pipeline),\n (\"linear\", LinearRegression())\n ])\n\nfull_pipeline_with_predictor.fit(housing, housing_labels)\nfull_pipeline_with_predictor.predict(some_data)",
"_____no_output_____"
]
],
[
[
"## Model persistence using joblib",
"_____no_output_____"
]
],
[
[
"my_model = full_pipeline_with_predictor",
"_____no_output_____"
],
[
"import joblib\njoblib.dump(my_model, \"my_model.pkl\") # DIFF\n#...\nmy_model_loaded = joblib.load(\"my_model.pkl\") # DIFF",
"_____no_output_____"
]
],
[
[
"## Example SciPy distributions for `RandomizedSearchCV`",
"_____no_output_____"
]
],
[
[
"from scipy.stats import geom, expon\ngeom_distrib=geom(0.5).rvs(10000, random_state=42)\nexpon_distrib=expon(scale=1).rvs(10000, random_state=42)\nplt.hist(geom_distrib, bins=50)\nplt.show()\nplt.hist(expon_distrib, bins=50)\nplt.show()",
"_____no_output_____"
]
],
[
[
"# Exercise solutions",
"_____no_output_____"
],
[
"## 1.",
"_____no_output_____"
],
[
"Question: Try a Support Vector Machine regressor (`sklearn.svm.SVR`), with various hyperparameters such as `kernel=\"linear\"` (with various values for the `C` hyperparameter) or `kernel=\"rbf\"` (with various values for the `C` and `gamma` hyperparameters). Don't worry about what these hyperparameters mean for now. How does the best `SVR` predictor perform?",
"_____no_output_____"
]
],
[
[
"from sklearn.model_selection import GridSearchCV\n\nparam_grid = [\n {'kernel': ['linear'], 'C': [10., 30., 100., 300., 1000., 3000., 10000., 30000.0]},\n {'kernel': ['rbf'], 'C': [1.0, 3.0, 10., 30., 100., 300., 1000.0],\n 'gamma': [0.01, 0.03, 0.1, 0.3, 1.0, 3.0]},\n ]\n\nsvm_reg = SVR()\ngrid_search = GridSearchCV(svm_reg, param_grid, cv=5, scoring='neg_mean_squared_error', verbose=2)\ngrid_search.fit(housing_prepared, housing_labels)",
"_____no_output_____"
]
],
[
[
"The best model achieves the following score (evaluated using 5-fold cross validation):",
"_____no_output_____"
]
],
[
[
"negative_mse = grid_search.best_score_\nrmse = np.sqrt(-negative_mse)\nrmse",
"_____no_output_____"
]
],
[
[
"That's much worse than the `RandomForestRegressor`. Let's check the best hyperparameters found:",
"_____no_output_____"
]
],
[
[
"grid_search.best_params_",
"_____no_output_____"
]
],
[
[
"The linear kernel seems better than the RBF kernel. Notice that the value of `C` is the maximum tested value. When this happens you definitely want to launch the grid search again with higher values for `C` (removing the smallest values), because it is likely that higher values of `C` will be better.",
"_____no_output_____"
],
[
"## 2.",
"_____no_output_____"
],
[
"Question: Try replacing `GridSearchCV` with `RandomizedSearchCV`.",
"_____no_output_____"
]
],
[
[
"from sklearn.model_selection import RandomizedSearchCV\nfrom scipy.stats import expon, reciprocal\n\n# see https://docs.scipy.org/doc/scipy/reference/stats.html\n# for `expon()` and `reciprocal()` documentation and more probability distribution functions.\n\n# Note: gamma is ignored when kernel is \"linear\"\nparam_distribs = {\n 'kernel': ['linear', 'rbf'],\n 'C': reciprocal(20, 200000),\n 'gamma': expon(scale=1.0),\n }\n\nsvm_reg = SVR()\nrnd_search = RandomizedSearchCV(svm_reg, param_distributions=param_distribs,\n n_iter=50, cv=5, scoring='neg_mean_squared_error',\n verbose=2, random_state=42)\nrnd_search.fit(housing_prepared, housing_labels)",
"_____no_output_____"
]
],
[
[
"The best model achieves the following score (evaluated using 5-fold cross validation):",
"_____no_output_____"
]
],
[
[
"negative_mse = rnd_search.best_score_\nrmse = np.sqrt(-negative_mse)\nrmse",
"_____no_output_____"
]
],
[
[
"Now this is much closer to the performance of the `RandomForestRegressor` (but not quite there yet). Let's check the best hyperparameters found:",
"_____no_output_____"
]
],
[
[
"rnd_search.best_params_",
"_____no_output_____"
]
],
[
[
"This time the search found a good set of hyperparameters for the RBF kernel. Randomized search tends to find better hyperparameters than grid search in the same amount of time.",
"_____no_output_____"
],
[
"Let's look at the exponential distribution we used, with `scale=1.0`. Note that some samples are much larger or smaller than 1.0, but when you look at the log of the distribution, you can see that most values are actually concentrated roughly in the range of exp(-2) to exp(+2), which is about 0.1 to 7.4.",
"_____no_output_____"
]
],
[
[
"expon_distrib = expon(scale=1.)\nsamples = expon_distrib.rvs(10000, random_state=42)\nplt.figure(figsize=(10, 4))\nplt.subplot(121)\nplt.title(\"Exponential distribution (scale=1.0)\")\nplt.hist(samples, bins=50)\nplt.subplot(122)\nplt.title(\"Log of this distribution\")\nplt.hist(np.log(samples), bins=50)\nplt.show()",
"_____no_output_____"
]
],
[
[
"The distribution we used for `C` looks quite different: the scale of the samples is picked from a uniform distribution within a given range, which is why the right graph, which represents the log of the samples, looks roughly constant. This distribution is useful when you don't have a clue of what the target scale is:",
"_____no_output_____"
]
],
[
[
"reciprocal_distrib = reciprocal(20, 200000)\nsamples = reciprocal_distrib.rvs(10000, random_state=42)\nplt.figure(figsize=(10, 4))\nplt.subplot(121)\nplt.title(\"Reciprocal distribution (scale=1.0)\")\nplt.hist(samples, bins=50)\nplt.subplot(122)\nplt.title(\"Log of this distribution\")\nplt.hist(np.log(samples), bins=50)\nplt.show()",
"_____no_output_____"
]
],
[
[
"The reciprocal distribution is useful when you have no idea what the scale of the hyperparameter should be (indeed, as you can see on the figure on the right, all scales are equally likely, within the given range), whereas the exponential distribution is best when you know (more or less) what the scale of the hyperparameter should be.",
"_____no_output_____"
],
[
"## 3.",
"_____no_output_____"
],
[
"Question: Try adding a transformer in the preparation pipeline to select only the most important attributes.",
"_____no_output_____"
]
],
[
[
"from sklearn.base import BaseEstimator, TransformerMixin\n\ndef indices_of_top_k(arr, k):\n return np.sort(np.argpartition(np.array(arr), -k)[-k:])\n\nclass TopFeatureSelector(BaseEstimator, TransformerMixin):\n def __init__(self, feature_importances, k):\n self.feature_importances = feature_importances\n self.k = k\n def fit(self, X, y=None):\n self.feature_indices_ = indices_of_top_k(self.feature_importances, self.k)\n return self\n def transform(self, X):\n return X[:, self.feature_indices_]",
"_____no_output_____"
]
],
[
[
"Note: this feature selector assumes that you have already computed the feature importances somehow (for example using a `RandomForestRegressor`). You may be tempted to compute them directly in the `TopFeatureSelector`'s `fit()` method, however this would likely slow down grid/randomized search since the feature importances would have to be computed for every hyperparameter combination (unless you implement some sort of cache).",
"_____no_output_____"
],
[
"Let's define the number of top features we want to keep:",
"_____no_output_____"
]
],
[
[
"k = 5",
"_____no_output_____"
]
],
[
[
"Now let's look for the indices of the top k features:",
"_____no_output_____"
]
],
[
[
"top_k_feature_indices = indices_of_top_k(feature_importances, k)\ntop_k_feature_indices",
"_____no_output_____"
],
[
"np.array(attributes)[top_k_feature_indices]",
"_____no_output_____"
]
],
[
[
"Let's double check that these are indeed the top k features:",
"_____no_output_____"
]
],
[
[
"sorted(zip(feature_importances, attributes), reverse=True)[:k]",
"_____no_output_____"
]
],
[
[
"Looking good... Now let's create a new pipeline that runs the previously defined preparation pipeline, and adds top k feature selection:",
"_____no_output_____"
]
],
[
[
"preparation_and_feature_selection_pipeline = Pipeline([\n ('preparation', full_pipeline),\n ('feature_selection', TopFeatureSelector(feature_importances, k))\n])",
"_____no_output_____"
],
[
"housing_prepared_top_k_features = preparation_and_feature_selection_pipeline.fit_transform(housing)",
"_____no_output_____"
]
],
[
[
"Let's look at the features of the first 3 instances:",
"_____no_output_____"
]
],
[
[
"housing_prepared_top_k_features[0:3]",
"_____no_output_____"
]
],
[
[
"Now let's double check that these are indeed the top k features:",
"_____no_output_____"
]
],
[
[
"housing_prepared[0:3, top_k_feature_indices]",
"_____no_output_____"
]
],
[
[
"Works great! :)",
"_____no_output_____"
],
[
"## 4.",
"_____no_output_____"
],
[
"Question: Try creating a single pipeline that does the full data preparation plus the final prediction.",
"_____no_output_____"
]
],
[
[
"prepare_select_and_predict_pipeline = Pipeline([\n ('preparation', full_pipeline),\n ('feature_selection', TopFeatureSelector(feature_importances, k)),\n ('svm_reg', SVR(**rnd_search.best_params_))\n])",
"_____no_output_____"
],
[
"prepare_select_and_predict_pipeline.fit(housing, housing_labels)",
"_____no_output_____"
]
],
[
[
"Let's try the full pipeline on a few instances:",
"_____no_output_____"
]
],
[
[
"some_data = housing.iloc[:4]\nsome_labels = housing_labels.iloc[:4]\n\nprint(\"Predictions:\\t\", prepare_select_and_predict_pipeline.predict(some_data))\nprint(\"Labels:\\t\\t\", list(some_labels))",
"_____no_output_____"
]
],
[
[
"Well, the full pipeline seems to work fine. Of course, the predictions are not fantastic: they would be better if we used the best `RandomForestRegressor` that we found earlier, rather than the best `SVR`.",
"_____no_output_____"
],
[
"## 5.",
"_____no_output_____"
],
[
"Question: Automatically explore some preparation options using `GridSearchCV`.",
"_____no_output_____"
]
],
[
[
"param_grid = [{\n 'preparation__num__imputer__strategy': ['mean', 'median', 'most_frequent'],\n 'feature_selection__k': list(range(1, len(feature_importances) + 1))\n}]\n\ngrid_search_prep = GridSearchCV(prepare_select_and_predict_pipeline, param_grid, cv=5,\n scoring='neg_mean_squared_error', verbose=2)\ngrid_search_prep.fit(housing, housing_labels)",
"_____no_output_____"
],
[
"grid_search_prep.best_params_",
"_____no_output_____"
]
],
[
[
"The best imputer strategy is `most_frequent` and apparently almost all features are useful (15 out of 16). The last one (`ISLAND`) seems to just add some noise.",
"_____no_output_____"
],
[
"Congratulations! You already know quite a lot about Machine Learning. :)",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
]
] |
e7eb3f45603d146b634f4eb94cb3f5047570b914 | 7,799 | ipynb | Jupyter Notebook | Chapter04/.ipynb_checkpoints/4.03. Extracting embeddings with ALBERT-checkpoint.ipynb | shizukanaskytree/Getting-Started-with-Google-BERT | 644d074574100b1357c0bc88bb212c212d70e030 | [
"MIT"
] | 111 | 2021-01-12T16:21:20.000Z | 2022-03-20T19:19:17.000Z | Chapter04/.ipynb_checkpoints/4.03. Extracting embeddings with ALBERT-checkpoint.ipynb | kimCheongBin/Getting-Started-with-Google-BERT | 644d074574100b1357c0bc88bb212c212d70e030 | [
"MIT"
] | 3 | 2021-11-22T06:51:51.000Z | 2022-02-13T08:51:05.000Z | Chapter04/.ipynb_checkpoints/4.03. Extracting embeddings with ALBERT-checkpoint.ipynb | kimCheongBin/Getting-Started-with-Google-BERT | 644d074574100b1357c0bc88bb212c212d70e030 | [
"MIT"
] | 53 | 2021-01-12T12:32:46.000Z | 2022-03-23T01:53:38.000Z | 35.940092 | 311 | 0.625208 | [
[
[
"# Extracting embeddings with ALBERT\nWith Hugging Face transformers, we can use the ALBERT model just like how we used BERT. Let's explore this with a small example. Suppose, we need to get the contextual word embedding of every word in the sentence Paris is a beautiful city. Let's see how to that with ALBERT. \n\nImport the necessary modules: ",
"_____no_output_____"
]
],
[
[
"!pip install transformers==3.5.1",
"Requirement already satisfied: transformers==3.5.1 in /usr/local/lib/python3.6/dist-packages (3.5.1)\nRequirement already satisfied: dataclasses; python_version < \"3.7\" in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (0.8)\nRequirement already satisfied: sentencepiece==0.1.91 in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (0.1.91)\nRequirement already satisfied: tqdm>=4.27 in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (4.41.1)\nRequirement already satisfied: sacremoses in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (0.0.43)\nRequirement already satisfied: regex!=2019.12.17 in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (2019.12.20)\nRequirement already satisfied: filelock in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (3.0.12)\nRequirement already satisfied: requests in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (2.23.0)\nRequirement already satisfied: packaging in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (20.8)\nRequirement already satisfied: tokenizers==0.9.3 in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (0.9.3)\nRequirement already satisfied: numpy in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (1.19.4)\nRequirement already satisfied: protobuf in /usr/local/lib/python3.6/dist-packages (from transformers==3.5.1) (3.12.4)\nRequirement already satisfied: click in /usr/local/lib/python3.6/dist-packages (from sacremoses->transformers==3.5.1) (7.1.2)\nRequirement already satisfied: six in /usr/local/lib/python3.6/dist-packages (from sacremoses->transformers==3.5.1) (1.15.0)\nRequirement already satisfied: joblib in /usr/local/lib/python3.6/dist-packages (from sacremoses->transformers==3.5.1) (1.0.0)\nRequirement already satisfied: chardet<4,>=3.0.2 in /usr/local/lib/python3.6/dist-packages (from requests->transformers==3.5.1) (3.0.4)\nRequirement already satisfied: certifi>=2017.4.17 in /usr/local/lib/python3.6/dist-packages (from requests->transformers==3.5.1) (2020.12.5)\nRequirement already satisfied: idna<3,>=2.5 in /usr/local/lib/python3.6/dist-packages (from requests->transformers==3.5.1) (2.10)\nRequirement already satisfied: urllib3!=1.25.0,!=1.25.1,<1.26,>=1.21.1 in /usr/local/lib/python3.6/dist-packages (from requests->transformers==3.5.1) (1.24.3)\nRequirement already satisfied: pyparsing>=2.0.2 in /usr/local/lib/python3.6/dist-packages (from packaging->transformers==3.5.1) (2.4.7)\nRequirement already satisfied: setuptools in /usr/local/lib/python3.6/dist-packages (from protobuf->transformers==3.5.1) (51.0.0)\n"
],
[
"from transformers import AlbertTokenizer, AlbertModel",
"_____no_output_____"
]
],
[
[
"\nDownload and load the pre-trained Albert model and tokenizer. In this tutorial, we use the ALBERT-base model: \n",
"_____no_output_____"
]
],
[
[
"model = AlbertModel.from_pretrained('albert-base-v2')\ntokenizer = AlbertTokenizer.from_pretrained('albert-base-v2')",
"_____no_output_____"
]
],
[
[
"\nNow, feed the sentence to the tokenizer and get the preprocessed input: ",
"_____no_output_____"
]
],
[
[
"sentence = \"Paris is a beautiful city\" \ninputs = tokenizer(sentence, return_tensors=\"pt\")",
"_____no_output_____"
]
],
[
[
"\nLet's print the inputs:",
"_____no_output_____"
]
],
[
[
"print(inputs)",
"{'input_ids': tensor([[ 2, 1162, 25, 21, 1632, 136, 3]]), 'token_type_ids': tensor([[0, 0, 0, 0, 0, 0, 0]]), 'attention_mask': tensor([[1, 1, 1, 1, 1, 1, 1]])}\n"
]
],
[
[
"\nNow we just feed the inputs to the model and get the result. The model returns the hidden_rep which contains the hidden state representation of all the tokens from the final encoder layer and cls_head which contains the hidden state representation of the [CLS] token from the final encoder layer:\n",
"_____no_output_____"
]
],
[
[
"hidden_rep, cls_head = model(**inputs)",
"_____no_output_____"
]
],
[
[
"\n\nWe can obtain the contextual word embedding of each word in the sentence just like BERT as:\n\n- hidden_rep[0][0] contains the contextual embedding of the token [CLS]\n- hidden_rep[0][1] contains the contextual embedding of the token 'Paris' \n- hidden_rep[0][2] contains the contextual embedding of the token 'is' \n\nSimilarly in this manner, hidden_rep[0][7] contains the contextual embedding of the token 'city'. \n\nIn this way, we can use the ALBERT model just like how we used the BERT model. We can also fine-tune the ALBERT model similar to how we fine-tuned the BERT model on any downstream task. Now that we learned how ALBERT works, in the next section, let us explore RoBERTa, another interesting variant of BERT.",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
]
] |
e7eb61aa2f88545cbd07495ee18885143a351956 | 17,658 | ipynb | Jupyter Notebook | assignments/HW2.2.2.3-shahzaib.ipynb | shahzaib-sheikh/deep-learning | 1240c986e17ec24ccbb2c5738caad5c73da5764c | [
"MIT"
] | null | null | null | assignments/HW2.2.2.3-shahzaib.ipynb | shahzaib-sheikh/deep-learning | 1240c986e17ec24ccbb2c5738caad5c73da5764c | [
"MIT"
] | null | null | null | assignments/HW2.2.2.3-shahzaib.ipynb | shahzaib-sheikh/deep-learning | 1240c986e17ec24ccbb2c5738caad5c73da5764c | [
"MIT"
] | null | null | null | 27.984152 | 90 | 0.323706 | [
[
[
"import os\nimport pandas as pd\nimport numpy as np\n\ndf = pd.read_csv(\n \"https://data.heatonresearch.com/data/t81-558/auto-mpg.csv\", \n na_values=['NA', '?'])\n\n#np.random.seed(42) # Uncomment this line to get the same shuffle each time\ndf = df.reindex(np.random.permutation(df.index))\n\npd.set_option('display.max_columns', 7)\npd.set_option('display.max_rows', 5)\ndisplay(df)",
"_____no_output_____"
],
[
"pd.set_option('display.max_columns', 7)\npd.set_option('display.max_rows', 5)\n\ndf.reset_index(inplace=True, drop=True)\ndisplay(df)",
"_____no_output_____"
],
[
"import os\nimport pandas as pd\n\ndf = pd.read_csv(\n \"https://data.heatonresearch.com/data/t81-558/auto-mpg.csv\", \n na_values=['NA', '?'])\n\ndf = df.sort_values(by='name', ascending=True)\nprint(f\"The first car is: {df['name'].iloc[0]}\")\n \npd.set_option('display.max_columns', 7)\npd.set_option('display.max_rows', 5)\ndisplay(df)",
"The first car is: amc ambassador brougham\n"
],
[
"import os\nimport pandas as pd\n\ndf = pd.read_csv(\n \"https://data.heatonresearch.com/data/t81-558/auto-mpg.csv\", \n na_values=['NA', '?'])\n\npd.set_option('display.max_columns', 7)\npd.set_option('display.max_rows', 5)\ndisplay(df)",
"_____no_output_____"
],
[
"g = df.groupby('cylinders')['mpg'].mean()\ng",
"_____no_output_____"
],
[
"d = g.to_dict()\nd",
"_____no_output_____"
],
[
"d[6]",
"_____no_output_____"
],
[
"df.groupby('cylinders')['mpg'].count().to_dict()",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7eb62c08d764c2dcb51a5aa45d1d99532711bbb | 1,855 | ipynb | Jupyter Notebook | 00_core.ipynb | VladislavYak/test_repo | 7b781ac1a7ed655f0051fb2dd28c47bc3b387558 | [
"Apache-2.0"
] | null | null | null | 00_core.ipynb | VladislavYak/test_repo | 7b781ac1a7ed655f0051fb2dd28c47bc3b387558 | [
"Apache-2.0"
] | null | null | null | 00_core.ipynb | VladislavYak/test_repo | 7b781ac1a7ed655f0051fb2dd28c47bc3b387558 | [
"Apache-2.0"
] | null | null | null | 17.666667 | 73 | 0.450674 | [
[
[
"# default_exp core",
"_____no_output_____"
]
],
[
[
"# module name here\n\n> API details.",
"_____no_output_____"
]
],
[
[
"#hide\nfrom nbdev.showdoc import *",
"_____no_output_____"
],
[
"#export\nimport numpy\n\nclass Matrix():\n \"\"\"\n Class generates a zero matrix\n \"\"\"\n def __init__(self, n_matrix:int, m_matrix:int):\n self.n = n_matrix\n self.m = m_matrix\n def make_matrix(self):\n return numpy.zeros(self.n * self.m).reshape(self.n, self.m)",
"_____no_output_____"
]
],
[
[
"Matrix",
"_____no_output_____"
]
],
[
[
"a = Matrix(2, 5)\na.make_matrix()",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7eb6453f773ed80db6a01f10fa2ae6684e51c8e | 860,039 | ipynb | Jupyter Notebook | main.ipynb | AlessandroTaglieri/ADM-HW4 | cdf37319c6ff0fbb6d8b7d1d3da9d1f6428b4d58 | [
"MIT"
] | null | null | null | main.ipynb | AlessandroTaglieri/ADM-HW4 | cdf37319c6ff0fbb6d8b7d1d3da9d1f6428b4d58 | [
"MIT"
] | null | null | null | main.ipynb | AlessandroTaglieri/ADM-HW4 | cdf37319c6ff0fbb6d8b7d1d3da9d1f6428b4d58 | [
"MIT"
] | 1 | 2021-04-06T10:14:49.000Z | 2021-04-06T10:14:49.000Z | 461.146917 | 345,932 | 0.932657 | [
[
[
"## 1. Hashing task!",
"_____no_output_____"
],
[
"* We've put all passwords from passwords1.txt and passwords2.txt in two lists: listPasswords1 and listPasswords2",
"_____no_output_____"
]
],
[
[
"listPasswords1=[]\nlistPasswords2=[]\nfilePassword1 = open(\"passwords1.txt\", 'r')\nlines_p1 = filePassword1.readlines()\nfor line in lines_p1:\n listPasswords1.append(line.strip())\n\nfilePassword2 = open(\"passwords2.txt\", 'r')\nlines_p2 = filePassword2.readlines()\nfor line in lines_p2:\n listPasswords2.append(line.strip())\n\n",
"_____no_output_____"
]
],
[
[
"We have set all variables that we need to build our Bloom Filter:\n* n = Number of items in the filter\n* p = Probability of false positives, fraction between 0 and 1\n* m = Number of bits in the filter (size of Bloom Filter bit-array)\n* k = Number of hash functions\n\nWe did these following considerations:\nN is the amount of passwords that are in passwordss1.txt (i.e. in listPasswords1).\nWe need to calculate p,m and k from following formulas:\n\\begin{align*}\nk = (m/n)*ln(2)\n\\end{align*}\n\n\\begin{align*}\nm = ((n*ln(p)) / (ln(2))^2\n\\end{align*}\n\nSince we don't know the value of any previous varaibles, from https://hackernoon.com/probabilistic-data-structures-bloom-filter-5374112a7832 (suggested link in track) we have set initially p=0.01. P must be a value between 0 and 1. With smaller value of p, we have a lower probability to have a false positives in search on bloom filter. \nBloom filter can have false positives and NOT false negatives. We have choosen this value (0.01) from a different test that we have tryed. We think that it should be an optimal value to have also optimal values about k and m.\nFinally we calculated k and m (with previous formulas).",
"_____no_output_____"
]
],
[
[
"n=len(listPasswords1)\np=0.01\nimport math\nm=math.ceil((n * math.log(p)) / math.log(1 / pow(2, math.log(2))))\nk = round((m / n) * math.log(2))",
"_____no_output_____"
]
],
[
[
"This following function is our hash function. We have written from scratch our fnv function based on fnv hash function. FNV hashes are designed to be fast while maintaining a low collision rate. The FNV speed allows one to quickly hash lots of data while maintaining a reasonable collision rate. \nTo build bloom filter, we need of various hash functions that should be independent, uniformly distributed and fast.\nFrom previous formulas, we have calculated the number of different hash functions that we should use. We set a variable, called 'seed' that is an integer from 0 to k(number of hash functions); in this way we'll have k different hash fucntions that transform our input string in k different number. These k numbers will be the index of our bit-array that represent respective inout password.\n\nThe initial values of 'FNV_prime' and 'offset_basis' are taken from this wikipedia page: https://en.wikipedia.org/wiki/Fowler–Noll–Vo_hash_function in 'FNV hash parameters'.\n",
"_____no_output_____"
]
],
[
[
"def fnv1_64(password, seed=0):\n \"\"\"\n Returns: The FNV-1 hash of a given string. \n \"\"\"\n #Constants\n FNV_prime = 1099511628211\n offset_basis = 14695981039346656037\n\n #FNV-1a Hash Function\n hash = offset_basis + seed\n for char in password:\n hash = hash * FNV_prime\n hash = hash ^ ord(char)\n return hash\n\n\n",
"_____no_output_____"
]
],
[
[
"This following class 'BloomFilter' represent our Bloom Filter. \n\nIt has three attributes:\n* sizeArray = dimension of bit-array\n* number_HashFucntion = number of hash functions\n* array_BloomFilter = bit-array of bloom filter\n\nIt has also two methods:\n* init: it takes, as parameteers, our bloom filter, k (number of hash functions that we have calculated from previous formula) and m (size of bit-array that we have calculated with previous function). It initializes the bit-array of Bloom filter (size=m) with all '0', its size with m and its number of hash functions with k.\n* add: this method allows to add an element from input list to our bloom filter. It takes this list and our bloom filter as parameters and it calculates the effective bit-array with right '1' and '0'.",
"_____no_output_____"
]
],
[
[
"class BloomFilter:\n\n sizeArray=0\n number_HashFucntion=0\n array_BloomFilter=[]\n \n @property\n def size(self):\n return self.sizeArray\n @property\n def numHash(self):\n return self.number_HashFucntion\n @property\n def arrayBloom(self):\n return self.array_BloomFilter\n \n def init(self,k,m):\n self.sizeArray=m\n self.number_HashFucntion=k\n \n for i in range(m):\n self.array_BloomFilter.append(0)\n \n def add(self,strings):\n #print(self.number_HashFucntion)\n #print(self.sizeArray)\n h=0\n for psw in strings:\n \n for seed in range(self.number_HashFucntion):\n index=fnv1_64(psw,seed) % self.sizeArray\n \n self.array_BloomFilter[index]=1",
"_____no_output_____"
]
],
[
[
"Then we made a function 'checkPassw' that checks how many password in listPasswords2 (i.e. in passowrds2.txt) are in our Bloom Filter. It takes our bloom filter and the list of passwords that are in passwords2.txt and it returns how many passwords are in bloom filter. \nA password is in our bloom filter if and only if its conversion with hash functions coresponds to all '1' in the bit-array. If there is only one '0', current password is not in bloom filter.\n\n'countCheck' is the number of occurences of passwords (from passwords2.txt) in bloom filter. This number represents the occurences of passwords from passwords2.txt that are 'probably' in password1.txt",
"_____no_output_____"
]
],
[
[
"def checkPassw(BloomFilter, listPasswords2):\n countCheck=0\n for psw in listPasswords2:\n count=0\n for seed in range(BloomFilter.number_HashFucntion):\n index=fnv1_64(psw,seed) % BloomFilter.sizeArray\n if BloomFilter.array_BloomFilter[index]==1:\n count+=1\n if count==BloomFilter.number_HashFucntion:\n countCheck+=1\n \n return countCheck",
"_____no_output_____"
]
],
[
[
"## Bonus section: \n###### We calculate the number of false positive\n\nThis following function 'falsePositives' allows to calculate the exact number of false positive. It takes these following data, as parameter:\n* BloomFilter = our bloom filter\n* listPassowrds1 = passwords from passwords1.txt\n* listPassowrds2 = passwords from passwords2.txt\n\nFor every password in listPasswords2, we check if current password is in the bloom filter. Then if this password is in bloom filter, it means that it is 'probably' in listPasswords2. \nWith conversion, through hash functions, If there is only one '0' in bloom filter, it means that this current password is not in bloom filter.\n\nTo check if it's a false positive (i.e. it is in bloom fileter but it is not in listPasswords1), we'll continue to verify if this current password is in listPassword1. If it's true, it means that it is a False positive and we increment our counter 'countFalsePositives'; if it's True it means this password is actually in listPassword1 and we continue with next password in listPassword2.\nWe used a SET structure to verify if a password is in listPassword1, beacuse it's faster to find an element in large structure than in a list. So we get a set 's' from listPassword1. This following figure shows the reason to use a set to find an element in a large collection of data than a simple list. \n\n<img src=\"png/HSRgg.png\">\n \nhttps://stackoverflow.com/questions/7571635/fastest-way-to-check-if-a-value-exists-in-a-list",
"_____no_output_____"
]
],
[
[
"def falsePositives(BloomFilter, listPasswords1, listPasswords2):\n s= set(listPasswords1)\n countFalsePositives=0\n \n for psw in listPasswords2:\n count=0\n for seed in range(BloomFilter.number_HashFucntion):\n index=fnv1_64(psw,seed) % BloomFilter.sizeArray\n if (BloomFilter.array_BloomFilter[index]==1):\n count+=1\n else:\n break\n if count==BloomFilter.number_HashFucntion:\n if not(psw in s):\n countFalsePositives+=1\n #print(psw)\n return countFalsePositives\n ",
"_____no_output_____"
]
],
[
[
"Then we did the main function, as wirtten in homework track. With this function we did these following steps:\n* init bit-array, its size adn number of hash functions of our bloom filter, with 'BloomFilter.init(BloomFilter,k,m)'\n* add passowrds from listPasswords1 into our bloom filter, with 'BloomFilter.add(BloomFilter,listPasswords1)'\n* calculate how many passwords (from passwords2.txt) are present in our bloom filter, with 'checkPassw(BloomFilter,listPasswords2)'\n\n###### Finally we print these following functions:\n* Number of hash function used\n* Number of duplicates detected\n* Probability of false positives\n* Execution time",
"_____no_output_____"
]
],
[
[
"import time\n\ndef BloomFilterFunc(listPasswords1, listPasswords2):\n start = time.time()\n #init our bloom filter\n BloomFilter.init(BloomFilter,k,m)\n #add all passowrd from listPassowrds1 to our bloom filter\n BloomFilter.add(BloomFilter,listPasswords1)\n #check and save into 'countPassw' the number of occurences of password (from passwords2) in the bloom filter\n countPassw=checkPassw(BloomFilter,listPasswords2)\n end = time.time()\n \n #print output data\n \n \n print('Number of hash function used: ', k)\n print('Number of duplicates detected: ', countPassw)\n print('Probability of false positives: ', p)\n print('Execution time: ', end-start)\n \n\n",
"_____no_output_____"
]
],
[
[
"* Execute main function",
"_____no_output_____"
]
],
[
[
"BloomFilterFunc(listPasswords1, listPasswords2)",
"Number of hash function used: 7\nNumber of duplicates detected: 14251447\nProbability of false positives: 0.01\nExecution time: 4041.6586632728577\n"
]
],
[
[
"* Execute bonus section",
"_____no_output_____"
]
],
[
[
"falsPositive=falsePositives(BloomFilter,listPasswords1,listPasswords2)",
"_____no_output_____"
],
[
"print('Number of false positive: ', falsPositive)",
"Number of false positive: 251447\n"
]
],
[
[
"# 2. Alphabetical Sort",
"_____no_output_____"
],
[
"Given a set of words, a common natural task is the one of sorting them in alphabetical order. It is something that you have for sure already done once in your life, using your own algorithm without maybe knowing it.\n\nIn order to be everyone on the same page, we will refer to the rules defined here. As for multi-word string, let stick with the first plicy proposed there.\n\nWhat you might know is that we can relate this task to a simple algorithm that runs in linear time: Counting Sort. Counting Sort is based on a simple assumption: you know the range of the possible values of the instances you have to sort. In this exercise you are asked to perform Alphabetical Sort exploiting the algorithm of Counting Sort.",
"_____no_output_____"
]
],
[
[
"import string\nimprot numpay as np\nlower = list(string.ascii_lowercase)\nupper = string.ascii_uppercase",
"_____no_output_____"
],
[
"lower",
"_____no_output_____"
]
],
[
[
"Build your own implementation of Counting Sort...",
"_____no_output_____"
],
[
"Here is the counting sort algorithm with a bite innovation and fewer assignments than the original one explained in the attached website to the hw",
"_____no_output_____"
]
],
[
[
"def s_counting(A): \n m = max(A)\n sorted_A = []\n temp = 0\n d = [0]*(m+1)\n \n for i in range(len(A)):\n d[A[i]]+=1\n\n for x,y in enumerate(d):\n sorted_A += [x]*y \n return sorted_A ",
"_____no_output_____"
],
[
"A = [0,3,2,3,3,0,5,2,3]\nsort_counting(A)",
"_____no_output_____"
]
],
[
[
"Build an algorithm, based on your implementation of Counting Sort, that receives in input a list with all the letters of the alphabet (not in alphabetical order), and returns the list ordered according to alphabetical order",
"_____no_output_____"
],
[
"We continue the same approch as first part here and keep ",
"_____no_output_____"
]
],
[
[
"def sort_letters(B):\n B = list( ''.join(B).lower())\n m = len(B)\n d = []\n sorted_letters = []\n for i in B:\n d.append(lower.index(i)) \n c = s_counting(d)\n for j in c:\n sorted_letters += lower[j]\n return sorted_letters ",
"_____no_output_____"
],
[
"B = ['p' , 'w' , 'x', 'k' , 'p' , 'a' , 'c' ,'a' , 'b' , 'a' , 'c']\nsort_letters(B)",
"_____no_output_____"
]
],
[
[
"Build an algorithm, based on your implementation of Counting Sort, that receives in input a list of length m, that contains words with maximum length equal to n, and returns the list ordered according to alphabetical order.",
"_____no_output_____"
],
[
"Here is the first algurithim try to follow the counting sort algurithim order, is our first approch to do it.. ",
"_____no_output_____"
]
],
[
[
"Lower = lower.copy()\nLower.insert(0,'') \n\nC = ['words' , 'amount' , 'efficiently' , 'thumb' , 'rule' , 'solvable' , 'Another' ,'open' ,'problem', 'is' ,'whether']\nC = [a.lower() for a in C]\nm = max([len(e) for e in C])\nd = [[] for _ in C]\n\n\nfor i in range(len(C)):\n w2l = list(C[i])\n l2num = [Lower.index(a) for a in w2l ]\n d[i] = l2num + [0]*(m-len(l2num))\n\nsorted_d =[[] for _ in d] \nfor x in reversed(range(m)):\n count = [0]*len(Lower)\n cum_count = [0]*len(Lower)\n \n \n for e in range(len(d)):\n count[d[e][x]]+=1\n \n \n for r in range(len(count)):\n if count[r]>0:\n for g in range(len(d)):\n if r ==d[g][x]:\n sorted_d.append( d[g])\nfinal_sort = [] \nfor h in sorted_d[-len(d):]:\n final_sort.append( ''.join([Lower[a] for a in h]))\n \n \n \nprint(final_sort) ",
"['amount', 'another', 'efficiently', 'is', 'open', 'problem', 'rule', 'solvable', 'thumb', 'words', 'whether']\n"
]
],
[
[
"Here in this algorithm, we have some limitation due to the big numbers, We tried to fix the problem with normalizing the numbers between and 1000 however this will only could distinguish the words which are equal at 3 to 5 letters and after that, it would be really time-consuming to calculate and normally end up with errors",
"_____no_output_____"
]
],
[
[
"x = ['words' , 'amount' , 'are' , 'efficiently' , 'thumb' , 'rule' , 'solvable' , 'Another' ,'open' ,'problem', 'is' ,'whether']\n#x = ['asd', 'bedf','mog','zor','bze']\n#x = input('Enter the words, seperated by comma(,)').split(',')\nx_2 = x.copy()\n\n# Finding the longest word\nmax_length = 0\nfor i in x:\n if max_length < len(list(i)):\n max_length = len(list(i))\n \n\n# Turning words to numbers\nfor i in range(len(x)):\n x[i] = convert_w_2_n(x[i], max_length)\n \n\n#Normalize between 0 to 1000\n\nmx = max(x)\nmn = min(x)\nfor i,j in enumerate(x):\n x[i] = int((10**(3))*(j-mn)/(mx-mn))\n \n \n\nmax_int = max(x)\naux_array = [0]*(max_int+1)\naux_array_2 = aux_array\n\n\n# Step 1 : counting\nfor i in x:\n aux_array[i] = aux_array[i] + 1\n \n# Step 2 : comulating\naux_array_2[0] = aux_array[0]\nfor i in range(1,max_int+1):\n aux_array_2[i] = aux_array_2[i-1] + aux_array[i]\n \n# Sorting\nfinal_list = ['']*len(x)\nfor i in reversed(range(len(x))): #e.g: i = 2, 1, 0 \n final_list[aux_array_2[x[i]]-1] = x_2[i]\n aux_array_2[x[i]] = aux_array_2[x[i]] -1\n \n \n \nprint(final_list)",
"['Another', 'amount', 'are', 'efficiently', 'is', 'open', 'problem', 'rule', 'solvable', 'thumb', 'whether', 'words']\n"
]
],
[
[
"## Final version with best performance and without any limitation",
"_____no_output_____"
],
[
"Here we rewirte the count sorting algurithim again and we keep the order\nOrder is the origilanl list index wich if we use order list as a index we can soert it, it shows how the elements moves from where to wher to have the final arry",
"_____no_output_____"
]
],
[
[
"#sorting\n\ndef sort_counting(A): \n m = max(A)\n sorted_A = []\n temp = 0\n d = [0]*(m+1)\n \n for i in range(len(A)):\n d[A[i]]+=1\n \n cum_d =[0]*(m+1)\n for i in d:\n cum_d.append(i) \n \n order = list()\n for x,y in enumerate(d):\n sorted_A += [x]*y\n for i in range(y):\n order.append(A.index(x))\n A[order[-1]] = -1\n\n\n return sorted_A , order ",
"_____no_output_____"
]
],
[
[
"## Now we use the counting sort algruthin and it's order to make this work\nWe have used pure counting sort algurtim and keep the $big O$ time still in $O(n)$ \nHowerver the total time it use is $T(n) = kn+c$ ",
"_____no_output_____"
],
[
"the one is exatly same big O and principle as counting sort",
"_____no_output_____"
]
],
[
[
"Lower = lower.copy()\nLower.insert(0,'') \nimport numpy as np\n\nC = [ 'Alessio', 'Alessandro' , 'Angela', 'Alessand','Anita', 'Anna','Alessandrx', 'Arianna' ,'Alessandra']\nC = [a.lower() for a in C]\nm = max([len(e) for e in C])\nd = [[] for _ in C]\nfinal_sort = C.copy()\n\nfor i in range(len(C)):\n w2l = list(C[i])\n l2num = [Lower.index(a) for a in w2l ]\n d[i] = l2num + [0]*(m-len(l2num))\n\ndd = np.array(d) \nfor x in reversed(range(m)):\n count, order = sort_counting(list(dd[: ,x]))\n temp = [[] for _ in d]\n temp_sort = final_sort.copy()\n \n for i,j in enumerate(order):\n temp[i] += list(dd[j])\n \n final_sort[i] =temp_sort[j]\n \n dd = np.array(temp) \n \n \nfinal_sort ",
"_____no_output_____"
]
],
[
[
"# 3. Find similar wines!",
"_____no_output_____"
],
[
"#### Imports",
"_____no_output_____"
]
],
[
[
"import random\n\nimport pandas as pd\n\nimport numpy as np\n\nfrom collections import defaultdict\n\nimport matplotlib\n\nimport matplotlib.pyplot as plt",
"_____no_output_____"
]
],
[
[
"## Functions created for implementing the kMeans ",
"_____no_output_____"
],
[
"#### Create the Euclidean distance ",
"_____no_output_____"
]
],
[
[
"def distance_2(vec1, vec2):\n \n if len(vec1) == len(vec2):\n \n if len(vec1) > 1:\n \n add = 0\n \n for i in range(len(vec1)):\n \n add = add + (vec1[i] - vec2[i])**2\n \n return add**(1/2)\n \n else:\n \n return abs(vec1 - vec2)\n \n else:\n \n return \"Wrong Input\"\n ",
"_____no_output_____"
]
],
[
[
"#### We will now check the dissimilarity of our clusters. To do that we need to define the variability of every cluster. Meaning the sum of the distances of every element in the cluster from the mean(centroid). ",
"_____no_output_____"
]
],
[
[
"def dissimilarity(cluster):\n \n def kmeansreduce(centroid, dictionary):\n \n a = dictionary[centroid] \n \n if len(a) > 0 :\n \n vector = a[0]\n \n for i in range(1,len(a)):\n \n vector = np.add(vector, a[i])\n \n return vector\n \n else:\n \n pass\n \n var = []\n \n add = 0\n \n for i in range(len(cluster.keys())):\n \n if len(cluster[i]) > 0:\n \n m = kmeansreduce(i, cluster) / len(cluster[i])\n \n for j in range(len(cluster[i])):\n \n add = add + distance_2(m, cluster[i][j])\n \n return(add)",
"_____no_output_____"
]
],
[
[
"#### Compute the sum of the squared distance between data points and all centroids (distance_2). \n#### Assign each data point to the closest cluster (clusters dictionary).\n#### Compute the centroids for the clusters by taking the average of the all data points that belong to each cluster.(initial centroids)\n#### We define also two functions to show that this algorithm can be done by MapReduce method",
"_____no_output_____"
]
],
[
[
"def kmeans(data, k):\n \n def kmeansmap(information, num_centroids, centroids):\n \n clusters = defaultdict(list)\n \n for i in range(num_centroids):\n \n clusters[i] = []\n\n classes = defaultdict(list)\n \n for i in range(information.shape[0]): \n \n d = []\n \n for j in range(num_centroids):\n \n d.append(distance_2(information[i,], centroids[j]))\n \n clusters[np.argmin(d,axis=0)].append(information[i,])\n \n classes[i].append(np.argmin(d,axis=0))\n \n return [clusters, classes]\n \n \n def kmeansreduce(centroid, dictionary):\n \n a = dictionary[centroid] \n \n if len(a) > 0 :\n \n vector = a[0]\n \n for i in range(1,len(a)):\n \n vector = np.add(vector, a[i])\n \n return vector\n \n else:\n \n pass\n\n \n # ===================================================================================\n \n initial_centroids = random.sample(list(data), k)\n\n while True:\n \n dict1 = kmeansmap(data, k, initial_centroids)[0]\n \n dict2 = kmeansmap(data, k, initial_centroids)[1]\n \n dict3 = defaultdict(list)\n \n for i in range(k):\n \n dict3[i] = []\n \n old_clusters = initial_centroids\n \n for i in range(k):\n \n dict3[i] = kmeansreduce(i, dict1)\n \n if len(dict3[i]) > 0:\n \n initial_centroids[i] = dict3[i]/len(dict3[i]) \n \n if old_clusters == initial_centroids:\n \n break\n \n return [dict1, dict2]\n ",
"_____no_output_____"
]
],
[
[
"# Now we will implement the algorithms and functions to the data\n#### To implement the algorithms we will clean a bit the data",
"_____no_output_____"
]
],
[
[
"url = r\"C:\\Users\\HP\\Documents\\ADM\\HW 4\\wine.data\"\n\nheader = [\"Class\", \"Alcohol\", \"Malic acid\", \"Ash\",\"Alcalinity of ash\", \"Magnesium\", \"Total phenols\", \"Flavanoids\", \"Nonflavanoid phenols\", \n \"Proanthocyanins\", \"Color intensity\", \"Hue\", \"OD280/OD315 of diluted wines\", \"Proline\"]\n\ndata = pd.read_table(url, delimiter = \",\", names = header )\n\ndata.head(3)",
"_____no_output_____"
]
],
[
[
"#### We normalize the values of the DataFrame, so we can measure the distance\n\n#### Some columns are not saved as floats , so we will have an error normalizing them, so we make them floats and then normalize them",
"_____no_output_____"
]
],
[
[
"for col in data.columns[1:]:\n \n if data[col].dtype == 'int64':\n \n data[col] = data[col].astype(\"float64\")\n \nfor col in data.columns[1:]:\n \n r = (max(data[col]) - min(data[col]))\n \n minimum = min(data[col])\n \n for i in range(len(data[col])):\n \n data[col][i] = (float(data[col][i]) - minimum)/r\n \ndata.head(3)",
"C:\\Users\\HP\\Anaconda3\\lib\\site-packages\\ipykernel_launcher.py:15: SettingWithCopyWarning: \nA value is trying to be set on a copy of a slice from a DataFrame\n\nSee the caveats in the documentation: http://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy\n from ipykernel import kernelapp as app\n"
]
],
[
[
"#### We will not test the variable class, since this is the classification we target. So we are going to save it in a file called target and work with the other variables.",
"_____no_output_____"
]
],
[
[
"target = data[\"Class\"]\n\ndata = data.drop(columns = [\"Class\"])\n\ndata = data.to_numpy()\n\ndata",
"_____no_output_____"
]
],
[
[
"#### This way the elements of each row are to be taken as a vector",
"_____no_output_____"
]
],
[
[
"data[1,]",
"_____no_output_____"
]
],
[
[
"#### Now we will implement the kmeans algorithm, with an unknown number of clusters. We will use the elbow method to figure whats the best number of clusters for our data. We will run the method for up to k = 10 clusters",
"_____no_output_____"
]
],
[
[
"elbow = {}\n\nfor k in range(1, 11):\n \n best = kmeans(data, k)\n \n for t in range(100):\n \n C = kmeans(data, k)\n \n if dissimilarity(C[0]) < dissimilarity(best[0]):\n \n best = C\n \n elbow[k] = dissimilarity(best[0])",
"_____no_output_____"
],
[
"plt.plot(list(elbow.keys()), list(elbow.values()))",
"_____no_output_____"
]
],
[
[
"#### From the previous plot we can figure out what's the best k for me... We will implement the kmeans algorithm for the specific k",
"_____no_output_____"
]
],
[
[
"best = kmeans(data, 3)\n\nfor t in range(100):\n \n C = kmeans(data, 3)\n \n if dissimilarity(C[0]) < dissimilarity(best[0]):\n \n best = C\n \noutcome = []\n\nfor i in range(data.shape[0]):\n \n outcome.append(best[1][i][0] + 1)\n ",
"_____no_output_____"
]
],
[
[
"#### We did the following commands for all the columns, and we observed that two columns/features have a big effect on the clustering of the other features. Here we will show the distribution of the features, when plotted with Magnesium and Total Phenols",
"_____no_output_____"
]
],
[
[
"f, axes = plt.subplots(4,3,figsize=(20,20))\n\naxes[0][0].scatter(data[:, 5], data[:, 1], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[0][0].set_xlabel(header[1])\n\naxes[0][1].scatter(data[:, 5], data[:, 2], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[0][1].set_xlabel(header[2])\n\naxes[0][2].scatter(data[:, 5], data[:, 3], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[0][2].set_xlabel(header[3])\n\naxes[1][0].scatter(data[:, 5], data[:, 4], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[1][0].set_xlabel(header[4])\n\naxes[1][1].scatter(data[:, 5], data[:, 6], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[1][1].set_xlabel(header[6])\n\naxes[1][2].scatter(data[:, 5], data[:, 7], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[1][2].set_xlabel(header[7])\n\naxes[2][0].scatter(data[:, 5], data[:, 8], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[2][0].set_xlabel(header[8])\n\naxes[2][1].scatter(data[:, 5], data[:, 9], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[2][1].set_xlabel(header[9])\n\naxes[2][2].scatter(data[:, 5], data[:, 10], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[2][2].set_xlabel(header[10])\n\naxes[3][0].scatter(data[:, 5], data[:, 11], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[3][0].set_xlabel(header[11])\n\naxes[3][1].scatter(data[:, 5], data[:, 12], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[3][1].set_xlabel(header[12])\n\nplt.suptitle(header[5])\n\nplt.show()",
"_____no_output_____"
],
[
"f, axes = plt.subplots(4,3,figsize=(20,20))\n\naxes[0][0].scatter(data[:, 6], data[:, 1], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[0][0].set_xlabel(header[1])\n\naxes[0][1].scatter(data[:, 6], data[:, 2], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[0][1].set_xlabel(header[2])\n\naxes[0][2].scatter(data[:, 6], data[:, 3], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[0][2].set_xlabel(header[3])\n\naxes[1][0].scatter(data[:, 6], data[:, 4], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[1][0].set_xlabel(header[4])\n\naxes[1][1].scatter(data[:, 6], data[:, 5], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[1][1].set_xlabel(header[5])\n\naxes[1][2].scatter(data[:, 6], data[:, 7], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[1][2].set_xlabel(header[7])\n\naxes[2][0].scatter(data[:, 6], data[:, 8], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[2][0].set_xlabel(header[8])\n\naxes[2][1].scatter(data[:, 6], data[:, 9], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[2][1].set_xlabel(header[9])\n\naxes[2][2].scatter(data[:, 6], data[:, 10], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[2][2].set_xlabel(header[10])\n\naxes[3][0].scatter(data[:, 6], data[:, 11], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[3][0].set_xlabel(header[11])\n\naxes[3][1].scatter(data[:, 6], data[:, 12], c=outcome, cmap=matplotlib.colors.ListedColormap([\"purple\", \"blue\", \"red\"]))\naxes[3][1].set_xlabel(header[12])\n\nplt.suptitle(header[6])\n\nplt.show()",
"_____no_output_____"
]
],
[
[
"# 4. K-means can go wrong!",
"_____no_output_____"
],
[
"Clustring problem is non-linear over the $S \\in (R^{d})^{K}$ where $d$ is the number of features in the dataset and $K$ is the number clusters. This problem can turn to linear form with binary variables while the $\\hat{S} \\in S$ only cosists of the points in the dataset. This problem can be formulated as K-median problem that is in familiy of MILP (Mixed Integer Linear Programming)\nSince the K-median is MILP, algorithm like Branch & Bound can quarntee the global optimum over $\\hat{S}$. In K-median, each centroid is one of the point in the dataset and it minimize the summation of the distance to corresponding centroids. So if K-median find the solution $\\hat{X}$ with $Cost(\\hat{X})$ that is less than $Cost(X)$ where $X$ is the solution stem from K-means we can say that K-means fails to find the global optimum ($Global\\: optimum \\: \\leq Cost(\\hat{X})\\:< Cost(X)$ [Link](https://stats.stackexchange.com/questions/48757/why-doesnt-k-means-give-the-global-minimum). \n\n### K-median formulation\n\n$x_{i,j} = 1 \\: \\: \\text{if point i is in cluster} \\: c_j \\: \\text{that is} \\: \\: p_j \\: \\text{otherwise} \\: 0$\n\n$z_{j} = 1 \\: \\: \\text{if} \\: p_j \\: \\text{is a centroid otherwise} \\: 0$\n\n$d_{i,j} = \\text{squared distance of point} \\: i \\: \\text{and} \\: j$\n\n$Cost = \\sum_{i}\\sum_{j}{x_{i,j}d_{i,j}}$\n\nsubject to:\n\n$x_{i,j} \\leq z_{j} \\:\\:\\forall i,j$\n\n$\\sum_{j}{x_{i,j}} = 1 \\:\\:\\forall i$\n\n$\\sum_{j}{z_{j}} = K$",
"_____no_output_____"
],
[
"<img src=\"png/k-means-algorithm.png\">",
"_____no_output_____"
],
[
"The above example shows the True clusters and possible solution we can get from K-means with different seed value. However K-median yeilds to the centroids with a bit higher Cost, but it found the true clusters while K-means fails.",
"_____no_output_____"
],
[
"### Example - Exercise 4",
"_____no_output_____"
]
],
[
[
"import numpy as np\nimport matplotlib.pyplot as plt\nimport pandas as pd\nfrom sklearn.datasets import load_wine\nimport json\nfrom sklearn.cluster import KMeans\nfrom pandas.io.json import json_normalize\n\nwine = load_wine()\nwine.target[[10, 80, 140]]\n\nlist(wine.target_names)",
"_____no_output_____"
],
[
"wine_df = pd.DataFrame(wine.data , columns= wine.feature_names)\n\nwine_df['target'] = wine.target",
"_____no_output_____"
]
],
[
[
"The real classes of the wines",
"_____no_output_____"
]
],
[
[
"plt.scatter(wine_df['alcohol'] , wine_df['od280/od315_of_diluted_wines'] , c = wine_df['target'])\nplt.show()",
"_____no_output_____"
],
[
"kmean = KMeans(n_clusters= 3, init = 'random' ).fit(wine_df[['alcohol', 'od280/od315_of_diluted_wines']])\nplt.scatter(wine_df['alcohol'] , wine_df['od280/od315_of_diluted_wines'] , c = kmean.labels_)\nplt.scatter( kmean.cluster_centers_[: , 0], kmean.cluster_centers_[: , 1] , marker='X' , c = 'r')\nplt.title(label = '# of iretations: {}'.format(kmean.n_iter_ ) ,loc='center')\nplt.show()",
"_____no_output_____"
]
],
[
[
"We can see after choosing approximate points of initializations it would be faster to do the clustering",
"_____no_output_____"
]
],
[
[
"kmean = KMeans(n_clusters= 3, init = np.array([[12,3] ,[13,3] ,[13,1.7]]) ).fit(wine_df[['alcohol', 'od280/od315_of_diluted_wines']])\nplt.scatter(wine_df['alcohol'] , wine_df['od280/od315_of_diluted_wines'] , c = kmean.labels_)\nplt.scatter( kmean.cluster_centers_[: , 0], kmean.cluster_centers_[: , 1] , marker='X' , c = 'r')\nplt.title(label = '# of iretations: {}'.format(kmean.n_iter_ ) ,loc='center')\nplt.show()",
"C:\\ProgramData\\Anaconda3\\lib\\site-packages\\sklearn\\cluster\\k_means_.py:971: RuntimeWarning: Explicit initial center position passed: performing only one init in k-means instead of n_init=10\n return_n_iter=True)\n"
]
],
[
[
"## We can easily show how random initialization can effect on the number of iteration and the cost increases",
"_____no_output_____"
]
],
[
[
"for k in range(12):\n kmean = KMeans(n_clusters= 3, init = 'random' ).fit(wine_df[['alcohol', 'od280/od315_of_diluted_wines']])\n print('# of iretations: {}'.format(kmean.n_iter_ ) , 'And inertia: {}'.format(kmean.inertia_))",
"# of iretations: 7 And inertia: 58.32594553894382\n# of iretations: 10 And inertia: 58.32594553894382\n# of iretations: 9 And inertia: 58.32594553894382\n# of iretations: 10 And inertia: 58.32594553894382\n# of iretations: 8 And inertia: 58.32594553894382\n# of iretations: 8 And inertia: 58.32594553894382\n# of iretations: 8 And inertia: 58.32594553894382\n# of iretations: 6 And inertia: 58.32594553894382\n# of iretations: 12 And inertia: 58.32594553894382\n# of iretations: 8 And inertia: 58.32594553894382\n# of iretations: 6 And inertia: 58.32594553894382\n# of iretations: 4 And inertia: 58.32594553894382\n"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7eb6fbfcc8ea075293975fdcd1418576619f95d | 7,165 | ipynb | Jupyter Notebook | Python4Scientitsts/4 thu.pdf/17. Understanding Python errors.ipynb | kbroaders/F20-Chem-291 | 3297e1b37c52e1bff246b654eac97d8a064f7d7a | [
"CC-BY-4.0"
] | null | null | null | Python4Scientitsts/4 thu.pdf/17. Understanding Python errors.ipynb | kbroaders/F20-Chem-291 | 3297e1b37c52e1bff246b654eac97d8a064f7d7a | [
"CC-BY-4.0"
] | null | null | null | Python4Scientitsts/4 thu.pdf/17. Understanding Python errors.ipynb | kbroaders/F20-Chem-291 | 3297e1b37c52e1bff246b654eac97d8a064f7d7a | [
"CC-BY-4.0"
] | null | null | null | 27.557692 | 378 | 0.54515 | [
[
[
"empty"
]
]
] | [
"empty"
] | [
[
"empty"
]
] |
e7eb70a956b3b7f955eda9f757ae9410b7ac4447 | 678,426 | ipynb | Jupyter Notebook | ML_Code_30-11_LSTM.ipynb | stephen447/ML-group-project | f8d596afae008efc9d71e6300a3ce18d30f1d165 | [
"MIT"
] | null | null | null | ML_Code_30-11_LSTM.ipynb | stephen447/ML-group-project | f8d596afae008efc9d71e6300a3ce18d30f1d165 | [
"MIT"
] | null | null | null | ML_Code_30-11_LSTM.ipynb | stephen447/ML-group-project | f8d596afae008efc9d71e6300a3ce18d30f1d165 | [
"MIT"
] | null | null | null | 724.814103 | 78,708 | 0.948093 | [
[
[
"#General imports for Plotting and Data manipulation \nimport matplotlib.pyplot as plt\nimport pandas as pd\nimport datetime as dt\nimport urllib.request, json\nimport os\nimport numpy as np\n%matplotlib inline\nfrom matplotlib.ticker import MaxNLocator\nfrom IPython.display import clear_output\nfrom packaging import version\nfrom datetime import datetime, timedelta\n\n#Tensorflow imports\nimport tensorflow as tf \nfrom tensorflow.keras.models import Model, Sequential\nfrom tensorflow.keras.layers import LSTM, Dense, Dropout, Bidirectional, Flatten\nfrom tensorflow.keras.callbacks import ModelCheckpoint, TensorBoard\n\n#Sklearn imports\nfrom sklearn.preprocessing import MinMaxScaler\nfrom sklearn.model_selection import train_test_split, KFold\nfrom sklearn import metrics\n\n#Keras imports\nimport keras\nfrom keras import datasets\nfrom keras.layers import Dense, Flatten, Dropout, Activation\nfrom keras.layers import PReLU, LeakyReLU, Conv2D, MaxPool2D, Lambda\nfrom keras.regularizers import l2\nfrom keras.models import model_from_json",
"_____no_output_____"
],
[
"# Define some useful functions for plotting \nclass PlotLossAccuracy(keras.callbacks.Callback):\n def on_train_begin(self, logs={}):\n self.i = 0\n self.x = []\n self.acc = []\n self.losses = []\n self.val_losses = []\n self.val_acc = []\n self.logs = []\n\n def on_epoch_end(self, epoch, logs={}):\n \n self.logs.append(logs)\n self.x.append(int(self.i))\n self.losses.append(logs.get('loss'))\n self.val_losses.append(logs.get('val_loss'))\n self.acc.append(logs.get('acc'))\n self.val_acc.append(logs.get('val_acc'))\n \n self.i += 1\n \n clear_output(wait=True)\n plt.figure(figsize=(16, 6))\n plt.plot([1, 2])\n plt.subplot(121) \n plt.plot(self.x, self.losses, label=\"train loss\")\n plt.plot(self.x, self.val_losses, label=\"validation loss\")\n plt.gca().xaxis.set_major_locator(MaxNLocator(integer=True))\n plt.ylabel('loss')\n plt.xlabel('epoch')\n plt.title('Model Loss')\n plt.legend()\n plt.subplot(122) \n plt.plot(self.x, self.acc, label=\"training accuracy\")\n plt.plot(self.x, self.val_acc, label=\"validation accuracy\")\n plt.legend()\n plt.ylabel('accuracy')\n plt.xlabel('epoch')\n plt.title('Model Accuracy')\n plt.gca().xaxis.set_major_locator(MaxNLocator(integer=True))\n plt.show();\n",
"_____no_output_____"
],
[
"#Import Stock Data\ndata = pd.read_csv(r\"C:\\Users\\Harry\\Desktop\\College\\Year 5\\CS7CS4 - MACHINE LEARNING\\ISEQ_data.csv\")\nstock_open_prices = data['Open'].astype(np.float32)\nstock_dates = data['Date']\n\nstock_open_prices = np.array(stock_open_prices)\nstock_dates = np.array(stock_dates)\n\nstock = data.iloc[:, 1:2].values\n\n#Adding day of week Number as feature\ndata['Date'] = pd.to_datetime(data['Date'],format='%Y-%m-%d')\ndata['dayofweek_num'] = data['Date'].dt.dayofweek \ndata['DateIndex'] = np.arange(data.shape[0])\n\ndata.head(100)\n\ncl = data.iloc[:, 2].values\nscl = MinMaxScaler()\n#Reshape the data\ncl = cl.reshape(cl.shape[0],1)\ncl = scl.fit_transform(cl)",
"_____no_output_____"
],
[
"#Create a function to process the data into 7 day look back slices \ndef processData(data,lb):\n X,Y = [],[]\n for i in range(len(data)-lb-1):\n X.append(data[i:(i+lb),0])\n Y.append(data[(i+lb),0])\n return np.array(X),np.array(Y)\n\nprint(\"cl shape is: \", cl.shape)\n#Process the data into the 7 day look back slices\nX,y = processData(cl,50)\n\n#Split data into into training and testing and into axes (With 80:20 Split)\nX_train, X_test = train_test_split(X, test_size=0.2, shuffle=False)\ny_train, y_test = train_test_split(y, test_size=0.2, shuffle=False)\n\ncl2 = data['dayofweek_num']\ncl2 = cl2.values.astype('float32')\ncl2 = cl2.reshape(cl2.shape[0], 1)\nprint(\"cl2 shape is: \",cl2.shape)\n\nX1, y1 = processData(cl2,50)\n\nX_trainDN, X_testDN = train_test_split(X1, test_size=0.2, shuffle=False)\ny_trainDN, y_testDN = train_test_split(y1, test_size=0.2, shuffle=False)\n\n#X_train = X_train.reshape(X_train.shape[0])\n#X_test = X_test.reshape(X_test.shape[0])\n\nX_train = np.dstack((X_train, X_trainDN))\nX_test = np.dstack((X_test, X_testDN))\n\n\nprint(\"X train shape is\", X_train.shape)\nprint(\"X train DN shape is\", X_trainDN.shape)\n\nprint(\"\")\n\nprint(\"X test shape is\", X_test.shape)\nprint(\"X test DN shape is\", X_testDN.shape)\nprint(\"\")\n\nprint(\"y test shape is\",y_test.shape)",
"cl shape is: (468, 1)\ncl2 shape is: (468, 1)\nX train shape is (333, 50)\nX train DN shape is (333, 50)\n\nX test shape is (84, 50)\nX test DN shape is (84, 50)\n\ny test shape is (84,)\n"
],
[
"#visualise data\n\nfig, ax = plt.subplots(figsize=(10, 10))\n\n# Add x-axis and y-axis\nax.plot(stock_dates,\n data['Open'],\n color='darkblue')\n\n# Set title and labels for axes\nax.set(xlabel=\"Date\",\n ylabel=\"Opening Price\",\n title=\"ISEQ Daily Opening Price for 02/01/2020 - 29/10/2021\")\n\nax.set_ylim(ymin=0)\n\nplt.setp(ax.get_xticklabels(), rotation=45)\ntemp = ax.xaxis.get_ticklabels()\ntemp = list(set(temp) - set(temp[::50]))\nfor label in temp:\n label.set_visible(False)\n\nplt.show()",
"_____no_output_____"
],
[
"input_shape = (50, 1)\n\n#Now we create our LSTM model \nmodel = Sequential()\n\n#Add layers \nmodel.add(LSTM(units = 96, return_sequences = True, input_shape = (50, 1))) #n lags, n features\nmodel.add(Dropout(0.2))\nmodel.add(LSTM(units = 96, return_sequences = True))\nmodel.add(Dropout(0.2))\nmodel.add(LSTM(units = 96, return_sequences = True))\nmodel.add(Dropout(0.2))\nmodel.add(LSTM(units = 96))\nmodel.add(Dropout(0.2))\n\nmodel.add(Dense(1))\nmodel.compile(optimizer = 'adam', loss = 'mean_squared_error')\n\n#Reshape Train and test sets to fit correctly with LSTM \nX_train = X_train.reshape((X_train.shape[0],X_train.shape[1], 1))\nX_test = X_test.reshape((X_test.shape[0],X_test.shape[1], 1))\n\nmodel.summary()\nmodel.save_weights('model.h5')",
"Model: \"sequential_7\"\n_________________________________________________________________\nLayer (type) Output Shape Param # \n=================================================================\nlstm_28 (LSTM) (None, 50, 192) 148992 \n_________________________________________________________________\ndropout_28 (Dropout) (None, 50, 192) 0 \n_________________________________________________________________\nlstm_29 (LSTM) (None, 50, 192) 295680 \n_________________________________________________________________\ndropout_29 (Dropout) (None, 50, 192) 0 \n_________________________________________________________________\nlstm_30 (LSTM) (None, 50, 192) 295680 \n_________________________________________________________________\ndropout_30 (Dropout) (None, 50, 192) 0 \n_________________________________________________________________\nlstm_31 (LSTM) (None, 192) 295680 \n_________________________________________________________________\ndropout_31 (Dropout) (None, 192) 0 \n_________________________________________________________________\ndense_7 (Dense) (None, 1) 193 \n=================================================================\nTotal params: 1,036,225\nTrainable params: 1,036,225\nNon-trainable params: 0\n_________________________________________________________________\n"
],
[
"#Establish number of epochs and train model \nnum_epochs = 125\npltCallBack = PlotLossAccuracy()\nhistory = model.fit(X_train, y_train, validation_data=(X_test, y_test),\n epochs=num_epochs, callbacks=[PlotLossAccuracy()], batch_size=64, verbose=1,shuffle=False) \nprint(\"\\tModel Loss based on ISEQ stock data \\n\\tafter %i Epochs in training\" %num_epochs)",
"_____no_output_____"
],
[
"#We now visualise the training data and prediction\nXt = model.predict(X_test)\n\nplt.plot(scl.inverse_transform(y_test.reshape(-1,1)) , label = 'Test Data')\nprint(\"Prediction Shape is: \", Xt.shape)\n\nplt.plot(scl.inverse_transform(Xt), label = 'Prediction')\nprediction = scl.inverse_transform(Xt)\nplt.legend()\nplt.title('Prediction Vs Test Data')\nplt.show()",
"Prediction Shape is: (84, 1)\n"
],
[
"#We visualise the original data vs the Prediction of the final 20% of the stock data\nprediction_main = np.zeros([468, 1], dtype = float) \nfor i in range(0, 468):\n prediction_main[i] = None\n\nfor i in range(84, 168):\n prediction_main[i+300] = prediction[i - 84]\n\nfinal40NKE = np.zeros([84, 1], dtype = float) \nfor i in range(384, 468):\n final40NKE[i-384] = stock[i] \n \nprediction_main2 = np.zeros([84, 1], dtype = float) \nfor i in range(0, 84):\n prediction_main2[i] = prediction_main[i+384]\n\nfig, ax = plt.subplots(figsize=(12,9))\nplt.plot(stock, color = 'black', label = 'ISEQ Stock Price')\nplt.plot(prediction_main, color = 'green', label = 'Predicted Stock Price')\nplt.title('Stock Price Prediction after %i Epochs in training' %num_epochs)\nplt.xlabel('Time')\nplt.ylabel('ISEQ Stock Price')\nplt.legend()\nplt.show()\n\nfig, ax = plt.subplots(figsize=(12,9)) \nplt.plot(stock[375:468, :], color = 'black', label = 'ISEQ Stock Price (Final 20 %)')\nplt.plot(prediction_main2, color = 'green', label = 'Predicted Stock Price')\nplt.title('Stock Price Prediction on Final 20%')\nplt.xlabel('Time')\nplt.ylabel('ISEQ Stock Price')\nplt.legend()\n\nplt.show()\n",
"_____no_output_____"
],
[
"#Model Params\nprint(\"Model intercept (LSTM):\", model) # Printing model intercept\nprint()\nprint(\"Mean absolute error (LSTM): \\t\", metrics.mean_absolute_error(y_test, Xt)) # Printing Mean absolute error\nprint(\"Mean squared error (LSTM): \\t\", metrics.mean_squared_error(y_test, Xt)) # Printing mean squared error\nprint(\"R squared (LSTM): \\t\\t\", metrics.r2_score(y_test, Xt)) # Printing R Squared score",
"Model intercept (LSTM): <tensorflow.python.keras.engine.sequential.Sequential object at 0x00000241CCD2F5B0>\n\nMean absolute error (LSTM): \t 0.040431557667772004\nMean squared error (LSTM): \t 0.002155448945449976\nR squared (LSTM): \t\t 0.23496505483040597\n"
],
[
"#We now visualise the training data and prediction\nXt = model.predict(X_train)\n\nplt.plot(scl.inverse_transform(y_train.reshape(-1,1)) , label = 'Train Data')\nprint(\"Prediction Shape is: \", Xt.shape)\n\nplt.plot(scl.inverse_transform(Xt), label = 'Prediction on Test Data')\nprediction = scl.inverse_transform(Xt)\nplt.legend()\nplt.title('Training data prediction Vs Train Data')\nplt.show()",
"Prediction Shape is: (333, 1)\n"
],
[
"#We visualise the original data vs the Prediction of the final 20% of the stock data\nprediction_main = np.zeros([468, 1], dtype = float) \nfor i in range(0, 468):\n prediction_main[i] = None\n\nfor i in range(0, 333):\n prediction_main[i] = prediction[i]\n\nfinal40NKE = np.zeros([84, 1], dtype = float) \nfor i in range(384, 468):\n final40NKE[i-384] = stock[i] \n \nprediction_main2 = np.zeros([333, 1], dtype = float) \nfor i in range(0, 333):\n prediction_main2[i] = prediction_main[i]\n\nfig, ax = plt.subplots(figsize=(12,9))\nplt.plot(stock, color = 'black', label = 'Nike Stock Price')\nplt.plot(prediction_main, color = 'green', label = 'Predicted Stock Price')\nplt.title('Stock Price Prediction after %i Epochs in training' %num_epochs)\n#plt.title('Stock Price Prediction after 100 Epochs in training')\nplt.xlabel('Time')\nplt.ylabel('ISEQ Stock Price')\nplt.legend()\nplt.show()\n\nfig, ax = plt.subplots(figsize=(12,9)) \nplt.plot(stock[0:333, :], color = 'black', label = 'ISEQ Stock Price (First 80 %)')\nplt.plot(prediction_main2, color = 'green', label = 'Predicted Stock Price')\nplt.title('Stock Price Prediction on First 80%')\nplt.xlabel('Time')\nplt.ylabel('ISEQ Stock Price')\nplt.legend()\n\nplt.show()\n",
"_____no_output_____"
],
[
"#Model Params\nprint(\"Model intercept (LSTM):\", model) # Printing model intercept\nprint()\n#print(\"Model intercept (LSTM):\", metrics.accuracy_score(y_test, Xt)) # Printing R Squared score\nprint(\"Mean absolute error (LSTM): \\t\", metrics.mean_absolute_error(y_train, Xt)) # Printing Mean absolute error\nprint(\"Mean squared error (LSTM): \\t\", metrics.mean_squared_error(y_train, Xt)) # Printing mean squared error\nprint(\"R squared (LSTM): \\t\\t\", metrics.r2_score(y_train, Xt)) # Printing R Squared score",
"Model intercept (LSTM): <tensorflow.python.keras.engine.sequential.Sequential object at 0x00000241CCD2F5B0>\n\nMean absolute error (LSTM): \t 0.03571302622802288\nMean squared error (LSTM): \t 0.0020214749425989257\nR squared (LSTM): \t\t 0.9608498765513941\n"
],
[
"####################################\n## TESTING and Cross validation ##\n####################################",
"_____no_output_____"
],
[
"#Cross Validation ~For Batch size\n\nnum_epochs = 50\n\nmean_error=[]\nstd_error=[]\nmean_absolute_error=[]\nr_squared=[]\nbatchSize_range = [8, 16, 24, 32, 40, 48, 56, 64, 72, 80, 88, 96, 128, 160]\n\nfor batchSizeX in batchSize_range:\n pltCallBack = PlotLossAccuracy()\n #Model already established above \n #print(\"\\tModel Loss based on ISEQ stock data \\n\\tafter %i Epochs in training\" %num_epochs)\n temp=[]\n temp2=[]\n temp3=[]\n kf = KFold(n_splits=5)\n #Clear model before retraining \n model.load_weights('model.h5') \n history = model.fit(X_train, y_train, validation_data=(X_test, y_test),\n epochs=num_epochs, callbacks=[PlotLossAccuracy()], batch_size=batchSizeX, verbose=1,shuffle=False) \n print(\"\\tModel Loss based on ISEQ stock data \\n\\tbased on batch size %i \" %batchSizeX)\n ypred = model.predict(X_test)\n temp.append(metrics.mean_squared_error(y_test,ypred))\n temp2.append(metrics.mean_absolute_error(y_test,ypred))\n temp3.append(metrics.r2_score(y_test,ypred))\n mean_error.append(np.array(temp).mean())\n mean_absolute_error.append(np.array(temp2))\n r_squared.append(np.array(temp3))\n std_error.append(np.array(temp).std())",
"_____no_output_____"
],
[
"#Plot results of cross validation \nfig = plt.figure(figsize=(7, 7))\nplt.errorbar(batchSize_range, mean_error, yerr=std_error)\nplt.title('Mean square error Plot for Batch Size cross validation', fontsize=15, fontweight= 'bold')\nplt.xlabel('Batch Size', fontsize=15, fontweight= 'bold')\nplt.ylabel('Mean square error', fontsize=15, fontweight= 'bold')\nplt.xlim((7,161))\nplt.show()\n\nfig = plt.figure(figsize=(7, 7))\nplt.errorbar(batchSize_range, mean_absolute_error, yerr=std_error)\nplt.title('Mean absolute error Plot for Batch Size cross validation', fontsize=15, fontweight= 'bold')\nplt.xlabel('Batch Size', fontsize=15, fontweight= 'bold')\nplt.ylabel('Mean absolute error', fontsize=15, fontweight= 'bold')\nplt.xlim((7,161))\nplt.show()\n\nr_squared = scl.inverse_transform(r_squared)\nfig = plt.figure(figsize=(7, 7))\nplt.errorbar(batchSize_range, r_squared, yerr=std_error)\nplt.title('R Squared Plot for Batch Size cross validation', fontsize=15, fontweight= 'bold')\nplt.xlabel('Number of Epochs in training', fontsize=15, fontweight= 'bold')\nplt.ylabel('R Squared', fontsize=15, fontweight= 'bold')\nplt.xlim((7,161))\nplt.show()",
"_____no_output_____"
],
[
"#Cross Validation ~For Epochs\n\nnum_epochs = 25\n\nmean_error=[]\nstd_error=[]\nr_squared=[]\nmean_absolute_error=[]\npredictions=[]\nepochs_range = [5, 10, 15, 25, 35, 50, 60, 75, 90, 100, 115, 125, 150]\n\nfor Nepochs in epochs_range:\n pltCallBack = PlotLossAccuracy()\n #Model already established above \n temp=[]\n temp2=[]\n temp3=[]\n temp4=[]\n kf = KFold(n_splits=5)\n #Clear model before retraining \n model.load_weights('model.h5') \n history = model.fit(X_train, y_train, validation_data=(X_test, y_test),\n epochs=Nepochs, callbacks=[PlotLossAccuracy()], batch_size=24, verbose=1,shuffle=False) \n print(\"\\tModel Loss based on ISEQ stock data \\n\\tbased on %i Epochs\" %Nepochs)\n ypred = model.predict(X_test)\n temp4.append(ypred)\n temp.append(metrics.mean_squared_error(y_test,ypred))\n temp2.append(metrics.mean_absolute_error(y_test,ypred))\n temp3.append(metrics.r2_score(y_test,ypred))\n mean_error.append(np.array(temp).mean())\n mean_absolute_error.append(np.array(temp2).mean())\n r_squared.append(np.array(temp3))\n std_error.append(np.array(temp).std())\n predictions.append(np.array(temp4))\n",
"_____no_output_____"
],
[
"#Plot results of cross validation \nfig = plt.figure(figsize=(7, 7))\nplt.errorbar(epochs_range, mean_error, yerr=std_error)\nplt.title('Plot for number of Epochs cross validation', fontsize=15, fontweight= 'bold')\nplt.xlabel('Number of epochs in training', fontsize=15, fontweight= 'bold')\nplt.ylabel('Mean square error', fontsize=15, fontweight= 'bold')\nplt.xlim((4,160))\nplt.show()\n\nfig = plt.figure(figsize=(7, 7))\nplt.errorbar(epochs_range, mean_absolute_error, yerr=std_error)\nplt.title('Plot for number of Epochs cross validation', fontsize=15, fontweight= 'bold')\nplt.xlabel('Number of Epochs in training', fontsize=15, fontweight= 'bold')\nplt.ylabel('Mean absolute error', fontsize=15, fontweight= 'bold')\nplt.xlim((4,160))\nplt.show()\n\nfig = plt.figure(figsize=(7, 7))\nplt.errorbar(epochs_range, r_squared, yerr=std_error)\nplt.title('Plot for number of Epochs cross validation', fontsize=15, fontweight= 'bold')\nplt.xlabel('Number of Epochs in training', fontsize=15, fontweight= 'bold')\nplt.ylabel('R Squared', fontsize=15, fontweight= 'bold')\nplt.xlim((4,160))\nplt.show()",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7eb77d3ea00427b33bd27f31c9125450cb70392 | 5,584 | ipynb | Jupyter Notebook | colabs/smartsheet_report_to_bigquery.ipynb | quan/starthinker | 4e392415d77affd4a3d91165d1141ab38efd3b8b | [
"Apache-2.0"
] | null | null | null | colabs/smartsheet_report_to_bigquery.ipynb | quan/starthinker | 4e392415d77affd4a3d91165d1141ab38efd3b8b | [
"Apache-2.0"
] | null | null | null | colabs/smartsheet_report_to_bigquery.ipynb | quan/starthinker | 4e392415d77affd4a3d91165d1141ab38efd3b8b | [
"Apache-2.0"
] | null | null | null | 34.9 | 230 | 0.523102 | [
[
[
"#1. Install Dependencies\nFirst install the libraries needed to execute recipes, this only needs to be done once, then click play.\n",
"_____no_output_____"
]
],
[
[
"!pip install git+https://github.com/google/starthinker\n",
"_____no_output_____"
]
],
[
[
"#2. Get Cloud Project ID\nTo run this recipe [requires a Google Cloud Project](https://github.com/google/starthinker/blob/master/tutorials/cloud_project.md), this only needs to be done once, then click play.\n",
"_____no_output_____"
]
],
[
[
"CLOUD_PROJECT = 'PASTE PROJECT ID HERE'\n\nprint(\"Cloud Project Set To: %s\" % CLOUD_PROJECT)\n",
"_____no_output_____"
]
],
[
[
"#3. Get Client Credentials\nTo read and write to various endpoints requires [downloading client credentials](https://github.com/google/starthinker/blob/master/tutorials/cloud_client_installed.md), this only needs to be done once, then click play.\n",
"_____no_output_____"
]
],
[
[
"CLIENT_CREDENTIALS = 'PASTE CREDENTIALS HERE'\n\nprint(\"Client Credentials Set To: %s\" % CLIENT_CREDENTIALS)\n",
"_____no_output_____"
]
],
[
[
"#4. Enter SmartSheet Report To BigQuery Parameters\nMove report data into a BigQuery table.\n 1. Specify <a href='https://smartsheet-platform.github.io/api-docs/' target='_blank'>SmartSheet Report</a> token.\n 1. Locate the ID of a report by viewing its properties.\n 1. Provide a BigQuery dataset ( must exist ) and table to write the data into.\n 1. StarThinker will automatically map the correct schema.\nModify the values below for your use case, can be done multiple times, then click play.\n",
"_____no_output_____"
]
],
[
[
"FIELDS = {\n 'auth_read': 'user', # Credentials used for reading data.\n 'auth_write': 'service', # Credentials used for writing data.\n 'token': '', # Retrieve from SmartSheet account settings.\n 'report': '', # Retrieve from report properties.\n 'dataset': '', # Existing BigQuery dataset.\n 'table': '', # Table to create from this report.\n 'schema': '', # Schema provided in JSON list format or leave empty to auto detect.\n}\n\nprint(\"Parameters Set To: %s\" % FIELDS)\n",
"_____no_output_____"
]
],
[
[
"#5. Execute SmartSheet Report To BigQuery\nThis does NOT need to be modified unles you are changing the recipe, click play.\n",
"_____no_output_____"
]
],
[
[
"from starthinker.util.project import project\nfrom starthinker.script.parse import json_set_fields\n\nUSER_CREDENTIALS = '/content/user.json'\n\nTASKS = [\n {\n 'smartsheet': {\n 'auth': 'user',\n 'report': {'field': {'kind': 'string','name': 'report','order': 3,'description': 'Retrieve from report properties.'}},\n 'token': {'field': {'description': 'Retrieve from SmartSheet account settings.','name': 'token','order': 2,'default': '','kind': 'string'}},\n 'out': {\n 'bigquery': {\n 'auth': 'user',\n 'dataset': {'field': {'description': 'Existing BigQuery dataset.','name': 'dataset','order': 4,'default': '','kind': 'string'}},\n 'table': {'field': {'description': 'Table to create from this report.','name': 'table','order': 5,'default': '','kind': 'string'}},\n 'schema': {'field': {'kind': 'json','name': 'schema','order': 6,'description': 'Schema provided in JSON list format or leave empty to auto detect.'}}\n }\n }\n }\n }\n]\n\njson_set_fields(TASKS, FIELDS)\n\nproject.initialize(_recipe={ 'tasks':TASKS }, _project=CLOUD_PROJECT, _user=USER_CREDENTIALS, _client=CLIENT_CREDENTIALS, _verbose=True, _force=True)\nproject.execute(_force=True)\n",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7eb7b9dd9abd0dc7f511a18f40017c8b15f0d16 | 7,264 | ipynb | Jupyter Notebook | cross_analsysis.ipynb | robbierobinette/rcv-tensorflow | 984852902f465bb6f61ba863e4b76092249911d0 | [
"MIT"
] | null | null | null | cross_analsysis.ipynb | robbierobinette/rcv-tensorflow | 984852902f465bb6f61ba863e4b76092249911d0 | [
"MIT"
] | null | null | null | cross_analsysis.ipynb | robbierobinette/rcv-tensorflow | 984852902f465bb6f61ba863e4b76092249911d0 | [
"MIT"
] | null | null | null | 43.238095 | 1,770 | 0.572687 | [
[
[
"\nfrom NDPopulation import NDPopulation\nfrom ProcessResult import ProcessResult",
"_____no_output_____"
],
[
"import glob\nfrom ElectionConstructor import *\nimport pickle\nfrom NDPopulation import NDPopulation\n\ndef load_path(path: str) -> any:\n with open(path, \"rb\") as f:\n return pickle.load(f)\n\n\n\ndef generate_results(candidates: List[Candidate], processes: List[ElectionConstructor]) -> List[ElectionResult]:\n dim = candidates[0].ideology.dim\n population = NDPopulation(np.zeros((dim,)), np.ones((dim,)))\n voters = population.generate_unit_voters(1000)\n ballots = [Ballot(v, candidates, unit_election_config) for v in voters]\n return [p.run(ballots, set(candidates)).winner() for p in processes]\n\ndef compare_outcomes():\n irv_results: List[ProcessResult] = [load_path(s) for s in glob.glob(\"process_results-2/Hare*.p\")]\n\n irv_results.sort(key=lambda r: r.dim)\n processes = [\n ElectionConstructor(constructor=construct_irv, name=\"Hare\"),\n ElectionConstructor(constructor=construct_h2h, name=\"Minimax\")\n ]\n\n print(\"dimensionality agree disagree %agree\")\n for ir in irv_results:\n dim = ir.dim\n agree_count = 0\n disagree_count = 0\n for w, cc in ir.stats.results[0:1000]:\n o = generate_results(cc, processes)\n if o[0].name == o[1].name:\n agree_count += 1\n else:\n disagree_count += 1\n\n print(\"%d %4d %4d %5.2f%%\" % (dim, agree_count, disagree_count, 100 * agree_count / (agree_count + disagree_count)))\n\ncompare_outcomes()",
"dimensionality agree disagree %agree\n1 573 427 57.30%\n2 808 192 80.80%\n3 905 95 90.50%\n4 932 68 93.20%\n"
],
[
"def print_winner_stats():\n irv_results: List[ProcessResult] = [load_path(s) for s in glob.glob(\"process_results-2/Hare*.p\")]\n irv_results.sort(key=lambda r: r.dim)\n\n h2h_results: List[ProcessResult] = [load_path(s) for s in glob.glob(\"process_results-2/Minimax*.p\")]\n h2h_results.sort(key=lambda r: r.dim)\n\n def get_winner_distance(result: ProcessResult) -> np.array:\n v = [w.ideology.distance_from_o() for w, cc in result.stats.results]\n return np.array(v)\n\n print(\"Dimensionality Hare Minimax\")\n dim = 1\n for ir, hr in zip(irv_results, h2h_results):\n ird = get_winner_distance(ir)\n hd = get_winner_distance(hr)\n i_mean: float = np.mean(ird)\n h_mean: float = np.mean(hd)\n print(\"%d %5.2f %5.2f\" % (dim, i_mean, h_mean))\n dim += 1\n\nprint_winner_stats()",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code"
]
] |
e7eba6c91631064b055ac18017b5a9a082536ce5 | 419,899 | ipynb | Jupyter Notebook | Notebooks/define_TreeMaze_Zones.ipynb | alexgonzl/TreeMazeAnalyses | a834dc6b59beffe6bce59cdd9749b761fab3fe08 | [
"MIT"
] | null | null | null | Notebooks/define_TreeMaze_Zones.ipynb | alexgonzl/TreeMazeAnalyses | a834dc6b59beffe6bce59cdd9749b761fab3fe08 | [
"MIT"
] | null | null | null | Notebooks/define_TreeMaze_Zones.ipynb | alexgonzl/TreeMazeAnalyses | a834dc6b59beffe6bce59cdd9749b761fab3fe08 | [
"MIT"
] | null | null | null | 846.570565 | 77,476 | 0.951174 | [
[
[
"%matplotlib inline\n\nimport numpy as np\nfrom scipy import signal, ndimage, interpolate\nfrom scipy.interpolate import CubicSpline\n\nimport matplotlib.pyplot as plt\nimport matplotlib.ticker as ticker\nimport seaborn as sns\nfont = {'family' : 'sans-serif',\n 'size' : 20}\n\nplt.rc('font', **font)\n\nfrom pathlib import Path\nimport os,sys\nimport h5py\nimport sys \n\nfrom circus.shared.parser import CircusParser\nfrom circus.shared.files import load_data\nimport time\n\nimport nept\nsys.path.append('../PreProcessing/') \nfrom pre_process_neuralynx import *\nfrom shapely.geometry import Point\nfrom shapely.geometry.polygon import LinearRing, Polygon\nfrom collections import Counter\nfrom descartes import PolygonPatch",
"_____no_output_____"
],
[
"# path to tracking data\ndatPath = '/Users/alexgonzalez/Google Drive/PostDoc/Code/TreeMazeAnalyses/Lib/Resources/VT1.nvt'",
"_____no_output_____"
],
[
"t,x,y = get_position(datPath)\nx = signal.medfilt(x,5)\ny = signal.medfilt(y,5)\nb = signal.firwin(10, cutoff = 0.2, window = \"hanning\")\nx = signal.filtfilt(b,1,x)\ny = signal.filtfilt(b,1,y)\nx2=(y-278)/269*1308\ny2=(-x+530)/305*1358\n\nrad=0.03\nx3 = x2*np.cos(rad)+y2*np.sin(rad)\ny3 = -x2*np.sin(rad)+y2*np.cos(rad)",
"_____no_output_____"
],
[
"# plot and get a sense of the tilt\nf,a1=plt.subplots(1,1, figsize=(10,10))\nx2=y-275\ny2=-x+530\na1.plot(x2,y2)\na1.axis('equal')\na1.axvline(0,color='k')\na1.axvline(18,color='k')\na1.axvline(-52,color='k')\na1.axhline(300,color='k')\na1.axhline(308,color='k')\na1.grid()\na1.set_ylim(0,350)",
"_____no_output_____"
],
[
"# compute the angle of the tilt\ndeg=np.arctan(8/70)/np.pi*180\nrad = np.arctan(8/70)\nz = np.sqrt(8**2+70**2)\nprint(z,deg,rad)\n70/z",
"70.45565981523414 6.519801751656987 0.11379200714370806\n"
],
[
"# plot and get a sense of the tilt\nf,a1=plt.subplots(1,1, figsize=(10,10))\ndims = [1358,1308]\nx2=(y-278)/269*1308\ny2=(-x+530)/305*1358\n\nrad=0.03\nx3 = x2*np.cos(rad)+y2*np.sin(rad)\ny3 = -x2*np.sin(rad)+y2*np.cos(rad)\na1.plot(x3,y3)\na1.axis('equal')\n#a1.axvline(0,color='k')\n#a1.axvline(-40,color='k')\n\n#a1.axhline(280,color='k')\na1.axhline(80,color='k')\na1.axhline(1358,color='k')\na1.axvline(-585,color='k')\na1.axvline(545,color='k')\na1.plot([0,-600],[600,1358])\na1.grid()\n#a1.set_ylim(0,350)",
"_____no_output_____"
],
[
"def getPoly(coords):\n return Polygon(coords)\ndef plotPoly(poly,ax): \n p1x,p1y = poly.exterior.xy\n ax.plot(p1x, p1y, color='k', alpha=0.3,\n linewidth=3,)\n ring_patch = PolygonPatch(poly, fc='r', ec='none', alpha=0.2)\n ax.add_patch(ring_patch)\n #ax.fill(p1x, p1x, alpha=1, fc='r', ec='none')",
"_____no_output_____"
],
[
"MazeZonesCoords ={'Home':[(-300, -80), (-300, 50),(300,50),(300, -80)],\n 'Center': [(-80,500),(-95,400),(-150,400),(-150,645),\n (-75,550),(0,600),(75,550),(150,660),(150,400),(95,400),(80,500)],\n 'SegA': [(-150,50),(-80,500),(80,500),(150,50)],\n 'SegB': [(0,600),(0,700),(200,1000),(300,900),(75, 550)],\n 'SegC': [(330,1060),(520,1330),(520,800),(300,900)],\n 'SegD': [(200,1000),(50,1250),(520,1330),(330,1060)],\n 'SegE': [(0,600),(0,700),(-200,1000),(-330,900),(-75, 550)],\n 'SegF': [(-200,1000),(-50,1250),(-600,1300),(-360,1060)],\n 'SegG': [(-360,1060),(-600,1300),(-600,800),(-330,900)],\n \n 'G1': [(520,1330),(700,1200),(700,800),(520,800)],\n 'G2': [(50,1250),(50,1450),(400,1450),(520,1330)],\n 'G3': [(-50,1250),(-50,1450),(-400,1450),(-600,1300)],\n 'G4': [(-600,1300),(-750,1200),(-750,800),(-600,800)],\n \n 'I1': [(200,1000),(330,1060),(300,900)],\n 'I2': [(-330,900),(-360,1060),(-200,1000)],\n }\n# MazeZonesCoords ={'Home':[(-200, -60), (-200, 80),(200,80),(200, -60)],\n# 'Center': [(-100,500),(-200, 500), (-200, 600),(0,730),\n# (200, 600),(200,500),(100,500),(100,560),(-100,560)],\n# 'SegA': [(-100,80),(-100,560),(100,560),(100,80)],\n# 'SegB': [(0,730),(200,1000),(330,900),(200, 600)],\n# 'SegC': [(330,1060),(550,1200),(550,900),(330,900)],\n# 'SegD': [(200,1000),(100,1300),(400,1300),(330,1060)],\n# 'SegE': [(0,730),(-200,1000),(-330,900),(-200, 600)],\n# 'SegF': [(-200,1000),(-100,1300),(-400,1300),(-330,1060)],\n# 'SegG': [(-330,1060),(-600,1200),(-600,900),(-330,900)],\n \n# 'G1': [(550,1200),(700,1200),(700,800),(550,800)],\n# 'G2': [(50,1300),(50,1450),(400,1450),(400,1300)],\n# 'G3': [(-50,1300),(-50,1450),(-400,1450),(-400,1300)],\n# 'G4': [(-600,1200),(-750,1200),(-750,800),(-600,800)],\n \n# 'I1': [(200,1000),(330,1060),(400,1300),(550,1200),(330,1060),(330,900)],\n# 'I2': [(-330,900),(-330,1060),(-600,1200),(-400,1300),(-330,1060),(-200,1000)],\n# }\nMazeZonesGeom = {}\nfor zo in MazeZonesCoords.keys():\n MazeZonesGeom[zo] = getPoly(MazeZonesCoords[zo])\n\nf,a1=plt.subplots(1,1, figsize=(8,8))\na1.plot(x3,y3)\na1.grid() \n\nfor zo in MazeZonesGeom.keys():\n plotPoly(MazeZonesGeom[zo],a1)",
"_____no_output_____"
],
[
"ZoneOrd=['Home','SegA','Center','SegB','I1','SegC','G1','SegD','G2','SegE','I2','SegF','G3', 'SegG','G4']\nPosZones = []\nfor xp, yp in zip(x3,y3):\n cnt=0\n for zo in ZoneOrd:\n if MazeZonesGeom[zo].contains(Point(xp,yp)):\n PosZones.append(cnt)\n cnt+=1",
"_____no_output_____"
],
[
"f,a1=plt.subplots(1,1, figsize=(16,6))\na=Counter(PosZones)\nnames = list(a.keys())\nvalues = list(a.values())\na1.bar(names, values)\na1.set_yscale('log')\n#a1.set_yticks([100,200, 500, 1000])\na1.set_xticks(np.arange(len(ZoneOrd)))\na1.set_xticklabels(ZoneOrd)\na1.get_yaxis().set_major_formatter(ticker.ScalarFormatter())\na1.grid()\n\nfor tick in a1.get_xticklabels():\n tick.set_rotation(45)\n",
"_____no_output_____"
],
[
"ZoneOrd=['Home','SegA','Center','SegB','I1','SegC','G1','SegD','G2','SegE','I2','SegF','G3', 'SegG','G4']\n\nf,a1=plt.subplots(1,1, figsize=(10,6))\na1.plot(t-t[0],PosZones)\na1.set_yticks(np.arange(len(ZoneOrd)))\na1.set_yticklabels(ZoneOrd)\na1.grid()",
"_____no_output_____"
],
[
"unique(PosZones)\nZoneOrd=['Home','SegA','Center','SegB','I1','SegC','G1','SegD','G2','SegE','I2','SegF','G3', 'SegG','G4']\n",
"_____no_output_____"
],
[
"tp=np.arange(t[0],t[-1],0.02)\n# y_cs = CubicSpline(t, y3)\n# x_cs = CubicSpline(t, x3)\ny_ip = interpolate.interp1d(t, y3, kind=\"nearest\",fill_value=\"extrapolate\")\nx_ip = interpolate.interp1d(t, x3, kind=\"nearest\",fill_value=\"extrapolate\")\nyp = y_ip(tp)\nxp = x_ip(tp)\n\nf,a1=plt.subplots(1,1, figsize=(8,8))\na1.plot(x3,y3)\na1.plot(xp,yp)\n\na1.grid() \n\nfor zo in MazeZonesGeom.keys():\n plotPoly(MazeZonesGeom[zo],a1)",
"_____no_output_____"
],
[
"n = 10\nb = signal.firwin(n, cutoff = 0.5, window = \"blackman\")\nw,h = signal.freqz(b,1)\nh_dB = 20 * log10 (abs(h))\nplt.plot(w/max(w),h_dB)\n",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7eba8f0fae9616e74ec6031a2333295634e1a3d | 794,462 | ipynb | Jupyter Notebook | Day1_practice2.ipynb | andreYoo/Time-series-analysis-anomaly-detection | ba989b41fe588aa9ff95a7045dca5a7b2126d0bf | [
"MIT"
] | null | null | null | Day1_practice2.ipynb | andreYoo/Time-series-analysis-anomaly-detection | ba989b41fe588aa9ff95a7045dca5a7b2126d0bf | [
"MIT"
] | null | null | null | Day1_practice2.ipynb | andreYoo/Time-series-analysis-anomaly-detection | ba989b41fe588aa9ff95a7045dca5a7b2126d0bf | [
"MIT"
] | null | null | null | 1,021.159383 | 231,134 | 0.945809 | [
[
[
"<a href=\"https://colab.research.google.com/github/andreYoo/Time-series-analysis-anomaly-detection/blob/main/Day1_practice2.ipynb\" target=\"_parent\"><img src=\"https://colab.research.google.com/assets/colab-badge.svg\" alt=\"Open In Colab\"/></a>",
"_____no_output_____"
]
],
[
[
"import numpy as np\nimport pandas as pd\nimport seaborn as sns\nimport matplotlib.pyplot as plt\nimport plotly.graph_objs as go\nimport os\nimport warnings\n\nplt.style.use('ggplot')\n",
"_____no_output_____"
],
[
"# 구글 드라이브 마운트 \nfrom google.colab import drive\ndrive.mount('/content/drive')",
"Drive already mounted at /content/drive; to attempt to forcibly remount, call drive.mount(\"/content/drive\", force_remount=True).\n"
],
[
"weather_station_location = pd.read_csv(\"./drive/MyDrive/study/weather_station_locations.csv\")\nweather = pd.read_csv(\"./drive/MyDrive/study/summary_of_weather.csv\")\nweather_station_location = weather_station_location.loc[:,[\"WBAN\",\"NAME\",\"STATE/COUNTRY ID\",\"Latitude\",\"Longitude\"]]\nweather = weather.loc[:,[\"STA\",\"Date\",\"MeanTemp\"]]",
"/usr/local/lib/python3.7/dist-packages/IPython/core/interactiveshell.py:2718: DtypeWarning:\n\nColumns (7,8,18,25) have mixed types.Specify dtype option on import or set low_memory=False.\n\n"
],
[
"weather_station_id = weather_station_location[weather_station_location.NAME == \"BINDUKURI\"].WBAN\nweather_bin = weather[weather.STA == int(weather_station_id)]\nweather_bin[\"Date\"] = pd.to_datetime(weather_bin[\"Date\"])",
"/usr/local/lib/python3.7/dist-packages/ipykernel_launcher.py:3: SettingWithCopyWarning:\n\n\nA value is trying to be set on a copy of a slice from a DataFrame.\nTry using .loc[row_indexer,col_indexer] = value instead\n\nSee the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy\n\n"
],
[
"plt.figure(figsize=(22,8))\nplt.plot(weather_bin.Date,weather_bin.MeanTemp)\nplt.title(\"Mean Temperature of Bindukuri Area\")\nplt.xlabel(\"Date\")\nplt.ylabel(\"Mean Temperature\")\nplt.show()",
"_____no_output_____"
],
[
"# lets create time series from weather\ntimeSeries = weather_bin.loc[:, [\"Date\",\"MeanTemp\"]]\ntimeSeries.index = timeSeries.Date\nts = timeSeries.drop(\"Date\",axis=1)\nts",
"_____no_output_____"
],
[
"from statsmodels.tsa.seasonal import seasonal_decompose\nresult = seasonal_decompose(ts['MeanTemp'], model='additive', freq=7)\nfig = plt.figure()\nfig = result.plot()\nfig.set_size_inches(20,15)",
"_____no_output_____"
],
[
"import statsmodels.api as sm\nfig = plt.figure(figsize=(20,8))\nax1 = fig.add_subplot(211)\nfig = sm.graphics.tsa.plot_acf(ts, lags=20, ax=ax1)",
"_____no_output_____"
],
[
"ts_diff = ts - ts.shift()\nplt.figure(figsize=(22,8))\nplt.plot(ts_diff)\nplt.title(\"Differencing method\")\nplt.xlabel(\"Date\")\nplt.ylabel(\"Differencing Mean Temperature\")\nplt.show()",
"_____no_output_____"
],
[
"import statsmodels.api as sm\nfig = plt.figure(figsize=(20,8))\nax1 = fig.add_subplot(211)\nfig = sm.graphics.tsa.plot_acf(ts_diff[1:], lags=20, ax=ax1) #\nax2 = fig.add_subplot(212)\nfig = sm.graphics.tsa.plot_pacf(ts_diff[1:], lags=20, ax=ax2)# , lags=40",
"_____no_output_____"
],
[
"# fit model\nfrom statsmodels.tsa.arima_model import ARIMA\nfrom pandas import datetime\nmodel = ARIMA(ts, order=(2,1,2))\nmodel_fit = model.fit(disp=0)\n# predict\nstart_index = datetime(1944, 6, 25)\nend_index = datetime(1945, 5, 31)\nforecast = model_fit.predict(start=start_index, end=end_index,\ntyp='levels')\n# visualization\nplt.figure(figsize=(22,8))\nplt.plot(weather_bin.Date,weather_bin.MeanTemp,label = \"original\")\nplt.plot(forecast,label = \"predicted\")\nplt.title(\"Time Series Forecast\")\nplt.xlabel(\"Date\")\nplt.ylabel(\"Mean Temperature\")\nplt.legend()\nplt.show()",
"/usr/local/lib/python3.7/dist-packages/ipykernel_launcher.py:3: FutureWarning:\n\nThe pandas.datetime class is deprecated and will be removed from pandas in a future version. Import from datetime module instead.\n\n/usr/local/lib/python3.7/dist-packages/statsmodels/tsa/base/tsa_model.py:219: ValueWarning:\n\nA date index has been provided, but it has no associated frequency information and so will be ignored when e.g. forecasting.\n\n/usr/local/lib/python3.7/dist-packages/statsmodels/tsa/base/tsa_model.py:219: ValueWarning:\n\nA date index has been provided, but it has no associated frequency information and so will be ignored when e.g. forecasting.\n\n"
],
[
"resi = np.array(weather_bin[weather_bin.Date>=start_index].MeanTemp) - np.array(forecast)\nplt.figure(figsize=(22,8))\nplt.plot(weather_bin.Date[weather_bin.Date>=start_index],resi)\nplt.xlabel(\"Date\")\nplt.ylabel(\"Residual\")\nplt.legend()\nplt.show()",
"No handles with labels found to put in legend.\n"
],
[
"from sklearn import metrics\n\n\ndef scoring(y_true, y_pred):\n r2 = round(metrics.r2_score(y_true, y_pred) * 100, 3)\n # mae = round(metrics.mean_absolute_error(y_true, y_pred),3)\n corr = round(np.corrcoef(y_true, y_pred)[0, 1], 3)\n mape = round(\n metrics.mean_absolute_percentage_error(y_true, y_pred) * 100, 3)\n rmse = round(metrics.mean_squared_error(y_true, y_pred,squared=False), 3)\n df = pd.DataFrame({\n 'R2': r2,\n \"Corr\": corr,\n \"RMSE\": rmse,\n \"MAPE\": mape\n },index=[0])\n return df\n",
"_____no_output_____"
],
[
"\nscoring(np.array(weather_bin[weather_bin.Date>=start_index].MeanTemp),np.array(forecast))",
"_____no_output_____"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ebd0f03dcba71aef518b0a0f2ae3a62efcd582 | 15,409 | ipynb | Jupyter Notebook | Plotly_Express.ipynb | agahkarakuzu/datavis_edu | f56f27a3d2d1f181875976d8b53e01b90b3c1742 | [
"MIT"
] | 2 | 2020-05-27T20:21:07.000Z | 2021-05-03T18:36:07.000Z | Plotly_Express.ipynb | agahkarakuzu/datavis_edu | f56f27a3d2d1f181875976d8b53e01b90b3c1742 | [
"MIT"
] | null | null | null | Plotly_Express.ipynb | agahkarakuzu/datavis_edu | f56f27a3d2d1f181875976d8b53e01b90b3c1742 | [
"MIT"
] | 6 | 2019-09-25T12:21:46.000Z | 2021-05-03T18:40:11.000Z | 31.57582 | 303 | 0.591537 | [
[
[
"## Note\n\n### This notebook assumes that you are familiar with NumPy & Pandas. No worries if you are not! \n\nLike music & MRI? You can learn NumPy and SciPy as you are making music using MRI sounds: \n\nhttps://www.loom.com/share/4b08c4df903c40b397e87b2ec9de572d\n\nGitHub repo: https://github.com/agahkarakuzu/sunrise",
"_____no_output_____"
],
[
"## If you are using Plotly for the first time, lucky you! \n\n> `plotly.express` is to `plotly` what `seaborn` is to `matplotlib`\n\nIf you know what `seaborn` and `matplotlib` are, you won't need further explanation to understand what `plotly.express` has to offer. If you are not familiar with any of these, forget what I just said and focus on the examples. \n\nSee how you can create superb interactive figures with a single line of code! \n\nI assume that you are familiar with [tidy Pandas data frame](https://www.jeannicholashould.com/tidy-data-in-python.html). If you've never heard such a thing before, give it a quick read before proceeding, because this is the data format accepted by `plotly.express`. \n\n> Plotly Express supports a wide variety of charts, including otherwise verbose-to-create animations, facetted plots and multidimensional plots like Scatterplot Matrices (SPLOMs), Parallel Coordinates and Parallel Categories plots.",
"_____no_output_____"
]
],
[
[
"import plotly.express as px",
"_____no_output_____"
]
],
[
[
"In the older version of Plotly, at this stage, you had to `init_notebook_mode()` and tell plotly that you will be using it offline. Good news: \n\n* Now plotly can automatically detect which renderer to use! \n* Plus, you don't have to write extra code to tell Plotly you will be working offline. Plotly figures now have the ability to display themselves in the following contexts:\n * JupyterLab & classic Jupyter notebook\n * Other notebooks like Colab, nteract, Azure & Kaggle\n * IDEs and CLIs like VSCode, PyCharm, QtConsole & Spyder\n * Other contexts such as sphinx-gallery\n * Dash apps (with dash_core_components.Graph())\n * Static raster and vector files (with fig.write_image())\n * Standalone interactive HTML files (with fig.write_html())\n * Embedded into any website (with fig.to_json() and Plotly.js)",
"_____no_output_____"
],
[
"Now lets import the famous `iris` dataset, which comes with `plotly.express` and display it. \n\n\nHint: Plotly 4.0 supports tab completion as well! Type `px.` then hit the tab from your keyboard. Available methods and attributes will appear in a dropdown list. ",
"_____no_output_____"
]
],
[
[
"# Read iris data into the variable named iris \niris = px.data.iris()\n\n# Display first last 5 rows of the dataframe \niris.tail()",
"_____no_output_____"
]
],
[
[
"## Create scatter plots\n\nAs you see, `iris` dataset has 6 columns, each having their own label. Now let's take a look at how `sepal_width` is corralated with `sepal_length`. ",
"_____no_output_____"
]
],
[
[
"fig = px.scatter(iris, x=\"sepal_width\", y=\"sepal_length\")\nfig.show()",
"_____no_output_____"
]
],
[
[
"Yes, that easy! 🎉\n\nYou can change the column indexes to observe other correlations such as `petal_length` and `petal_height`. What if you were also able to color markers with respect to the `species` category? Well, all it takes is to pass another argument :) ",
"_____no_output_____"
]
],
[
[
"fig = px.scatter(iris, x=\"sepal_width\", y=\"sepal_length\",color='species')\nfig.show()",
"_____no_output_____"
]
],
[
[
"💬**Scatter plots are not enough! I want my histograms displayed on their respective axes.** \n\n👏Plotly express got you covered. ",
"_____no_output_____"
]
],
[
[
"fig = px.scatter(iris, x=\"sepal_width\", y=\"sepal_length\", color=\"species\", marginal_y=\"rug\", marginal_x=\"histogram\")\nfig.show()",
"_____no_output_____"
]
],
[
[
"🙄Of course scatter plots need their best fit line. \n\nAnd why not show `boxplots` or `violinpots` instead of histograms and rug lines? 🚀",
"_____no_output_____"
]
],
[
[
"fig = px.scatter(iris, x=\"sepal_width\", y=\"sepal_length\", color=\"species\", marginal_y=\"violin\",\n marginal_x=\"box\", trendline=\"ols\")\nfig.show()",
"_____no_output_____"
]
],
[
[
"- What is better than a scatter plot? \n> A scatter plot matrix! 🤯\n\nYou can explore cross-filtering ability of SPLOM charts in plotly. Hover your cursor over a point cloud in one of the panels, and select a poriton of them by left click + dragging. Selected data points will be highlighted in the remaining sub-panels! Double click to reset.",
"_____no_output_____"
]
],
[
[
"fig = px.scatter_matrix(iris, dimensions=[\"sepal_width\", \"sepal_length\", \"petal_width\", \"petal_length\"], color=\"species\")\nfig.show()",
"_____no_output_____"
]
],
[
[
"## Remember parallel sets? Let's create one\n\nIn [the presentation](https://zenodo.org/record/3841775#.XsqgFJ5Kg1I), we saw that parallel set can be useful for visualization of proportions if there are more than two grouping variables are present. \n\nIn this example, we will be working with the `tips` dataset, which has five grouping conditions: `sex`, `smoker`, `day`, `time`, `size`. Each of these will represent a column, and each column will be split into number of pieces equal to the unique entries listed in the corresponding category. \n\nEach row represents a restaurant bill.",
"_____no_output_____"
]
],
[
[
"tips = px.data.tips()\ntips.tail()",
"_____no_output_____"
],
[
"# Hint: You can change colorscale. Type px.colors.sequential. then hit tab :) \nfig = px.parallel_categories(tips, color=\"total_bill\", dimensions=['sex','smoker','day','time','size'], color_continuous_scale='viridis',template='plotly_dark')\nfig.show()",
"_____no_output_____"
]
],
[
[
"### Sunburst chart & Treemap\n**Data:** A `pandas.DataFrame` with 1704 rows and the following columns:\n\n`['country', 'continent', 'year', 'lifeExp', 'pop', 'gdpPercap',iso_alpha', 'iso_num']`.",
"_____no_output_____"
]
],
[
[
"df = px.data.gapminder().query(\"year == 2007\")\nfig = px.sunburst(df, path=['continent', 'country'], values='pop',\n color='lifeExp', hover_data=['iso_alpha'],color_continuous_scale='viridis',template='plotly_white')\nfig.show()",
"_____no_output_____"
]
],
[
[
"## Polar coordinates\n**Data**: Level of wind intensity in a cardinal direction, and its frequency.\n\n- Scatter polar\n- Line polar \n- Bar polar",
"_____no_output_____"
]
],
[
[
"df = px.data.wind()\nfig = px.scatter_polar(df, r=\"frequency\", theta=\"direction\", color=\"strength\", symbol=\"strength\",\n color_discrete_sequence=px.colors.sequential.Plasma_r, template='plotly_dark')\nfig.show()",
"_____no_output_____"
]
],
[
[
"## Ternary plot\n**Data:** Results for an electoral district in the 2013 Montreal mayoral election.",
"_____no_output_____"
]
],
[
[
"df = px.data.election()\nfig = px.scatter_ternary(df, a=\"Joly\", b=\"Coderre\", c=\"Bergeron\", color=\"winner\", size=\"total\", hover_name=\"district\",\n size_max=15, color_discrete_map = {\"Joly\": \"blue\", \"Bergeron\": \"green\", \"Coderre\":\"red\"}, template=\"plotly_dark\" )\nfig.show()",
"_____no_output_____"
]
],
[
[
"## See all available `px` charts, attributes and more \n\nPlotly express gives you the liberty to change visual attributes of the plots as you like! There are many other charts made available out of the box, all can be plotted with a single line of code. \n\n### Here is the [complete reference documentation](https://www.plotly.express/plotly_express/) for `plotly.express`.",
"_____no_output_____"
],
[
"## Saving the best for the last \n\nRemember I said \n\n> including otherwise verbose-to-create animations\n\nat the beginning of this notebook? Show time! \n\nLets load `gapminder` dataset and observe the relationship between life expectancy and gdp per capita from 1952 to 2017 for five continents. ",
"_____no_output_____"
]
],
[
[
"gapminder = px.data.gapminder()\ngapminder.tail()",
"_____no_output_____"
],
[
"fig = px.scatter(gapminder, x=\"gdpPercap\", y=\"lifeExp\", animation_frame=\"year\", animation_group=\"country\",\n size=\"pop\", color=\"continent\", hover_name=\"country\", facet_col=\"continent\",\n log_x=True, size_max=45, range_x=[100,100000], range_y=[25,90])\nfig.show()",
"_____no_output_____"
]
],
[
[
"👽I know you like dark themes. ",
"_____no_output_____"
]
],
[
[
"# See the last argument (template) I passed to the function. To see other alternatives \n# visit https://plot.ly/python/templates/ \n\nfig = px.scatter(gapminder, x=\"gdpPercap\", y=\"lifeExp\", animation_frame=\"year\", animation_group=\"country\",\n size=\"pop\", color=\"continent\", hover_name=\"country\", facet_col=\"continent\",\n log_x=True, size_max=45, range_x=[100,100000], range_y=[25,90], template=\"plotly_dark\")\nfig.show()",
"_____no_output_____"
]
],
[
[
"# Let's work with our own data\n\nWe will load raw MRI data (K-Space), which is saved in `ISMRM-RD` format. ",
"_____no_output_____"
]
],
[
[
"from ismrmrd import Dataset as read_ismrmrd\nfrom ismrmrd.xsd import CreateFromDocument as parse_ismrmd_header\nimport numpy as np\n# Here, we are just loading a 3D data into a numpy matrix, so that we can use plotly with it! \ndset = read_ismrmrd('Kspace/sub-ismrm_ses-sunrise_acq-chord1.h5', 'dataset')\nheader = parse_ismrmd_header(dset.read_xml_header())\nnX = header.encoding[0].encodedSpace.matrixSize.x\nnY = header.encoding[0].encodedSpace.matrixSize.y\nnZ = header.encoding[0].encodedSpace.matrixSize.z\nnCoils = header.acquisitionSystemInformation.receiverChannels\nraw = np.zeros((nCoils, nX, nY), dtype=np.complex64)\nfor tr in range(nY):\n raw[:,:,tr] = dset.read_acquisition(tr).data",
"_____no_output_____"
]
],
[
[
"## 100X100 matrix, 16 receive channels",
"_____no_output_____"
]
],
[
[
"raw.shape",
"_____no_output_____"
],
[
"fig = px.imshow(raw.real,color_continuous_scale='viridis',facet_col=0,facet_col_wrap=4,template='plotly_dark')\nfig.update_layout(title='Channel Raw')",
"_____no_output_____"
]
],
[
[
"## Simple image reconstruction",
"_____no_output_____"
]
],
[
[
"from scipy.fft import fft2, fftshift\nfrom scipy import ndimage\nim = np.zeros(raw.shape)\n# Let's apply some ellipsoid filter. \nraw = ndimage.fourier_ellipsoid(fftshift(raw),size=2)\n#raw = ndimage.fourier_ellipsoid(raw,size=2)\nfor ch in range(nCoils):\n # Comment in and see what it gives \n im[ch,:,:] = abs(fftshift(fft2(raw[ch,:,:])))\n # Normalize \n im[ch,:,:] /= im[ch,:,:].max()",
"_____no_output_____"
],
[
"fig = px.imshow(im,color_continuous_scale='viridis', animation_frame=0,template='plotly_dark')\nfig.update_layout(title='Channel Recon').show()",
"_____no_output_____"
]
],
[
[
"## SAVE HTML OUTPUT\n* This is the file under the `.docs` directory, from which a `GitHub page` is served:\n\n",
"_____no_output_____"
]
],
[
[
"fig.write_html('multichannel.html')",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7ebd6ebeedb2ea67ec2aa34f12a3bc1af125c4e | 6,662 | ipynb | Jupyter Notebook | ed_breadth_first_search.ipynb | devkosal/code_challenges | 0591c4839555376a231682db7cc12c8a70515b09 | [
"MIT"
] | null | null | null | ed_breadth_first_search.ipynb | devkosal/code_challenges | 0591c4839555376a231682db7cc12c8a70515b09 | [
"MIT"
] | null | null | null | ed_breadth_first_search.ipynb | devkosal/code_challenges | 0591c4839555376a231682db7cc12c8a70515b09 | [
"MIT"
] | null | null | null | 22.659864 | 188 | 0.439508 | [
[
[
"class TreeNode:\n def __init__(self, val):\n self.val = val\n self.left, self.right = None, None",
"_____no_output_____"
],
[
"root = TreeNode(12)\nroot.left = TreeNode(7)\nroot.right = TreeNode(1)\nroot.left.left = TreeNode(9)\nroot.right.left = TreeNode(10)\nroot.right.right = TreeNode(5)",
"_____no_output_____"
]
],
[
[
"### Binary Tree Level Order Traversal (easy)",
"_____no_output_____"
],
[
"Given a binary tree, populate an array to represent its level-by-level traversal. You should populate the values of all nodes of each level from left to right in separate sub-arrays.",
"_____no_output_____"
]
],
[
[
"def get_depth(root):\n def helper(root, i):\n if not root.left and not root.right:\n return i\n r,l = 0,0\n if root.left:\n l = helper(root.left, i+1)\n if root.right:\n r = helper(root.right, i+1)\n return max(l,r)\n return helper(root, 0)\n\ndef traverse(root):\n res = [[] for _ in range(get_depth(root)+1)]\n def helper(root, i):\n res[i].append(root.val)\n if root.left:\n helper(root.left, i+1)\n if root.right:\n helper(root.right, i+1)\n helper(root, 0)\n return res",
"_____no_output_____"
],
[
"get_depth(root)\ntraverse(root)",
"_____no_output_____"
],
[
"from collections import deque\ndef traverse(root):\n res = []\n q = deque([root])\n while q:\n num_vals = len(q)\n level_res = []\n for _ in range(num_vals):\n r = q.popleft()\n level_res.append(r.val)\n if r.left:\n q.append(r.left)\n if r.right:\n q.append(r.right)\n res.append(level_res)\n return res\n \n ",
"_____no_output_____"
],
[
"traverse(root)",
"_____no_output_____"
],
[
"def get_depth(root):\n q = deque([root])\n depth = 0\n while q:\n has_next_level = False\n num_items = len(q)\n for _ in range(num_items):\n r = q.popleft()\n if r.right:\n q.append(r.right)\n if r.left:\n q.append(r.left)\n depth += 1\n return depth\nget_depth(root)",
"_____no_output_____"
]
],
[
[
"### Reverse level order traversal",
"_____no_output_____"
]
],
[
[
"def reverse_traverse(root):\n q = deque([root])\n res = deque()\n while q:\n num_items = len(q)\n level_res = deque()\n for _ in range(num_items):\n r = q.popleft()\n level_res.append(r.val)\n if r.left:\n q.append(r.left)\n if r.right:\n q.append(r.right)\n res.appendleft(level_res)\n return res",
"_____no_output_____"
],
[
"reverse_traverse(root)",
"_____no_output_____"
]
],
[
[
"### zigzag order traversal",
"_____no_output_____"
]
],
[
[
"def zig_traverse(root):\n q = deque([root])\n res = deque()\n while q:\n num_items = len(q)\n level_res = deque()\n for _ in range(num_items):\n r = q.popleft()\n level_res.append(r.val)\n if r.left:\n q.append(r.left)\n if r.right:\n q.append(r.right)\n res.appendleft(level_res)\n return res",
"_____no_output_____"
]
],
[
[
"### Connect All Level Order Siblings (medium) -- Problem 1",
"_____no_output_____"
]
],
[
[
"def connect_level_order(tree):\n pass",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7ebe0bce2f32e345eced5de8efb2e24cfdd29a9 | 5,614 | ipynb | Jupyter Notebook | examples/plot_spectra.ipynb | ickc/TAIL | 3b4e49d6d5a4c2b57f7de5cbfcb2441e405efbeb | [
"BSD-3-Clause"
] | 1 | 2020-12-10T22:58:02.000Z | 2020-12-10T22:58:02.000Z | examples/plot_spectra.ipynb | ickc/TAIL | 3b4e49d6d5a4c2b57f7de5cbfcb2441e405efbeb | [
"BSD-3-Clause"
] | 1 | 2017-04-24T09:31:29.000Z | 2017-04-24T09:31:29.000Z | examples/plot_spectra.ipynb | ickc/TAIL | 3b4e49d6d5a4c2b57f7de5cbfcb2441e405efbeb | [
"BSD-3-Clause"
] | null | null | null | 18.467105 | 104 | 0.518703 | [
[
[
"Plotting final spectra with MC error-bar (without MCMC error-bar)",
"_____no_output_____"
]
],
[
[
"from pathlib import Path",
"_____no_output_____"
],
[
"import numpy as np\nimport pandas as pd",
"_____no_output_____"
],
[
"import matplotlib.pyplot as plt\nimport seaborn as sns",
"_____no_output_____"
],
[
"%matplotlib inline",
"_____no_output_____"
],
[
"import plotly.offline as py",
"_____no_output_____"
],
[
"import plotly.express as px",
"_____no_output_____"
],
[
"%load_ext autoreload\n%autoreload 2",
"_____no_output_____"
],
[
"%time from tail.analysis.container import Spectra, FSky, NullSpectra",
"_____no_output_____"
],
[
"basedir = Path('/scratch/largepatch_new')\npath = basedir / 'high_1_0_2.hdf5'\npath_cache = basedir / 'high_1_0_2_spectra_final.hdf5'\npath_mask = basedir / 'masks.hdf5'",
"_____no_output_____"
],
[
"save = False",
"_____no_output_____"
],
[
"# select spectra\nbin_width = 100",
"_____no_output_____"
],
[
"# narrow l-range for science\nl_min = 600\nl_max = 3000",
"_____no_output_____"
],
[
"ev_est = 'signal'\nddof = None",
"_____no_output_____"
],
[
"f_sky = FSky.load(path_mask)",
"_____no_output_____"
],
[
"%time spectra = Spectra.load(path_cache, bin_width, full=True, path_theory_spectra=path)",
"_____no_output_____"
],
[
"spectra.ev_est, spectra.ddof",
"_____no_output_____"
],
[
"spectra.ev_est = ev_est\nspectra.ddof = ddof",
"_____no_output_____"
],
[
"%time spectra = spectra.slicing_l(l_min, l_max, ascontiguousarray=True)",
"_____no_output_____"
],
[
"spectra.pte_to_frame",
"_____no_output_____"
]
],
[
[
"# Plotting code",
"_____no_output_____"
]
],
[
[
"fig = spectra.plot_spectra(subtract_leakage=True)",
"_____no_output_____"
],
[
"if save:\n Path('media').mkdir(exist_ok=True)\n py.plot(fig, filename='media/spectra.html', include_plotlyjs='cdn', include_mathjax='cdn')",
"_____no_output_____"
],
[
"fig",
"_____no_output_____"
],
[
"fig.update_layout(xaxis_type=\"log\", yaxis_type=\"log\")",
"_____no_output_____"
],
[
"if save:\n py.plot(fig, filename='media/spectra-log.html', include_plotlyjs='cdn', include_mathjax='cdn')",
"_____no_output_____"
]
],
[
[
"# Analytical Error-bar",
"_____no_output_____"
]
],
[
[
"err_analytic = spectra.err_analytic(f_sky)",
"_____no_output_____"
],
[
"df_err = spectra.to_frame_4d(err_analytic)",
"_____no_output_____"
],
[
"df_err.T",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
]
] |
e7ebfeab62866086f0d0899f2cb7ee39aa3bd239 | 13,281 | ipynb | Jupyter Notebook | vk_NLP - TF IDF.ipynb | vitthalkcontact/NLP | 97ecb4fe31bb43b97010e7ff7c503d833d816ce5 | [
"Unlicense"
] | 1 | 2020-10-14T14:18:55.000Z | 2020-10-14T14:18:55.000Z | vk_NLP - TF IDF.ipynb | vitthalkcontact/NLP | 97ecb4fe31bb43b97010e7ff7c503d833d816ce5 | [
"Unlicense"
] | null | null | null | vk_NLP - TF IDF.ipynb | vitthalkcontact/NLP | 97ecb4fe31bb43b97010e7ff7c503d833d816ce5 | [
"Unlicense"
] | null | null | null | 30.461009 | 132 | 0.422634 | [
[
[
"# TF-IDF",
"_____no_output_____"
],
[
"### Draw back of Bag of Words",
"_____no_output_____"
]
],
[
[
"# All the words have given same importance\n# No Semantic information preserved\n# For above two problems TF-IDF model is the solution",
"_____no_output_____"
]
],
[
[
"# Steps in TF-IDF",
"_____no_output_____"
]
],
[
[
"# 1. Lower case the corpus or paragraph.\n# 2. Tokenization.\n# 3. TF: Term Frequency, IDF: Inverse Document Frequency, TF-IDF = TF*log(IDF).\n# 4. TF = No. of occurance of a word in a document / No. of words in that document.\n# 5. IDF = log(No. of documents/No. of documents containing the word)\n# 6. TFIDF(word) = TF(Document, word) * IDF (word)",
"_____no_output_____"
],
[
"import nltk",
"_____no_output_____"
],
[
"nltk.download()",
"showing info https://raw.githubusercontent.com/nltk/nltk_data/gh-pages/index.xml\n"
],
[
"paragraph = '''In a country like India with a galloping population, unfortunately nobody is paying attention to the issue \nof population. Political parties are feeling shy, politicians are feeling shy, Parliament also does not adequately discuss \nabout the issue,” said Naidu while addressing the 58th convocation of Indian Agricultural Research Institute (IARI).\n\nHe said, “You know how population is growing, creating problems. See the problems in Delhi, traffic, more human beings, \nmore vehicles, more tension, less attention. If you have tension you cannot pay attention.” \nEmphasising on the need to increase food production to meet demand of growing population, Naidu said, \n“In future if population increases like this, and you are not able to adequately match it with increase in production, \nthere will be problem'''",
"_____no_output_____"
],
[
"# Cleaning the Text",
"_____no_output_____"
],
[
"import re\nfrom nltk.corpus import stopwords\nfrom nltk.stem.porter import PorterStemmer\nfrom nltk.stem import WordNetLemmatizer",
"_____no_output_____"
],
[
"ps = PorterStemmer()",
"_____no_output_____"
],
[
"wordnet = WordNetLemmatizer()",
"_____no_output_____"
],
[
"sentences = nltk.sent_tokenize(paragraph)",
"_____no_output_____"
],
[
"# sentences",
"_____no_output_____"
],
[
"corpus = []",
"_____no_output_____"
],
[
"for i in range(len(sentences)):\n review = re.sub(\"[^a-zA-Z]\", ' ', sentences[i])\n review = review.lower()\n review = review.split()\n review = [wordnet.lemmatize(word) for word in review if word not in set(stopwords.words('english'))]\n review = ' '.join(review)\n corpus.append(review)",
"_____no_output_____"
],
[
"# Creatung the TF-IDF Model",
"_____no_output_____"
],
[
"# # Creating the TF-IDF model\n# from sklearn.feature_extraction.text import TfidfVectorizer\n# cv = TfidfVectorizer()\n# X = cv.fit_transform(corpus).toarray()",
"_____no_output_____"
],
[
"from sklearn.feature_extraction.text import TfidfVectorizer\ntfidf = TfidfVectorizer()\nX = tfidf.fit_transform(corpus)",
"_____no_output_____"
],
[
"X.toarray()",
"_____no_output_____"
],
[
"type(X)",
"_____no_output_____"
],
[
"X.shape",
"_____no_output_____"
],
[
"print(X[:,0])",
" (4, 0)\t0.18444604729119288\n"
],
[
"print(X[:,:])",
" (0, 26)\t0.2611488808945384\n (0, 5)\t0.21677716168619507\n (0, 38)\t0.3236873066380182\n (0, 34)\t0.3236873066380182\n (0, 51)\t0.3236873066380182\n (0, 41)\t0.43355432337239014\n (0, 18)\t0.3236873066380182\n (0, 23)\t0.3236873066380182\n (0, 29)\t0.2611488808945384\n (0, 9)\t0.3236873066380182\n (1, 21)\t0.20243884765910772\n (1, 25)\t0.20243884765910772\n (1, 44)\t0.20243884765910772\n (1, 3)\t0.20243884765910772\n (1, 24)\t0.20243884765910772\n (1, 8)\t0.20243884765910772\n (1, 49)\t0.20243884765910772\n (1, 1)\t0.20243884765910772\n (1, 32)\t0.16332638763273197\n (1, 45)\t0.13557565561148596\n (1, 13)\t0.20243884765910772\n (1, 2)\t0.16332638763273197\n (1, 4)\t0.20243884765910772\n (1, 35)\t0.20243884765910772\n (1, 40)\t0.20243884765910772\n :\t:\n (3, 11)\t0.3420339209721722\n (3, 46)\t0.3420339209721722\n (3, 42)\t0.2290641031273583\n (3, 5)\t0.2290641031273583\n (4, 30)\t0.18444604729119288\n (4, 0)\t0.18444604729119288\n (4, 17)\t0.18444604729119288\n (4, 12)\t0.18444604729119288\n (4, 31)\t0.18444604729119288\n (4, 43)\t0.36889209458238575\n (4, 16)\t0.18444604729119288\n (4, 22)\t0.5533381418735785\n (4, 33)\t0.18444604729119288\n (4, 14)\t0.18444604729119288\n (4, 37)\t0.18444604729119288\n (4, 7)\t0.18444604729119288\n (4, 48)\t0.14880990958778192\n (4, 42)\t0.12352566750705657\n (4, 19)\t0.14880990958778192\n (4, 32)\t0.14880990958778192\n (4, 45)\t0.12352566750705657\n (4, 2)\t0.14880990958778192\n (4, 5)\t0.12352566750705657\n (4, 41)\t0.24705133501411314\n (4, 29)\t0.14880990958778192\n"
]
]
] | [
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ec030784fedd5d0b631ea9febc7cdb820ddfba | 21,698 | ipynb | Jupyter Notebook | .ipynb_checkpoints/Data_Analyst_ND_Project3-checkpoint.ipynb | jorcus/DAND-Wrangle-OpenStreetMap-Data | 6cf3480f3acc46e6080470fd3174d43c413fd0e4 | [
"MIT"
] | null | null | null | .ipynb_checkpoints/Data_Analyst_ND_Project3-checkpoint.ipynb | jorcus/DAND-Wrangle-OpenStreetMap-Data | 6cf3480f3acc46e6080470fd3174d43c413fd0e4 | [
"MIT"
] | null | null | null | .ipynb_checkpoints/Data_Analyst_ND_Project3-checkpoint.ipynb | jorcus/DAND-Wrangle-OpenStreetMap-Data | 6cf3480f3acc46e6080470fd3174d43c413fd0e4 | [
"MIT"
] | null | null | null | 29.76406 | 531 | 0.518527 | [
[
[
"# OpenStreetMap Data Case Study",
"_____no_output_____"
],
[
"## Problems Encountered in the Map\nDiscuss the main problems with the data in the following order:\n\n- Overabbreviated street names (“S Tryon St Ste 105”)\n\n- Second level “k” tags with the value \"type\"(which overwrites the element’s previously processed node[“type”]field).\n\n- Street names in second level “k” tags pulled from Tiger GPS data and divided into segments, in the following format:\n\n- Unstructure Unique ID (1, 42653, 2321, 5030230)\n\n\n### Map Area - Dataset\n\nIn this project, I choose San Jose which is a large city surrounded by rolling hills in Silicon Valley, a major technology hub in California's Bay Area. I want to learn more about the place to see what database querying reveals. This location is one of my dreams working area as it's all over the world-class Tech corporations around there. \n\nSan Jose, United States (OSM XML: 364.6 MB)\n- https://mapzen.com/data/metro-extracts/metro/san-jose_california/ \n",
"_____no_output_____"
]
],
[
[
"# -*- coding: utf-8 -*-\n\nimport pprint\nimport xml.etree.ElementTree as ET\nfrom collections import defaultdict\nimport re\nimport os\n\nDATASET = \"san-jose_california.osm\" # osm filename\nPATH = \"./\" # directory contain the osm file\nOSMFILE = PATH + DATASET\nprint('Dataset folder:', OSMFILE)",
"Dataset folder: ./san-jose_california.osm\n"
]
],
[
[
"### Iterative Parsing the OSM file.",
"_____no_output_____"
]
],
[
[
"# mapparser.py\n# iterative parsing\nfrom mapparser import count_tags, count_tags_total\n\ntags = count_tags(OSMFILE)\nprint('Numbers of tag: ', len(tags))\nprint('Numbers of tag elements: ', count_tags_total(tags))\npprint.pprint(tags)",
"Numbers of tag: 8\nNumbers of tag elements: 4599618\n{'bounds': 1,\n 'member': 18333,\n 'nd': 1965111,\n 'node': 1679378,\n 'osm': 1,\n 'relation': 1759,\n 'tag': 705634,\n 'way': 229401}\n"
]
],
[
[
"### Categorize the tag keys.\nCategorize the tag keys in the followings:\n- \"lower\", for tags that contain only lowercase letters and are valid,\n- \"lower_colon\", for otherwise valid tags with a colon in their names,\n- \"problemchars\", for tags with problematic characters, and\n- \"other\", for other tags that do not fall into the other three categories.",
"_____no_output_____"
]
],
[
[
"# tags.py\nfrom tags import key_type\ndef process_map_tags(filename):\n keys = {\"lower\": 0, \"lower_colon\": 0, \"problemchars\": 0, \"other\": 0}\n for _, element in ET.iterparse(filename):\n keys = key_type(element, keys)\n return keys\nkeys = process_map_tags(OSMFILE)\npprint.pprint(keys)",
"{'lower': 459030, 'lower_colon': 224633, 'other': 21969, 'problemchars': 2}\n"
]
],
[
[
"### Number of Unique Users\nAs you can see, each of the user has their own unique ID. However, the ID is unstructured likes 1, 1005885, 1030, 100744. I structured all the unique user id in the followings:\n- 25663 => 0025663\n- 951370 => 0951370",
"_____no_output_____"
]
],
[
[
"# users.py\nfrom users import unique_user_id, max_length_user_id, structure_user_id\n\ndef test():\n users = unique_user_id(OSMFILE)\n # structured = structure_user_id(users)\n # pprint.pprint(structured)\n max_length = max_length_user_id(users)\n print('Number of users: ', len(users))\n print('User ID maximum length', max_length)\n\n print_limit = 10\n for user_id in users:\n if len(user_id) < max_length:\n structured_id = user_id\n while len(structured_id) < max_length:\n structured_id = str('0' + structured_id)\n\n if print_limit > 0:\n print_limit -= 1\n print(user_id, \"=>\", structured_id)\n else:\n break\n\nif __name__ == '__main__':\n test()",
"Number of users: 1359\nUser ID maximum length 7\n25663 => 0025663\n951370 => 0951370\n199089 => 0199089\n637707 => 0637707\n28145 => 0028145\n941449 => 0941449\n281267 => 0281267\n41907 => 0041907\n166129 => 0166129\n173623 => 0173623\n"
]
],
[
[
"### Over-abbreviated Street Names\nSome basic query is over-abbreviated. I updated all the problematic address strings in the followings:\n\n- Seaboard Ave => Seaboard Avenue\n- Cherry Ave => Cherry Avenue",
"_____no_output_____"
]
],
[
[
"#audit.py\nfrom audit import audit, update_name, street_type_re, mapping\n\ndef test():\n st_types = audit(OSMFILE)\n # pprint.pprint(dict(st_types)) #print out dictonary of potentially incorrect street types\n print_limit = 10\n for st_type, ways in st_types.items(): # .iteritems() for python2\n for name in ways:\n if street_type_re.search(name).group() in mapping:\n better_name = update_name(name, mapping)\n if print_limit > 0:\n print_limit -= 1\n print (name, \"=>\", better_name)\n else:\n break\n \nif __name__ == '__main__':\n test()",
"Hillsdale Ave => Hillsdale Avenue\nMeridian Ave => Meridian Avenue\nWalsh Ave => Walsh Avenue\nSeaboard Ave => Seaboard Avenue\nN Blaney Ave => N Blaney Avenue\nSaratoga Ave => Saratoga Avenue\n1425 E Dunne Ave => 1425 E Dunne Avenue\nBlake Ave => Blake Avenue\nThe Alameda Ave => The Alameda Avenue\nHollenbeck Ave => Hollenbeck Avenue\n"
]
],
[
[
"### Insert data into Mongodb",
"_____no_output_____"
]
],
[
[
"# data.py\nfrom data import process_map\n\ndata = process_map(OSMFILE, True)",
"_____no_output_____"
],
[
"data[0]",
"_____no_output_____"
]
],
[
[
"# Data Overview",
"_____no_output_____"
]
],
[
[
"from pymongo import MongoClient\nclient = MongoClient('localhost:27017')\ndb = client.SanJose\ncollection = db.SanJoseMAP\n#collection.insert(data)",
"_____no_output_____"
],
[
"collection",
"_____no_output_____"
],
[
"print('Size of the original xml file: ',os.path.getsize(OSMFILE)/(1024*1024.0), 'MB')\nprint('Size of the processed json file: ',os.path.getsize(os.path.join(PATH, \"san-jose_california.osm.json\"))/(1024*1024.0), 'MB')\nprint('Number of documents: ' + str(collection.find().count()))\nprint('Number of nodes: ' + str(collection.find({\"type\":\"node\"}).count()))\nprint('Number of ways: ' + str(collection.find({\"type\":\"way\"}).count()))\nprint('Number of relations: ' + str(collection.find({\"type\":\"relation\"}).count()))\nprint('Number of unique users: ' + str(len(collection.distinct(\"created.user\"))))\nprint('Number of pizza places: ' + str(collection.find({\"cuisine\":\"pizza\"}).count()))",
"Size of the original xml file: 348.08773612976074 MB\nSize of the processed json file: 512.8097190856934 MB\nNumber of documents: 19761132\nNumber of nodes: 17470242\nNumber of ways: 2290674\nNumber of relations: 0\nNumber of unique users: 1356\nNumber of pizza places: 636\n"
]
],
[
[
"### Contributor statistics and gamification suggestio\n\nThe contributions of users seems incredibly skewed, possibly due to automated versus manual map editing (the word “bot” appears in some usernames). Here are some user percentage statistics:\n- Top user contribution percentage (“nmixter”) - 15.08%\n- Combined top 2 users' contribution (“nmixter” and “andygol”) - 30.07%\n- Combined Top 10 users contribution - 64.12%\n\nThinking about these user percentages, I’m reminded of “gamification” as a motivating force for contribution. In the context of the OpenStreetMap, if user data were more prominently displayed, perhaps others would take an initiative in submitting more edits to the map. It's so surprise, the only top 10 users that contributed over than 50% of this dataset. That might spur the creation of more efficient bots, especially if certain gamification elements were present, such as rewards, badges, or a leaderboard. \n\n\n\n\n\n#### Top 10 users with most contributions",
"_____no_output_____"
]
],
[
[
"# Top 10 users with most contributions\npipeline = [{\"$group\":{\"_id\": \"$created.user\", \"count\": {\"$sum\": 1}}},\n {\"$sort\": {\"count\": -1}},\n {\"$limit\": 10}]\nresult = collection.aggregate(pipeline)\nfor r in range(10):\n print (result.next())",
"{'_id': 'nmixter', 'count': 2980568}\n{'_id': 'andygol', 'count': 2961664}\n{'_id': 'mk408', 'count': 1615791}\n{'_id': 'Bike Mapper', 'count': 969105}\n{'_id': 'samely', 'count': 813227}\n{'_id': 'RichRico', 'count': 768741}\n{'_id': 'dannykath', 'count': 752101}\n{'_id': 'MustangBuyer', 'count': 646129}\n{'_id': 'karitotp', 'count': 645535}\n{'_id': 'Minh Nguyen', 'count': 517383}\n"
]
],
[
[
"#### Number of users appearing only once (having 1 post)\n##### There's only one user appearing only once. Which means most the the user appear at least once.",
"_____no_output_____"
]
],
[
[
"# Number of users appearing only once (having 1 post)\npipeline = [{\"$group\":{\"_id\":\"$created.user\", \"count\":{\"$sum\":1}}},\n {\"$group\":{\"_id\":\"$count\", \"num_users\":{\"$sum\":1}}},\n {\"$sort\":{\"_id\":1}}, {\"$limit\":1}]\n\nresult = collection.aggregate(pipeline)\nfor r in range(1):\n print (result.next())",
"{'_id': 1, 'num_users': 1}\n"
]
],
[
[
"#### Top 10 Biggest religion\n##### The result show in Sanjose area, the Christian is one of the biggest religion. The the seconds largest is Unknown, seem like the record is missing. Then coming to the third largest religion is jewish.",
"_____no_output_____"
]
],
[
[
"# Top 10 Biggest religion\npipeline = [{\"$match\":{\"amenity\":{\"$exists\":1}, \"amenity\":\"place_of_worship\"}},\n {\"$group\":{\"_id\":\"$religion\", \"count\":{\"$sum\":1}}},\n {\"$sort\":{\"count\":-1}}, {\"$limit\":10}]\nresult = collection.aggregate(pipeline)\nfor r in range(10):\n print (result.next())",
"{'_id': 'christian', 'count': 1996}\n{'_id': None, 'count': 139}\n{'_id': 'jewish', 'count': 33}\n{'_id': 'buddhist', 'count': 26}\n{'_id': 'muslim', 'count': 18}\n{'_id': 'hindu', 'count': 14}\n{'_id': 'unitarian_universalist', 'count': 13}\n{'_id': 'sikh', 'count': 7}\n{'_id': 'caodaism', 'count': 7}\n{'_id': 'zoroastrian', 'count': 7}\n"
]
],
[
[
"#### Top 10 appearing amenities",
"_____no_output_____"
]
],
[
[
"# Top 10 appearing amenities\npipeline = [{\"$match\":{\"amenity\":{\"$exists\":1}}}, \n {\"$group\":{\"_id\":\"$amenity\",\"count\":{\"$sum\":1}}},\n {\"$sort\":{\"count\":-1}}, {\"$limit\":10}]\n\nresult = collection.aggregate(pipeline)\nfor r in range(10):\n print (result.next())",
"{'_id': '', 'count': 3798126}\n{'_id': 'parking', 'count': 12302}\n{'_id': 'restaurant', 'count': 6573}\n{'_id': 'fast_food', 'count': 3406}\n{'_id': 'school', 'count': 3321}\n{'_id': 'place_of_worship', 'count': 2298}\n{'_id': 'bench', 'count': 1807}\n{'_id': 'cafe', 'count': 1753}\n{'_id': 'fuel', 'count': 1580}\n{'_id': 'bicycle_parking', 'count': 1347}\n"
]
],
[
[
"#### Top 10 popular cuisines",
"_____no_output_____"
]
],
[
[
"# Top 10 popular cuisines\npipeline = [{\"$match\":{\"amenity\":{\"$exists\":1}, \"amenity\":\"restaurant\"}},\n {\"$group\":{\"_id\":\"$cuisine\", \"count\":{\"$sum\":1}}},\n {\"$sort\":{\"count\":-1}}, {\"$limit\":10}]\n\nresult = collection.aggregate(pipeline)\nfor r in range(10):\n print (result.next())",
"{'_id': None, 'count': 1296}\n{'_id': '', 'count': 572}\n{'_id': 'mexican', 'count': 570}\n{'_id': 'chinese', 'count': 504}\n{'_id': 'vietnamese', 'count': 459}\n{'_id': 'pizza', 'count': 390}\n{'_id': 'japanese', 'count': 293}\n{'_id': 'american', 'count': 283}\n{'_id': 'italian', 'count': 222}\n{'_id': 'indian', 'count': 214}\n"
]
],
[
[
"#### Sort postcodes by count, descending",
"_____no_output_____"
]
],
[
[
"# Sort postcodes by count, descending\npipeline = [{\"$match\":{\"address.postcode\":{\"$exists\":1}}}, \n {\"$group\":{\"_id\":\"$address.postcode\", \"count\":{\"$sum\":1}}}, \n {\"$sort\":{\"count\":-1}}]\n\nresult = collection.aggregate(pipeline)\nfor r in range(10):\n print (result.next())",
"{'_id': '', 'count': 1893917}\n{'_id': '95014', 'count': 3503}\n{'_id': '95070', 'count': 2438}\n{'_id': '94087', 'count': 2205}\n{'_id': '94086', 'count': 2052}\n{'_id': '95051', 'count': 1772}\n{'_id': '95129', 'count': 1397}\n{'_id': '95127', 'count': 1130}\n{'_id': '95054', 'count': 1023}\n{'_id': '95035', 'count': 1018}\n"
]
],
[
[
"#### Sort street by count, descending",
"_____no_output_____"
]
],
[
[
"# Sort street by count, descending\npipeline = [{\"$match\":{\"address.street\":{\"$exists\":1}}},\n {\"$group\":{\"_id\":\"$address.street\", \"count\":{\"$sum\":1}}},\n {\"$sort\":{\"count\":-1}}]\n\nresult = collection.aggregate(pipeline)\nfor r in range(10):\n print (result.next())",
"{'_id': '', 'count': 1885122}\n{'_id': 'Stevens Creek Boulevard', 'count': 2898}\n{'_id': 'Hollenbeck Avenue', 'count': 1745}\n{'_id': 'South Stelling Road', 'count': 1300}\n{'_id': 'East Estates Drive', 'count': 1230}\n{'_id': 'Johnson Avenue', 'count': 1200}\n{'_id': 'Miller Avenue', 'count': 1170}\n{'_id': 'Bollinger Road', 'count': 1170}\n{'_id': 'North Santa Cruz Avenue', 'count': 1160}\n{'_id': 'South De Anza Boulevard', 'count': 1127}\n"
]
],
[
[
"## Conclusion\nI believe it has been cleaned for the purposes of this exercise. However, some area of the San Jose data is obviously far from being complete. There's still some data haven't clean likes Inconsistent postal codes (“NC28226”, “282260783”, “28226”) and “Incorrect” postal codes (Charlotte area zip codes all begin with “282” however a large portion of all documented zip codes were outside this region.)\n\n## References\n\n[1] OpenStreetMap Sample Project Data Wrangling with MongoDB (Matthew Banbury). Available from: <https://docs.google.com/document/d/1F0Vs14oNEs2idFJR3C_OPxwS6L0HPliOii-QpbmrMo4/pub>",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
]
] |
e7ec03afdb7edc33f64181caf355066fada18ef6 | 129,534 | ipynb | Jupyter Notebook | example/vae/VAE_example.ipynb | dkuspawono/incubator-mxnet | 235f032776a73a2e07e8ee65e0ea74c556ac71de | [
"Apache-2.0"
] | 116 | 2018-01-01T03:53:49.000Z | 2021-09-06T07:09:09.000Z | example/vae/VAE_example.ipynb | shreelola/incubator-mxnet | 210e6d49e859fe5f8aa7304c66a18f3eb065a471 | [
"Apache-2.0"
] | 27 | 2017-07-04T17:45:51.000Z | 2019-09-12T06:56:27.000Z | example/vae/VAE_example.ipynb | shreelola/incubator-mxnet | 210e6d49e859fe5f8aa7304c66a18f3eb065a471 | [
"Apache-2.0"
] | 47 | 2016-04-19T22:46:09.000Z | 2020-09-30T08:09:16.000Z | 107.497095 | 17,196 | 0.818001 | [
[
[
"import mxnet as mx\nimport numpy as np\nimport os\nimport logging\nimport matplotlib.pyplot as plt\nimport matplotlib.cm as cm",
"_____no_output_____"
]
],
[
[
"# Building a Variational Autoencoder in MXNet\n\n#### Xiaoyu Lu, July 5th, 2017\n\nThis tutorial guides you through the process of building a variational encoder in MXNet. In this notebook we'll focus on an example using the MNIST handwritten digit recognition dataset. Refer to [Auto-Encoding Variational Bayes](https://arxiv.org/abs/1312.6114/) for more details on the model description.\n\n",
"_____no_output_____"
],
[
"## Prerequisites\n\nTo complete this tutorial, we need following python packages:\n\n- numpy, matplotlib ",
"_____no_output_____"
],
[
"## 1. Loading the Data\n\nWe first load the MNIST dataset, which contains 60000 training and 10000 test examples. The following code imports required modules and loads the data. These images are stored in a 4-D matrix with shape (`batch_size, num_channels, width, height`). For the MNIST dataset, there is only one color channel, and both width and height are 28, so we reshape each image as a 28x28 array. See below for a visualization:\n",
"_____no_output_____"
]
],
[
[
"mnist = mx.test_utils.get_mnist()\nimage = np.reshape(mnist['train_data'],(60000,28*28))\nlabel = image\nimage_test = np.reshape(mnist['test_data'],(10000,28*28))\nlabel_test = image_test\n[N,features] = np.shape(image) #number of examples and features",
"_____no_output_____"
],
[
"f, (ax1, ax2, ax3, ax4) = plt.subplots(1,4, sharex='col', sharey='row',figsize=(12,3))\nax1.imshow(np.reshape(image[0,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax2.imshow(np.reshape(image[1,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax3.imshow(np.reshape(image[2,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax4.imshow(np.reshape(image[3,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nplt.show()",
"_____no_output_____"
]
],
[
[
"We can optionally save the parameters in the directory variable 'model_prefix'. We first create data iterators for MXNet, with each batch of data containing 100 images.",
"_____no_output_____"
]
],
[
[
"model_prefix = None\n\nbatch_size = 100\nnd_iter = mx.io.NDArrayIter(data={'data':image},label={'loss_label':label},\n batch_size = batch_size)\nnd_iter_test = mx.io.NDArrayIter(data={'data':image_test},label={'loss_label':label_test},\n batch_size = batch_size)",
"_____no_output_____"
]
],
[
[
"## 2. Building the Network Architecture\n\n### 2.1 Gaussian MLP as encoder\nNext we constuct the neural network, as in the [paper](https://arxiv.org/abs/1312.6114/), we use *Multilayer Perceptron (MLP)* for both the encoder and decoder. For encoder, a Gaussian MLP is used as follows:\n\n\\begin{align}\n\\log q_{\\phi}(z|x) &= \\log \\mathcal{N}(z:\\mu,\\sigma^2I) \\\\\n\\textit{ where } \\mu &= W_2h+b_2, \\log \\sigma^2 = W_3h+b_3\\\\\nh &= \\tanh(W_1x+b_1)\n\\end{align}\n\nwhere $\\{W_1,W_2,W_3,b_1,b_2,b_3\\}$ are the weights and biases of the MLP.\nNote below that `encoder_mu`(`mu`) and `encoder_logvar`(`logvar`) are symbols. So, we can use `get_internals()` to get the values of them, after which we can sample the latent variable $z$.\n\n\n\n",
"_____no_output_____"
]
],
[
[
"## define data and loss labels as symbols \ndata = mx.sym.var('data')\nloss_label = mx.sym.var('loss_label')\n\n## define fully connected and activation layers for the encoder, where we used tanh activation function.\nencoder_h = mx.sym.FullyConnected(data=data, name=\"encoder_h\",num_hidden=400)\nact_h = mx.sym.Activation(data=encoder_h, act_type=\"tanh\",name=\"activation_h\")\n\n## define mu and log variance which are the fully connected layers of the previous activation layer\nmu = mx.sym.FullyConnected(data=act_h, name=\"mu\",num_hidden = 5)\nlogvar = mx.sym.FullyConnected(data=act_h, name=\"logvar\",num_hidden = 5)\n\n## sample the latent variables z according to Normal(mu,var)\nz = mu + np.multiply(mx.symbol.exp(0.5 * logvar), \n mx.symbol.random_normal(loc=0, scale=1, shape=np.shape(logvar.get_internals()[\"logvar_output\"])))",
"_____no_output_____"
]
],
[
[
"### 2.2 Bernoulli MLP as decoder\n\nIn this case let $p_\\theta(x|z)$ be a multivariate Bernoulli whose probabilities are computed from $z$ with a feed forward neural network with a single hidden layer:\n\n\\begin{align}\n\\log p(x|z) &= \\sum_{i=1}^D x_i\\log y_i + (1-x_i)\\log (1-y_i) \\\\\n\\textit{ where } y &= f_\\sigma(W_5\\tanh (W_4z+b_4)+b_5)\n\\end{align}\n\nwhere $f_\\sigma(\\dot)$ is the elementwise sigmoid activation function, $\\{W_4,W_5,b_4,b_5\\}$ are the weights and biases of the decoder MLP. A Bernouilli likelihood is suitable for this type of data but you can easily extend it to other likelihood types by parsing into the argument `likelihood` in the `VAE` class, see section 4 for details.",
"_____no_output_____"
]
],
[
[
"# define fully connected and tanh activation layers for the decoder\ndecoder_z = mx.sym.FullyConnected(data=z, name=\"decoder_z\",num_hidden=400)\nact_z = mx.sym.Activation(data=decoder_z, act_type=\"tanh\",name=\"activation_z\")\n\n# define the output layer with sigmoid activation function, where the dimension is equal to the input dimension\ndecoder_x = mx.sym.FullyConnected(data=act_z, name=\"decoder_x\",num_hidden=features)\ny = mx.sym.Activation(data=decoder_x, act_type=\"sigmoid\",name='activation_x')",
"_____no_output_____"
]
],
[
[
"### 2.3 Joint Loss Function for the Encoder and the Decoder\n\nThe variational lower bound also called evidence lower bound (ELBO) can be estimated as:\n\n\\begin{align}\n\\mathcal{L}(\\theta,\\phi;x_{(i)}) \\approx \\frac{1}{2}\\left(1+\\log ((\\sigma_j^{(i)})^2)-(\\mu_j^{(i)})^2-(\\sigma_j^{(i)})^2\\right) + \\log p_\\theta(x^{(i)}|z^{(i)})\n\\end{align}\n\nwhere the first term is the KL divergence of the approximate posterior from the prior, and the second term is an expected negative reconstruction error. We would like to maximize this lower bound, so we can define the loss to be $-\\mathcal{L}$(minus ELBO) for MXNet to minimize.",
"_____no_output_____"
]
],
[
[
"# define the objective loss function that needs to be minimized\nKL = 0.5*mx.symbol.sum(1+logvar-pow( mu,2)-mx.symbol.exp(logvar),axis=1)\nloss = -mx.symbol.sum(mx.symbol.broadcast_mul(loss_label,mx.symbol.log(y)) \n + mx.symbol.broadcast_mul(1-loss_label,mx.symbol.log(1-y)),axis=1)-KL\noutput = mx.symbol.MakeLoss(sum(loss),name='loss')",
"_____no_output_____"
]
],
[
[
"## 3. Training the model\n\nNow, we can define the model and train it. First we will initilize the weights and the biases to be Gaussian(0,0.01), and then use stochastic gradient descent for optimization. To warm start the training, one may also initilize with pre-trainined parameters `arg_params` using `init=mx.initializer.Load(arg_params)`. \n\nTo save intermediate results, we can optionally use `epoch_end_callback = mx.callback.do_checkpoint(model_prefix, 1)` which saves the parameters to the path given by model_prefix, and with period every $1$ epoch. To assess the performance, we output $-\\mathcal{L}$(minus ELBO) after each epoch, with the command `eval_metric = 'Loss'` which is defined above. We will also plot the training loss for mini batches by accessing the log and saving it to a list, and then parsing it to the argument `batch_end_callback`.",
"_____no_output_____"
]
],
[
[
"# set up the log\nnd_iter.reset()\nlogging.getLogger().setLevel(logging.DEBUG) \n\n# define function to trave back training loss\ndef log_to_list(period, lst):\n def _callback(param):\n \"\"\"The checkpoint function.\"\"\"\n if param.nbatch % period == 0:\n name, value = param.eval_metric.get()\n lst.append(value)\n return _callback\n\n# define the model\nmodel = mx.mod.Module(\n symbol = output ,\n data_names=['data'],\n label_names = ['loss_label'])",
"_____no_output_____"
],
[
"# training the model, save training loss as a list.\ntraining_loss=list()\n\n# initilize the parameters for training using Normal.\ninit = mx.init.Normal(0.01)\nmodel.fit(nd_iter, # train data\n initializer=init,\n # if eval_data is supplied, test loss will also be reported\n # eval_data = nd_iter_test,\n optimizer='sgd', # use SGD to train\n optimizer_params={'learning_rate':1e-3,'wd':1e-2}, \n # save parameters for each epoch if model_prefix is supplied\n epoch_end_callback = None if model_prefix==None else mx.callback.do_checkpoint(model_prefix, 1),\n batch_end_callback = log_to_list(N/batch_size,training_loss), \n num_epoch=100,\n eval_metric = 'Loss')",
"INFO:root:Epoch[0] Train-loss=375.023381\nINFO:root:Epoch[0] Time cost=6.127\nINFO:root:Epoch[1] Train-loss=212.780315\nINFO:root:Epoch[1] Time cost=6.409\nINFO:root:Epoch[2] Train-loss=208.209400\nINFO:root:Epoch[2] Time cost=6.619\nINFO:root:Epoch[3] Train-loss=206.146854\nINFO:root:Epoch[3] Time cost=6.648\nINFO:root:Epoch[4] Train-loss=204.530598\nINFO:root:Epoch[4] Time cost=7.000\nINFO:root:Epoch[5] Train-loss=202.799992\nINFO:root:Epoch[5] Time cost=6.778\nINFO:root:Epoch[6] Train-loss=200.333474\nINFO:root:Epoch[6] Time cost=7.187\nINFO:root:Epoch[7] Train-loss=197.506393\nINFO:root:Epoch[7] Time cost=6.712\nINFO:root:Epoch[8] Train-loss=195.969775\nINFO:root:Epoch[8] Time cost=6.896\nINFO:root:Epoch[9] Train-loss=195.418288\nINFO:root:Epoch[9] Time cost=6.887\nINFO:root:Epoch[10] Train-loss=194.739763\nINFO:root:Epoch[10] Time cost=6.745\nINFO:root:Epoch[11] Train-loss=194.380536\nINFO:root:Epoch[11] Time cost=6.706\nINFO:root:Epoch[12] Train-loss=193.955462\nINFO:root:Epoch[12] Time cost=6.592\nINFO:root:Epoch[13] Train-loss=193.493671\nINFO:root:Epoch[13] Time cost=6.775\nINFO:root:Epoch[14] Train-loss=192.958739\nINFO:root:Epoch[14] Time cost=6.600\nINFO:root:Epoch[15] Train-loss=191.928542\nINFO:root:Epoch[15] Time cost=6.586\nINFO:root:Epoch[16] Train-loss=189.797939\nINFO:root:Epoch[16] Time cost=6.700\nINFO:root:Epoch[17] Train-loss=186.672446\nINFO:root:Epoch[17] Time cost=6.869\nINFO:root:Epoch[18] Train-loss=184.616599\nINFO:root:Epoch[18] Time cost=7.144\nINFO:root:Epoch[19] Train-loss=183.305978\nINFO:root:Epoch[19] Time cost=6.997\nINFO:root:Epoch[20] Train-loss=181.944634\nINFO:root:Epoch[20] Time cost=6.481\nINFO:root:Epoch[21] Train-loss=181.005329\nINFO:root:Epoch[21] Time cost=6.754\nINFO:root:Epoch[22] Train-loss=178.363118\nINFO:root:Epoch[22] Time cost=7.000\nINFO:root:Epoch[23] Train-loss=176.363421\nINFO:root:Epoch[23] Time cost=6.923\nINFO:root:Epoch[24] Train-loss=174.573954\nINFO:root:Epoch[24] Time cost=6.510\nINFO:root:Epoch[25] Train-loss=173.245940\nINFO:root:Epoch[25] Time cost=6.926\nINFO:root:Epoch[26] Train-loss=172.082522\nINFO:root:Epoch[26] Time cost=6.733\nINFO:root:Epoch[27] Train-loss=171.123084\nINFO:root:Epoch[27] Time cost=6.616\nINFO:root:Epoch[28] Train-loss=170.239300\nINFO:root:Epoch[28] Time cost=7.004\nINFO:root:Epoch[29] Train-loss=169.538416\nINFO:root:Epoch[29] Time cost=6.341\nINFO:root:Epoch[30] Train-loss=168.952901\nINFO:root:Epoch[30] Time cost=6.736\nINFO:root:Epoch[31] Train-loss=168.169076\nINFO:root:Epoch[31] Time cost=6.616\nINFO:root:Epoch[32] Train-loss=167.208973\nINFO:root:Epoch[32] Time cost=6.446\nINFO:root:Epoch[33] Train-loss=165.732213\nINFO:root:Epoch[33] Time cost=6.405\nINFO:root:Epoch[34] Train-loss=163.606801\nINFO:root:Epoch[34] Time cost=6.139\nINFO:root:Epoch[35] Train-loss=161.985880\nINFO:root:Epoch[35] Time cost=6.678\nINFO:root:Epoch[36] Train-loss=160.763072\nINFO:root:Epoch[36] Time cost=8.749\nINFO:root:Epoch[37] Train-loss=160.025193\nINFO:root:Epoch[37] Time cost=6.519\nINFO:root:Epoch[38] Train-loss=159.319723\nINFO:root:Epoch[38] Time cost=7.584\nINFO:root:Epoch[39] Train-loss=158.670701\nINFO:root:Epoch[39] Time cost=6.874\nINFO:root:Epoch[40] Train-loss=158.225733\nINFO:root:Epoch[40] Time cost=6.402\nINFO:root:Epoch[41] Train-loss=157.741337\nINFO:root:Epoch[41] Time cost=8.617\nINFO:root:Epoch[42] Train-loss=157.301411\nINFO:root:Epoch[42] Time cost=6.515\nINFO:root:Epoch[43] Train-loss=156.765170\nINFO:root:Epoch[43] Time cost=6.447\nINFO:root:Epoch[44] Train-loss=156.389668\nINFO:root:Epoch[44] Time cost=6.130\nINFO:root:Epoch[45] Train-loss=155.815434\nINFO:root:Epoch[45] Time cost=6.155\nINFO:root:Epoch[46] Train-loss=155.432254\nINFO:root:Epoch[46] Time cost=6.158\nINFO:root:Epoch[47] Train-loss=155.114027\nINFO:root:Epoch[47] Time cost=6.749\nINFO:root:Epoch[48] Train-loss=154.612441\nINFO:root:Epoch[48] Time cost=6.255\nINFO:root:Epoch[49] Train-loss=154.137659\nINFO:root:Epoch[49] Time cost=7.813\nINFO:root:Epoch[50] Train-loss=153.634072\nINFO:root:Epoch[50] Time cost=7.408\nINFO:root:Epoch[51] Train-loss=153.417397\nINFO:root:Epoch[51] Time cost=7.747\nINFO:root:Epoch[52] Train-loss=152.851887\nINFO:root:Epoch[52] Time cost=8.587\nINFO:root:Epoch[53] Train-loss=152.575068\nINFO:root:Epoch[53] Time cost=7.554\nINFO:root:Epoch[54] Train-loss=152.084419\nINFO:root:Epoch[54] Time cost=6.628\nINFO:root:Epoch[55] Train-loss=151.724836\nINFO:root:Epoch[55] Time cost=6.535\nINFO:root:Epoch[56] Train-loss=151.302525\nINFO:root:Epoch[56] Time cost=7.148\nINFO:root:Epoch[57] Train-loss=150.960916\nINFO:root:Epoch[57] Time cost=7.195\nINFO:root:Epoch[58] Train-loss=150.603895\nINFO:root:Epoch[58] Time cost=6.649\nINFO:root:Epoch[59] Train-loss=150.237795\nINFO:root:Epoch[59] Time cost=6.222\nINFO:root:Epoch[60] Train-loss=149.936080\nINFO:root:Epoch[60] Time cost=8.450\nINFO:root:Epoch[61] Train-loss=149.514617\nINFO:root:Epoch[61] Time cost=6.113\nINFO:root:Epoch[62] Train-loss=149.229345\nINFO:root:Epoch[62] Time cost=6.088\nINFO:root:Epoch[63] Train-loss=148.893769\nINFO:root:Epoch[63] Time cost=6.558\nINFO:root:Epoch[64] Train-loss=148.526837\nINFO:root:Epoch[64] Time cost=7.590\nINFO:root:Epoch[65] Train-loss=148.249951\nINFO:root:Epoch[65] Time cost=6.180\nINFO:root:Epoch[66] Train-loss=147.940414\nINFO:root:Epoch[66] Time cost=6.242\nINFO:root:Epoch[67] Train-loss=147.621304\nINFO:root:Epoch[67] Time cost=8.501\nINFO:root:Epoch[68] Train-loss=147.294314\nINFO:root:Epoch[68] Time cost=7.645\nINFO:root:Epoch[69] Train-loss=147.074479\nINFO:root:Epoch[69] Time cost=7.092\nINFO:root:Epoch[70] Train-loss=146.796387\nINFO:root:Epoch[70] Time cost=6.914\nINFO:root:Epoch[71] Train-loss=146.508842\nINFO:root:Epoch[71] Time cost=6.606\nINFO:root:Epoch[72] Train-loss=146.230444\nINFO:root:Epoch[72] Time cost=7.755\nINFO:root:Epoch[73] Train-loss=145.970296\nINFO:root:Epoch[73] Time cost=6.409\nINFO:root:Epoch[74] Train-loss=145.711610\nINFO:root:Epoch[74] Time cost=6.334\nINFO:root:Epoch[75] Train-loss=145.460053\nINFO:root:Epoch[75] Time cost=7.269\nINFO:root:Epoch[76] Train-loss=145.156451\nINFO:root:Epoch[76] Time cost=6.744\nINFO:root:Epoch[77] Train-loss=144.957674\nINFO:root:Epoch[77] Time cost=7.100\nINFO:root:Epoch[78] Train-loss=144.729749\nINFO:root:Epoch[78] Time cost=6.242\nINFO:root:Epoch[79] Train-loss=144.481728\nINFO:root:Epoch[79] Time cost=6.865\nINFO:root:Epoch[80] Train-loss=144.236061\nINFO:root:Epoch[80] Time cost=6.632\nINFO:root:Epoch[81] Train-loss=144.030473\nINFO:root:Epoch[81] Time cost=6.764\nINFO:root:Epoch[82] Train-loss=143.776374\nINFO:root:Epoch[82] Time cost=6.564\nINFO:root:Epoch[83] Train-loss=143.538847\nINFO:root:Epoch[83] Time cost=6.181\nINFO:root:Epoch[84] Train-loss=143.326444\nINFO:root:Epoch[84] Time cost=6.220\nINFO:root:Epoch[85] Train-loss=143.078987\nINFO:root:Epoch[85] Time cost=6.823\nINFO:root:Epoch[86] Train-loss=142.877117\nINFO:root:Epoch[86] Time cost=7.755\nINFO:root:Epoch[87] Train-loss=142.667316\nINFO:root:Epoch[87] Time cost=6.068\nINFO:root:Epoch[88] Train-loss=142.461755\nINFO:root:Epoch[88] Time cost=6.111\nINFO:root:Epoch[89] Train-loss=142.270438\nINFO:root:Epoch[89] Time cost=6.221\nINFO:root:Epoch[90] Train-loss=142.047086\nINFO:root:Epoch[90] Time cost=8.061\nINFO:root:Epoch[91] Train-loss=141.855774\nINFO:root:Epoch[91] Time cost=6.433\nINFO:root:Epoch[92] Train-loss=141.688955\nINFO:root:Epoch[92] Time cost=7.153\nINFO:root:Epoch[93] Train-loss=141.442910\nINFO:root:Epoch[93] Time cost=7.113\nINFO:root:Epoch[94] Train-loss=141.279274\nINFO:root:Epoch[94] Time cost=7.152\nINFO:root:Epoch[95] Train-loss=141.086522\nINFO:root:Epoch[95] Time cost=6.472\nINFO:root:Epoch[96] Train-loss=140.901925\nINFO:root:Epoch[96] Time cost=6.767\nINFO:root:Epoch[97] Train-loss=140.722496\nINFO:root:Epoch[97] Time cost=7.044\nINFO:root:Epoch[98] Train-loss=140.579295\nINFO:root:Epoch[98] Time cost=7.040\nINFO:root:Epoch[99] Train-loss=140.386067\nINFO:root:Epoch[99] Time cost=6.669\n"
],
[
"ELBO = [-training_loss[i] for i in range(len(training_loss))]\nplt.plot(ELBO)\nplt.ylabel('ELBO');plt.xlabel('epoch');plt.title(\"training curve for mini batches\")\nplt.show()",
"_____no_output_____"
]
],
[
[
"As expected, the ELBO is monotonically increasing over epoch, and we reproduced the results given in the paper [Auto-Encoding Variational Bayes](https://arxiv.org/abs/1312.6114/). Now we can extract/load the parameters and then feed the network forward to calculate $y$ which is the reconstructed image, and we can also calculate the ELBO for the test set. ",
"_____no_output_____"
]
],
[
[
"arg_params = model.get_params()[0]\n\n# if saved the parameters, can load them using `load_checkpoint` method at e.g. 100th epoch\n# sym, arg_params, aux_params = mx.model.load_checkpoint(model_prefix, 100)\n# assert sym.tojson() == output.tojson()\n\ne = y.bind(mx.cpu(), {'data': nd_iter_test.data[0][1],\n 'encoder_h_weight': arg_params['encoder_h_weight'],\n 'encoder_h_bias': arg_params['encoder_h_bias'],\n 'mu_weight': arg_params['mu_weight'],\n 'mu_bias': arg_params['mu_bias'],\n 'logvar_weight':arg_params['logvar_weight'],\n 'logvar_bias':arg_params['logvar_bias'],\n 'decoder_z_weight':arg_params['decoder_z_weight'],\n 'decoder_z_bias':arg_params['decoder_z_bias'],\n 'decoder_x_weight':arg_params['decoder_x_weight'],\n 'decoder_x_bias':arg_params['decoder_x_bias'], \n 'loss_label':label})\n\nx_fit = e.forward()\nx_construction = x_fit[0].asnumpy()",
"_____no_output_____"
],
[
"# learning images on the test set\nf, ((ax1, ax2, ax3, ax4)) = plt.subplots(1,4, sharex='col', sharey='row',figsize=(12,3))\nax1.imshow(np.reshape(image_test[0,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax1.set_title('True image')\nax2.imshow(np.reshape(x_construction[0,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax2.set_title('Learned image')\nax3.imshow(np.reshape(x_construction[999,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax3.set_title('Learned image')\nax4.imshow(np.reshape(x_construction[9999,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax4.set_title('Learned image')\nplt.show()",
"_____no_output_____"
],
[
"# calculate the ELBO which is minus the loss for test set\nmetric = mx.metric.Loss()\nmodel.score(nd_iter_test, metric)",
"_____no_output_____"
]
],
[
[
"## 4. All together: MXNet-based class VAE",
"_____no_output_____"
]
],
[
[
"from VAE import VAE",
"_____no_output_____"
]
],
[
[
"One can directly call the class `VAE` to do the training:\n\n```VAE(n_latent=5,num_hidden_ecoder=400,num_hidden_decoder=400,x_train=None,x_valid=None,\nbatch_size=100,learning_rate=0.001,weight_decay=0.01,num_epoch=100,optimizer='sgd',model_prefix=None,\ninitializer = mx.init.Normal(0.01),likelihood=Bernoulli)```\n\nThe outputs are the learned model and training loss.",
"_____no_output_____"
]
],
[
[
"# can initilize weights and biases with the learned parameters as follows: \n# init = mx.initializer.Load(params)\n\n# call the VAE, output model contains the learned model and training loss\nout = VAE(n_latent=2, x_train=image, x_valid=None, num_epoch=200) ",
"INFO:root:Epoch[0] Train-loss=377.146422\nINFO:root:Epoch[0] Time cost=5.989\nINFO:root:Epoch[1] Train-loss=211.998043\nINFO:root:Epoch[1] Time cost=6.303\nINFO:root:Epoch[2] Train-loss=207.103096\nINFO:root:Epoch[2] Time cost=7.368\nINFO:root:Epoch[3] Train-loss=204.958183\nINFO:root:Epoch[3] Time cost=7.530\nINFO:root:Epoch[4] Train-loss=203.342700\nINFO:root:Epoch[4] Time cost=8.887\nINFO:root:Epoch[5] Train-loss=201.649251\nINFO:root:Epoch[5] Time cost=9.147\nINFO:root:Epoch[6] Train-loss=199.782661\nINFO:root:Epoch[6] Time cost=8.924\nINFO:root:Epoch[7] Train-loss=198.044015\nINFO:root:Epoch[7] Time cost=8.920\nINFO:root:Epoch[8] Train-loss=195.732077\nINFO:root:Epoch[8] Time cost=8.857\nINFO:root:Epoch[9] Train-loss=194.070547\nINFO:root:Epoch[9] Time cost=9.216\nINFO:root:Epoch[10] Train-loss=193.186871\nINFO:root:Epoch[10] Time cost=8.966\nINFO:root:Epoch[11] Train-loss=192.700208\nINFO:root:Epoch[11] Time cost=8.843\nINFO:root:Epoch[12] Train-loss=192.191504\nINFO:root:Epoch[12] Time cost=8.152\nINFO:root:Epoch[13] Train-loss=191.842837\nINFO:root:Epoch[13] Time cost=6.180\nINFO:root:Epoch[14] Train-loss=191.310450\nINFO:root:Epoch[14] Time cost=6.067\nINFO:root:Epoch[15] Train-loss=190.520681\nINFO:root:Epoch[15] Time cost=6.058\nINFO:root:Epoch[16] Train-loss=189.784146\nINFO:root:Epoch[16] Time cost=6.046\nINFO:root:Epoch[17] Train-loss=188.515020\nINFO:root:Epoch[17] Time cost=6.062\nINFO:root:Epoch[18] Train-loss=187.530712\nINFO:root:Epoch[18] Time cost=6.088\nINFO:root:Epoch[19] Train-loss=186.194826\nINFO:root:Epoch[19] Time cost=6.491\nINFO:root:Epoch[20] Train-loss=185.492288\nINFO:root:Epoch[20] Time cost=6.182\nINFO:root:Epoch[21] Train-loss=184.922654\nINFO:root:Epoch[21] Time cost=6.058\nINFO:root:Epoch[22] Train-loss=184.677911\nINFO:root:Epoch[22] Time cost=6.042\nINFO:root:Epoch[23] Train-loss=183.921396\nINFO:root:Epoch[23] Time cost=5.994\nINFO:root:Epoch[24] Train-loss=183.600690\nINFO:root:Epoch[24] Time cost=6.038\nINFO:root:Epoch[25] Train-loss=183.388476\nINFO:root:Epoch[25] Time cost=6.025\nINFO:root:Epoch[26] Train-loss=182.972208\nINFO:root:Epoch[26] Time cost=6.014\nINFO:root:Epoch[27] Train-loss=182.561678\nINFO:root:Epoch[27] Time cost=6.064\nINFO:root:Epoch[28] Train-loss=182.475261\nINFO:root:Epoch[28] Time cost=5.983\nINFO:root:Epoch[29] Train-loss=182.308808\nINFO:root:Epoch[29] Time cost=6.371\nINFO:root:Epoch[30] Train-loss=182.135900\nINFO:root:Epoch[30] Time cost=6.038\nINFO:root:Epoch[31] Train-loss=181.978367\nINFO:root:Epoch[31] Time cost=6.924\nINFO:root:Epoch[32] Train-loss=181.677153\nINFO:root:Epoch[32] Time cost=8.205\nINFO:root:Epoch[33] Train-loss=181.677775\nINFO:root:Epoch[33] Time cost=6.017\nINFO:root:Epoch[34] Train-loss=181.257998\nINFO:root:Epoch[34] Time cost=6.056\nINFO:root:Epoch[35] Train-loss=181.125288\nINFO:root:Epoch[35] Time cost=6.020\nINFO:root:Epoch[36] Train-loss=181.018858\nINFO:root:Epoch[36] Time cost=6.035\nINFO:root:Epoch[37] Train-loss=180.785110\nINFO:root:Epoch[37] Time cost=6.049\nINFO:root:Epoch[38] Train-loss=180.452598\nINFO:root:Epoch[38] Time cost=6.083\nINFO:root:Epoch[39] Train-loss=180.362733\nINFO:root:Epoch[39] Time cost=6.198\nINFO:root:Epoch[40] Train-loss=180.060788\nINFO:root:Epoch[40] Time cost=6.049\nINFO:root:Epoch[41] Train-loss=180.022728\nINFO:root:Epoch[41] Time cost=6.135\nINFO:root:Epoch[42] Train-loss=179.648499\nINFO:root:Epoch[42] Time cost=6.055\nINFO:root:Epoch[43] Train-loss=179.507952\nINFO:root:Epoch[43] Time cost=6.108\nINFO:root:Epoch[44] Train-loss=179.303132\nINFO:root:Epoch[44] Time cost=6.020\nINFO:root:Epoch[45] Train-loss=178.945211\nINFO:root:Epoch[45] Time cost=6.004\nINFO:root:Epoch[46] Train-loss=178.808598\nINFO:root:Epoch[46] Time cost=6.016\nINFO:root:Epoch[47] Train-loss=178.550906\nINFO:root:Epoch[47] Time cost=6.050\nINFO:root:Epoch[48] Train-loss=178.403674\nINFO:root:Epoch[48] Time cost=6.115\nINFO:root:Epoch[49] Train-loss=178.237544\nINFO:root:Epoch[49] Time cost=6.004\nINFO:root:Epoch[50] Train-loss=178.033747\nINFO:root:Epoch[50] Time cost=6.051\nINFO:root:Epoch[51] Train-loss=177.802884\nINFO:root:Epoch[51] Time cost=6.028\nINFO:root:Epoch[52] Train-loss=177.533980\nINFO:root:Epoch[52] Time cost=6.052\nINFO:root:Epoch[53] Train-loss=177.490143\nINFO:root:Epoch[53] Time cost=6.019\nINFO:root:Epoch[54] Train-loss=177.136637\nINFO:root:Epoch[54] Time cost=6.014\nINFO:root:Epoch[55] Train-loss=177.062524\nINFO:root:Epoch[55] Time cost=6.024\nINFO:root:Epoch[56] Train-loss=176.869033\nINFO:root:Epoch[56] Time cost=6.065\nINFO:root:Epoch[57] Train-loss=176.704606\nINFO:root:Epoch[57] Time cost=6.037\nINFO:root:Epoch[58] Train-loss=176.470091\nINFO:root:Epoch[58] Time cost=6.012\nINFO:root:Epoch[59] Train-loss=176.261440\nINFO:root:Epoch[59] Time cost=6.215\nINFO:root:Epoch[60] Train-loss=176.133904\nINFO:root:Epoch[60] Time cost=6.042\nINFO:root:Epoch[61] Train-loss=175.941920\nINFO:root:Epoch[61] Time cost=6.000\nINFO:root:Epoch[62] Train-loss=175.731296\nINFO:root:Epoch[62] Time cost=6.025\nINFO:root:Epoch[63] Train-loss=175.613303\nINFO:root:Epoch[63] Time cost=6.002\nINFO:root:Epoch[64] Train-loss=175.438844\nINFO:root:Epoch[64] Time cost=5.982\nINFO:root:Epoch[65] Train-loss=175.254716\nINFO:root:Epoch[65] Time cost=6.016\nINFO:root:Epoch[66] Train-loss=175.090210\nINFO:root:Epoch[66] Time cost=6.008\nINFO:root:Epoch[67] Train-loss=174.895443\nINFO:root:Epoch[67] Time cost=6.008\nINFO:root:Epoch[68] Train-loss=174.701321\nINFO:root:Epoch[68] Time cost=6.418\nINFO:root:Epoch[69] Train-loss=174.553292\nINFO:root:Epoch[69] Time cost=6.072\nINFO:root:Epoch[70] Train-loss=174.349379\nINFO:root:Epoch[70] Time cost=6.048\nINFO:root:Epoch[71] Train-loss=174.174641\nINFO:root:Epoch[71] Time cost=6.036\nINFO:root:Epoch[72] Train-loss=173.966333\nINFO:root:Epoch[72] Time cost=6.017\nINFO:root:Epoch[73] Train-loss=173.798454\nINFO:root:Epoch[73] Time cost=6.018\nINFO:root:Epoch[74] Train-loss=173.635657\nINFO:root:Epoch[74] Time cost=5.985\nINFO:root:Epoch[75] Train-loss=173.423795\nINFO:root:Epoch[75] Time cost=6.016\nINFO:root:Epoch[76] Train-loss=173.273981\nINFO:root:Epoch[76] Time cost=6.018\nINFO:root:Epoch[77] Train-loss=173.073401\nINFO:root:Epoch[77] Time cost=5.996\nINFO:root:Epoch[78] Train-loss=172.888044\nINFO:root:Epoch[78] Time cost=6.035\nINFO:root:Epoch[79] Train-loss=172.694943\nINFO:root:Epoch[79] Time cost=8.492\nINFO:root:Epoch[80] Train-loss=172.504260\nINFO:root:Epoch[80] Time cost=7.380\nINFO:root:Epoch[81] Train-loss=172.323245\nINFO:root:Epoch[81] Time cost=6.063\nINFO:root:Epoch[82] Train-loss=172.131274\nINFO:root:Epoch[82] Time cost=6.209\nINFO:root:Epoch[83] Train-loss=171.932986\nINFO:root:Epoch[83] Time cost=6.060\nINFO:root:Epoch[84] Train-loss=171.755262\nINFO:root:Epoch[84] Time cost=6.068\nINFO:root:Epoch[85] Train-loss=171.556803\nINFO:root:Epoch[85] Time cost=6.004\nINFO:root:Epoch[86] Train-loss=171.384773\nINFO:root:Epoch[86] Time cost=6.059\nINFO:root:Epoch[87] Train-loss=171.185034\nINFO:root:Epoch[87] Time cost=6.001\nINFO:root:Epoch[88] Train-loss=170.995980\nINFO:root:Epoch[88] Time cost=6.143\nINFO:root:Epoch[89] Train-loss=170.818701\nINFO:root:Epoch[89] Time cost=6.690\nINFO:root:Epoch[90] Train-loss=170.629929\nINFO:root:Epoch[90] Time cost=6.869\nINFO:root:Epoch[91] Train-loss=170.450824\nINFO:root:Epoch[91] Time cost=7.156\nINFO:root:Epoch[92] Train-loss=170.261806\nINFO:root:Epoch[92] Time cost=6.972\nINFO:root:Epoch[93] Train-loss=170.070318\nINFO:root:Epoch[93] Time cost=6.595\nINFO:root:Epoch[94] Train-loss=169.906993\nINFO:root:Epoch[94] Time cost=6.561\nINFO:root:Epoch[95] Train-loss=169.734455\nINFO:root:Epoch[95] Time cost=6.744\nINFO:root:Epoch[96] Train-loss=169.564318\nINFO:root:Epoch[96] Time cost=6.601\nINFO:root:Epoch[97] Train-loss=169.373926\nINFO:root:Epoch[97] Time cost=6.725\nINFO:root:Epoch[98] Train-loss=169.215408\nINFO:root:Epoch[98] Time cost=6.391\nINFO:root:Epoch[99] Train-loss=169.039854\nINFO:root:Epoch[99] Time cost=6.677\nINFO:root:Epoch[100] Train-loss=168.869222\nINFO:root:Epoch[100] Time cost=6.370\nINFO:root:Epoch[101] Train-loss=168.703175\nINFO:root:Epoch[101] Time cost=6.607\nINFO:root:Epoch[102] Train-loss=168.523054\nINFO:root:Epoch[102] Time cost=6.368\nINFO:root:Epoch[103] Train-loss=168.365964\nINFO:root:Epoch[103] Time cost=10.267\nINFO:root:Epoch[104] Train-loss=168.181174\nINFO:root:Epoch[104] Time cost=11.132\nINFO:root:Epoch[105] Train-loss=168.021498\nINFO:root:Epoch[105] Time cost=10.187\nINFO:root:Epoch[106] Train-loss=167.858251\nINFO:root:Epoch[106] Time cost=10.676\nINFO:root:Epoch[107] Train-loss=167.690670\nINFO:root:Epoch[107] Time cost=10.973\nINFO:root:Epoch[108] Train-loss=167.535069\nINFO:root:Epoch[108] Time cost=10.108\nINFO:root:Epoch[109] Train-loss=167.373971\nINFO:root:Epoch[109] Time cost=11.013\nINFO:root:Epoch[110] Train-loss=167.207507\nINFO:root:Epoch[110] Time cost=11.427\nINFO:root:Epoch[111] Train-loss=167.043077\nINFO:root:Epoch[111] Time cost=10.349\nINFO:root:Epoch[112] Train-loss=166.884060\nINFO:root:Epoch[112] Time cost=13.129\nINFO:root:Epoch[113] Train-loss=166.746976\nINFO:root:Epoch[113] Time cost=11.255\nINFO:root:Epoch[114] Train-loss=166.572499\nINFO:root:Epoch[114] Time cost=10.037\nINFO:root:Epoch[115] Train-loss=166.445170\nINFO:root:Epoch[115] Time cost=10.406\nINFO:root:Epoch[116] Train-loss=166.284912\nINFO:root:Epoch[116] Time cost=10.170\nINFO:root:Epoch[117] Train-loss=166.171475\nINFO:root:Epoch[117] Time cost=10.034\nINFO:root:Epoch[118] Train-loss=166.015457\nINFO:root:Epoch[118] Time cost=10.047\nINFO:root:Epoch[119] Train-loss=165.882208\nINFO:root:Epoch[119] Time cost=10.008\nINFO:root:Epoch[120] Train-loss=165.753836\nINFO:root:Epoch[120] Time cost=10.056\nINFO:root:Epoch[121] Train-loss=165.626045\nINFO:root:Epoch[121] Time cost=10.704\nINFO:root:Epoch[122] Train-loss=165.492859\nINFO:root:Epoch[122] Time cost=10.609\nINFO:root:Epoch[123] Train-loss=165.361132\nINFO:root:Epoch[123] Time cost=10.027\nINFO:root:Epoch[124] Train-loss=165.256487\nINFO:root:Epoch[124] Time cost=11.225\nINFO:root:Epoch[125] Train-loss=165.119995\nINFO:root:Epoch[125] Time cost=11.266\nINFO:root:Epoch[126] Train-loss=165.012773\nINFO:root:Epoch[126] Time cost=10.547\nINFO:root:Epoch[127] Train-loss=164.898748\nINFO:root:Epoch[127] Time cost=10.339\nINFO:root:Epoch[128] Train-loss=164.775702\nINFO:root:Epoch[128] Time cost=10.875\nINFO:root:Epoch[129] Train-loss=164.692449\nINFO:root:Epoch[129] Time cost=8.412\nINFO:root:Epoch[130] Train-loss=164.564323\nINFO:root:Epoch[130] Time cost=7.239\nINFO:root:Epoch[131] Train-loss=164.468273\nINFO:root:Epoch[131] Time cost=10.096\nINFO:root:Epoch[132] Train-loss=164.328320\nINFO:root:Epoch[132] Time cost=9.680\nINFO:root:Epoch[133] Train-loss=164.256156\nINFO:root:Epoch[133] Time cost=10.707\nINFO:root:Epoch[134] Train-loss=164.151625\nINFO:root:Epoch[134] Time cost=13.835\nINFO:root:Epoch[135] Train-loss=164.046402\nINFO:root:Epoch[135] Time cost=10.049\nINFO:root:Epoch[136] Train-loss=163.960676\nINFO:root:Epoch[136] Time cost=9.625\nINFO:root:Epoch[137] Train-loss=163.873193\nINFO:root:Epoch[137] Time cost=9.845\nINFO:root:Epoch[138] Train-loss=163.783837\nINFO:root:Epoch[138] Time cost=9.618\nINFO:root:Epoch[139] Train-loss=163.658903\nINFO:root:Epoch[139] Time cost=10.411\nINFO:root:Epoch[140] Train-loss=163.588920\nINFO:root:Epoch[140] Time cost=9.633\nINFO:root:Epoch[141] Train-loss=163.493254\nINFO:root:Epoch[141] Time cost=10.668\nINFO:root:Epoch[142] Train-loss=163.401188\nINFO:root:Epoch[142] Time cost=10.644\nINFO:root:Epoch[143] Train-loss=163.334470\nINFO:root:Epoch[143] Time cost=9.665\nINFO:root:Epoch[144] Train-loss=163.235133\nINFO:root:Epoch[144] Time cost=9.612\nINFO:root:Epoch[145] Train-loss=163.168029\nINFO:root:Epoch[145] Time cost=9.578\nINFO:root:Epoch[146] Train-loss=163.092392\nINFO:root:Epoch[146] Time cost=10.215\nINFO:root:Epoch[147] Train-loss=163.014362\nINFO:root:Epoch[147] Time cost=12.296\nINFO:root:Epoch[148] Train-loss=162.891574\nINFO:root:Epoch[148] Time cost=9.578\nINFO:root:Epoch[149] Train-loss=162.831664\nINFO:root:Epoch[149] Time cost=9.536\nINFO:root:Epoch[150] Train-loss=162.768784\nINFO:root:Epoch[150] Time cost=9.607\nINFO:root:Epoch[151] Train-loss=162.695416\nINFO:root:Epoch[151] Time cost=9.681\nINFO:root:Epoch[152] Train-loss=162.620814\nINFO:root:Epoch[152] Time cost=9.464\nINFO:root:Epoch[153] Train-loss=162.527031\nINFO:root:Epoch[153] Time cost=9.518\nINFO:root:Epoch[154] Train-loss=162.466575\nINFO:root:Epoch[154] Time cost=9.562\nINFO:root:Epoch[155] Train-loss=162.409388\nINFO:root:Epoch[155] Time cost=9.483\nINFO:root:Epoch[156] Train-loss=162.308957\nINFO:root:Epoch[156] Time cost=9.545\nINFO:root:Epoch[157] Train-loss=162.211725\nINFO:root:Epoch[157] Time cost=9.542\nINFO:root:Epoch[158] Train-loss=162.141098\nINFO:root:Epoch[158] Time cost=9.768\nINFO:root:Epoch[159] Train-loss=162.124311\nINFO:root:Epoch[159] Time cost=7.155\nINFO:root:Epoch[160] Train-loss=162.013039\nINFO:root:Epoch[160] Time cost=6.147\nINFO:root:Epoch[161] Train-loss=161.954485\nINFO:root:Epoch[161] Time cost=9.121\nINFO:root:Epoch[162] Train-loss=161.913859\nINFO:root:Epoch[162] Time cost=9.936\nINFO:root:Epoch[163] Train-loss=161.830799\nINFO:root:Epoch[163] Time cost=8.612\nINFO:root:Epoch[164] Train-loss=161.768672\nINFO:root:Epoch[164] Time cost=9.722\nINFO:root:Epoch[165] Train-loss=161.689120\nINFO:root:Epoch[165] Time cost=9.478\nINFO:root:Epoch[166] Train-loss=161.598279\nINFO:root:Epoch[166] Time cost=9.466\nINFO:root:Epoch[167] Train-loss=161.551172\nINFO:root:Epoch[167] Time cost=9.419\nINFO:root:Epoch[168] Train-loss=161.488880\nINFO:root:Epoch[168] Time cost=9.457\nINFO:root:Epoch[169] Train-loss=161.410458\nINFO:root:Epoch[169] Time cost=9.504\nINFO:root:Epoch[170] Train-loss=161.340681\nINFO:root:Epoch[170] Time cost=9.866\nINFO:root:Epoch[171] Train-loss=161.281700\nINFO:root:Epoch[171] Time cost=9.526\nINFO:root:Epoch[172] Train-loss=161.215523\nINFO:root:Epoch[172] Time cost=9.511\nINFO:root:Epoch[173] Train-loss=161.152452\nINFO:root:Epoch[173] Time cost=9.498\nINFO:root:Epoch[174] Train-loss=161.058544\nINFO:root:Epoch[174] Time cost=9.561\nINFO:root:Epoch[175] Train-loss=161.036475\nINFO:root:Epoch[175] Time cost=9.463\nINFO:root:Epoch[176] Train-loss=161.009996\nINFO:root:Epoch[176] Time cost=9.629\nINFO:root:Epoch[177] Train-loss=160.853546\nINFO:root:Epoch[177] Time cost=9.518\nINFO:root:Epoch[178] Train-loss=160.860520\nINFO:root:Epoch[178] Time cost=9.395\nINFO:root:Epoch[179] Train-loss=160.810621\nINFO:root:Epoch[179] Time cost=9.452\nINFO:root:Epoch[180] Train-loss=160.683071\nINFO:root:Epoch[180] Time cost=9.411\nINFO:root:Epoch[181] Train-loss=160.674101\nINFO:root:Epoch[181] Time cost=8.784\nINFO:root:Epoch[182] Train-loss=160.554823\nINFO:root:Epoch[182] Time cost=7.265\nINFO:root:Epoch[183] Train-loss=160.536528\nINFO:root:Epoch[183] Time cost=6.108\nINFO:root:Epoch[184] Train-loss=160.525913\nINFO:root:Epoch[184] Time cost=6.349\nINFO:root:Epoch[185] Train-loss=160.399412\nINFO:root:Epoch[185] Time cost=7.364\nINFO:root:Epoch[186] Train-loss=160.380027\nINFO:root:Epoch[186] Time cost=7.651\nINFO:root:Epoch[187] Train-loss=160.272921\nINFO:root:Epoch[187] Time cost=7.309\nINFO:root:Epoch[188] Train-loss=160.243907\nINFO:root:Epoch[188] Time cost=7.162\nINFO:root:Epoch[189] Train-loss=160.194351\nINFO:root:Epoch[189] Time cost=8.941\nINFO:root:Epoch[190] Train-loss=160.130400\nINFO:root:Epoch[190] Time cost=10.242\nINFO:root:Epoch[191] Train-loss=160.073841\nINFO:root:Epoch[191] Time cost=10.528\nINFO:root:Epoch[192] Train-loss=160.021623\nINFO:root:Epoch[192] Time cost=9.482\nINFO:root:Epoch[193] Train-loss=159.938673\nINFO:root:Epoch[193] Time cost=9.465\nINFO:root:Epoch[194] Train-loss=159.885823\nINFO:root:Epoch[194] Time cost=9.523\nINFO:root:Epoch[195] Train-loss=159.886516\nINFO:root:Epoch[195] Time cost=9.599\nINFO:root:Epoch[196] Train-loss=159.797400\nINFO:root:Epoch[196] Time cost=8.675\nINFO:root:Epoch[197] Train-loss=159.705562\nINFO:root:Epoch[197] Time cost=9.551\nINFO:root:Epoch[198] Train-loss=159.738354\nINFO:root:Epoch[198] Time cost=9.919\nINFO:root:Epoch[199] Train-loss=159.619932\nINFO:root:Epoch[199] Time cost=10.121\n"
],
[
"# encode test images to obtain mu and logvar which are used for sampling\n[mu,logvar] = VAE.encoder(out,image_test)\n# sample in the latent space\nz = VAE.sampler(mu,logvar)\n# decode from the latent space to obtain reconstructed images\nx_construction = VAE.decoder(out,z)\n",
"_____no_output_____"
],
[
"f, ((ax1, ax2, ax3, ax4)) = plt.subplots(1,4, sharex='col', sharey='row',figsize=(12,3))\nax1.imshow(np.reshape(image_test[0,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax1.set_title('True image')\nax2.imshow(np.reshape(x_construction[0,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax2.set_title('Learned image')\nax3.imshow(np.reshape(x_construction[999,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax3.set_title('Learned image')\nax4.imshow(np.reshape(x_construction[9999,:],(28,28)), interpolation='nearest', cmap=cm.Greys)\nax4.set_title('Learned image')\nplt.show()",
"_____no_output_____"
],
[
"z1 = z[:,0]\nz2 = z[:,1]\n\nfig = plt.figure()\nax = fig.add_subplot(111)\nax.plot(z1,z2,'ko')\nplt.title(\"latent space\")\n\n#np.where((z1>3) & (z2<2) & (z2>0))\n#select the points from the latent space\na_vec = [2,5,7,789,25,9993]\nfor i in range(len(a_vec)):\n ax.plot(z1[a_vec[i]],z2[a_vec[i]],'ro') \n ax.annotate('z%d' %i, xy=(z1[a_vec[i]],z2[a_vec[i]]), \n xytext=(z1[a_vec[i]],z2[a_vec[i]]),color = 'r',fontsize=15)\n\n\nf, ((ax0, ax1, ax2, ax3, ax4,ax5)) = plt.subplots(1,6, sharex='col', sharey='row',figsize=(12,2.5))\nfor i in range(len(a_vec)):\n eval('ax%d' %(i)).imshow(np.reshape(x_construction[a_vec[i],:],(28,28)), interpolation='nearest', cmap=cm.Greys)\n eval('ax%d' %(i)).set_title('z%d'%i)\n\nplt.show()",
"_____no_output_____"
]
],
[
[
"Above is a plot of points in the 2D latent space and their corresponding decoded images, it can be seen that points that are close in the latent space get mapped to the same digit from the decoder, and we can see how it evolves from left to right.",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
]
] |
e7ec187b0ee25e3c74252060b24f3d8c0a1275ac | 294,601 | ipynb | Jupyter Notebook | Qiskit Textbook Solutions/Chapter 1/1.4 - Single Qubit Gates.ipynb | kj3moraes/MyQiskitProgramming | 76a83b1463331d6d88d9e0b07fff1258715c195c | [
"MIT"
] | 2 | 2020-12-04T18:02:52.000Z | 2020-12-06T11:45:47.000Z | Qiskit Textbook Solutions/Chapter 1/1.4 - Single Qubit Gates.ipynb | kj3moraes/MyQiskitProgramming | 76a83b1463331d6d88d9e0b07fff1258715c195c | [
"MIT"
] | null | null | null | Qiskit Textbook Solutions/Chapter 1/1.4 - Single Qubit Gates.ipynb | kj3moraes/MyQiskitProgramming | 76a83b1463331d6d88d9e0b07fff1258715c195c | [
"MIT"
] | null | null | null | 375.766582 | 49,432 | 0.942366 | [
[
[
"%matplotlib inline\n# Importing standard Qiskit libraries\nfrom qiskit import QuantumCircuit, execute, Aer, IBMQ\nfrom qiskit.compiler import transpile, assemble\nfrom qiskit.visualization import plot_bloch_multivector, plot_histogram\nfrom qiskit.visualization import *\nfrom math import sqrt, pi\n\n# Loading your IBM Q account(s)\nprovider = IBMQ.load_account()",
"_____no_output_____"
]
],
[
[
"## Quick Exercises 1 ",
"_____no_output_____"
],
[
"### 1. Verify that |+⟩ and |−⟩ are in fact eigenstates of the X-gate.",
"_____no_output_____"
],
[
"First we need to define the |+⟩ and |−⟩ states in 2 different qubits. I will initalize the first qubit to 1 and the second to 0.",
"_____no_output_____"
]
],
[
[
"qCirc1 = QuantumCircuit(2, 2)\noneInit = [0, 1]\nqCirc1.initialize(oneInit, 0)\nzeroInit = [1, 0]\nqCirc1.initialize(zeroInit, 1)\nqCirc1.draw('mpl')",
"_____no_output_____"
],
[
"qCirc1.h(0)\nqCirc1.h(1)\nqCirc1.draw('mpl')",
"_____no_output_____"
]
],
[
[
"Now the first qubit is in the |-⟩ and the second qubit is in the |+⟩ state. We can now apply the X gates. If the |+⟩ and |−⟩ states are really eigenstates then a reapplication of the Hadamard gates and a measurement should give |0⟩ and |1⟩.",
"_____no_output_____"
]
],
[
[
"qCirc1.x([0,1])\nqCirc1.h([0,1])\nqCirc1.draw('mpl')",
"_____no_output_____"
],
[
"qCirc1.measure(0, 0)\nqCirc1.measure(1, 1)\nqCirc1.draw('mpl')",
"_____no_output_____"
]
],
[
[
"#### Simulating this circuit on QASM\n",
"_____no_output_____"
]
],
[
[
"nativeSim = Aer.get_backend('qasm_simulator')\nresult = execute(qCirc1, backend = nativeSim).result()\nplot_histogram(result.get_counts(qCirc1))",
"_____no_output_____"
]
],
[
[
"This proves the property we were looking for",
"_____no_output_____"
],
[
"### 4. Find the eigenstates of the Y-gate, and their co-ordinates on the Bloch sphere.",
"_____no_output_____"
],
[
"The eigenstates of the Y gate will simply be the y-basis vectors because the are unaffected by a rotation of $\\pi$ about the y-axis. These are \n$$ \\frac{1}{\\sqrt{2}}(|0⟩ + i|1⟩) \\text{ and } \\frac{1}{\\sqrt{2}}(|0⟩ - i|1⟩) $$",
"_____no_output_____"
],
[
"### Experiment with Pauli Y Gate",
"_____no_output_____"
],
[
"The following experiment is to test out the x-axis basis vectors under a y-axis transformation",
"_____no_output_____"
]
],
[
[
"qCircTest = QuantumCircuit(2, 2);\nqCircTest.initialize(zeroInit, 0);\nqCircTest.initialize(oneInit, 1)\nqCircTest.draw('mpl')",
"_____no_output_____"
],
[
"qCircTest.y([0,1])\nqCircTest.measure([0, 1], [0, 1])\nqCircTest.draw('mpl')",
"_____no_output_____"
],
[
"result = execute(qCircTest, backend = nativeSim, shots = 1024).result()\nplot_histogram(result.get_counts(qCircTest))",
"_____no_output_____"
]
],
[
[
"## Quick Exercises 2",
"_____no_output_____"
],
[
"### 2. Show that applying the sequence of gates: HZH, to any qubit state is equivalent to applying an X-gate.",
"_____no_output_____"
],
[
"Here we will define another circuit with 1 qubit and show that a HZH transforms |0⟩ to |1⟩ and |1⟩ and |0⟩. ",
"_____no_output_____"
]
],
[
[
"qCirc2 = QuantumCircuit(1, 1)\nqCirc2.initialize(zeroInit, 0)\nqCirc2.draw('mpl')",
"_____no_output_____"
],
[
"qCirc2.h(0)\nqCirc2.z(0)\nqCirc2.h(0)\nqCirc2.measure(0, 0)\nqCirc2.draw('mpl')",
"_____no_output_____"
]
],
[
[
"#### Simulating this circuit on QASM\n",
"_____no_output_____"
]
],
[
[
"result2 = execute(qCirc2, backend = nativeSim, shots = 1024).result()\nplot_histogram(result2.get_counts(qCirc2))",
"_____no_output_____"
],
[
"qCirc2 = QuantumCircuit(1, 1)\nqCirc2.initialize(oneInit, 0)\nqCirc2.draw('mpl')",
"_____no_output_____"
],
[
"qCirc2.h(0)\nqCirc2.z(0)\nqCirc2.h(0)\nqCirc2.measure(0, 0)\nqCirc2.draw('mpl')",
"_____no_output_____"
],
[
"result2 = execute(qCirc2, backend = nativeSim, shots = 1024).result()\nplot_histogram(result2.get_counts(qCirc2))",
"_____no_output_____"
]
],
[
[
"### 3. Find a combination of X, Z and H-gates that is equivalent to a Y-gate (ignoring global phase).",
"_____no_output_____"
],
[
"As we saw before, the Y gate does not affect the |0⟩ and |1⟩ basis vectors. Since we can ignore the global phase, we can take $-i$ outside. This yeilds the matrix\n\n\n$$ \\sigma_Y = -i \\begin{bmatrix} 0 & 1 \\\\ -1 & 0 \\end{bmatrix} $$\n\nBy shear trial and error the matrix given is ZX.",
"_____no_output_____"
]
],
[
[
"qCirc23 = QuantumCircuit(1, 1)\nqCirc23.initialize([1/sqrt(2), 1j/sqrt(2)], 0)\nqCirc23.draw('mpl')",
"_____no_output_____"
],
[
"svSim = Aer.get_backend('statevector_simulator')\nsvResult = execute(qCirc23, backend = svSim, shots = 1024).result()\nstateVectorBefore = svResult.get_statevector()\nplot_bloch_multivector(stateVectorBefore)",
"_____no_output_____"
],
[
"qCirc23.z(0)\nqCirc23.x(0)\nqCirc23.draw('mpl')",
"_____no_output_____"
],
[
"svResult = execute(qCirc23, backend = svSim, shots = 1024).result()\nstateVectorAfter = svResult.get_statevector()\nplot_bloch_multivector(stateVectorAfter)",
"_____no_output_____"
],
[
"qCirc23.measure(0, 0)\nresult = execute(qCirc23, backend = nativeSim, shots = 1024).result()\nplot_histogram(result.get_counts(qCirc23))",
"_____no_output_____"
]
],
[
[
"## Quick Exercises 3",
"_____no_output_____"
],
[
"### 1. If we initialise our qubit in the state |+⟩, what is the probability of measuring it in state |−⟩?",
"_____no_output_____"
],
[
"The probability would be 0.",
"_____no_output_____"
],
[
"### 2. Use Qiskit to display the probability of measuring a |0⟩ qubit in the states |+⟩ and |−⟩",
"_____no_output_____"
]
],
[
[
"qCirc32 = QuantumCircuit(1, 1) \nqCirc32.initialize([1/sqrt(2), 1/sqrt(2)], 0)\nqCirc32.measure(0, 0)\nqCirc32.draw('mpl')",
"_____no_output_____"
],
[
"result = execute(qCirc32, backend = nativeSim, shots = 1024).result()\nplot_histogram(result.get_counts(qCirc32))",
"_____no_output_____"
]
],
[
[
"### 3. Try to create a function that measures in the Y-basis.",
"_____no_output_____"
],
[
"To do this we would need to \n1. Convert Y-basis into Z-basis (this can simply be done by rotating the x-axis by $ \\pi/2$ )\n2. Use Qiskit measurement to measure in Z-basis\n3. Convert back into Y-basis (rotate the x-axis by $ -\\pi/2$)",
"_____no_output_____"
]
],
[
[
"def measure_y(qc, qubit, cbit):\n # STEP 1\n qc.sdg(qubit)\n qc.h(qubit)\n \n # STEP 2\n qc.measure(qubit, cbit)\n \n # STEP 3\n qc.h(qubit)\n qc.s(qubit)",
"_____no_output_____"
],
[
"circuit = QuantumCircuit(1, 1)\noutwardInit = [1/sqrt(2), -1j/sqrt(2)]\ncircuit.initialize(outwardInit, 0)\nmeasure_y(circuit, 0 ,0)\ncircuit.draw('mpl')",
"_____no_output_____"
],
[
"result = execute(circuit, backend = nativeSim, shots = 1024).result()\nplot_histogram(result.get_counts(circuit))",
"_____no_output_____"
],
[
"svResult = execute(circuit, backend = svSim, shots = 1024).result()\nplot_bloch_multivector(svResult.get_statevector())",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown",
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code"
]
] |
e7ec26bea66f988135ab1779ada4e893f8dbd8a2 | 217,171 | ipynb | Jupyter Notebook | 2.Improving Deep Neural Networks/Week 3/TensorFlow_Tutorial_v3b.ipynb | thesauravkarmakar/deeplearning.ai | 1758daed135e187f545e0ead6dbad5a44e5c3165 | [
"MIT"
] | null | null | null | 2.Improving Deep Neural Networks/Week 3/TensorFlow_Tutorial_v3b.ipynb | thesauravkarmakar/deeplearning.ai | 1758daed135e187f545e0ead6dbad5a44e5c3165 | [
"MIT"
] | null | null | null | 2.Improving Deep Neural Networks/Week 3/TensorFlow_Tutorial_v3b.ipynb | thesauravkarmakar/deeplearning.ai | 1758daed135e187f545e0ead6dbad5a44e5c3165 | [
"MIT"
] | null | null | null | 115.824533 | 118,292 | 0.833293 | [
[
[
"# TensorFlow Tutorial\n\nWelcome to this week's programming assignment. Until now, you've always used numpy to build neural networks. Now we will step you through a deep learning framework that will allow you to build neural networks more easily. Machine learning frameworks like TensorFlow, PaddlePaddle, Torch, Caffe, Keras, and many others can speed up your machine learning development significantly. All of these frameworks also have a lot of documentation, which you should feel free to read. In this assignment, you will learn to do the following in TensorFlow: \n\n- Initialize variables\n- Start your own session\n- Train algorithms \n- Implement a Neural Network\n\nPrograming frameworks can not only shorten your coding time, but sometimes also perform optimizations that speed up your code. ",
"_____no_output_____"
],
[
"## <font color='darkblue'>Updates</font>\n\n#### If you were working on the notebook before this update...\n* The current notebook is version \"v3b\".\n* You can find your original work saved in the notebook with the previous version name (it may be either TensorFlow Tutorial version 3\" or \"TensorFlow Tutorial version 3a.) \n* To view the file directory, click on the \"Coursera\" icon in the top left of this notebook.\n\n#### List of updates\n* forward_propagation instruction now says 'A1' instead of 'a1' in the formula for Z2; \n and are updated to say 'A2' instead of 'Z2' in the formula for Z3.\n* create_placeholders instruction refer to the data type \"tf.float32\" instead of float.\n* in the model function, the x axis of the plot now says \"iterations (per fives)\" instead of iterations(per tens)\n* In the linear_function, comments remind students to create the variables in the order suggested by the starter code. The comments are updated to reflect this order.\n* The test of the cost function now creates the logits without passing them through a sigmoid function (since the cost function will include the sigmoid in the built-in tensorflow function).\n* In the 'model' function, the minibatch_cost is now divided by minibatch_size (instead of num_minibatches).\n* Updated print statements and 'expected output that are used to check functions, for easier visual comparison.\n",
"_____no_output_____"
],
[
"## 1 - Exploring the Tensorflow Library\n\nTo start, you will import the library:",
"_____no_output_____"
]
],
[
[
"import math\nimport numpy as np\nimport h5py\nimport matplotlib.pyplot as plt\nimport tensorflow as tf\nfrom tensorflow.python.framework import ops\nfrom tf_utils import load_dataset, random_mini_batches, convert_to_one_hot, predict\n\n%matplotlib inline\nnp.random.seed(1)",
"_____no_output_____"
]
],
[
[
"Now that you have imported the library, we will walk you through its different applications. You will start with an example, where we compute for you the loss of one training example. \n$$loss = \\mathcal{L}(\\hat{y}, y) = (\\hat y^{(i)} - y^{(i)})^2 \\tag{1}$$",
"_____no_output_____"
]
],
[
[
"y_hat = tf.constant(36, name='y_hat') # Define y_hat constant. Set to 36.\ny = tf.constant(39, name='y') # Define y. Set to 39\n\nloss = tf.Variable((y - y_hat)**2, name='loss') # Create a variable for the loss\n\ninit = tf.global_variables_initializer() # When init is run later (session.run(init)),\n # the loss variable will be initialized and ready to be computed\nwith tf.Session() as session: # Create a session and print the output\n session.run(init) # Initializes the variables\n print(session.run(loss)) # Prints the loss",
"9\n"
]
],
[
[
"Writing and running programs in TensorFlow has the following steps:\n\n1. Create Tensors (variables) that are not yet executed/evaluated. \n2. Write operations between those Tensors.\n3. Initialize your Tensors. \n4. Create a Session. \n5. Run the Session. This will run the operations you'd written above. \n\nTherefore, when we created a variable for the loss, we simply defined the loss as a function of other quantities, but did not evaluate its value. To evaluate it, we had to run `init=tf.global_variables_initializer()`. That initialized the loss variable, and in the last line we were finally able to evaluate the value of `loss` and print its value.\n\nNow let us look at an easy example. Run the cell below:",
"_____no_output_____"
]
],
[
[
"a = tf.constant(2)\nb = tf.constant(10)\nc = tf.multiply(a,b)\nprint(c)",
"Tensor(\"Mul:0\", shape=(), dtype=int32)\n"
]
],
[
[
"As expected, you will not see 20! You got a tensor saying that the result is a tensor that does not have the shape attribute, and is of type \"int32\". All you did was put in the 'computation graph', but you have not run this computation yet. In order to actually multiply the two numbers, you will have to create a session and run it.",
"_____no_output_____"
]
],
[
[
"sess = tf.Session()\nprint(sess.run(c))",
"20\n"
]
],
[
[
"Great! To summarize, **remember to initialize your variables, create a session and run the operations inside the session**. \n\nNext, you'll also have to know about placeholders. A placeholder is an object whose value you can specify only later. \nTo specify values for a placeholder, you can pass in values by using a \"feed dictionary\" (`feed_dict` variable). Below, we created a placeholder for x. This allows us to pass in a number later when we run the session. ",
"_____no_output_____"
]
],
[
[
"# Change the value of x in the feed_dict\n\nx = tf.placeholder(tf.int64, name = 'x')\nprint(sess.run(2 * x, feed_dict = {x: 3}))\nsess.close()",
"6\n"
]
],
[
[
"When you first defined `x` you did not have to specify a value for it. A placeholder is simply a variable that you will assign data to only later, when running the session. We say that you **feed data** to these placeholders when running the session. \n\nHere's what's happening: When you specify the operations needed for a computation, you are telling TensorFlow how to construct a computation graph. The computation graph can have some placeholders whose values you will specify only later. Finally, when you run the session, you are telling TensorFlow to execute the computation graph.",
"_____no_output_____"
],
[
"### 1.1 - Linear function\n\nLets start this programming exercise by computing the following equation: $Y = WX + b$, where $W$ and $X$ are random matrices and b is a random vector. \n\n**Exercise**: Compute $WX + b$ where $W, X$, and $b$ are drawn from a random normal distribution. W is of shape (4, 3), X is (3,1) and b is (4,1). As an example, here is how you would define a constant X that has shape (3,1):\n```python\nX = tf.constant(np.random.randn(3,1), name = \"X\")\n\n```\nYou might find the following functions helpful: \n- tf.matmul(..., ...) to do a matrix multiplication\n- tf.add(..., ...) to do an addition\n- np.random.randn(...) to initialize randomly\n",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: linear_function\n\ndef linear_function():\n \"\"\"\n Implements a linear function: \n Initializes X to be a random tensor of shape (3,1)\n Initializes W to be a random tensor of shape (4,3)\n Initializes b to be a random tensor of shape (4,1)\n Returns: \n result -- runs the session for Y = WX + b \n \"\"\"\n \n np.random.seed(1)\n \n \"\"\"\n Note, to ensure that the \"random\" numbers generated match the expected results,\n please create the variables in the order given in the starting code below.\n (Do not re-arrange the order).\n \"\"\"\n ### START CODE HERE ### (4 lines of code)\n X = tf.constant(np.random.randn(3,1), name = \"X\")\n W = tf.constant(np.random.randn(4,3), name = \"W\")\n b = tf.constant(np.random.randn(4,1), name = \"b\")\n Y = tf.add(tf.matmul(W,X),b)\n ### END CODE HERE ### \n \n # Create the session using tf.Session() and run it with sess.run(...) on the variable you want to calculate\n \n ### START CODE HERE ###\n sess = tf.Session()\n result = sess.run(Y)\n ### END CODE HERE ### \n \n # close the session \n sess.close()\n\n return result",
"_____no_output_____"
],
[
"print( \"result = \\n\" + str(linear_function()))",
"result = \n[[-2.15657382]\n [ 2.95891446]\n [-1.08926781]\n [-0.84538042]]\n"
]
],
[
[
"*** Expected Output ***: \n\n```\nresult = \n[[-2.15657382]\n [ 2.95891446]\n [-1.08926781]\n [-0.84538042]]\n```",
"_____no_output_____"
],
[
"### 1.2 - Computing the sigmoid \nGreat! You just implemented a linear function. Tensorflow offers a variety of commonly used neural network functions like `tf.sigmoid` and `tf.softmax`. For this exercise lets compute the sigmoid function of an input. \n\nYou will do this exercise using a placeholder variable `x`. When running the session, you should use the feed dictionary to pass in the input `z`. In this exercise, you will have to (i) create a placeholder `x`, (ii) define the operations needed to compute the sigmoid using `tf.sigmoid`, and then (iii) run the session. \n\n** Exercise **: Implement the sigmoid function below. You should use the following: \n\n- `tf.placeholder(tf.float32, name = \"...\")`\n- `tf.sigmoid(...)`\n- `sess.run(..., feed_dict = {x: z})`\n\n\nNote that there are two typical ways to create and use sessions in tensorflow: \n\n**Method 1:**\n```python\nsess = tf.Session()\n# Run the variables initialization (if needed), run the operations\nresult = sess.run(..., feed_dict = {...})\nsess.close() # Close the session\n```\n**Method 2:**\n```python\nwith tf.Session() as sess: \n # run the variables initialization (if needed), run the operations\n result = sess.run(..., feed_dict = {...})\n # This takes care of closing the session for you :)\n```\n",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: sigmoid\n\ndef sigmoid(z):\n \"\"\"\n Computes the sigmoid of z\n \n Arguments:\n z -- input value, scalar or vector\n \n Returns: \n results -- the sigmoid of z\n \"\"\"\n \n ### START CODE HERE ### ( approx. 4 lines of code)\n # Create a placeholder for x. Name it 'x'.\n x = tf.placeholder(tf.float32, name = \"x\")\n\n # compute sigmoid(x)\n sigmoid = tf.sigmoid(x)\n\n # Create a session, and run it. Please use the method 2 explained above. \n # You should use a feed_dict to pass z's value to x. \n None\n # Run session and call the output \"result\"\n with tf.Session() as sess:\n result = sess.run(sigmoid, feed_dict = {x:z})\n \n ### END CODE HERE ###\n \n return result",
"_____no_output_____"
],
[
"print (\"sigmoid(0) = \" + str(sigmoid(0)))\nprint (\"sigmoid(12) = \" + str(sigmoid(12)))",
"sigmoid(0) = 0.5\nsigmoid(12) = 0.999994\n"
]
],
[
[
"*** Expected Output ***: \n\n<table> \n<tr> \n<td>\n**sigmoid(0)**\n</td>\n<td>\n0.5\n</td>\n</tr>\n<tr> \n<td>\n**sigmoid(12)**\n</td>\n<td>\n0.999994\n</td>\n</tr> \n\n</table> ",
"_____no_output_____"
],
[
"<font color='blue'>\n**To summarize, you how know how to**:\n1. Create placeholders\n2. Specify the computation graph corresponding to operations you want to compute\n3. Create the session\n4. Run the session, using a feed dictionary if necessary to specify placeholder variables' values. ",
"_____no_output_____"
],
[
"### 1.3 - Computing the Cost\n\nYou can also use a built-in function to compute the cost of your neural network. So instead of needing to write code to compute this as a function of $a^{[2](i)}$ and $y^{(i)}$ for i=1...m: \n$$ J = - \\frac{1}{m} \\sum_{i = 1}^m \\large ( \\small y^{(i)} \\log a^{ [2] (i)} + (1-y^{(i)})\\log (1-a^{ [2] (i)} )\\large )\\small\\tag{2}$$\n\nyou can do it in one line of code in tensorflow!\n\n**Exercise**: Implement the cross entropy loss. The function you will use is: \n\n\n- `tf.nn.sigmoid_cross_entropy_with_logits(logits = ..., labels = ...)`\n\nYour code should input `z`, compute the sigmoid (to get `a`) and then compute the cross entropy cost $J$. All this can be done using one call to `tf.nn.sigmoid_cross_entropy_with_logits`, which computes\n\n$$- \\frac{1}{m} \\sum_{i = 1}^m \\large ( \\small y^{(i)} \\log \\sigma(z^{[2](i)}) + (1-y^{(i)})\\log (1-\\sigma(z^{[2](i)})\\large )\\small\\tag{2}$$\n\n",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: cost\n\ndef cost(logits, labels):\n \"\"\"\n Computes the cost using the sigmoid cross entropy\n \n Arguments:\n logits -- vector containing z, output of the last linear unit (before the final sigmoid activation)\n labels -- vector of labels y (1 or 0) \n \n Note: What we've been calling \"z\" and \"y\" in this class are respectively called \"logits\" and \"labels\" \n in the TensorFlow documentation. So logits will feed into z, and labels into y. \n \n Returns:\n cost -- runs the session of the cost (formula (2))\n \"\"\"\n \n ### START CODE HERE ### \n \n # Create the placeholders for \"logits\" (z) and \"labels\" (y) (approx. 2 lines)\n z = tf.placeholder(tf.float32,name=\"z\")\n y = tf.placeholder(tf.float32,name=\"y\")\n \n # Use the loss function (approx. 1 line)\n cost = tf.nn.sigmoid_cross_entropy_with_logits(logits=z,labels=y)\n \n # Create a session (approx. 1 line). See method 1 above.\n sess = tf.Session()\n \n # Run the session (approx. 1 line).\n cost = sess.run(cost,feed_dict = {z:logits, y:labels})\n \n # Close the session (approx. 1 line). See method 1 above.\n sess.close()\n \n ### END CODE HERE ###\n \n return cost",
"_____no_output_____"
],
[
"logits = np.array([0.2,0.4,0.7,0.9])\n\ncost = cost(logits, np.array([0,0,1,1]))\nprint (\"cost = \" + str(cost))",
"cost = [ 0.79813886 0.91301525 0.40318605 0.34115386]\n"
]
],
[
[
"** Expected Output** : \n\n```\ncost = [ 0.79813886 0.91301525 0.40318605 0.34115386]\n```",
"_____no_output_____"
],
[
"### 1.4 - Using One Hot encodings\n\nMany times in deep learning you will have a y vector with numbers ranging from 0 to C-1, where C is the number of classes. If C is for example 4, then you might have the following y vector which you will need to convert as follows:\n\n\n<img src=\"images/onehot.png\" style=\"width:600px;height:150px;\">\n\nThis is called a \"one hot\" encoding, because in the converted representation exactly one element of each column is \"hot\" (meaning set to 1). To do this conversion in numpy, you might have to write a few lines of code. In tensorflow, you can use one line of code: \n\n- tf.one_hot(labels, depth, axis) \n\n**Exercise:** Implement the function below to take one vector of labels and the total number of classes $C$, and return the one hot encoding. Use `tf.one_hot()` to do this. ",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: one_hot_matrix\n\ndef one_hot_matrix(labels, C):\n \"\"\"\n Creates a matrix where the i-th row corresponds to the ith class number and the jth column\n corresponds to the jth training example. So if example j had a label i. Then entry (i,j) \n will be 1. \n \n Arguments:\n labels -- vector containing the labels \n C -- number of classes, the depth of the one hot dimension\n \n Returns: \n one_hot -- one hot matrix\n \"\"\"\n \n ### START CODE HERE ###\n \n # Create a tf.constant equal to C (depth), name it 'C'. (approx. 1 line)\n C = tf.constant(C,name='C')\n \n # Use tf.one_hot, be careful with the axis (approx. 1 line)\n one_hot_matrix = tf.one_hot(labels,C,axis=0)\n \n # Create the session (approx. 1 line)\n sess = tf.Session()\n \n # Run the session (approx. 1 line)\n one_hot = sess.run(one_hot_matrix)\n \n # Close the session (approx. 1 line). See method 1 above.\n sess.close()\n \n ### END CODE HERE ###\n \n return one_hot",
"_____no_output_____"
],
[
"labels = np.array([1,2,3,0,2,1])\none_hot = one_hot_matrix(labels, C = 4)\nprint (\"one_hot = \\n\" + str(one_hot))",
"one_hot = \n[[ 0. 0. 0. 1. 0. 0.]\n [ 1. 0. 0. 0. 0. 1.]\n [ 0. 1. 0. 0. 1. 0.]\n [ 0. 0. 1. 0. 0. 0.]]\n"
]
],
[
[
"**Expected Output**: \n\n```\none_hot = \n[[ 0. 0. 0. 1. 0. 0.]\n [ 1. 0. 0. 0. 0. 1.]\n [ 0. 1. 0. 0. 1. 0.]\n [ 0. 0. 1. 0. 0. 0.]]\n```",
"_____no_output_____"
],
[
"### 1.5 - Initialize with zeros and ones\n\nNow you will learn how to initialize a vector of zeros and ones. The function you will be calling is `tf.ones()`. To initialize with zeros you could use tf.zeros() instead. These functions take in a shape and return an array of dimension shape full of zeros and ones respectively. \n\n**Exercise:** Implement the function below to take in a shape and to return an array (of the shape's dimension of ones). \n\n - tf.ones(shape)\n",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: ones\n\ndef ones(shape):\n \"\"\"\n Creates an array of ones of dimension shape\n \n Arguments:\n shape -- shape of the array you want to create\n \n Returns: \n ones -- array containing only ones\n \"\"\"\n \n ### START CODE HERE ###\n \n # Create \"ones\" tensor using tf.ones(...). (approx. 1 line)\n ones = tf.ones(shape)\n \n # Create the session (approx. 1 line)\n sess = tf.Session()\n \n # Run the session to compute 'ones' (approx. 1 line)\n ones = sess.run(ones)\n \n # Close the session (approx. 1 line). See method 1 above.\n sess.close()\n \n ### END CODE HERE ###\n return ones",
"_____no_output_____"
],
[
"print (\"ones = \" + str(ones([3])))",
"ones = [ 1. 1. 1.]\n"
]
],
[
[
"**Expected Output:**\n\n<table> \n <tr> \n <td>\n **ones**\n </td>\n <td>\n [ 1. 1. 1.]\n </td>\n </tr>\n\n</table>",
"_____no_output_____"
],
[
"# 2 - Building your first neural network in tensorflow\n\nIn this part of the assignment you will build a neural network using tensorflow. Remember that there are two parts to implement a tensorflow model:\n\n- Create the computation graph\n- Run the graph\n\nLet's delve into the problem you'd like to solve!\n\n### 2.0 - Problem statement: SIGNS Dataset\n\nOne afternoon, with some friends we decided to teach our computers to decipher sign language. We spent a few hours taking pictures in front of a white wall and came up with the following dataset. It's now your job to build an algorithm that would facilitate communications from a speech-impaired person to someone who doesn't understand sign language.\n\n- **Training set**: 1080 pictures (64 by 64 pixels) of signs representing numbers from 0 to 5 (180 pictures per number).\n- **Test set**: 120 pictures (64 by 64 pixels) of signs representing numbers from 0 to 5 (20 pictures per number).\n\nNote that this is a subset of the SIGNS dataset. The complete dataset contains many more signs.\n\nHere are examples for each number, and how an explanation of how we represent the labels. These are the original pictures, before we lowered the image resolutoion to 64 by 64 pixels.\n<img src=\"images/hands.png\" style=\"width:800px;height:350px;\"><caption><center> <u><font color='purple'> **Figure 1**</u><font color='purple'>: SIGNS dataset <br> <font color='black'> </center>\n\n\nRun the following code to load the dataset.",
"_____no_output_____"
]
],
[
[
"# Loading the dataset\nX_train_orig, Y_train_orig, X_test_orig, Y_test_orig, classes = load_dataset()",
"_____no_output_____"
]
],
[
[
"Change the index below and run the cell to visualize some examples in the dataset.",
"_____no_output_____"
]
],
[
[
"# Example of a picture\nindex = 0\nplt.imshow(X_train_orig[index])\nprint (\"y = \" + str(np.squeeze(Y_train_orig[:, index])))",
"y = 5\n"
]
],
[
[
"As usual you flatten the image dataset, then normalize it by dividing by 255. On top of that, you will convert each label to a one-hot vector as shown in Figure 1. Run the cell below to do so.",
"_____no_output_____"
]
],
[
[
"# Flatten the training and test images\nX_train_flatten = X_train_orig.reshape(X_train_orig.shape[0], -1).T\nX_test_flatten = X_test_orig.reshape(X_test_orig.shape[0], -1).T\n# Normalize image vectors\nX_train = X_train_flatten/255.\nX_test = X_test_flatten/255.\n# Convert training and test labels to one hot matrices\nY_train = convert_to_one_hot(Y_train_orig, 6)\nY_test = convert_to_one_hot(Y_test_orig, 6)\n\nprint (\"number of training examples = \" + str(X_train.shape[1]))\nprint (\"number of test examples = \" + str(X_test.shape[1]))\nprint (\"X_train shape: \" + str(X_train.shape))\nprint (\"Y_train shape: \" + str(Y_train.shape))\nprint (\"X_test shape: \" + str(X_test.shape))\nprint (\"Y_test shape: \" + str(Y_test.shape))",
"number of training examples = 1080\nnumber of test examples = 120\nX_train shape: (12288, 1080)\nY_train shape: (6, 1080)\nX_test shape: (12288, 120)\nY_test shape: (6, 120)\n"
]
],
[
[
"**Note** that 12288 comes from $64 \\times 64 \\times 3$. Each image is square, 64 by 64 pixels, and 3 is for the RGB colors. Please make sure all these shapes make sense to you before continuing.",
"_____no_output_____"
],
[
"**Your goal** is to build an algorithm capable of recognizing a sign with high accuracy. To do so, you are going to build a tensorflow model that is almost the same as one you have previously built in numpy for cat recognition (but now using a softmax output). It is a great occasion to compare your numpy implementation to the tensorflow one. \n\n**The model** is *LINEAR -> RELU -> LINEAR -> RELU -> LINEAR -> SOFTMAX*. The SIGMOID output layer has been converted to a SOFTMAX. A SOFTMAX layer generalizes SIGMOID to when there are more than two classes. ",
"_____no_output_____"
],
[
"### 2.1 - Create placeholders\n\nYour first task is to create placeholders for `X` and `Y`. This will allow you to later pass your training data in when you run your session. \n\n**Exercise:** Implement the function below to create the placeholders in tensorflow.",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: create_placeholders\n\ndef create_placeholders(n_x, n_y):\n \"\"\"\n Creates the placeholders for the tensorflow session.\n \n Arguments:\n n_x -- scalar, size of an image vector (num_px * num_px = 64 * 64 * 3 = 12288)\n n_y -- scalar, number of classes (from 0 to 5, so -> 6)\n \n Returns:\n X -- placeholder for the data input, of shape [n_x, None] and dtype \"tf.float32\"\n Y -- placeholder for the input labels, of shape [n_y, None] and dtype \"tf.float32\"\n \n Tips:\n - You will use None because it let's us be flexible on the number of examples you will for the placeholders.\n In fact, the number of examples during test/train is different.\n \"\"\"\n\n ### START CODE HERE ### (approx. 2 lines)\n X = tf.placeholder(tf.float32, [n_x,None], name= \"X\")\n Y = tf.placeholder(tf.float32,[n_y, None], name = \"Y\")\n ### END CODE HERE ###\n \n return X, Y",
"_____no_output_____"
],
[
"X, Y = create_placeholders(12288, 6)\nprint (\"X = \" + str(X))\nprint (\"Y = \" + str(Y))",
"X = Tensor(\"X_2:0\", shape=(12288, ?), dtype=float32)\nY = Tensor(\"Y:0\", shape=(6, ?), dtype=float32)\n"
]
],
[
[
"**Expected Output**: \n\n<table> \n <tr> \n <td>\n **X**\n </td>\n <td>\n Tensor(\"Placeholder_1:0\", shape=(12288, ?), dtype=float32) (not necessarily Placeholder_1)\n </td>\n </tr>\n <tr> \n <td>\n **Y**\n </td>\n <td>\n Tensor(\"Placeholder_2:0\", shape=(6, ?), dtype=float32) (not necessarily Placeholder_2)\n </td>\n </tr>\n\n</table>",
"_____no_output_____"
],
[
"### 2.2 - Initializing the parameters\n\nYour second task is to initialize the parameters in tensorflow.\n\n**Exercise:** Implement the function below to initialize the parameters in tensorflow. You are going use Xavier Initialization for weights and Zero Initialization for biases. The shapes are given below. As an example, to help you, for W1 and b1 you could use: \n\n```python\nW1 = tf.get_variable(\"W1\", [25,12288], initializer = tf.contrib.layers.xavier_initializer(seed = 1))\nb1 = tf.get_variable(\"b1\", [25,1], initializer = tf.zeros_initializer())\n```\nPlease use `seed = 1` to make sure your results match ours.",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: initialize_parameters\n\ndef initialize_parameters():\n \"\"\"\n Initializes parameters to build a neural network with tensorflow. The shapes are:\n W1 : [25, 12288]\n b1 : [25, 1]\n W2 : [12, 25]\n b2 : [12, 1]\n W3 : [6, 12]\n b3 : [6, 1]\n \n Returns:\n parameters -- a dictionary of tensors containing W1, b1, W2, b2, W3, b3\n \"\"\"\n \n tf.set_random_seed(1) # so that your \"random\" numbers match ours\n \n ### START CODE HERE ### (approx. 6 lines of code)\n W1 = tf.get_variable(\"W1\", [25,12288], initializer = tf.contrib.layers.xavier_initializer(seed = 1))\n b1 = tf.get_variable(\"b1\", [25,1], initializer = tf.zeros_initializer())\n W2 = tf.get_variable(\"W2\", [12,25], initializer = tf.contrib.layers.xavier_initializer(seed = 1))\n b2 = tf.get_variable(\"b2\", [12,1], initializer = tf.zeros_initializer())\n W3 = tf.get_variable(\"W3\", [6,12], initializer = tf.contrib.layers.xavier_initializer(seed = 1))\n b3 = tf.get_variable(\"b3\", [6,1], initializer = tf.zeros_initializer())\n ### END CODE HERE ###\n\n parameters = {\"W1\": W1,\n \"b1\": b1,\n \"W2\": W2,\n \"b2\": b2,\n \"W3\": W3,\n \"b3\": b3}\n \n return parameters",
"_____no_output_____"
],
[
"tf.reset_default_graph()\nwith tf.Session() as sess:\n parameters = initialize_parameters()\n print(\"W1 = \" + str(parameters[\"W1\"]))\n print(\"b1 = \" + str(parameters[\"b1\"]))\n print(\"W2 = \" + str(parameters[\"W2\"]))\n print(\"b2 = \" + str(parameters[\"b2\"]))",
"W1 = <tf.Variable 'W1:0' shape=(25, 12288) dtype=float32_ref>\nb1 = <tf.Variable 'b1:0' shape=(25, 1) dtype=float32_ref>\nW2 = <tf.Variable 'W2:0' shape=(12, 25) dtype=float32_ref>\nb2 = <tf.Variable 'b2:0' shape=(12, 1) dtype=float32_ref>\n"
]
],
[
[
"**Expected Output**: \n\n<table> \n <tr> \n <td>\n **W1**\n </td>\n <td>\n < tf.Variable 'W1:0' shape=(25, 12288) dtype=float32_ref >\n </td>\n </tr>\n <tr> \n <td>\n **b1**\n </td>\n <td>\n < tf.Variable 'b1:0' shape=(25, 1) dtype=float32_ref >\n </td>\n </tr>\n <tr> \n <td>\n **W2**\n </td>\n <td>\n < tf.Variable 'W2:0' shape=(12, 25) dtype=float32_ref >\n </td>\n </tr>\n <tr> \n <td>\n **b2**\n </td>\n <td>\n < tf.Variable 'b2:0' shape=(12, 1) dtype=float32_ref >\n </td>\n </tr>\n\n</table>",
"_____no_output_____"
],
[
"As expected, the parameters haven't been evaluated yet.",
"_____no_output_____"
],
[
"### 2.3 - Forward propagation in tensorflow \n\nYou will now implement the forward propagation module in tensorflow. The function will take in a dictionary of parameters and it will complete the forward pass. The functions you will be using are: \n\n- `tf.add(...,...)` to do an addition\n- `tf.matmul(...,...)` to do a matrix multiplication\n- `tf.nn.relu(...)` to apply the ReLU activation\n\n**Question:** Implement the forward pass of the neural network. We commented for you the numpy equivalents so that you can compare the tensorflow implementation to numpy. It is important to note that the forward propagation stops at `z3`. The reason is that in tensorflow the last linear layer output is given as input to the function computing the loss. Therefore, you don't need `a3`!\n\n",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: forward_propagation\n\ndef forward_propagation(X, parameters):\n \"\"\"\n Implements the forward propagation for the model: LINEAR -> RELU -> LINEAR -> RELU -> LINEAR -> SOFTMAX\n \n Arguments:\n X -- input dataset placeholder, of shape (input size, number of examples)\n parameters -- python dictionary containing your parameters \"W1\", \"b1\", \"W2\", \"b2\", \"W3\", \"b3\"\n the shapes are given in initialize_parameters\n\n Returns:\n Z3 -- the output of the last LINEAR unit\n \"\"\"\n \n # Retrieve the parameters from the dictionary \"parameters\" \n W1 = parameters['W1']\n b1 = parameters['b1']\n W2 = parameters['W2']\n b2 = parameters['b2']\n W3 = parameters['W3']\n b3 = parameters['b3']\n \n ### START CODE HERE ### (approx. 5 lines) # Numpy Equivalents:\n Z1 = tf.add(tf.matmul(W1,X),b1) # Z1 = np.dot(W1, X) + b1\n A1 = tf.nn.relu(Z1) # A1 = relu(Z1)\n Z2 = tf.add(tf.matmul(W2,A1),b2) # Z2 = np.dot(W2, A1) + b2\n A2 = tf.nn.relu(Z2) # A2 = relu(Z2)\n Z3 = tf.add(tf.matmul(W3,A2),b3) # Z3 = np.dot(W3, A2) + b3\n ### END CODE HERE ###\n \n return Z3",
"_____no_output_____"
],
[
"tf.reset_default_graph()\n\nwith tf.Session() as sess:\n X, Y = create_placeholders(12288, 6)\n parameters = initialize_parameters()\n Z3 = forward_propagation(X, parameters)\n print(\"Z3 = \" + str(Z3))",
"Z3 = Tensor(\"Add_2:0\", shape=(6, ?), dtype=float32)\n"
]
],
[
[
"**Expected Output**: \n\n<table> \n <tr> \n <td>\n **Z3**\n </td>\n <td>\n Tensor(\"Add_2:0\", shape=(6, ?), dtype=float32)\n </td>\n </tr>\n\n</table>",
"_____no_output_____"
],
[
"You may have noticed that the forward propagation doesn't output any cache. You will understand why below, when we get to brackpropagation.",
"_____no_output_____"
],
[
"### 2.4 Compute cost\n\nAs seen before, it is very easy to compute the cost using:\n```python\ntf.reduce_mean(tf.nn.softmax_cross_entropy_with_logits(logits = ..., labels = ...))\n```\n**Question**: Implement the cost function below. \n- It is important to know that the \"`logits`\" and \"`labels`\" inputs of `tf.nn.softmax_cross_entropy_with_logits` are expected to be of shape (number of examples, num_classes). We have thus transposed Z3 and Y for you.\n- Besides, `tf.reduce_mean` basically does the summation over the examples.",
"_____no_output_____"
]
],
[
[
"# GRADED FUNCTION: compute_cost \n\ndef compute_cost(Z3, Y):\n \"\"\"\n Computes the cost\n \n Arguments:\n Z3 -- output of forward propagation (output of the last LINEAR unit), of shape (6, number of examples)\n Y -- \"true\" labels vector placeholder, same shape as Z3\n \n Returns:\n cost - Tensor of the cost function\n \"\"\"\n \n # to fit the tensorflow requirement for tf.nn.softmax_cross_entropy_with_logits(...,...)\n logits = tf.transpose(Z3)\n labels = tf.transpose(Y)\n \n ### START CODE HERE ### (1 line of code)\n cost = tf.reduce_mean(tf.nn.softmax_cross_entropy_with_logits(logits=logits,labels=labels))\n ### END CODE HERE ###\n \n return cost",
"_____no_output_____"
],
[
"tf.reset_default_graph()\n\nwith tf.Session() as sess:\n X, Y = create_placeholders(12288, 6)\n parameters = initialize_parameters()\n Z3 = forward_propagation(X, parameters)\n cost = compute_cost(Z3, Y)\n print(\"cost = \" + str(cost))",
"cost = Tensor(\"Mean:0\", shape=(), dtype=float32)\n"
]
],
[
[
"**Expected Output**: \n\n<table> \n <tr> \n <td>\n **cost**\n </td>\n <td>\n Tensor(\"Mean:0\", shape=(), dtype=float32)\n </td>\n </tr>\n\n</table>",
"_____no_output_____"
],
[
"### 2.5 - Backward propagation & parameter updates\n\nThis is where you become grateful to programming frameworks. All the backpropagation and the parameters update is taken care of in 1 line of code. It is very easy to incorporate this line in the model.\n\nAfter you compute the cost function. You will create an \"`optimizer`\" object. You have to call this object along with the cost when running the tf.session. When called, it will perform an optimization on the given cost with the chosen method and learning rate.\n\nFor instance, for gradient descent the optimizer would be:\n```python\noptimizer = tf.train.GradientDescentOptimizer(learning_rate = learning_rate).minimize(cost)\n```\n\nTo make the optimization you would do:\n```python\n_ , c = sess.run([optimizer, cost], feed_dict={X: minibatch_X, Y: minibatch_Y})\n```\n\nThis computes the backpropagation by passing through the tensorflow graph in the reverse order. From cost to inputs.\n\n**Note** When coding, we often use `_` as a \"throwaway\" variable to store values that we won't need to use later. Here, `_` takes on the evaluated value of `optimizer`, which we don't need (and `c` takes the value of the `cost` variable). ",
"_____no_output_____"
],
[
"### 2.6 - Building the model\n\nNow, you will bring it all together! \n\n**Exercise:** Implement the model. You will be calling the functions you had previously implemented.",
"_____no_output_____"
]
],
[
[
"def model(X_train, Y_train, X_test, Y_test, learning_rate = 0.0001,\n num_epochs = 1500, minibatch_size = 32, print_cost = True):\n \"\"\"\n Implements a three-layer tensorflow neural network: LINEAR->RELU->LINEAR->RELU->LINEAR->SOFTMAX.\n \n Arguments:\n X_train -- training set, of shape (input size = 12288, number of training examples = 1080)\n Y_train -- test set, of shape (output size = 6, number of training examples = 1080)\n X_test -- training set, of shape (input size = 12288, number of training examples = 120)\n Y_test -- test set, of shape (output size = 6, number of test examples = 120)\n learning_rate -- learning rate of the optimization\n num_epochs -- number of epochs of the optimization loop\n minibatch_size -- size of a minibatch\n print_cost -- True to print the cost every 100 epochs\n \n Returns:\n parameters -- parameters learnt by the model. They can then be used to predict.\n \"\"\"\n \n ops.reset_default_graph() # to be able to rerun the model without overwriting tf variables\n tf.set_random_seed(1) # to keep consistent results\n seed = 3 # to keep consistent results\n (n_x, m) = X_train.shape # (n_x: input size, m : number of examples in the train set)\n n_y = Y_train.shape[0] # n_y : output size\n costs = [] # To keep track of the cost\n \n # Create Placeholders of shape (n_x, n_y)\n ### START CODE HERE ### (1 line)\n X, Y = create_placeholders(n_x, n_y)\n ### END CODE HERE ###\n\n # Initialize parameters\n ### START CODE HERE ### (1 line)\n parameters = initialize_parameters()\n ### END CODE HERE ###\n \n # Forward propagation: Build the forward propagation in the tensorflow graph\n ### START CODE HERE ### (1 line)\n Z3 = forward_propagation(X, parameters)\n ### END CODE HERE ###\n \n # Cost function: Add cost function to tensorflow graph\n ### START CODE HERE ### (1 line)\n cost = compute_cost(Z3, Y)\n ### END CODE HERE ###\n \n # Backpropagation: Define the tensorflow optimizer. Use an AdamOptimizer.\n ### START CODE HERE ### (1 line)\n optimizer = tf.train.AdamOptimizer(learning_rate = learning_rate).minimize(cost)\n ### END CODE HERE ###\n \n # Initialize all the variables\n init = tf.global_variables_initializer()\n\n # Start the session to compute the tensorflow graph\n with tf.Session() as sess:\n \n # Run the initialization\n sess.run(init)\n \n # Do the training loop\n for epoch in range(num_epochs):\n\n epoch_cost = 0. # Defines a cost related to an epoch\n num_minibatches = int(m / minibatch_size) # number of minibatches of size minibatch_size in the train set\n seed = seed + 1\n minibatches = random_mini_batches(X_train, Y_train, minibatch_size, seed)\n\n for minibatch in minibatches:\n\n # Select a minibatch\n (minibatch_X, minibatch_Y) = minibatch\n \n # IMPORTANT: The line that runs the graph on a minibatch.\n # Run the session to execute the \"optimizer\" and the \"cost\", the feedict should contain a minibatch for (X,Y).\n ### START CODE HERE ### (1 line)\n _ , minibatch_cost = sess.run([optimizer, cost], feed_dict={X: minibatch_X, Y: minibatch_Y})\n ### END CODE HERE ###\n \n epoch_cost += minibatch_cost / minibatch_size\n\n # Print the cost every epoch\n if print_cost == True and epoch % 100 == 0:\n print (\"Cost after epoch %i: %f\" % (epoch, epoch_cost))\n if print_cost == True and epoch % 5 == 0:\n costs.append(epoch_cost)\n \n # plot the cost\n plt.plot(np.squeeze(costs))\n plt.ylabel('cost')\n plt.xlabel('iterations (per fives)')\n plt.title(\"Learning rate =\" + str(learning_rate))\n plt.show()\n\n # lets save the parameters in a variable\n parameters = sess.run(parameters)\n print (\"Parameters have been trained!\")\n\n # Calculate the correct predictions\n correct_prediction = tf.equal(tf.argmax(Z3), tf.argmax(Y))\n\n # Calculate accuracy on the test set\n accuracy = tf.reduce_mean(tf.cast(correct_prediction, \"float\"))\n\n print (\"Train Accuracy:\", accuracy.eval({X: X_train, Y: Y_train}))\n print (\"Test Accuracy:\", accuracy.eval({X: X_test, Y: Y_test}))\n \n return parameters",
"_____no_output_____"
]
],
[
[
"Run the following cell to train your model! On our machine it takes about 5 minutes. Your \"Cost after epoch 100\" should be 1.048222. If it's not, don't waste time; interrupt the training by clicking on the square (⬛) in the upper bar of the notebook, and try to correct your code. If it is the correct cost, take a break and come back in 5 minutes!",
"_____no_output_____"
]
],
[
[
"parameters = model(X_train, Y_train, X_test, Y_test)",
"Cost after epoch 0: 1.913693\nCost after epoch 100: 1.048222\nCost after epoch 200: 0.756012\nCost after epoch 300: 0.590844\nCost after epoch 400: 0.483423\nCost after epoch 500: 0.392928\nCost after epoch 600: 0.323629\nCost after epoch 700: 0.262100\nCost after epoch 800: 0.210199\nCost after epoch 900: 0.171622\nCost after epoch 1000: 0.145907\nCost after epoch 1100: 0.110942\nCost after epoch 1200: 0.088966\nCost after epoch 1300: 0.061226\nCost after epoch 1400: 0.053860\n"
]
],
[
[
"**Expected Output**:\n\n<table> \n <tr> \n <td>\n **Train Accuracy**\n </td>\n <td>\n 0.999074\n </td>\n </tr>\n <tr> \n <td>\n **Test Accuracy**\n </td>\n <td>\n 0.716667\n </td>\n </tr>\n\n</table>\n\nAmazing, your algorithm can recognize a sign representing a figure between 0 and 5 with 71.7% accuracy.\n\n**Insights**:\n- Your model seems big enough to fit the training set well. However, given the difference between train and test accuracy, you could try to add L2 or dropout regularization to reduce overfitting. \n- Think about the session as a block of code to train the model. Each time you run the session on a minibatch, it trains the parameters. In total you have run the session a large number of times (1500 epochs) until you obtained well trained parameters.",
"_____no_output_____"
],
[
"### 2.7 - Test with your own image (optional / ungraded exercise)\n\nCongratulations on finishing this assignment. You can now take a picture of your hand and see the output of your model. To do that:\n 1. Click on \"File\" in the upper bar of this notebook, then click \"Open\" to go on your Coursera Hub.\n 2. Add your image to this Jupyter Notebook's directory, in the \"images\" folder\n 3. Write your image's name in the following code\n 4. Run the code and check if the algorithm is right!",
"_____no_output_____"
]
],
[
[
"import scipy\nfrom PIL import Image\nfrom scipy import ndimage\n\n## START CODE HERE ## (PUT YOUR IMAGE NAME) \nmy_image = \"thumbs_up.jpg\"\n## END CODE HERE ##\n\n# We preprocess your image to fit your algorithm.\nfname = \"images/\" + my_image\nimage = np.array(ndimage.imread(fname, flatten=False))\nimage = image/255.\nmy_image = scipy.misc.imresize(image, size=(64,64)).reshape((1, 64*64*3)).T\nmy_image_prediction = predict(my_image, parameters)\n\nplt.imshow(image)\nprint(\"Your algorithm predicts: y = \" + str(np.squeeze(my_image_prediction)))",
"Your algorithm predicts: y = 3\n"
]
],
[
[
"You indeed deserved a \"thumbs-up\" although as you can see the algorithm seems to classify it incorrectly. The reason is that the training set doesn't contain any \"thumbs-up\", so the model doesn't know how to deal with it! We call that a \"mismatched data distribution\" and it is one of the various of the next course on \"Structuring Machine Learning Projects\".",
"_____no_output_____"
],
[
"<font color='blue'>\n**What you should remember**:\n- Tensorflow is a programming framework used in deep learning\n- The two main object classes in tensorflow are Tensors and Operators. \n- When you code in tensorflow you have to take the following steps:\n - Create a graph containing Tensors (Variables, Placeholders ...) and Operations (tf.matmul, tf.add, ...)\n - Create a session\n - Initialize the session\n - Run the session to execute the graph\n- You can execute the graph multiple times as you've seen in model()\n- The backpropagation and optimization is automatically done when running the session on the \"optimizer\" object.",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
]
] |
e7ec34fad9fe056caf49770a8aa8ab6a2514c618 | 141,442 | ipynb | Jupyter Notebook | AI.ipynb | wonjongchurl/github_test | 3e249ca0e88fadd6040f3796a0110e8d11e889ec | [
"MIT"
] | null | null | null | AI.ipynb | wonjongchurl/github_test | 3e249ca0e88fadd6040f3796a0110e8d11e889ec | [
"MIT"
] | null | null | null | AI.ipynb | wonjongchurl/github_test | 3e249ca0e88fadd6040f3796a0110e8d11e889ec | [
"MIT"
] | 1 | 2021-07-31T10:29:23.000Z | 2021-07-31T10:29:23.000Z | 152.580367 | 60,754 | 0.817176 | [
[
[
"<a href=\"https://colab.research.google.com/github/jongchurlwon/github_test/blob/main/AI.ipynb\" target=\"_parent\"><img src=\"https://colab.research.google.com/assets/colab-badge.svg\" alt=\"Open In Colab\"/></a>",
"_____no_output_____"
]
],
[
[
"!pip install pyupbit",
"Collecting pyupbit\n Downloading pyupbit-0.2.19-py3-none-any.whl (19 kB)\nRequirement already satisfied: pandas in /usr/local/lib/python3.7/dist-packages (from pyupbit) (1.1.5)\nCollecting websockets\n Downloading websockets-9.1-cp37-cp37m-manylinux2010_x86_64.whl (103 kB)\n\u001b[K |████████████████████████████████| 103 kB 36.0 MB/s \n\u001b[?25hRequirement already satisfied: requests in /usr/local/lib/python3.7/dist-packages (from pyupbit) (2.23.0)\nCollecting pyjwt>=2.0.0\n Downloading PyJWT-2.1.0-py3-none-any.whl (16 kB)\nRequirement already satisfied: numpy>=1.15.4 in /usr/local/lib/python3.7/dist-packages (from pandas->pyupbit) (1.19.5)\nRequirement already satisfied: pytz>=2017.2 in /usr/local/lib/python3.7/dist-packages (from pandas->pyupbit) (2018.9)\nRequirement already satisfied: python-dateutil>=2.7.3 in /usr/local/lib/python3.7/dist-packages (from pandas->pyupbit) (2.8.1)\nRequirement already satisfied: six>=1.5 in /usr/local/lib/python3.7/dist-packages (from python-dateutil>=2.7.3->pandas->pyupbit) (1.15.0)\nRequirement already satisfied: idna<3,>=2.5 in /usr/local/lib/python3.7/dist-packages (from requests->pyupbit) (2.10)\nRequirement already satisfied: certifi>=2017.4.17 in /usr/local/lib/python3.7/dist-packages (from requests->pyupbit) (2021.5.30)\nRequirement already satisfied: urllib3!=1.25.0,!=1.25.1,<1.26,>=1.21.1 in /usr/local/lib/python3.7/dist-packages (from requests->pyupbit) (1.24.3)\nRequirement already satisfied: chardet<4,>=3.0.2 in /usr/local/lib/python3.7/dist-packages (from requests->pyupbit) (3.0.4)\nInstalling collected packages: websockets, pyjwt, pyupbit\nSuccessfully installed pyjwt-2.1.0 pyupbit-0.2.19 websockets-9.1\n"
],
[
"import pyupbit",
"_____no_output_____"
],
[
"#BTC 최근 200시간의 데이터 불러옴\ndf = pyupbit.get_ohlcv(\"KRW-ETH\", interval=\"minute60\")\ndf",
"_____no_output_____"
],
[
"#시간(ds)와 종가(y)값만 남김\ndf = df.reset_index()\ndf['ds'] = df['index']\ndf['y'] = df['close']\ndata = df[['ds','y']]\ndata",
"_____no_output_____"
],
[
"#prophet 불러옴\nfrom fbprophet import Prophet",
"_____no_output_____"
],
[
"#학습\nmodel = Prophet()\nmodel.fit(data)",
"INFO:fbprophet:Disabling yearly seasonality. Run prophet with yearly_seasonality=True to override this.\nINFO:fbprophet:Disabling weekly seasonality. Run prophet with weekly_seasonality=True to override this.\n"
],
[
"\n#24시간 미래 예측\nfuture = model.make_future_dataframe(periods=24, freq='H')\nforecast = model.predict(future)",
"_____no_output_____"
],
[
"#그래프1\nfig1 = model.plot(forecast)\n",
"_____no_output_____"
],
[
"#그래프2\nfig2 = model.plot_components(forecast)",
"_____no_output_____"
],
[
"#매수 시점의 가격\nnowValue = pyupbit.get_current_price(\"KRW-ETH\")\nnowValue",
"_____no_output_____"
],
[
"\n#종가의 가격을 구함\n\n#현재 시간이 자정 이전\ncloseDf = forecast[forecast['ds'] == forecast.iloc[-1]['ds'].replace(hour=9)]\n\n#현재 시간이 자정 이후\nif len(closeDf) == 0:\n closeDf = forecast[forecast['ds'] == data.iloc[-1]['ds'].replace(hour=9)]\n\n#어쨋든 당일 종가\ncloseValue = closeDf['yhat'].values[0]\ncloseValue",
"_____no_output_____"
],
[
"\n#구체적인 가격\nprint(\"현재 시점 가격: \", nowValue)\nprint(\"종가의 가격: \", closeValue)",
"현재 시점 가격: 2727000.0\n종가의 가격: 2755170.7701194733\n"
],
[
"forecast",
"_____no_output_____"
],
[
"",
"_____no_output_____"
],
[
"",
"_____no_output_____"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ec38132310fab626fe4bb7a209096e30953a44 | 55,881 | ipynb | Jupyter Notebook | Association_rules.ipynb | anagha0397/Association-Rules | a33f35c1b1830c3c9f1fcccfc7bb3cfa2781fbc2 | [
"MIT"
] | null | null | null | Association_rules.ipynb | anagha0397/Association-Rules | a33f35c1b1830c3c9f1fcccfc7bb3cfa2781fbc2 | [
"MIT"
] | null | null | null | Association_rules.ipynb | anagha0397/Association-Rules | a33f35c1b1830c3c9f1fcccfc7bb3cfa2781fbc2 | [
"MIT"
] | null | null | null | 36.595285 | 199 | 0.378268 | [
[
[
"!pip install conda",
"Requirement already satisfied: conda in c:\\users\\dell\\anaconda3\\lib\\site-packages (4.10.1)\nRequirement already satisfied: pycosat>=0.6.3 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from conda) (0.6.3)\nRequirement already satisfied: requests>=2.12.4 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from conda) (2.25.1)\nRequirement already satisfied: ruamel_yaml_conda>=0.11.14 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from conda) (0.15.100)\nRequirement already satisfied: menuinst in c:\\users\\dell\\anaconda3\\lib\\site-packages (from conda) (1.4.16)\nRequirement already satisfied: certifi>=2017.4.17 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from requests>=2.12.4->conda) (2020.12.5)\nRequirement already satisfied: chardet<5,>=3.0.2 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from requests>=2.12.4->conda) (4.0.0)\nRequirement already satisfied: idna<3,>=2.5 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from requests>=2.12.4->conda) (2.10)\nRequirement already satisfied: urllib3<1.27,>=1.21.1 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from requests>=2.12.4->conda) (1.26.4)\n"
],
[
"!pip install mlxtend",
"Collecting mlxtend\n Downloading mlxtend-0.18.0-py2.py3-none-any.whl (1.3 MB)\nRequirement already satisfied: joblib>=0.13.2 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from mlxtend) (1.0.1)\nRequirement already satisfied: pandas>=0.24.2 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from mlxtend) (1.2.4)\nRequirement already satisfied: setuptools in c:\\users\\dell\\anaconda3\\lib\\site-packages (from mlxtend) (52.0.0.post20210125)\nRequirement already satisfied: scikit-learn>=0.20.3 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from mlxtend) (0.24.1)\nRequirement already satisfied: matplotlib>=3.0.0 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from mlxtend) (3.3.4)\nRequirement already satisfied: numpy>=1.16.2 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from mlxtend) (1.20.1)\nRequirement already satisfied: scipy>=1.2.1 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from mlxtend) (1.6.2)\nRequirement already satisfied: python-dateutil>=2.1 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from matplotlib>=3.0.0->mlxtend) (2.8.1)\nRequirement already satisfied: kiwisolver>=1.0.1 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from matplotlib>=3.0.0->mlxtend) (1.3.1)\nRequirement already satisfied: pillow>=6.2.0 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from matplotlib>=3.0.0->mlxtend) (8.2.0)\nRequirement already satisfied: cycler>=0.10 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from matplotlib>=3.0.0->mlxtend) (0.10.0)\nRequirement already satisfied: pyparsing!=2.0.4,!=2.1.2,!=2.1.6,>=2.0.3 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from matplotlib>=3.0.0->mlxtend) (2.4.7)\nRequirement already satisfied: six in c:\\users\\dell\\anaconda3\\lib\\site-packages (from cycler>=0.10->matplotlib>=3.0.0->mlxtend) (1.15.0)\nRequirement already satisfied: pytz>=2017.3 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from pandas>=0.24.2->mlxtend) (2021.1)\nRequirement already satisfied: threadpoolctl>=2.0.0 in c:\\users\\dell\\anaconda3\\lib\\site-packages (from scikit-learn>=0.20.3->mlxtend) (2.1.0)\nInstalling collected packages: mlxtend\nSuccessfully installed mlxtend-0.18.0\n"
],
[
"import pandas as pd\nfrom mlxtend.frequent_patterns import apriori,association_rules\nfrom mlxtend.preprocessing import TransactionEncoder",
"_____no_output_____"
],
[
"tit = pd.read_csv(\"G:/data sceince/Python/Association Rules/Titanic.csv\")",
"_____no_output_____"
],
[
"tit.head()",
"_____no_output_____"
],
[
"tit.shape",
"_____no_output_____"
],
[
"# Creating dummy variables as our data is categorical in nature",
"_____no_output_____"
],
[
"titanic = pd.get_dummies(tit)",
"_____no_output_____"
],
[
"titanic.head()",
"_____no_output_____"
],
[
"# Now in case of this data set so many combinations are possible so, very large number of rules will be created and it is not desired.\n# In order to get less number of rules we will calculate frequent sets using Apriori Algorithm. ",
"_____no_output_____"
],
[
"frequent_sets = apriori(titanic, min_support=0.1, use_colnames=True)\nfrequent_sets",
"_____no_output_____"
],
[
"# Creating rules by using association rules function",
"_____no_output_____"
],
[
"rules = association_rules(frequent_sets, metric = 'lift', min_threshold = 0.9) # Min_threshold is the value of lift ratio we are assuming\nrules",
"_____no_output_____"
],
[
"# Based on the lift ratio calculated we will decide which rule is more important for finding the consequent items. If the lift ratio is greater than 1 that means the rule is very important. \n# Now in order to see which rule has the highest lift ratio we will sort the data in descending order using sort_values function",
"_____no_output_____"
],
[
"rules.sort_values('lift',ascending=False)[0:20] # [0:20] means we are viewing only 1st 21 rules",
"_____no_output_____"
],
[
"# Final step",
"_____no_output_____"
],
[
"# We will only extract the rules where the lift ratio is greater than 1",
"_____no_output_____"
],
[
"[rules.lift>1]\nrules",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ec43c67d8aee0867da47d2283a97ff878d9a71 | 2,799 | ipynb | Jupyter Notebook | examples/notebooks/watch_notebook.ipynb | dix000p/kubernetes-client-python | 22e473e02883aca1058606092c86311f02f42be2 | [
"Apache-2.0"
] | 3 | 2019-05-19T05:05:37.000Z | 2020-03-20T04:56:20.000Z | examples/notebooks/watch_notebook.ipynb | Rohit-0505/python | 4a204fa4cd1ae6f8bfe2e1eea15145ba72532456 | [
"Apache-2.0"
] | 1 | 2019-08-15T14:27:17.000Z | 2019-08-15T14:28:07.000Z | examples/notebooks/watch_notebook.ipynb | Rohit-0505/python | 4a204fa4cd1ae6f8bfe2e1eea15145ba72532456 | [
"Apache-2.0"
] | 2 | 2021-07-09T08:49:05.000Z | 2021-08-03T18:08:36.000Z | 21.530769 | 290 | 0.559486 | [
[
[
"How to watch changes to an object\n==================\n\nIn this notebook, we learn how kubernetes API resource Watch endpoint is used to observe resource changes. It can be used to get information about changes to any kubernetes object.",
"_____no_output_____"
]
],
[
[
"from kubernetes import client, config, watch",
"_____no_output_____"
]
],
[
[
"### Load config from default location.",
"_____no_output_____"
]
],
[
[
"config.load_kube_config()",
"_____no_output_____"
]
],
[
[
"### Create API instance",
"_____no_output_____"
]
],
[
[
"api_instance = client.CoreV1Api()",
"_____no_output_____"
]
],
[
[
"### Run a Watch on the Pods endpoint. \nWatch would be executed and produce output about changes to any Pod. After running the cell below, You can test this by running the Pod notebook [create_pod.ipynb](create_pod.ipynb) and observing the additional output here. You can stop the cell from running by restarting the kernel.",
"_____no_output_____"
]
],
[
[
"w = watch.Watch()\nfor event in w.stream(api_instance.list_pod_for_all_namespaces):\n print(\"Event: %s %s %s\" % (event['type'],event['object'].kind, event['object'].metadata.name))",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7ec47c544bc6fe8ae897b8015b47cc809ad91b6 | 21,451 | ipynb | Jupyter Notebook | notebooks/image_models/labs/3_tf_hub_transfer_learning.ipynb | henrypurbreadcom/asl-ml-immersion | d74d919be561f97d19f4af62b90fd67944cee403 | [
"Apache-2.0"
] | 11 | 2021-09-08T05:39:02.000Z | 2022-03-25T14:35:22.000Z | notebooks/image_models/labs/3_tf_hub_transfer_learning.ipynb | henrypurbreadcom/asl-ml-immersion | d74d919be561f97d19f4af62b90fd67944cee403 | [
"Apache-2.0"
] | 118 | 2021-08-28T03:09:44.000Z | 2022-03-31T00:38:44.000Z | notebooks/image_models/labs/3_tf_hub_transfer_learning.ipynb | henrypurbreadcom/asl-ml-immersion | d74d919be561f97d19f4af62b90fd67944cee403 | [
"Apache-2.0"
] | 110 | 2021-09-02T15:01:35.000Z | 2022-03-31T12:32:48.000Z | 39.504604 | 658 | 0.631952 | [
[
[
"# TensorFlow Transfer Learning\n\nThis notebook shows how to use pre-trained models from [TensorFlowHub](https://www.tensorflow.org/hub). Sometimes, there is not enough data, computational resources, or time to train a model from scratch to solve a particular problem. We'll use a pre-trained model to classify flowers with better accuracy than a new model for use in a mobile application.\n\n## Learning Objectives\n1. Know how to apply image augmentation\n2. Know how to download and use a TensorFlow Hub module as a layer in Keras.",
"_____no_output_____"
]
],
[
[
"import os\nimport pathlib\n\nimport IPython.display as display\nimport matplotlib.pylab as plt\nimport numpy as np\nimport tensorflow as tf\nimport tensorflow_hub as hub\nfrom PIL import Image\nfrom tensorflow.keras import Sequential\nfrom tensorflow.keras.layers import (\n Conv2D,\n Dense,\n Dropout,\n Flatten,\n MaxPooling2D,\n Softmax,\n)",
"_____no_output_____"
]
],
[
[
"## Exploring the data\n\nAs usual, let's take a look at the data before we start building our model. We'll be using a creative-commons licensed flower photo dataset of 3670 images falling into 5 categories: 'daisy', 'roses', 'dandelion', 'sunflowers', and 'tulips'.\n\nThe below [tf.keras.utils.get_file](https://www.tensorflow.org/api_docs/python/tf/keras/utils/get_file) command downloads a dataset to the local Keras cache. To see the files through a terminal, copy the output of the cell below.",
"_____no_output_____"
]
],
[
[
"data_dir = tf.keras.utils.get_file(\n \"flower_photos\",\n \"https://storage.googleapis.com/download.tensorflow.org/example_images/flower_photos.tgz\",\n untar=True,\n)\n\n# Print data path\nprint(\"cd\", data_dir)",
"_____no_output_____"
]
],
[
[
"We can use python's built in [pathlib](https://docs.python.org/3/library/pathlib.html) tool to get a sense of this unstructured data.",
"_____no_output_____"
]
],
[
[
"data_dir = pathlib.Path(data_dir)\n\nimage_count = len(list(data_dir.glob(\"*/*.jpg\")))\nprint(\"There are\", image_count, \"images.\")\n\nCLASS_NAMES = np.array(\n [item.name for item in data_dir.glob(\"*\") if item.name != \"LICENSE.txt\"]\n)\nprint(\"These are the available classes:\", CLASS_NAMES)",
"_____no_output_____"
]
],
[
[
"Let's display the images so we can see what our model will be trying to learn.",
"_____no_output_____"
]
],
[
[
"roses = list(data_dir.glob(\"roses/*\"))\n\nfor image_path in roses[:3]:\n display.display(Image.open(str(image_path)))",
"_____no_output_____"
]
],
[
[
"## Building the dataset\n\nKeras has some convenient methods to read in image data. For instance [tf.keras.preprocessing.image.ImageDataGenerator](https://www.tensorflow.org/api_docs/python/tf/keras/preprocessing/image/ImageDataGenerator) is great for small local datasets. A tutorial on how to use it can be found [here](https://www.tensorflow.org/tutorials/load_data/images), but what if we have so many images, it doesn't fit on a local machine? We can use [tf.data.datasets](https://www.tensorflow.org/api_docs/python/tf/data/Dataset) to build a generator based on files in a Google Cloud Storage Bucket.\n\nWe have already prepared these images to be stored on the cloud in `gs://cloud-ml-data/img/flower_photos/`. The images are randomly split into a training set with 90% data and an iterable with 10% data listed in CSV files:\n\nTraining set: [train_set.csv](https://storage.cloud.google.com/cloud-ml-data/img/flower_photos/train_set.csv) \nEvaluation set: [eval_set.csv](https://storage.cloud.google.com/cloud-ml-data/img/flower_photos/eval_set.csv) \n\nExplore the format and contents of the train.csv by running:",
"_____no_output_____"
]
],
[
[
"!gsutil cat gs://cloud-ml-data/img/flower_photos/train_set.csv | head -5 > /tmp/input.csv\n!cat /tmp/input.csv",
"_____no_output_____"
],
[
"!gsutil cat gs://cloud-ml-data/img/flower_photos/train_set.csv | sed 's/,/ /g' | awk '{print $2}' | sort | uniq > /tmp/labels.txt\n!cat /tmp/labels.txt",
"_____no_output_____"
]
],
[
[
"Let's figure out how to read one of these images from the cloud. TensorFlow's [tf.io.read_file](https://www.tensorflow.org/api_docs/python/tf/io/read_file) can help us read the file contents, but the result will be a [Base64 image string](https://en.wikipedia.org/wiki/Base64). Hmm... not very readable for humans or Tensorflow.\n\nThankfully, TensorFlow's [tf.image.decode_jpeg](https://www.tensorflow.org/api_docs/python/tf/io/decode_jpeg) function can decode this string into an integer array, and [tf.image.convert_image_dtype](https://www.tensorflow.org/api_docs/python/tf/image/convert_image_dtype) can cast it into a 0 - 1 range float. Finally, we'll use [tf.image.resize](https://www.tensorflow.org/api_docs/python/tf/image/resize) to force image dimensions to be consistent for our neural network.\n\nWe'll wrap these into a function as we'll be calling these repeatedly. While we're at it, let's also define our constants for our neural network.",
"_____no_output_____"
]
],
[
[
"IMG_HEIGHT = 224\nIMG_WIDTH = 224\nIMG_CHANNELS = 3\n\nBATCH_SIZE = 32\n# 10 is a magic number tuned for local training of this dataset.\nSHUFFLE_BUFFER = 10 * BATCH_SIZE\nAUTOTUNE = tf.data.experimental.AUTOTUNE\n\nVALIDATION_IMAGES = 370\nVALIDATION_STEPS = VALIDATION_IMAGES // BATCH_SIZE",
"_____no_output_____"
],
[
"def decode_img(img, reshape_dims):\n # Convert the compressed string to a 3D uint8 tensor.\n img = tf.image.decode_jpeg(img, channels=IMG_CHANNELS)\n # Use `convert_image_dtype` to convert to floats in the [0,1] range.\n img = tf.image.convert_image_dtype(img, tf.float32)\n # Resize the image to the desired size.\n return tf.image.resize(img, reshape_dims)",
"_____no_output_____"
]
],
[
[
"Is it working? Let's see!\n\n**TODO 1.a:** Run the `decode_img` function and plot it to see a happy looking daisy.",
"_____no_output_____"
]
],
[
[
"img = tf.io.read_file(\n \"gs://cloud-ml-data/img/flower_photos/daisy/754296579_30a9ae018c_n.jpg\"\n)\n\n# Uncomment to see the image string.\n# print(img)\n# TODO: decode image and plot it",
"_____no_output_____"
]
],
[
[
"One flower down, 3669 more of them to go. Rather than load all the photos in directly, we'll use the file paths given to us in the csv and load the images when we batch. [tf.io.decode_csv](https://www.tensorflow.org/api_docs/python/tf/io/decode_csv) reads in csv rows (or each line in a csv file), while [tf.math.equal](https://www.tensorflow.org/api_docs/python/tf/math/equal) will help us format our label such that it's a boolean array with a truth value corresponding to the class in `CLASS_NAMES`, much like the labels for the MNIST Lab.",
"_____no_output_____"
]
],
[
[
"def decode_csv(csv_row):\n record_defaults = [\"path\", \"flower\"]\n filename, label_string = tf.io.decode_csv(csv_row, record_defaults)\n image_bytes = tf.io.read_file(filename=filename)\n label = tf.math.equal(CLASS_NAMES, label_string)\n return image_bytes, label",
"_____no_output_____"
]
],
[
[
"Next, we'll transform the images to give our network more variety to train on. There are a number of [image manipulation functions](https://www.tensorflow.org/api_docs/python/tf/image). We'll cover just a few:\n\n* [tf.image.random_crop](https://www.tensorflow.org/api_docs/python/tf/image/random_crop) - Randomly deletes the top/bottom rows and left/right columns down to the dimensions specified.\n* [tf.image.random_flip_left_right](https://www.tensorflow.org/api_docs/python/tf/image/random_flip_left_right) - Randomly flips the image horizontally\n* [tf.image.random_brightness](https://www.tensorflow.org/api_docs/python/tf/image/random_brightness) - Randomly adjusts how dark or light the image is.\n* [tf.image.random_contrast](https://www.tensorflow.org/api_docs/python/tf/image/random_contrast) - Randomly adjusts image contrast.\n\n**TODO 1.b:** Augment the image using the random functions.",
"_____no_output_____"
]
],
[
[
"MAX_DELTA = 63.0 / 255.0 # Change brightness by at most 17.7%\nCONTRAST_LOWER = 0.2\nCONTRAST_UPPER = 1.8\n\n\ndef read_and_preprocess(image_bytes, label, random_augment=False):\n if random_augment:\n img = decode_img(image_bytes, [IMG_HEIGHT + 10, IMG_WIDTH + 10])\n # TODO: augment the image.\n else:\n img = decode_img(image_bytes, [IMG_WIDTH, IMG_HEIGHT])\n return img, label\n\n\ndef read_and_preprocess_with_augment(image_bytes, label):\n return read_and_preprocess(image_bytes, label, random_augment=True)",
"_____no_output_____"
]
],
[
[
"Finally, we'll make a function to craft our full dataset using [tf.data.dataset](https://www.tensorflow.org/api_docs/python/tf/data/Dataset). The [tf.data.TextLineDataset](https://www.tensorflow.org/api_docs/python/tf/data/TextLineDataset) will read in each line in our train/eval csv files to our `decode_csv` function.\n\n[.cache](https://www.tensorflow.org/api_docs/python/tf/data/Dataset#cache) is key here. It will store the dataset in memory",
"_____no_output_____"
]
],
[
[
"def load_dataset(csv_of_filenames, batch_size, training=True):\n dataset = (\n tf.data.TextLineDataset(filenames=csv_of_filenames)\n .map(decode_csv)\n .cache()\n )\n\n if training:\n dataset = (\n dataset.map(read_and_preprocess_with_augment)\n .shuffle(SHUFFLE_BUFFER)\n .repeat(count=None)\n ) # Indefinately.\n else:\n dataset = dataset.map(read_and_preprocess).repeat(\n count=1\n ) # Each photo used once.\n\n # Prefetch prepares the next set of batches while current batch is in use.\n return dataset.batch(batch_size=batch_size).prefetch(buffer_size=AUTOTUNE)",
"_____no_output_____"
]
],
[
[
"We'll test it out with our training set. A batch size of one will allow us to easily look at each augmented image.",
"_____no_output_____"
]
],
[
[
"train_path = \"gs://cloud-ml-data/img/flower_photos/train_set.csv\"\ntrain_data = load_dataset(train_path, 1)\nitr = iter(train_data)",
"_____no_output_____"
]
],
[
[
"**TODO 1.c:** Run the below cell repeatedly to see the results of different batches. The images have been un-normalized for human eyes. Can you tell what type of flowers they are? Is it fair for the AI to learn on?",
"_____no_output_____"
]
],
[
[
"image_batch, label_batch = next(itr)\nimg = image_batch[0]\nplt.imshow(img)\nprint(label_batch[0])",
"_____no_output_____"
]
],
[
[
"**Note:** It may take a 4-5 minutes to see result of different batches. \n",
"_____no_output_____"
],
[
"## MobileNetV2\n\nThese flower photos are much larger than handwritting recognition images in MNIST. They are about 10 times as many pixels per axis **and** there are three color channels, making the information here over 200 times larger!\n\nHow do our current techniques stand up? Copy your best model architecture over from the <a href=\"2_mnist_models.ipynb\">MNIST models lab</a> and see how well it does after training for 5 epochs of 50 steps.\n\n**TODO 2.a** Copy over the most accurate model from 2_mnist_models.ipynb or build a new CNN Keras model.",
"_____no_output_____"
]
],
[
[
"eval_path = \"gs://cloud-ml-data/img/flower_photos/eval_set.csv\"\nnclasses = len(CLASS_NAMES)\nhidden_layer_1_neurons = 400\nhidden_layer_2_neurons = 100\ndropout_rate = 0.25\nnum_filters_1 = 64\nkernel_size_1 = 3\npooling_size_1 = 2\nnum_filters_2 = 32\nkernel_size_2 = 3\npooling_size_2 = 2\n\nlayers = [\n # TODO: Add your image model.\n]\n\nold_model = Sequential(layers)\nold_model.compile(\n optimizer=\"adam\", loss=\"categorical_crossentropy\", metrics=[\"accuracy\"]\n)\n\ntrain_ds = load_dataset(train_path, BATCH_SIZE)\neval_ds = load_dataset(eval_path, BATCH_SIZE, training=False)",
"_____no_output_____"
],
[
"old_model.fit_generator(\n train_ds,\n epochs=5,\n steps_per_epoch=5,\n validation_data=eval_ds,\n validation_steps=VALIDATION_STEPS,\n)",
"_____no_output_____"
]
],
[
[
"If your model is like mine, it learns a little bit, slightly better then random, but *ugh*, it's too slow! With a batch size of 32, 5 epochs of 5 steps is only getting through about a quarter of our images. Not to mention, this is a much larger problem then MNIST, so wouldn't we need a larger model? But how big do we need to make it?\n\nEnter Transfer Learning. Why not take advantage of someone else's hard work? We can take the layers of a model that's been trained on a similar problem to ours and splice it into our own model.\n\n[Tensorflow Hub](https://tfhub.dev/s?module-type=image-augmentation,image-classification,image-others,image-style-transfer,image-rnn-agent) is a database of models, many of which can be used for Transfer Learning. We'll use a model called [MobileNet](https://tfhub.dev/google/imagenet/mobilenet_v2_035_224/feature_vector/4) which is an architecture optimized for image classification on mobile devices, which can be done with [TensorFlow Lite](https://github.com/tensorflow/hub/blob/master/examples/colab/tf2_image_retraining.ipynb). Let's compare how a model trained on [ImageNet](http://www.image-net.org/) data compares to one built from scratch.\n\nThe `tensorflow_hub` python package has a function to include a Hub model as a [layer in Keras](https://www.tensorflow.org/hub/api_docs/python/hub/KerasLayer). We'll set the weights of this model as un-trainable. Even though this is a compressed version of full scale image classification models, it still has over four hundred thousand paramaters! Training all these would not only add to our computation, but it is also prone to over-fitting. We'll add some L2 regularization and Dropout to prevent that from happening to our trainable weights.\n\n**TODO 2.b**: Add a Hub Keras Layer at the top of the model using the handle provided.",
"_____no_output_____"
]
],
[
[
"module_selection = \"mobilenet_v2_100_224\"\nmodule_handle = \"https://tfhub.dev/google/imagenet/{}/feature_vector/4\".format(\n module_selection\n)\n\ntransfer_model = tf.keras.Sequential(\n [\n # TODO\n tf.keras.layers.Dropout(rate=0.2),\n tf.keras.layers.Dense(\n nclasses,\n activation=\"softmax\",\n kernel_regularizer=tf.keras.regularizers.l2(0.0001),\n ),\n ]\n)\ntransfer_model.build((None,) + (IMG_HEIGHT, IMG_WIDTH, IMG_CHANNELS))\ntransfer_model.summary()",
"_____no_output_____"
]
],
[
[
"Even though we're only adding one more `Dense` layer in order to get the probabilities for each of the 5 flower types, we end up with over six thousand parameters to train ourselves. Wow!\n\nMoment of truth. Let's compile this new model and see how it compares to our MNIST architecture.",
"_____no_output_____"
]
],
[
[
"transfer_model.compile(\n optimizer=\"adam\", loss=\"categorical_crossentropy\", metrics=[\"accuracy\"]\n)\n\ntrain_ds = load_dataset(train_path, BATCH_SIZE)\neval_ds = load_dataset(eval_path, BATCH_SIZE, training=False)",
"_____no_output_____"
],
[
"transfer_model.fit(\n train_ds,\n epochs=5,\n steps_per_epoch=5,\n validation_data=eval_ds,\n validation_steps=VALIDATION_STEPS,\n)",
"_____no_output_____"
]
],
[
[
"Alright, looking better!\n\nStill, there's clear room to improve. Data bottlenecks are especially prevalent with image data due to the size of the image files. There's much to consider such as the computation of augmenting images and the bandwidth to transfer images between machines.\n\nThink life is too short, and there has to be a better way? In the next lab, we'll blast away these problems by developing a cloud strategy to train with TPUs!\n\n## Bonus Exercise\n\nKeras has a [local way](https://keras.io/models/sequential/) to do distributed training, but we'll be using a different technique in the next lab. Want to give the local way a try? Check out this excellent [blog post](https://stanford.edu/~shervine/blog/keras-how-to-generate-data-on-the-fly) to get started. Or want to go full-blown Keras? It also has a number of [pre-trained models](https://keras.io/applications/) ready to use.",
"_____no_output_____"
],
[
"Copyright 2019 Google Inc.\nLicensed under the Apache License, Version 2.0 (the \"License\"); you may not use this file except in compliance with the License. You may obtain a copy of the License at\nhttp://www.apache.org/licenses/LICENSE-2.0\nUnless required by applicable law or agreed to in writing, software distributed under the License is distributed on an \"AS IS\" BASIS, WITHOUT WARRANTIES OR CONDITIONS OF ANY KIND, either express or implied. See the License for the specific language governing permissions and limitations under the License.",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown",
"markdown"
]
] |
e7ec4829e00e8c48cd647d130d28896c6665ac98 | 219,861 | ipynb | Jupyter Notebook | figures/figure02.ipynb | UBC-MOAD/Saldias_et_al_2021 | b3736d17bb72455897b0fa52f143836ae12eecce | [
"Apache-2.0"
] | null | null | null | figures/figure02.ipynb | UBC-MOAD/Saldias_et_al_2021 | b3736d17bb72455897b0fa52f143836ae12eecce | [
"Apache-2.0"
] | 1 | 2021-05-13T01:47:33.000Z | 2021-05-13T01:47:33.000Z | figures/figure02.ipynb | UBC-MOAD/Saldias_et_al_2021 | b3736d17bb72455897b0fa52f143836ae12eecce | [
"Apache-2.0"
] | null | null | null | 505.427586 | 201,116 | 0.929919 | [
[
[
"### Saldías et al. Figure 02\n\nWaves - ssh anomaly (canyon minus no-canyon), allowed and scattered waves",
"_____no_output_____"
]
],
[
[
"from brokenaxes import brokenaxes\nimport cmocean as cmo\nimport matplotlib.pyplot as plt\nfrom matplotlib.gridspec import GridSpec\nimport matplotlib.gridspec as gspec\nimport matplotlib.patches as patches\nfrom netCDF4 import Dataset\nimport numpy as np\nimport pandas as pd\nimport scipy as sc\nimport scipy.io as sio\nimport xarray as xr\n\nimport matplotlib.colors as mcolors\nimport matplotlib.lines as mlines\nfrom matplotlib.lines import Line2D\n\n%matplotlib inline\n%matplotlib inline",
"_____no_output_____"
],
[
"def get_fig_file(file_fig):\n # Brink mode\n file = sio.loadmat(file_fig)\n z, xpl, xxx, zzz = file['z'][0,:], file['xpl'][0,:], file['xxx'][0,:], file['zzz'][0,:]\n \n # (u is cross-shore and v is alongshore in Brink.)\n p0, u0, v0, w0, r0 = file['p_profile'], file['u_profile'],file['v_profile'], file['w_profile'], file['r_profile']\n\n scale=0.2\n w = w0 * 0.01 * scale # cms-1 to ms-1 and normalization (?)\n u = u0 * 0.01 * scale # cms-1 to ms-1 and normalization \n v = v0 * 0.01 * scale # cms-1 to ms-1 and normalization \n r = r0 * 1.0 * scale # mg/cm³ to kg/m³ and normalization\n p = p0 * 0.1 * scale # dyn/cm² to 0.1 Pa (or kg m-1 s-2) and normalization\n return(u,v,w,r,p,z,xpl, xxx, zzz)\n\ndef plot_Brink(ax,fld,z,xpl,xxx,zzz,minp,maxp,nlev=15):\n landc='#8b7765'\n levels=np.linspace(minp,maxp,nlev)\n cnf = ax.contourf(xpl, z, fld, levels=levels, cmap=cmo.cm.delta, vmin=minp, \n vmax=maxp, zorder=1)\n ax.contour(xpl, z, fld, levels=levels, linewidths=1, linestyles='-', colors='0.4', zorder=2)\n ax.contour(xpl, z, fld, levels=[0], linewidths=2, linestyles='-', colors='k', zorder=2)\n ax.fill_between(xxx, zzz.min(), zzz, facecolor=landc, zorder=3)\n levels=np.linspace(np.nanmin(v),np.nanmax(v),nlev)\n return(cnf, ax) ",
"_____no_output_____"
],
[
"runs = ['DS','IS','SS']",
"_____no_output_____"
],
[
"fig = plt.figure(figsize=(7.48,9))\nplt.rcParams.update({'font.size': 8})\n\n# Set up subplot grid\ngs = GridSpec(4, 3, width_ratios=[1,1,1], height_ratios=[0.6,1.3,1.5,1.3],\n wspace=0.1,hspace=0.3, figure=fig)\n\nax1 = fig.add_subplot(gs[0, 0])\nax2 = fig.add_subplot(gs[0, 1])\nax3 = fig.add_subplot(gs[0, 2])\nax4 = fig.add_subplot(gs[1, 0])\nax5 = fig.add_subplot(gs[1, 1])\nax6 = fig.add_subplot(gs[1, 2])\nax7 = fig.add_subplot(gs[2, 0])\nax8 = fig.add_subplot(gs[2, 1:])\nax9 = fig.add_subplot(gs[3, 0])\nax10 = fig.add_subplot(gs[3, 1])\nax11 = fig.add_subplot(gs[3, 2])\n\nfor ax in [ax2,ax3,ax5,ax6,ax10,ax11]:\n ax.set_yticks([])\n \nfor ax,run in zip([ax1,ax2,ax3],runs):\n ax.set_xlabel('x (km)', labelpad=0)\n ax.set_title(run)\n\nfor ax in [ax4,ax5,ax6,ax7]:\n ax.set_xlabel('Days', labelpad=0)\n\nfor ax in [ax9,ax10,ax11]:\n ax.set_xlabel('x (km)', labelpad=0)\n\nax1.set_ylabel('Depth (m)', labelpad=0)\nax4.set_ylabel('y (km)', labelpad=0)\nax7.set_ylabel('y (km)', labelpad=0)\nax9.set_ylabel('Depth (m)', labelpad=0)\nax8.set_xlabel(r'$k$ ($10^{-5}$ rad m$^{-1}$)', labelpad=0)\nax8.set_ylabel(r'$\\omega$ ($10^{-5}$ rad s$^{-1}$)', labelpad=0.5)\nax8.yaxis.set_label_position(\"right\")\nax8.yaxis.tick_right()\n\n# Shelf profiles\nfor run, ax in zip(runs, [ax1,ax2,ax3]):\n can_file = '/Volumes/MOBY/ROMS-CTW/ocean_his_ctw_CR_'+run+'_7d.nc'\n yshelf = 400\n yaxis = int(579/2)\n with Dataset(can_file, 'r') as nbl:\n hshelf = -nbl.variables['h'][yshelf,:]\n haxis = -nbl.variables['h'][yaxis,:]\n x_rho = (nbl.variables['x_rho'][:]-400E3)/1000\n y_rho = (nbl.variables['y_rho'][:]-400E3)/1000 \n ax.plot(x_rho[yshelf,:], hshelf,'k-', label='shelf')\n ax.plot(x_rho[yaxis,:], haxis,'k:', label='canyon \\n axis')\n ax.set_xlim(-50,0)\n ax.set_ylim(-500,0)\nax1.legend(labelspacing=0) \n \n#SSH hovmöller plots (canyon-no canyon)\nxind = 289\nfor run, ax in zip(runs,(ax4,ax5,ax6)):\n nc_file = '/Volumes/MOBY/ROMS-CTW/ocean_his_ctw_NCR_'+run+'_7d.nc'\n can_file = '/Volumes/MOBY/ROMS-CTW/ocean_his_ctw_CR_'+run+'_7d.nc'\n\n with Dataset(can_file, 'r') as nbl:\n y_rho = nbl.variables['y_rho'][:]\n time = nbl.variables['ocean_time'][:]\n zeta = nbl.variables['zeta'][:,:,xind]\n\n with Dataset(nc_file, 'r') as nbl:\n y_rho_nc = nbl.variables['y_rho'][:]\n time_nc = nbl.variables['ocean_time'][:]\n zeta_nc = nbl.variables['zeta'][:,:,xind]\n \n pc2 = ax.pcolormesh((time_nc)/(3600*24),(y_rho_nc[:,xind]/1000)-400,\n np.transpose((zeta[:,:]-zeta_nc[:,:]))*1000,\n cmap=cmo.cm.balance, vmax=4.0, vmin=-4.0)\n if run == 'IS':\n rect = patches.Rectangle((5,-20),15,160,linewidth=2,edgecolor='k',facecolor='none')\n ax.add_patch(rect)\n\n ax.axhline(0.0, color='k', alpha=0.5)\n ax.set_ylim(-400,400)\ncbar_ax = fig.add_axes([0.92, 0.585, 0.025, 0.17])\ncb = fig.colorbar(pc2, cax=cbar_ax, orientation='vertical', format='%1.0f')\ncb.set_label(r'Surface elevation (10$^{-3}$ m)')\n\n# Zoomed-in SSH hovmöller plot of IS (canyon-no canyon)\nyind = 420\nxlim = 100\nxind = 289\ny1 = 189\ny2 = 389\ny3 = 526\ny4 = 540\ny5 = 315\n\nrun = 'IS'\nax = ax7\nnc_file = '/Volumes/MOBY/ROMS-CTW/ocean_his_ctw_NCR_'+run+'_7d.nc'\ncan_file = '/Volumes/MOBY/ROMS-CTW/ocean_his_ctw_CR_'+run+'_7d.nc'\n\nwith Dataset(can_file, 'r') as nbl:\n y_rho = nbl.variables['y_rho'][:]\n time = nbl.variables['ocean_time'][:]\n zeta = nbl.variables['zeta'][:,:,xind]\n\nwith Dataset(nc_file, 'r') as nbl:\n y_rho_nc = nbl.variables['y_rho'][:]\n time_nc = nbl.variables['ocean_time'][:]\n zeta_nc = nbl.variables['zeta'][:,:,xind]\n \npc2 = ax.pcolormesh((time_nc)/(3600*24),(y_rho_nc[:,xind]/1000)-400,\n np.transpose((zeta[:,:]-zeta_nc[:,:]))*1000,\n cmap=cmo.cm.balance, vmax=4.0, vmin=-4.0)\n\nt1_IS = (time_nc[47])/(3600*24)\ny1_IS = (y_rho_nc[y2,xind]/1000)-400\nt2_IS = (time_nc[65])/(3600*24)\ny2_IS = (y_rho_nc[y4,xind]/1000)-400\nax.plot([t1_IS, t2_IS],[y1_IS, y2_IS], '.-', color='k')\n\nt1_IS = (time_nc[47])/(3600*24)\ny1_IS = (y_rho_nc[289,xind]/1000)-400\nt2_IS = (time_nc[55])/(3600*24)\ny2_IS = (y_rho_nc[y2,xind]/1000)-400\nax.plot([t1_IS, t2_IS],[y1_IS, y2_IS], '.-',color='k')\n\n\nax.axhline(0.0, color='k', alpha=0.5)\nax.axhline(-5.0, color='0.5', alpha=0.5)\nax.axhline(5.0, color='0.5', alpha=0.5)\nax.set_ylim(-20,140)\nax.set_xlim(5,20)\nrect = patches.Rectangle((5.1,-19),14.85,158,linewidth=2,edgecolor='k',facecolor='none')\nax.add_patch(rect)\n\n# Dispersion curves\ng = 9.81 # gravitational accel. m/s^2\nHs = 100 # m shelf break depth\nf = 1.028E-4 # inertial frequency\nomega_fw = 1.039E-5 # fw = forcing wave\nk_fw = 6.42E-6# rad/m\ndomain_length = 800E3 # m\ncanyon_width = 10E3 # m\n\ncol1 = '#254441' #'#23022e'\ncol2 = '#43AA8B' #'#573280'\ncol3 = '#B2B09B' #'#ada8b6'\ncol4 = '#FF6F59' #'#58A4B0'\n\nfiles = ['../dispersion_curves/DS/dispc_DS_mode1_KRM.dat',\n '../dispersion_curves/IS/dispc_IS_mode1_KRM.dat',\n '../dispersion_curves/SS/dispc_SS_mode1_KRM.dat',\n '../dispersion_curves/DS/dispc_DS_mode2_KRM.dat',\n '../dispersion_curves/IS/dispc_IS_mode2_KRM.dat',\n '../dispersion_curves/SS/dispc_SS_mode2_KRM.dat',\n '../dispersion_curves/DS/dispc_DS_mode3_KRM.dat',\n '../dispersion_curves/IS/dispc_IS_mode3_KRM.dat',\n '../dispersion_curves/SS/dispc_SS_mode3_KRM.dat',\n '../dispersion_curves/IS/dispc_IS_mode4_KRM.dat',\n '../dispersion_curves/SS/dispc_SS_mode4_KRM.dat',\n '../dispersion_curves/DS/dispc_DS_mode5_KRM.dat', \n '../dispersion_curves/IS/dispc_IS_mode5_KRM.dat',\n '../dispersion_curves/SS/dispc_SS_mode5_KRM.dat',\n '../dispersion_curves/IS/dispc_IS_mode6_KRM.dat',\n '../dispersion_curves/SS/dispc_SS_mode6_KRM.dat',\n ]\ncolors = [col1,\n col2,\n col3,\n col1,\n col2,\n col3,\n col1,\n col2,\n col3,\n col2,\n col3,\n col1,\n col2,\n col3,\n #col1,\n col2,\n col3,\n ]\nlinestyles = ['-','-','-','--','--','--',':',':',':','-.','-.','-','-','-','--','--']\nlabels = [ r'DS $\\bar{c_1}$',r'IS $\\bar{c_1}$',r'SS $\\bar{c_1}$',\n r'DS $\\bar{c_2}$',r'IS $\\bar{c_2}$',r'SS $\\bar{c_2}$',\n r'DS $\\bar{c_3}$',r'IS $\\bar{c_3}$',r'SS $\\bar{c_3}$',\n r'IS $\\bar{c_4}$',r'SS $\\bar{c_4}$',\n r'DS $\\bar{c_5}$',r'IS $\\bar{c_5}$',r'SS $\\bar{c_5}$',\n r'IS $\\bar{c_6}$',r'SS $\\bar{c_6}$']\n\nax8.axhline(omega_fw*1E5, color='0.5', label='1/7 days')\nax8.axhline(f*1E5, color='gold', label='f')\nax8.axvline((1E5*(2*np.pi))/domain_length, linestyle='-', color=col4, alpha=1, label='domain length')\n\nfor file, col, lab, line in zip(files, colors, labels, linestyles):\n data_mode = pd.read_csv(file, delim_whitespace=True, header=None, names=['wavenum', 'freq', 'perturbation'])\n omega = data_mode['freq'][:]\n k = data_mode['wavenum'][:]*100\n ax8.plot(k*1E5, omega*1E5, linestyle=line,\n color=col,linewidth=2,alpha=0.9,\n label=lab+r'=%1.2f ms$^{-1}$' % (np.mean(omega/k)))\n\nax8.plot((omega_fw/1.59)*1E5, omega_fw*1E5, '^',color=col1, \n markersize=9, label = 'incident DS %1.2f' %(1.59),\n markeredgecolor='0.2',markeredgewidth=1)\n\nax8.plot((omega_fw/1.39)*1E5, omega_fw*1E5, '^',color=col2, \n markersize=9, label = 'incident IS %1.2f' %(1.39),\n markeredgecolor='0.2',markeredgewidth=1)\n\nax8.plot((omega_fw/1.29)*1E5, omega_fw*1E5, '^',color=col3, \n markersize=9, label = 'incident SS %1.2f' %(1.29),\n markeredgecolor='0.2',markeredgewidth=1)\n\nax8.plot((omega_fw/0.32)*1E5, omega_fw*1E5, 'o',color=col1, \n markersize=9, label = 'DS model c=%1.2f m/s' %(0.32),\n markeredgecolor='0.2',markeredgewidth=1)\n\nax8.plot((omega_fw/0.23)*1E5, omega_fw*1E5, 'o',color=col2, \n markersize=9, label = 'IS model c=%1.2f m/s' %(0.23),\n markeredgecolor='0.2',markeredgewidth=1)\n\nax8.plot((omega_fw/1.04)*1E5, omega_fw*1E5, 'o',color=col3, \n markersize=9, label = 'SS model c=%1.2f m/s' %(1.04),\n markeredgecolor='0.2',markeredgewidth=1)\n\nax8.plot((omega_fw/0.14)*1E5, omega_fw*1E5, 'd',color=col1, \n markersize=11, label = 'DS model c=%1.2f m/s' %(0.14),\n markeredgecolor='0.2',markeredgewidth=1)\n\nax8.plot((omega_fw/0.14)*1E5, omega_fw*1E5, 'd',color=col2, \n markersize=9, label = 'IS model c=%1.2f m/s' %(0.14),\n markeredgecolor='0.2',markeredgewidth=1)\n\nax8.set_ylim(0, 1.5)\nax8.set_xlim(0,8)\n\nlegend_elements=[]\n\nlegend_elements.append(Line2D([0], [0], marker='^',color='w', label='incident',\n markerfacecolor='k', mec='k',markersize=6))\nlegend_elements.append(Line2D([0], [0], marker='o',color='w', label='1$^{st}$ scattered',\n markerfacecolor='k', mec='k',markersize=6))\nlegend_elements.append(Line2D([0], [0], marker='d',color='w', label='2$^{nd}$ scattered',\n markerfacecolor='k', mec='k',markersize=6))\n\nfor col, run in zip([col1,col2,col3], runs):\n legend_elements.append(Line2D([0], [0], marker='s',color=col, linewidth=4,label=run,\n markerfacecolor=col, mec=col, markersize=0))\n\nax8.legend(handles=legend_elements, bbox_to_anchor=(0.65,0.32),frameon=False, handlelength=0.7,\n handletextpad=0.5, ncol=2,columnspacing=0.25, framealpha=0, edgecolor='w',labelspacing=0.2)\n\n# Mode structure (Modes 1, 4 and 6 IS run)\nrun='IS'\nmodes = ['mode1','mode3', 'mode5']\nfor mode, ax in zip(modes, [ax9,ax10,ax11]):\n u,v,w,r,p,z,xpl,xxx,zzz = get_fig_file('../dispersion_curves/'+run+'/figures_'+run+'_'+mode+'_KRM.mat')\n minp = -(1.66e-06)*1E6\n maxp = (1.66e-06)*1E6\n cntf, ax = plot_Brink(ax, p*1E6, z, xpl, xxx, zzz, minp, maxp)\n ax.set_xlim(0,50)\ncbar_ax = fig.add_axes([0.92, 0.125, 0.025, 0.17])\ncb = fig.colorbar(cntf, cax=cbar_ax, orientation='vertical', format='%1.1f')\ncb.set_label(r'Pressure (10$^{-6}$ Pa)')\n\nax9.text(0.5,0.9,'Incident wave',transform=ax9.transAxes, fontweight='bold')\nax10.text(0.5,0.9,'Mode 3 (IS)',transform=ax10.transAxes, fontweight='bold')\nax11.text(0.5,0.9,'Mode 5 (IS)',transform=ax11.transAxes, fontweight='bold')\n\nax8.text(0.09,0.75,'mode 1',transform=ax8.transAxes,rotation=70 )\nax8.text(0.27,0.75,'mode 2',transform=ax8.transAxes,rotation=51 )\nax8.text(0.43,0.75,'mode 3',transform=ax8.transAxes,rotation=41 )\nax8.text(0.65,0.75,'mode 4',transform=ax8.transAxes,rotation=30 )\nax8.text(0.87,0.72,'mode 5',transform=ax8.transAxes,rotation=25 )\nax8.text(0.87,0.47,'mode 6',transform=ax8.transAxes,rotation=18 )\n\nax1.text(0.95,0.05,'a',transform=ax1.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\nax2.text(0.95,0.05,'b',transform=ax2.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\nax3.text(0.95,0.05,'c',transform=ax3.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\n\nax4.text(0.95,0.03,'d',transform=ax4.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\nax5.text(0.95,0.03,'e',transform=ax5.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\nax6.text(0.96,0.03,'f',transform=ax6.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\n\n\nax7.text(0.01,0.94,'g',transform=ax7.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\nax8.text(0.01,0.03,'h',transform=ax8.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\n\nax9.text(0.97,0.03,'i',transform=ax9.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\nax10.text(0.97,0.03,'j',transform=ax10.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\nax11.text(0.95,0.03,'k',transform=ax11.transAxes, fontsize=8, fontweight='bold',\n color='w', bbox={'facecolor': 'black', 'alpha': 1, 'pad': 1})\n\nplt.savefig('Figure2.png',format='png',bbox_inches='tight', dpi=300)\nplt.show()",
"_____no_output_____"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code"
]
] |
e7ec505f8e53e6d090873ccb2910ce2ec84a53c4 | 13,655 | ipynb | Jupyter Notebook | Car Data Analysis.ipynb | elaesme/Car-Data-Analysis | ff63229cbdcb915d6311fed352a52cb75114993e | [
"MIT"
] | null | null | null | Car Data Analysis.ipynb | elaesme/Car-Data-Analysis | ff63229cbdcb915d6311fed352a52cb75114993e | [
"MIT"
] | null | null | null | Car Data Analysis.ipynb | elaesme/Car-Data-Analysis | ff63229cbdcb915d6311fed352a52cb75114993e | [
"MIT"
] | null | null | null | 34.834184 | 289 | 0.458733 | [
[
[
"## Rating and Review Analysis of Car Brands Project \n\nAuthor: Sabriye Ela Esme\n\nThis notebook includes the codes for analyzing rating score and review score of different brands of cars according to 'Edmunds-Consumer Car Ratings and Reviews' data retrieved from https://www.kaggle.com/ankkur13/edmundsconsumer-car-ratings-and-reviews.\n\nFirstly, for every brand, the data converted into a dataframe, and from that dataframe, the ratings given to the cars of the brand (between 0-5) summed up and divided by the number of reviews to get an average rating score. Let's start with Porsche.",
"_____no_output_____"
]
],
[
[
"import pandas as pd\nimport numpy as np\nlst=[]\n\ndata = pd.read_csv('C:\\\\Users\\\\Ela\\\\Desktop\\\\Cars_review_project\\\\Scraped_Car_Review_porsche.csv', lineterminator='\\n')\nbrand0= 'Porsche'",
"_____no_output_____"
]
],
[
[
"Here's a glimpse of dataframe of Porsche.",
"_____no_output_____"
]
],
[
[
"data.head()",
"_____no_output_____"
],
[
"score0=sum(data['Rating\\r'])/data.shape[0]\nlst.append([brand0, score0])",
"_____no_output_____"
]
],
[
[
"## Calculation of the score for other brands",
"_____no_output_____"
]
],
[
[
"#Calculation of the score for other brands\ndata1 = pd.read_csv('C://Users//Ela//Desktop//Cars_review_project//Scrapped_Car_Reviews_Audi.csv', lineterminator='\\n')\nbrand1= 'Audi'\nscore1=sum(data1['Rating\\r'])/data1.shape[0]\nlst.append([brand1, score1])\n\ndata2 = pd.read_csv('C://Users//Ela//Desktop//Cars_review_project//Scrapped_Car_Reviews_BMW.csv', lineterminator='\\n')\nbrand2= 'BMW'\nscore2=sum(data2['Rating\\r'])/data2.shape[0]\nlst.append([brand2, score2])\n\ndata3 = pd.read_csv('C:\\\\Users\\\\Ela\\\\Desktop\\\\Cars_review_project\\\\Scraped_Car_Review_mercedes-benz.csv', lineterminator='\\n')\nbrand3=' Mercedes-Benz'\nscore3=sum(data3['Rating\\r'])/data3.shape[0]\nlst.append([brand3, score3])\n\ndata4 = pd.read_csv('C:\\\\Users\\\\Ela\\\\Desktop\\\\Cars_review_project\\\\Scraped_Car_Review_jaguar.csv', lineterminator='\\n')\nbrand4='Jaguar'\nscore4=sum(data4['Rating\\r'])/data4.shape[0]\nlst.append([brand4, score4])",
"_____no_output_____"
]
],
[
[
"## Scores for Reviews\n\nIn this part of the code, there are two different functions to find the total review score of a brand. First the review function gets every review as a single sentence and checks the words to give the sentence a score based on how positive or negative the words in the review are.\n\nThe second function called total_score is to find the average review score for a single brand. It checks every review from the dataframes created earlier to calculate an average review score",
"_____no_output_____"
]
],
[
[
"def review_score(liste):\n words=[\"love\", \"amazing\", \"happy\", \"great\", \"best\", \"win\", \"powerful\", \"beatiful\"]\n negwords=[\"hate\", \"regret\", \"bad\", \"weak\", \"disappointed\", \"sad\"]\n scorepos=0\n scoreneg=0\n for i in liste:\n if i in words:\n scorepos+=1\n elif i in negwords:\n scoreneg+=1\n \n tot= scorepos - scoreneg\n return tot\n",
"_____no_output_____"
],
[
"def total_score(datframe):\n rlist=[]\n rscore=0\n for k in range(len(datframe[\"Review\"])):\n bb= datframe[\"Review\"][k].split()\n aa = [strings.lower() for strings in bb]\n res= review_score(aa)\n rscore+=res\n\n return rscore/datframe.shape[0]",
"_____no_output_____"
]
],
[
[
"From this line, the code is about creating a new dataframe which shows the average rating score, average review score and the total score (sum of the 2 different averages) for every brand, in a descending order according to total score.",
"_____no_output_____"
]
],
[
[
"totlist=[total_score(data), total_score(data1), total_score(data2), total_score(data3),total_score(data4)]\nfor i in range(len(lst)):\n lst[i].append(totlist[i])\n lst[i].append(totlist[i]+lst[i][1])\n\nlst.sort(key=lambda x:x[3], reverse= 1)\ndf = pd.DataFrame(lst[0:],columns=['Brand', 'Average Rating Score', 'Average Review Score', 'Total Score'])\ndf",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7ec5203639ed1170e5b195cfbfd64e13c536b4b | 88,339 | ipynb | Jupyter Notebook | quchem_examples/Simulating Quantum Circuit.ipynb | AlexisRalli/VQE-code | 4112d2bba4c327360e95dfd7cb6120b2ce67bf29 | [
"MIT"
] | 1 | 2021-04-01T14:01:46.000Z | 2021-04-01T14:01:46.000Z | quchem_examples/Simulating Quantum Circuit.ipynb | AlexisRalli/VQE-code | 4112d2bba4c327360e95dfd7cb6120b2ce67bf29 | [
"MIT"
] | 5 | 2019-11-13T16:23:54.000Z | 2021-04-07T11:03:06.000Z | quchem_examples/Simulating Quantum Circuit.ipynb | AlexisRalli/VQE-code | 4112d2bba4c327360e95dfd7cb6120b2ce67bf29 | [
"MIT"
] | null | null | null | 128.586608 | 31,188 | 0.845085 | [
[
[
"from quchem.Simulating_Quantum_Circuit import *\nfrom quchem.Ansatz_Generator_Functions import *\nfrom openfermion.ops import QubitOperator\nHF_circ = [cirq.X.on(cirq.LineQubit(0)), cirq.X.on(cirq.LineQubit(1))]\nx = QubitOperator('X0 Y1 Z2 Y3', 0.25j)\ntheta = np.pi\nfull_exp_circ_obj = full_exponentiated_PauliWord_circuit(x, theta)\nUCCSD_circ = cirq.Circuit(cirq.decompose_once((full_exp_circ_obj(*cirq.LineQubit.range(full_exp_circ_obj.num_qubits())))))\nYY = QubitOperator('X0 X1 Y3', 0.25j)\npauliword_change_and_measure_obj = change_pauliword_to_Z_basis_then_measure(YY)\nP_measure = cirq.Circuit(cirq.decompose_once((pauliword_change_and_measure_obj(*cirq.LineQubit.range(pauliword_change_and_measure_obj.num_qubits())))))\n\nfull_circuit = cirq.Circuit([*HF_circ, *UCCSD_circ.all_operations(), *P_measure.all_operations()])\nprint(full_circuit)",
"0: ───X──────────H──────────@───────────────────────────────────────@──────────H───────────Ry(-0.5π)───M───\n │ │ │\n1: ───X──────────Rx(0.5π)───X───@───────────────────────@───────────X──────────Rx(-0.5π)───Ry(-0.5π)───M───\n │ │ │\n2: ─────────────────────────────X───@───────────────@───X──────────────────────────────────────────────┼───\n │ │ │\n3: ───Rx(0.5π)──────────────────────X───Rz(-0.5π)───X───Rx(-0.5π)───Rx(0.5π)───────────────────────────M───\n"
]
],
[
[
"## Get_Histogram_key",
"_____no_output_____"
]
],
[
[
"YY = QubitOperator('X0 X1 Y3', 0.25j)\nGet_Histogram_key(YY)",
"_____no_output_____"
]
],
[
[
"## Simulate_Quantum_Circuit",
"_____no_output_____"
]
],
[
[
"num_shots = 1000\nYY = QubitOperator('X0 X1 Y3', 0.25j)\n\nhistogram_string= Get_Histogram_key(YY)\nSimulate_Quantum_Circuit(full_circuit, num_shots, histogram_string)",
"_____no_output_____"
]
],
[
[
"## Get_wavefunction",
"_____no_output_____"
]
],
[
[
"YY = QubitOperator('X0 X1 Y3', 0.25j)\n\ncirq_NO_M = cirq.Circuit([*HF_circ, *UCCSD_circ.all_operations()])\n\nhistogram_string= Get_Histogram_key(YY)\nGet_wavefunction(cirq_NO_M, sig_figs=3)",
"_____no_output_____"
]
],
[
[
"## Return_as_binary",
"_____no_output_____"
]
],
[
[
"num_shots = 1000\nYY = QubitOperator('X0 X1 Y3', 0.25j)\n\nhistogram_string= Get_Histogram_key(YY)\nc_result = Simulate_Quantum_Circuit(full_circuit, num_shots, histogram_string)\nReturn_as_binary(c_result, histogram_string)",
"_____no_output_____"
]
],
[
[
"## expectation_value_by_parity",
"_____no_output_____"
]
],
[
[
"num_shots = 1000\nYY = QubitOperator('X0 X1 Y3', 0.25j)\n\nhistogram_string= Get_Histogram_key(YY)\nc_result = Simulate_Quantum_Circuit(full_circuit, num_shots, histogram_string)\nb_result=Return_as_binary(c_result, histogram_string)\n\nexpectation_value_by_parity(b_result)",
"_____no_output_____"
],
[
"from quchem.Hamiltonian_Generator_Functions import *\n### Parameters\nMolecule = 'H2'\ngeometry = [('H', (0., 0., 0.)), ('H', (0., 0., 0.74))]\nbasis = 'sto-3g'\n\n### Get Hamiltonian\nHamilt = Hamiltonian(Molecule,\n run_scf=1, run_mp2=1, run_cisd=1, run_ccsd=1, run_fci=1,\n basis=basis,\n multiplicity=1,\n geometry=geometry) # normally None!\n\nHamilt.Get_Molecular_Hamiltonian(Get_H_matrix=False)\nQubitHam = Hamilt.Get_Qubit_Hamiltonian(transformation='JW')\n\nansatz_obj = Ansatz(Hamilt.molecule.n_electrons, Hamilt.molecule.n_qubits)\n\nSec_Quant_CC_ia_ops, Sec_Quant_CC_ijab_ops, theta_parameters_ia, theta_parameters_ijab = ansatz_obj.Get_ia_and_ijab_terms()\n\nQubit_Op_list_Second_Quant_CC_Ops_ia, Qubit_Op_list_Second_Quant_CC_Ops_ijab = ansatz_obj.UCCSD_single_trotter_step(Sec_Quant_CC_ia_ops, Sec_Quant_CC_ijab_ops,\n transformation='JW')\n\nfull_ansatz_Q_Circ = Ansatz_Circuit(Qubit_Op_list_Second_Quant_CC_Ops_ia, Qubit_Op_list_Second_Quant_CC_Ops_ijab,\n Hamilt.molecule.n_qubits, Hamilt.molecule.n_electrons)\n\nansatz_cirq_circuit = full_ansatz_Q_Circ.Get_Full_HF_UCCSD_QC(theta_parameters_ia, theta_parameters_ijab)",
"_____no_output_____"
],
[
"QubitHam",
"_____no_output_____"
]
],
[
[
"$$\\begin{aligned} H &=h_{0} I+h_{1} Z_{0}+h_{2} Z_{1}+h_{3} Z_{2}+h_{4} Z_{3} \\\\ &+h_{5} Z_{0} Z_{1}+h_{6} Z_{0} Z_{2}+h_{7} Z_{1} Z_{2}+h_{8} Z_{0} Z_{3}+h_{9} Z_{1} Z_{3} \\\\ &+h_{10} Z_{2} Z_{3}+h_{11} Y_{0} Y_{1} X_{2} X_{3}+h_{12} X_{0} Y_{1} Y_{2} X_{3} \\\\ &+h_{13} Y_{0} X_{1} X_{2} Y_{3}+h_{14} X_{0} X_{1} Y_{2} Y_{3} \\end{aligned}$$",
"_____no_output_____"
]
],
[
[
"n_shots=1000\n\ndef GIVE_ENERGY(theta_ia_theta_jab_list):\n theta_ia = theta_ia_theta_jab_list[:len(theta_parameters_ia)]\n theta_ijab = theta_ia_theta_jab_list[len(theta_parameters_ia):]\n \n ansatz_cirq_circuit = full_ansatz_Q_Circ.Get_Full_HF_UCCSD_QC(theta_parameters_ia, theta_parameters_ijab)\n\n VQE_exp = VQE_Experiment(QubitHam, ansatz_cirq_circuit, n_shots)\n \n\n return VQE_exp.Calc_Energy()\n\n### optimizer\nfrom quchem.Scipy_Optimizer import *\n# THETA_params = [*theta_parameters_ia, *theta_parameters_ijab]\nTHETA_params=[0,0,0]\nGG = Optimizer(GIVE_ENERGY, THETA_params, 'Nelder-Mead', store_values=True, display_iter_steps=True,\n tol=1e-5,\n display_convergence_message=True)\nGG.get_env(50)\nGG.plot_convergence()\nplt.show()",
"0: Input_to_Funct: [ 0.00025 -0.0005 0.00025]: Output: -1.1188432276915568\n1: Input_to_Funct: [ 0.00025 -0.0005 0.00025]: Output: -1.11630628122307\n2: Input_to_Funct: [ 0.00025 -0.0005 0.00025]: Output: -1.1183902015364695\n3: Input_to_Funct: [ 0.00025 -0.0005 0.00025]: Output: -1.1133163085994964\n4: Input_to_Funct: [ 0.00025 -0.0005 0.00025]: Output: -1.1117760196722009\n5: Input_to_Funct: [ 0.00025 -0.0005 0.00025]: Output: -1.123735910166495\n6: Input_to_Funct: [ 0.00023032 -0.00044907 0.00021644]: Output: -1.1175747544573131\n7: Input_to_Funct: [ 0.00023032 -0.00044907 0.00021644]: Output: -1.1210177532359737\n8: Input_to_Funct: [ 0.00023032 -0.00044907 0.00021644]: Output: -1.1144941766027223\n9: Input_to_Funct: [ 0.00023032 -0.00044907 0.00021644]: Output: -1.1121384405962706\n10: Input_to_Funct: [ 0.00023032 -0.00044907 0.00021644]: Output: -1.119205648615626\n11: Input_to_Funct: [ 0.00023032 -0.00044907 0.00021644]: Output: -1.1167593073781568\n12: Input_to_Funct: [ 0.00023032 -0.00044907 0.00021644]: Output: -1.1130444929064445\n13: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1159438602990006\n14: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1169405178401917\n15: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1147659922957747\n16: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1143129661406876\n17: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1210177532359737\n18: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.11630628122307\n19: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1145847818337398\n20: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1138599399856008\n21: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1135881242925485\n22: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1180277806124002\n23: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.111504203979149\n24: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1162156759920525\n25: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.119386859077661\n26: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1177559649193478\n27: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1166687021471393\n28: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1212895689290259\n29: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.114403571371705\n30: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1145847818337398\n31: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1159438602990006\n32: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1166687021471393\n33: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1108699673620273\n34: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1158532550679832\n35: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1178465701503653\n36: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1144941766027223\n37: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1183902015364695\n38: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1152190184508615\n39: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1150378079888266\n40: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1178465701503653\n41: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.114856597526792\n42: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1156720446059483\n43: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1197492800017304\n44: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1250949886317558\n45: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1154908341439138\n46: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1201117009258\n47: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1155814393749308\n48: Input_to_Funct: [ 0.00023327 -0.00046107 0.00021931]: Output: -1.1125914667513574\nWarning: Maximum number of iterations has been exceeded.\nReason for termination is Maximum number of iterations has been exceeded.\n"
]
],
[
[
"## REDUCED H2 ansatz:",
"_____no_output_____"
]
],
[
[
"from quchem.Simulating_Quantum_Circuit import *\nfrom quchem.Ansatz_Generator_Functions import *\nfrom openfermion.ops import QubitOperator\n\ndef H2_ansatz(theta):\n HF_circ = [cirq.X.on(cirq.LineQubit(0)), cirq.X.on(cirq.LineQubit(1))]\n \n full_exp_circ_obj = full_exponentiated_PauliWord_circuit(QubitOperator('Y0 X1 X2 X3', -1j), theta)\n UCCSD_circ = cirq.Circuit(cirq.decompose_once((full_exp_circ_obj(*cirq.LineQubit.range(full_exp_circ_obj.num_qubits())))))\n full_circuit = cirq.Circuit([*HF_circ, *UCCSD_circ.all_operations()])\n \n return full_circuit\n \nH2_ansatz(np.pi) ",
"_____no_output_____"
],
[
"n_shots=1000\n\ndef GIVE_ENERGY(THETA):\n \n ansatz_cirq_circuit = H2_ansatz(THETA)\n\n VQE_exp = VQE_Experiment(QubitHam, ansatz_cirq_circuit, n_shots)\n\n return VQE_exp.Calc_Energy()\n",
"_____no_output_____"
],
[
"### full angle scan\n\nimport matplotlib.pyplot as plt\n%matplotlib inline\n\ntheta_list = np.arange(0,2*np.pi, 0.1)\n\nE_list = [GIVE_ENERGY(theta) for theta in theta_list]\n\nplt.plot(E_list)\nprint(min(E_list))",
"-1.1373435841146557\n"
],
[
"## optimzer\n\nfrom quchem.Scipy_Optimizer import *\nTHETA_params=[2]\nGG = Optimizer(GIVE_ENERGY, THETA_params, 'Nelder-Mead', store_values=True, display_iter_steps=True,\n tol=1e-5,\n display_convergence_message=True)\nGG.get_env(50)\nGG.plot_convergence()\nplt.show()",
"0: Input_to_Funct: [2.3]: Output: -0.058458098910411926\n1: Input_to_Funct: [2.7]: Output: -0.694898287710437\n2: Input_to_Funct: [3.1]: Output: -1.1000942099698485\n3: Input_to_Funct: [3.3]: Output: -1.1330803222454058\n4: Input_to_Funct: [3.2]: Output: -1.1337591613565454\n5: Input_to_Funct: [3.2]: Output: -1.1301545473871317\n6: Input_to_Funct: [3.225]: Output: -1.1345419890439155\n7: Input_to_Funct: [3.225]: Output: -1.1404383941335479\n8: Input_to_Funct: [3.225]: Output: -1.128671036255271\n9: Input_to_Funct: [3.225]: Output: -1.1382749867085218\n10: Input_to_Funct: [3.225]: Output: -1.1339395718439667\n11: Input_to_Funct: [3.225]: Output: -1.1361616630655416\n12: Input_to_Funct: [3.225]: Output: -1.1390548673765042\n13: Input_to_Funct: [3.225]: Output: -1.13499106417041\n14: Input_to_Funct: [3.225]: Output: -1.1356301538080331\n15: Input_to_Funct: [3.225]: Output: -1.138522252092495\n16: Input_to_Funct: [3.225]: Output: -1.1313059590801648\n17: Input_to_Funct: [3.225]: Output: -1.1386437747487763\n18: Input_to_Funct: [3.225]: Output: -1.1382235720200673\n19: Input_to_Funct: [3.225]: Output: -1.128951533134805\n20: Input_to_Funct: [3.225]: Output: -1.1429763446112393\n21: Input_to_Funct: [3.225]: Output: -1.1402707178784968\n22: Input_to_Funct: [3.225]: Output: -1.1330395805980908\n23: Input_to_Funct: [3.225]: Output: -1.1400624390024947\n24: Input_to_Funct: [3.225]: Output: -1.1383862932373212\n25: Input_to_Funct: [3.225]: Output: -1.1320356539488055\n26: Input_to_Funct: [3.225]: Output: -1.1363870191079366\n27: Input_to_Funct: [3.225]: Output: -1.1365044887183298\n28: Input_to_Funct: [3.225]: Output: -1.1370038725067781\n29: Input_to_Funct: [3.225]: Output: -1.1373786195940347\n30: Input_to_Funct: [3.225]: Output: -1.142082618464253\n31: Input_to_Funct: [3.225]: Output: -1.1383349805661624\n32: Input_to_Funct: [3.225]: Output: -1.1339406778704673\n33: Input_to_Funct: [3.225]: Output: -1.1337058406669769\n34: Input_to_Funct: [3.225]: Output: -1.1348785495978142\n35: Input_to_Funct: [3.225]: Output: -1.1325838114582079\n36: Input_to_Funct: [3.225]: Output: -1.1355385445678108\n37: Input_to_Funct: [3.225]: Output: -1.136172679167637\n38: Input_to_Funct: [3.225]: Output: -1.1396156779462974\n39: Input_to_Funct: [3.225]: Output: -1.1357319791757372\n40: Input_to_Funct: [3.225]: Output: -1.1382493303729424\n41: Input_to_Funct: [3.225]: Output: -1.1367678272594914\n42: Input_to_Funct: [3.225]: Output: -1.130790298099937\n43: Input_to_Funct: [3.225]: Output: -1.1319463587788803\n44: Input_to_Funct: [3.225]: Output: -1.1387219517993106\n45: Input_to_Funct: [3.225]: Output: -1.1412342459414229\n46: Input_to_Funct: [3.225]: Output: -1.1366722669906766\n47: Input_to_Funct: [3.225]: Output: -1.1293238641344692\n48: Input_to_Funct: [3.225]: Output: -1.1391956792521796\nWarning: Maximum number of iterations has been exceeded.\nReason for termination is Maximum number of iterations has been exceeded.\n"
],
[
"ansatz_cirq_circuit = H2_ansatz(3.22500077)\nVQE_exp = VQE_Experiment(QubitHam, ansatz_cirq_circuit, 1000)\nprint('Energy = ', VQE_exp.Calc_Energy())\nprint('')\nprint('state:')\nVQE_exp.Get_wavefunction_of_state(sig_figs=4)",
"Energy = -1.1349360253505665\n\nstate:\n"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7ec58ba4c0c799c4318d95ca9e5b998e30bb9a1 | 364,054 | ipynb | Jupyter Notebook | training/notebooks/New_Weight_Optimization.ipynb | MIPT-Oulu/3DHistoGrading | b779a154d0e5b104fc152c8952124768fb7b1dc6 | [
"MIT"
] | 1 | 2021-11-04T18:46:25.000Z | 2021-11-04T18:46:25.000Z | training/notebooks/New_Weight_Optimization.ipynb | MIPT-Oulu/3DHistoGrading | b779a154d0e5b104fc152c8952124768fb7b1dc6 | [
"MIT"
] | 7 | 2018-08-14T07:35:53.000Z | 2018-09-07T12:17:10.000Z | training/notebooks/New_Weight_Optimization.ipynb | MIPT-Oulu/3D-Histo-Grading | b779a154d0e5b104fc152c8952124768fb7b1dc6 | [
"MIT"
] | null | null | null | 44.850807 | 104 | 0.496209 | [
[
[
"import sys\n\nsys.path.append('/home/tuomas/Desktop/GITS/mCTSegmentation/mctseg/unet/')\n\nimport numpy as np\nimport os\nimport h5py\nimport time\nimport gc\nimport copy\n\nfrom collections import OrderedDict\n\nimport torch\nimport torch.nn as nn\nimport torch.functional as F\n\nfrom model import UNet",
"_____no_output_____"
],
[
"def fuse_bn_sequential(block):\n \"\"\"\n This function takes a sequential block and fuses the batch normalization with convolution\n :param model: nn.Sequential. Source resnet model\n :return: nn.Sequential. Converted block\n \"\"\"\n print(block)\n if not isinstance(block, nn.Sequential):\n return block\n stack = []\n for m in block.children():\n if isinstance(m, nn.BatchNorm2d):\n if isinstance(stack[-1], nn.Conv2d):\n bn_st_dict = m.state_dict()\n conv_st_dict = stack[-1].state_dict()\n\n # BatchNorm params\n eps = m.eps\n mu = bn_st_dict['running_mean']\n var = bn_st_dict['running_var']\n gamma = bn_st_dict['weight']\n\n if 'bias' in bn_st_dict:\n beta = bn_st_dict['bias']\n else:\n beta = torch.zeros(gamma.size(0)).float().to(gamma.device)\n\n # Conv params\n W = conv_st_dict['weight']\n if 'bias' in conv_st_dict:\n bias = conv_st_dict['bias']\n else:\n bias = torch.zeros(W.size(0)).float().to(gamma.device)\n\n denom = torch.sqrt(var + eps)\n b = beta - gamma.mul(mu).div(denom)\n A = gamma.div(denom)\n bias *= A\n A = A.expand_as(W.transpose(0, -1)).transpose(0, -1)\n\n W.mul_(A)\n bias.add_(b)\n\n stack[-1].weight.data.copy_(W)\n if stack[-1].bias is None:\n stack[-1].bias = torch.nn.Parameter(bias)\n else:\n stack[-1].bias.data.copy_(bias)\n\n else:\n stack.append(m)\n \n\n if len(stack) > 1:\n return nn.Sequential(*stack)\n else:\n return stack[0]\n\n\ndef fuse_bn_recursively(model):\n for module_name in model._modules:\n model._modules[module_name] = fuse_bn_sequential(model._modules[module_name])\n if len(model._modules[module_name]._modules) > 0:\n fuse_bn_recursively(model._modules[module_name])\n if len(model._modules[module_name]._modules) > 0:\n fuse_bn_recursively(model._modules[module_name])\n\n return model",
"_____no_output_____"
],
[
"path = '/home/tuomas/Desktop/GITS/snapshots/2018_12_03_15_25'\nfiles = os.listdir(path)\nfiles.sort()\n\nnames = []\nnets = []\n\nfor file in files:\n print(file) \n f = file.split('_')\n if f[0] == 'fold':\n name = f[0]+'_'+f[1]+'_'+f[2]+'_'+f[3][:-4]\n names.append(name)\n net1 = UNet(24,6,2);\n net1.load_state_dict(torch.load(os.path.join(path,file)));\n net2 = fuse_bn_recursively(net1);\n #net2._modules['center'] = fuse_bn_sequential(net2._modules['center'])\n \n nets.append(copy.deepcopy(net2));\n \n net2 = None\n net1 = None\n gc.collect()\n \n",
"fold_0_epoch_26.pth\nEncoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(1, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nEncoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nEncoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nEncoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nEncoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nEncoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(768, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 768, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nDecoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(384, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(1536, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 384, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nDecoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(192, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(768, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 192, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nDecoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(96, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(384, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 96, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nDecoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(48, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(192, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 48, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nDecoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(96, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nDecoder(\n (layers): Sequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n )\n)\nSequential(\n (conv_3x3_0): Sequential(\n (0): Sequential(\n (0): Conv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n (conv_3x3_1): Sequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n )\n)\nSequential(\n (0): Sequential(\n (0): Conv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n )\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nBatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): BatchNorm2d(24, eps=1e-05, momentum=0.1, affine=True, track_running_stats=True)\n (2): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Sequential(\n (0): Conv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n (1): Sequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n )\n)\nSequential(\n (0): Conv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(48, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nSequential(\n (0): Conv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\n (1): ReLU(inplace)\n)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 24, kernel_size=(3, 3), stride=(1, 1), padding=(1, 1))\nReLU(inplace)\nConv2d(24, 1, kernel_size=(1, 1), stride=(1, 1))\n"
],
[
"for key,w in nets[0].state_dict().items():\n print(key)\n print(w.shape)",
"down1.layers.0.0.weight\ntorch.Size([24, 1, 3, 3])\ndown1.layers.0.0.bias\ntorch.Size([24])\ndown1.layers.1.0.weight\ntorch.Size([24, 24, 3, 3])\ndown1.layers.1.0.bias\ntorch.Size([24])\ndown2.layers.0.0.weight\ntorch.Size([48, 24, 3, 3])\ndown2.layers.0.0.bias\ntorch.Size([48])\ndown2.layers.1.0.weight\ntorch.Size([48, 48, 3, 3])\ndown2.layers.1.0.bias\ntorch.Size([48])\ndown3.layers.0.0.weight\ntorch.Size([96, 48, 3, 3])\ndown3.layers.0.0.bias\ntorch.Size([96])\ndown3.layers.1.0.weight\ntorch.Size([96, 96, 3, 3])\ndown3.layers.1.0.bias\ntorch.Size([96])\ndown4.layers.0.0.weight\ntorch.Size([192, 96, 3, 3])\ndown4.layers.0.0.bias\ntorch.Size([192])\ndown4.layers.1.0.weight\ntorch.Size([192, 192, 3, 3])\ndown4.layers.1.0.bias\ntorch.Size([192])\ndown5.layers.0.0.weight\ntorch.Size([384, 192, 3, 3])\ndown5.layers.0.0.bias\ntorch.Size([384])\ndown5.layers.1.0.weight\ntorch.Size([384, 384, 3, 3])\ndown5.layers.1.0.bias\ntorch.Size([384])\ndown6.layers.0.0.weight\ntorch.Size([768, 384, 3, 3])\ndown6.layers.0.0.bias\ntorch.Size([768])\ndown6.layers.1.0.weight\ntorch.Size([768, 768, 3, 3])\ndown6.layers.1.0.bias\ntorch.Size([768])\ncenter.0.0.weight\ntorch.Size([768, 768, 3, 3])\ncenter.0.0.bias\ntorch.Size([768])\ncenter.1.0.weight\ntorch.Size([768, 768, 3, 3])\ncenter.1.0.bias\ntorch.Size([768])\nup6.layers.0.0.weight\ntorch.Size([384, 1536, 3, 3])\nup6.layers.0.0.bias\ntorch.Size([384])\nup6.layers.1.0.weight\ntorch.Size([384, 384, 3, 3])\nup6.layers.1.0.bias\ntorch.Size([384])\nup5.layers.0.0.weight\ntorch.Size([192, 768, 3, 3])\nup5.layers.0.0.bias\ntorch.Size([192])\nup5.layers.1.0.weight\ntorch.Size([192, 192, 3, 3])\nup5.layers.1.0.bias\ntorch.Size([192])\nup4.layers.0.0.weight\ntorch.Size([96, 384, 3, 3])\nup4.layers.0.0.bias\ntorch.Size([96])\nup4.layers.1.0.weight\ntorch.Size([96, 96, 3, 3])\nup4.layers.1.0.bias\ntorch.Size([96])\nup3.layers.0.0.weight\ntorch.Size([48, 192, 3, 3])\nup3.layers.0.0.bias\ntorch.Size([48])\nup3.layers.1.0.weight\ntorch.Size([48, 48, 3, 3])\nup3.layers.1.0.bias\ntorch.Size([48])\nup2.layers.0.0.weight\ntorch.Size([24, 96, 3, 3])\nup2.layers.0.0.bias\ntorch.Size([24])\nup2.layers.1.0.weight\ntorch.Size([24, 24, 3, 3])\nup2.layers.1.0.bias\ntorch.Size([24])\nup1.layers.0.0.weight\ntorch.Size([24, 48, 3, 3])\nup1.layers.0.0.bias\ntorch.Size([24])\nup1.layers.1.0.weight\ntorch.Size([24, 24, 3, 3])\nup1.layers.1.0.bias\ntorch.Size([24])\nmixer.weight\ntorch.Size([1, 24, 1, 1])\nmixer.bias\ntorch.Size([1])\n"
],
[
"savename = 'UNetE3New.h5'\n#h5 = h5py.File(savename,'w')\n\nfor name,net in zip(names,nets):\n W = OrderedDict()\n for key,w in net.state_dict().items():\n _name = key[:]\n W[_name] = w\n savename = '/media/tuomas/data/WRKNew/UNet_'+name[:6]+'_new.h5';\n print(savename)\n h5 = h5py.File(savename,'w')\n for key in W:\n if key.endswith('0.bias') or key.endswith('0.weight'):\n N = key.split('.')\n #print(key)\n if N[1] == 'layers':\n npart = N[2]\n npart = npart.split('_')\n dname = N[0]+'_'+npart[-1]+'_'+N[-1]\n #print(dname)\n else:\n npart = N[1]\n npart = npart.split('_')\n dname = N[0]+'_'+npart[-1]+'_'+N[-1]\n #print(dname)\n print(dname)\n T = W[key]\n #T = T.view(T.numel())\n T = T.cpu().numpy().astype('float64')\n #D = T.shape\n #if len(D) == 4:\n # T = np.swapaxes(T,3,2)\n T = T.flatten()\n #print(T) \n h5.create_dataset(dname,data=T)\n if key.startswith('mixer'):\n dname = key \n T = W[key]\n T = T.cpu().numpy().astype('float64')\n T = T.flatten()\n print(dname)\n print(T.shape)\n h5.create_dataset(dname,data=T)\n \n h5.close()\n ",
"/media/tuomas/data/WRKNew/UNet_fold_0_new.h5\ndown1_0_weight\ndown1_0_bias\ndown1_1_weight\ndown1_1_bias\ndown2_0_weight\ndown2_0_bias\ndown2_1_weight\ndown2_1_bias\ndown3_0_weight\ndown3_0_bias\ndown3_1_weight\ndown3_1_bias\ndown4_0_weight\ndown4_0_bias\ndown4_1_weight\ndown4_1_bias\ndown5_0_weight\ndown5_0_bias\ndown5_1_weight\ndown5_1_bias\ndown6_0_weight\ndown6_0_bias\ndown6_1_weight\ndown6_1_bias\ncenter_0_weight\ncenter_0_bias\ncenter_1_weight\ncenter_1_bias\nup6_0_weight\nup6_0_bias\nup6_1_weight\nup6_1_bias\nup5_0_weight\nup5_0_bias\nup5_1_weight\nup5_1_bias\nup4_0_weight\nup4_0_bias\nup4_1_weight\nup4_1_bias\nup3_0_weight\nup3_0_bias\nup3_1_weight\nup3_1_bias\nup2_0_weight\nup2_0_bias\nup2_1_weight\nup2_1_bias\nup1_0_weight\nup1_0_bias\nup1_1_weight\nup1_1_bias\nmixer.weight\n(24,)\nmixer.bias\n(1,)\n/media/tuomas/data/WRKNew/UNet_fold_1_new.h5\ndown1_0_weight\ndown1_0_bias\ndown1_1_weight\ndown1_1_bias\ndown2_0_weight\ndown2_0_bias\ndown2_1_weight\ndown2_1_bias\ndown3_0_weight\ndown3_0_bias\ndown3_1_weight\ndown3_1_bias\ndown4_0_weight\ndown4_0_bias\ndown4_1_weight\ndown4_1_bias\ndown5_0_weight\ndown5_0_bias\ndown5_1_weight\ndown5_1_bias\ndown6_0_weight\ndown6_0_bias\ndown6_1_weight\ndown6_1_bias\ncenter_0_weight\ncenter_0_bias\ncenter_1_weight\ncenter_1_bias\nup6_0_weight\nup6_0_bias\nup6_1_weight\nup6_1_bias\nup5_0_weight\nup5_0_bias\nup5_1_weight\nup5_1_bias\nup4_0_weight\nup4_0_bias\nup4_1_weight\nup4_1_bias\nup3_0_weight\nup3_0_bias\nup3_1_weight\nup3_1_bias\nup2_0_weight\nup2_0_bias\nup2_1_weight\nup2_1_bias\nup1_0_weight\nup1_0_bias\nup1_1_weight\nup1_1_bias\nmixer.weight\n(24,)\nmixer.bias\n(1,)\n/media/tuomas/data/WRKNew/UNet_fold_2_new.h5\ndown1_0_weight\ndown1_0_bias\ndown1_1_weight\ndown1_1_bias\ndown2_0_weight\ndown2_0_bias\ndown2_1_weight\ndown2_1_bias\ndown3_0_weight\ndown3_0_bias\ndown3_1_weight\ndown3_1_bias\ndown4_0_weight\ndown4_0_bias\ndown4_1_weight\ndown4_1_bias\ndown5_0_weight\ndown5_0_bias\ndown5_1_weight\ndown5_1_bias\ndown6_0_weight\ndown6_0_bias\ndown6_1_weight\ndown6_1_bias\ncenter_0_weight\ncenter_0_bias\ncenter_1_weight\ncenter_1_bias\nup6_0_weight\nup6_0_bias\nup6_1_weight\nup6_1_bias\nup5_0_weight\nup5_0_bias\nup5_1_weight\nup5_1_bias\nup4_0_weight\nup4_0_bias\nup4_1_weight\nup4_1_bias\nup3_0_weight\nup3_0_bias\nup3_1_weight\nup3_1_bias\nup2_0_weight\nup2_0_bias\nup2_1_weight\nup2_1_bias\nup1_0_weight\nup1_0_bias\nup1_1_weight\nup1_1_bias\nmixer.weight\n(24,)\nmixer.bias\n(1,)\n/media/tuomas/data/WRKNew/UNet_fold_3_new.h5\ndown1_0_weight\ndown1_0_bias\ndown1_1_weight\ndown1_1_bias\ndown2_0_weight\ndown2_0_bias\ndown2_1_weight\ndown2_1_bias\ndown3_0_weight\ndown3_0_bias\ndown3_1_weight\ndown3_1_bias\ndown4_0_weight\ndown4_0_bias\ndown4_1_weight\ndown4_1_bias\ndown5_0_weight\ndown5_0_bias\ndown5_1_weight\ndown5_1_bias\ndown6_0_weight\ndown6_0_bias\ndown6_1_weight\ndown6_1_bias\ncenter_0_weight\ncenter_0_bias\ncenter_1_weight\ncenter_1_bias\nup6_0_weight\nup6_0_bias\nup6_1_weight\nup6_1_bias\nup5_0_weight\nup5_0_bias\nup5_1_weight\nup5_1_bias\nup4_0_weight\nup4_0_bias\nup4_1_weight\nup4_1_bias\nup3_0_weight\nup3_0_bias\nup3_1_weight\nup3_1_bias\nup2_0_weight\nup2_0_bias\nup2_1_weight\nup2_1_bias\nup1_0_weight\nup1_0_bias\nup1_1_weight\nup1_1_bias\nmixer.weight\n(24,)\nmixer.bias\n(1,)\n/media/tuomas/data/WRKNew/UNet_fold_4_new.h5\ndown1_0_weight\ndown1_0_bias\ndown1_1_weight\ndown1_1_bias\ndown2_0_weight\ndown2_0_bias\ndown2_1_weight\ndown2_1_bias\ndown3_0_weight\ndown3_0_bias\ndown3_1_weight\ndown3_1_bias\ndown4_0_weight\ndown4_0_bias\ndown4_1_weight\ndown4_1_bias\ndown5_0_weight\ndown5_0_bias\ndown5_1_weight\ndown5_1_bias\ndown6_0_weight\ndown6_0_bias\ndown6_1_weight\ndown6_1_bias\ncenter_0_weight\ncenter_0_bias\ncenter_1_weight\ncenter_1_bias\nup6_0_weight\nup6_0_bias\nup6_1_weight\nup6_1_bias\nup5_0_weight\nup5_0_bias\nup5_1_weight\nup5_1_bias\nup4_0_weight\nup4_0_bias\nup4_1_weight\nup4_1_bias\nup3_0_weight\nup3_0_bias\nup3_1_weight\nup3_1_bias\nup2_0_weight\nup2_0_bias\nup2_1_weight\nup2_1_bias\nup1_0_weight\nup1_0_bias\nup1_1_weight\nup1_1_bias\nmixer.weight\n(24,)\nmixer.bias\n(1,)\n"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7ec62975efc7c9d6adddeab4660d89ea9a883df | 350,835 | ipynb | Jupyter Notebook | faers_multilabel_outcome_ml_pipeline_dask_joblib_dd_3_5_2021.ipynb | briangriner/OSTF-FAERS | 4af97c85d43950704bfb7f1695873e1809f6f43c | [
"MIT"
] | null | null | null | faers_multilabel_outcome_ml_pipeline_dask_joblib_dd_3_5_2021.ipynb | briangriner/OSTF-FAERS | 4af97c85d43950704bfb7f1695873e1809f6f43c | [
"MIT"
] | null | null | null | faers_multilabel_outcome_ml_pipeline_dask_joblib_dd_3_5_2021.ipynb | briangriner/OSTF-FAERS | 4af97c85d43950704bfb7f1695873e1809f6f43c | [
"MIT"
] | 1 | 2021-02-18T03:34:54.000Z | 2021-02-18T03:34:54.000Z | 216.431215 | 54,144 | 0.885604 | [
[
[
"# FAERS AE Multilabel Outcomes ML pipeline - Dask Distributed + Joblib + Dask DataFrames",
"_____no_output_____"
],
[
"## Methodology\n\n### Objective\n**Use FAERS data on drug safety to identify possible risk factors associated with patient mortality and other serious adverse events associated with approved used of a drug or drug class** \n\n### Data\n**_Outcome table_** \n1. Start with outcome_c table to define unit of analysis (primaryid)\n2. Reshape outcome_c to one row per primaryid\n3. Outcomes grouped into 3 categories: a. death, b. serious, c. other \n4. Multiclass model target format: each outcome grp coded into separate columns\n\n**_Demo table_**\n1. Drop fields not used in model input to reduce table size (preferably before import to notebook)\n2. Check if demo table one row per primaryid (if NOT then need to reshape / clean - TBD)\n\n**_Model input and targets_**\n1. Merge clean demo table with reshaped multilabel outcome targets (rows: primaryid, cols: outcome grps)\n2. Inspect merged file to check for anomalies (outliers, bad data, ...)\n\n### Model\n**_Multilabel Classifier_**\n1. Since each primaryid has multiple outcomes coded in the outcome_c table, the ML model should predict the probability of each possible outcome.\n2. In scikit-learn lib most/all classifiers can predict multilabel outcomes by coding target outputs into array\n\n### Results\nTBD\n\n### Insights\nTBD",
"_____no_output_____"
]
],
[
[
"# scale sklearn dask example setup - compare to multi thread below\nfrom dask.distributed import Client, progress\nclient = Client(n_workers=4, threads_per_worker=1, memory_limit='2GB')\nclient",
"_____no_output_____"
],
[
"#import libraries\nimport numpy as np\nprint('The numpy version is {}.'.format(np.__version__))\nimport pandas as pd\nprint('The pandas version is {}.'.format(pd.__version__))\nfrom pandas import read_csv, DataFrame\n# dask dataframe\nimport dask\nimport dask.dataframe as dd\nfrom dask.diagnostics import ProgressBar\nprint('The dask version is {}.'.format(dask.__version__))\n#from random import random\nimport sklearn\n#from sklearn.base import (BaseEstimator, TransformerMixin)\nprint('The scikit-learn version is {}.'.format(sklearn.__version__))\nimport joblib\nfrom joblib import dump, load\nprint('The joblib version is {}.'.format(joblib.__version__))\n\n# preprocess + model selection + pipeline\nfrom sklearn.preprocessing import OneHotEncoder, StandardScaler #, LabelBinarizer, MultiLabelBinarizer\nfrom sklearn.impute import SimpleImputer, KNNImputer, MissingIndicator\nfrom sklearn.model_selection import train_test_split, GridSearchCV #, cross_val_score, \nfrom sklearn.compose import ColumnTransformer\nfrom sklearn.pipeline import Pipeline\nfrom sklearn.multioutput import MultiOutputClassifier\n\n# models supporting multilabel classification\nfrom sklearn.tree import DecisionTreeClassifier, ExtraTreeClassifier\nfrom sklearn.ensemble import ExtraTreesClassifier, RandomForestClassifier\nfrom sklearn.neighbors import KNeighborsClassifier, RadiusNeighborsClassifier\nfrom sklearn.neural_network import MLPClassifier\nfrom sklearn.linear_model import RidgeClassifierCV\n\n# libs for managing imbalanced classes\nfrom sklearn.utils import resample\n\n# metrics appropriate for multilabel classification\nfrom sklearn.metrics import jaccard_score, hamming_loss, accuracy_score, roc_auc_score, average_precision_score\nfrom sklearn.metrics import multilabel_confusion_matrix, ConfusionMatrixDisplay \n\n# feature importance\nfrom sklearn.inspection import permutation_importance, partial_dependence, plot_partial_dependence\n\n# visualization\nimport matplotlib as mpl\nprint('The matplotlib version is {}.'.format(mpl.__version__))\nfrom matplotlib import pyplot as plt\nfrom matplotlib.ticker import PercentFormatter\nfrom mpl_toolkits.mplot3d import Axes3D\nimport seaborn as sns\nprint('The seaborn version is {}.'.format(sns.__version__))\nsns.set()\n\n# dtree viz libs\nimport pydotplus\nfrom io import StringIO\nfrom sklearn import tree\nfrom sklearn.tree import export_graphviz\nfrom IPython.display import Image\n\n%matplotlib inline\n\nimport warnings\nwarnings.filterwarnings('ignore')\n\n# utilities for timing decorator\nimport time\nfrom functools import wraps\n\ndef timefn(fn):\n @wraps(fn)\n def measure_time(*args, **kwargs):\n t1 = time.time()\n result = fn(*args, **kwargs)\n t2 = time.time()\n print(f'@timefn: {fn.__name__} took {t2 - t1} seconds')\n return result\n return measure_time\n\n",
"The numpy version is 1.19.5.\nThe pandas version is 1.2.0.\nThe dask version is 2020.12.0.\nThe scikit-learn version is 0.24.0.\nThe joblib version is 0.13.0.\nThe matplotlib version is 3.0.2.\nThe seaborn version is 0.9.0.\n"
],
[
"%%time\n# read data into dask dataframe\n\n# provide path to datafile\nfile_in = '../input/demo-outc_cod-multilabel-wt_lbs-age_yrs.csv'\n# provide list of fields to import (alt-read in all cols and use df.drop(['cols2drop'],axis = 1)\ncols_in = ['primaryid', 'i_f_code', 'rept_cod', 'sex', 'occp_cod', 'outc_cod__CA', \n 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO', 'outc_cod__LT', 'outc_cod__OT', \n 'outc_cod__RI', 'n_outc', 'wt_lbs', 'age_yrs']\n#@timefn\ndef data_in(infile, incols):\n \"\"\"Used to time reading data from source\"\"\" \n ddf = dd.read_csv(infile, usecols=incols)\n print(ddf.columns, '\\n')\n print(ddf.head(),'\\n')\n print(f'Total number of rows: {len(ddf):,}\\n')\n ddf2 = ddf.primaryid.nunique().compute()\n print(f'Unique number of primaryids: {ddf2}')\n return ddf\n \n \nif __name__=='__main__':\n ddf = data_in(file_in, cols_in)",
"Index(['primaryid', 'i_f_code', 'rept_cod', 'sex', 'occp_cod', 'outc_cod__CA',\n 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO', 'outc_cod__LT',\n 'outc_cod__OT', 'outc_cod__RI', 'n_outc', 'wt_lbs', 'age_yrs'],\n dtype='object') \n\n primaryid i_f_code rept_cod sex occp_cod outc_cod__CA outc_cod__DE \\\n0 100046942 F EXP F LW 0 0 \n1 100048206 F EXP F MD 0 0 \n2 100048622 F EXP F LW 0 0 \n3 100051352 F EXP F LW 0 0 \n4 100051382 F EXP F LW 0 0 \n\n outc_cod__DS outc_cod__HO outc_cod__LT outc_cod__OT outc_cod__RI \\\n0 0 0 0 1 0 \n1 0 1 0 1 0 \n2 0 0 0 1 0 \n3 0 0 0 1 0 \n4 0 0 0 1 0 \n\n n_outc wt_lbs age_yrs \n0 1 178.574463 NaN \n1 2 NaN 68.0 \n2 1 NaN 57.0 \n3 1 NaN 51.0 \n4 1 182.983709 50.0 \n\nTotal number of rows: 260,715\n\nUnique number of primaryids: 260715\nCPU times: user 2.3 s, sys: 242 ms, total: 2.54 s\nWall time: 10.4 s\n"
],
[
"with ProgressBar():\n display(ddf.head())",
"_____no_output_____"
]
],
[
[
"## ML Pipeline - Preprocessing step",
"_____no_output_____"
]
],
[
[
"# dask: data prep + preprocessor + pipeline funcs\ndef df_prep(ddf):\n \"\"\"df_prep func used in pipeline to support select features and prep multilabel targets for clf\n assumes dask DataFrame is named 'ddf'\n \"\"\" \n # compute ddf\n #ddf2 = ddf.compute()\n # drop fields from df when defining model targets and features\n y_drop = ['primaryid', 'i_f_code', 'rept_cod', 'sex', 'occp_cod', 'n_outc', 'wt_lbs', 'age_yrs'] \n X_drop = ['primaryid', 'outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO', \n 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI']\n # convert target to ndarray for sklearn\n y = ddf.drop(y_drop, axis=1).compute()\n y_arr = y.to_numpy() # look into dask array\n X = ddf.drop(X_drop, axis=1).compute()\n print('Step 0: Create ndarray for multilabel targets + select model features','\\n')\n print('y_arr\\n', y_arr.shape, '\\n', y_arr.dtype, '\\n', y_arr[:2], y.columns, '\\n')\n print('X\\n', X.shape, '\\n', X.dtypes, '\\n', X[:2],'\\n', X.columns, '\\n')\n return X, y_arr\n\ndef preprocessor():\n \"make data preprocessor for pipeline\"\n\n # 2. group features by type for categorical vs numeric transformers\n num_features = ['n_outc', 'wt_lbs', 'age_yrs'] \n cat_features = ['i_f_code', 'rept_cod', 'sex', 'occp_cod'] \n feature_labels = num_features + cat_features\n print('Step 2: Group features by type for pipeline\\n')\n print('num_features\\n', num_features)\n print('cat_features\\n', cat_features)\n print('feature_labels\\n', feature_labels,'\\n')\n \n # 3. create transformers for model input features by type\n num_transformer = Pipeline(steps=[\n ('imputer', SimpleImputer(strategy='median')),\n ('scaler', StandardScaler())])\n cat_transformer = Pipeline(steps=[\n ('1_hot', OneHotEncoder(handle_unknown='ignore'))])\n print('Step 3: Column transformers by type for pipeline\\n')\n print('num_transformer\\n', num_transformer)\n print('cat_transformer\\n', cat_transformer,'\\n')\n\n # 4. combine transformers into preprocessing step\n preprocessor = ColumnTransformer(transformers=[\n #('dfprep', dfprep_transformer, all_features),\n ('num', num_transformer, num_features),\n ('cat', cat_transformer, cat_features)], remainder='passthrough') \n print('Step 4: Preprocessor for pipeline\\n')\n print('preprocessor\\n', preprocessor,'\\n')\n return preprocessor\n\ndef model_fit(X_, y_):\n # fit clfs using dask with joblib backend\n for classifier in classifiers:\n ml_pipe = Pipeline(steps=[('preprocessor', preprocessor()),\n ('classifier', MultiOutputClassifier(classifier))\n ]\n )\n # use context manager to run dask during training\n with joblib.parallel_backend('dask'):\n ml_clf = ml_pipe.fit(X_, y_)\n \n \n # save/load fited clf obj with joblib\n dump(ml_clf, 'ml_clf_obj.joblib') \n ml_clf_obj = load('ml_clf_obj.joblib')\n return ml_clf_obj\n \n \n# 6. ml clf model pipe\ndef ml_clf_pipe(clf_lst):\n \"\"\"Pipeline to evaluate multilabel classifiers prior to hyperparameter tuning.\"\"\"\n # 0. data prep\n X, y_arr = df_prep(ddf)\n # 1. train, test set split (can extend later to multiple train-test splits in pipeline)\n X_train, X_test, y_train, y_test = train_test_split(X, y_arr, test_size = 0.3)\n print('Step 1: Train-test set split\\n')\n print('X_train\\n', X_train.shape, '\\n', X_train[:2])\n print('y_train\\n', y_train.shape, '\\n', y_train[:2])\n print('X_test\\n', X_test.shape, '\\n', X_test[:2])\n print('y_test\\n', y_test.shape, '\\n', y_test[:2],'\\n')\n \n # train, dump & load model \n ml_clf_obj = model_fit(X_train, y_train)\n\n y_pred = ml_clf_obj.predict(X_test)\n print('y_pred\\n', y_pred.shape,'\\n',y_pred[3:],'\\n')\n\n print('Multilabel Classifier: Performance Metrics:\\n')\n # accuracy, hamming loss and jaccard score for mlabel\n print('accuracy: ', accuracy_score(y_test, y_pred))\n print('hamming loss: ', hamming_loss(y_test, y_pred))\n print('jaccard score: ', jaccard_score(y_test, y_pred, average='micro'))\n print('roc auc score: ', roc_auc_score(y_test, y_pred))\n print('average precision score: ', average_precision_score(y_test, y_pred),'\\n')\n\n # generate ml cm\n multilabel_cm = multilabel_confusion_matrix(y_test, y_pred)\n # calc tp, tn, fp, fn rates\n tp = multilabel_cm[:, 0, 0]\n tn = multilabel_cm[:, 1, 1]\n fp = multilabel_cm[:, 0, 1]\n fn = multilabel_cm[:, 1, 0]\n\n outc_labels = ['outc_cod__CA','outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO', 'outc_cod__LT', \n 'outc_cod__OT', 'outc_cod__RI']\n print('Recall, Specificity, Fall Out and Miss Rate for Multilabel Adverse Event Outcomes:\\n', outc_labels)\n # recall\n print('recall (true pos rate):\\n', tp / (tp + fn))\n # specificity\n print('specificity (true neg rate):\\n', tn / (tn + fp))\n # fall out\n print('fall out (false pos rate):\\n', fp / (fp + tn))\n # miss rate\n print('miss rate (false neg rate):\\n', fn / (fn + tp), '\\n')\n\n # plot multilabel confusion matrix\n def print_confusion_matrix(confusion_matrix, axes, class_label, class_names, fontsize=14):\n df_cm = pd.DataFrame(confusion_matrix, index=class_names, columns=class_names) \n try:\n heatmap = sns.heatmap(df_cm, annot=True, fmt=\"d\", cbar=False, ax=axes, cmap=\"YlGnBu\") #cmap='RdBu_r', \n except ValueError:\n raise ValueError(\"Confusion matrix values must be integers.\")\n heatmap.yaxis.set_ticklabels(heatmap.yaxis.get_ticklabels(), rotation=0, ha='right', \n fontsize=fontsize)\n heatmap.xaxis.set_ticklabels(heatmap.yaxis.get_ticklabels(), rotation=45, ha='right', \n fontsize=fontsize)\n axes.set_xlabel('True label')\n axes.set_ylabel('Predicted label')\n axes.set_title(\"Confusion matrix for class - \" + class_label)\n\n # plot grid of cm's - one per output - raw\n fig, ax = plt.subplots(3, 3, figsize=(12, 7))\n\n for axes, cfs_matrix, label in zip(ax.flatten(), multilabel_cm, outc_labels): # labels):\n print_confusion_matrix(cfs_matrix, axes, label, [\"N\", \"Y\"])\n\n fig.tight_layout()\n plt.show()\n\n # corr matrix heatmap \n y_df = pd.DataFrame(y_pred, columns=outc_labels)\n ncol = y_df.shape[1]\n fig, ax = plt.subplots(figsize=(ncol,ncol))\n ax = sns.heatmap(y_df.corr(), fmt='.2f', annot=True, ax=ax, cmap='RdBu_r', vmin=-1, vmax=1)\n imgname = str(classifiers[0]) + '-hmap_corr.png'\n fig.savefig(imgname, dpi=300, bbox_inches='tight')\n print('Correlation Matrix Heatmap of Predicted Multilabel Adverse Events\\n')\n plt.show()\n",
"_____no_output_____"
]
],
[
[
"## Decision Tree Classifier",
"_____no_output_____"
]
],
[
[
"# multilabel clfs\nclassifiers = [ \n #RidgeClassifierCV(class_weight='balanced'),\n DecisionTreeClassifier(class_weight='balanced'),\n #ExtraTreesClassifier(class_weight='balanced'),\n #RandomForestClassifier(class_weight='balanced'),\n #MLPClassifier(solver='sdg', learning_rate='adaptive', early_stopping=True),\n #KNeighborsClassifier(weights='distance'),\n #RadiusNeighborsClassifier(weights='distance')\n]\n \n# fit and eval model\nif __name__ == '__main__':\n ml_clf_pipe(classifiers)\n \n ",
"Step 0: Create ndarray for multilabel targets + select model features \n\ny_arr\n (260715, 7) \n int64 \n [[0 0 0 0 0 1 0]\n [0 0 0 1 0 1 0]] Index(['outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO',\n 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI'],\n dtype='object') \n\nX\n (260715, 7) \n i_f_code object\nrept_cod object\nsex object\noccp_cod object\nn_outc int64\nwt_lbs float64\nage_yrs float64\ndtype: object \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n0 F EXP F LW 1 178.574463 NaN\n1 F EXP F MD 2 NaN 68.0 \n Index(['i_f_code', 'rept_cod', 'sex', 'occp_cod', 'n_outc', 'wt_lbs',\n 'age_yrs'],\n dtype='object') \n\nStep 1: Train-test set split\n\nX_train\n (182500, 7) \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n122096 I EXP M CN 1 NaN 66.0\n130518 F EXP M MD 2 NaN 71.0\ny_train\n (182500, 7) \n [[0 0 0 0 0 1 0]\n [0 1 0 0 0 1 0]]\nX_test\n (78215, 7) \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n232434 I PER M NaN 1 NaN 24.0\n229686 F EXP F CN 1 NaN 23.0\ny_test\n (78215, 7) \n [[0 0 0 0 0 1 0]\n [0 0 0 0 0 1 0]] \n\nStep 2: Group features by type for pipeline\n\nnum_features\n ['n_outc', 'wt_lbs', 'age_yrs']\ncat_features\n ['i_f_code', 'rept_cod', 'sex', 'occp_cod']\nfeature_labels\n ['n_outc', 'wt_lbs', 'age_yrs', 'i_f_code', 'rept_cod', 'sex', 'occp_cod'] \n\nStep 3: Column transformers by type for pipeline\n\nnum_transformer\n Pipeline(steps=[('imputer', SimpleImputer(strategy='median')),\n ('scaler', StandardScaler())])\ncat_transformer\n Pipeline(steps=[('1_hot', OneHotEncoder(handle_unknown='ignore'))]) \n\nStep 4: Preprocessor for pipeline\n\npreprocessor\n ColumnTransformer(remainder='passthrough',\n transformers=[('num',\n Pipeline(steps=[('imputer',\n SimpleImputer(strategy='median')),\n ('scaler', StandardScaler())]),\n ['n_outc', 'wt_lbs', 'age_yrs']),\n ('cat',\n Pipeline(steps=[('1_hot',\n OneHotEncoder(handle_unknown='ignore'))]),\n ['i_f_code', 'rept_cod', 'sex', 'occp_cod'])]) \n\ny_pred\n (78215, 7) \n [[0 0 0 ... 0 1 0]\n [0 0 0 ... 0 0 0]\n [0 0 0 ... 0 1 0]\n ...\n [0 0 0 ... 0 0 0]\n [0 0 0 ... 0 1 0]\n [0 0 0 ... 0 0 0]] \n\nMultilabel Classifier: Performance Metrics:\n\naccuracy: 0.32413219970593876\nhamming loss: 0.17950338353074402\njaccard score: 0.39451683455010317\nroc auc score: 0.6927179362614825\naverage precision score: 0.2543793998817573 \n\nRecall, Specificity, Fall Out and Miss Rate for Multilabel Adverse Event Outcomes:\n ['outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO', 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI']\nrecall (true pos rate):\n [0.99828599 0.91875986 0.98189365 0.74417355 0.97659484 0.50211667\n 0.99902149]\nspecificity (true neg rate):\n [0.05172659 0.29873003 0.0913101 0.67611727 0.21260402 0.75668032\n 0.04395604]\nfall out (false pos rate):\n [0.94827341 0.70126997 0.9086899 0.32388273 0.78739598 0.24331968\n 0.95604396]\nmiss rate (false neg rate):\n [0.00171401 0.08124014 0.01810635 0.25582645 0.02340516 0.49788333\n 0.00097851] \n\n"
],
[
"# use model object to create predictions on new data\n\n# load fit clf\nml_clf_obj = load('ml_clf_obj.joblib')\ndir(ml_clf_obj)",
"_____no_output_____"
],
[
"# predict multilabel outcomes\n# prep new data\nX, y_arr = df_prep(ddf)\n# predict ml outcomes\ny_arr_pred = ml_clf_obj.predict(X)\nprint('Predicted AEs: DecisionTree Classifier\\n', y_arr_pred.shape, '\\n', y_arr_pred[5:])",
"Predicted AEs: DecisionTree Classifier\n (260715, 7) \n [[0 0 0 ... 0 1 0]\n [0 1 0 ... 1 1 0]\n [0 0 0 ... 0 1 0]\n ...\n [0 0 0 ... 0 1 0]\n [0 0 0 ... 0 1 0]\n [0 0 0 ... 0 1 0]]\n"
]
],
[
[
"## Random Forest Classifier",
"_____no_output_____"
]
],
[
[
"# multilabel clfs\nclassifiers = [ \n #RidgeClassifierCV(class_weight='balanced'),\n #DecisionTreeClassifier(class_weight='balanced'),\n #ExtraTreesClassifier(class_weight='balanced'),\n RandomForestClassifier(class_weight='balanced'),\n #MLPClassifier(solver='sdg', learning_rate='adaptive', early_stopping=True),\n #KNeighborsClassifier(weights='distance'),\n #RadiusNeighborsClassifier(weights='distance')\n]\n \n# fit and eval model\nif __name__ == '__main__':\n ml_clf_pipe(classifiers)",
"Step 0: Create ndarray for multilabel targets + select model features \n\ny_arr\n (260715, 7) \n int64 \n [[0 0 0 0 0 1 0]\n [0 0 0 1 0 1 0]] Index(['outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO',\n 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI'],\n dtype='object') \n\nX\n (260715, 7) \n i_f_code object\nrept_cod object\nsex object\noccp_cod object\nn_outc int64\nwt_lbs float64\nage_yrs float64\ndtype: object \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n0 F EXP F LW 1 178.574463 NaN\n1 F EXP F MD 2 NaN 68.0 \n Index(['i_f_code', 'rept_cod', 'sex', 'occp_cod', 'n_outc', 'wt_lbs',\n 'age_yrs'],\n dtype='object') \n\nStep 1: Train-test set split\n\nX_train\n (182500, 7) \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n215322 F EXP F HP 1 NaN 20.0\n147333 F EXP NaN CN 1 NaN NaN\ny_train\n (182500, 7) \n [[0 0 0 0 1 0 0]\n [0 0 0 0 0 1 0]]\nX_test\n (78215, 7) \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n101264 F EXP F MD 1 149.252977 57.0\n1121 F PER M HP 2 NaN 59.0\ny_test\n (78215, 7) \n [[0 0 0 0 0 1 0]\n [0 1 0 1 0 0 0]] \n\nStep 2: Group features by type for pipeline\n\nnum_features\n ['n_outc', 'wt_lbs', 'age_yrs']\ncat_features\n ['i_f_code', 'rept_cod', 'sex', 'occp_cod']\nfeature_labels\n ['n_outc', 'wt_lbs', 'age_yrs', 'i_f_code', 'rept_cod', 'sex', 'occp_cod'] \n\nStep 3: Column transformers by type for pipeline\n\nnum_transformer\n Pipeline(steps=[('imputer', SimpleImputer(strategy='median')),\n ('scaler', StandardScaler())])\ncat_transformer\n Pipeline(steps=[('1_hot', OneHotEncoder(handle_unknown='ignore'))]) \n\nStep 4: Preprocessor for pipeline\n\npreprocessor\n ColumnTransformer(remainder='passthrough',\n transformers=[('num',\n Pipeline(steps=[('imputer',\n SimpleImputer(strategy='median')),\n ('scaler', StandardScaler())]),\n ['n_outc', 'wt_lbs', 'age_yrs']),\n ('cat',\n Pipeline(steps=[('1_hot',\n OneHotEncoder(handle_unknown='ignore'))]),\n ['i_f_code', 'rept_cod', 'sex', 'occp_cod'])]) \n\ny_pred\n (78215, 7) \n [[0 0 0 ... 0 0 0]\n [0 1 0 ... 0 0 0]\n [0 0 0 ... 0 0 0]\n ...\n [0 0 0 ... 0 0 0]\n [0 0 0 ... 0 1 0]\n [0 1 0 ... 0 0 0]] \n\nMultilabel Classifier: Performance Metrics:\n\naccuracy: 0.36216838202390844\nhamming loss: 0.16593273120793417\njaccard score: 0.41249013800328516\nroc auc score: 0.6861288652766024\naverage precision score: 0.25951938521440965 \n\nRecall, Specificity, Fall Out and Miss Rate for Multilabel Adverse Event Outcomes:\n ['outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO', 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI']\nrecall (true pos rate):\n [0.99800847 0.91542261 0.9812189 0.7502746 0.97531649 0.50529399\n 0.99888193]\nspecificity (true neg rate):\n [0.04706676 0.31680418 0.11486486 0.69057324 0.26307299 0.75766912\n 0.05223881]\nfall out (false pos rate):\n [0.95293324 0.68319582 0.88513514 0.30942676 0.73692701 0.24233088\n 0.94776119]\nmiss rate (false neg rate):\n [0.00199153 0.08457739 0.0187811 0.2497254 0.02468351 0.49470601\n 0.00111807] \n\n"
],
[
"# use model object to create predictions on new data\n\n# load fit clf\nml_clf_obj = load('ml_clf_obj.joblib')\ndir(ml_clf_obj)",
"_____no_output_____"
],
[
"# predict multilabel outcomes\n# prep new data\nX, y_arr = df_prep(ddf)\n# predict ml outcomes\ny_arr_pred = ml_clf_obj.predict(X)\nprint('Predicted AEs: RandomForest Classifier\\n', y_arr_pred.shape, '\\n', y_arr_pred[5:])",
"Step 0: Create ndarray for multilabel targets + select model features \n\ny_arr\n (260715, 7) \n int64 \n [[0 0 0 0 0 1 0]\n [0 0 0 1 0 1 0]] Index(['outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO',\n 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI'],\n dtype='object') \n\nX\n (260715, 7) \n i_f_code object\nrept_cod object\nsex object\noccp_cod object\nn_outc int64\nwt_lbs float64\nage_yrs float64\ndtype: object \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n0 F EXP F LW 1 178.574463 NaN\n1 F EXP F MD 2 NaN 68.0 \n Index(['i_f_code', 'rept_cod', 'sex', 'occp_cod', 'n_outc', 'wt_lbs',\n 'age_yrs'],\n dtype='object') \n\nPredicted AEs: RandomForest Classifier\n (260715, 7) \n [[0 0 0 ... 0 1 0]\n [0 0 0 ... 0 1 0]\n [0 0 0 ... 0 1 0]\n ...\n [0 0 0 ... 0 1 0]\n [0 0 0 ... 0 1 0]\n [0 0 0 ... 0 1 0]]\n"
]
],
[
[
"## RidgeClassifierCV",
"_____no_output_____"
]
],
[
[
"# multilabel clfs\nclassifiers = [ \n RidgeClassifierCV(class_weight='balanced'),\n #DecisionTreeClassifier(class_weight='balanced'),\n #ExtraTreesClassifier(class_weight='balanced'),\n #RandomForestClassifier(class_weight='balanced'),\n #MLPClassifier(solver='sdg', learning_rate='adaptive', early_stopping=True),\n #KNeighborsClassifier(weights='distance'),\n #RadiusNeighborsClassifier(weights='distance')\n]\n \n# fit and eval model\nif __name__ == '__main__':\n ml_clf_pipe(classifiers)",
"Step 0: Create ndarray for multilabel targets + select model features \n\ny_arr\n (260715, 7) \n int64 \n [[0 0 0 0 0 1 0]\n [0 0 0 1 0 1 0]] Index(['outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO',\n 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI'],\n dtype='object') \n\nX\n (260715, 7) \n i_f_code object\nrept_cod object\nsex object\noccp_cod object\nn_outc int64\nwt_lbs float64\nage_yrs float64\ndtype: object \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n0 F EXP F LW 1 178.574463 NaN\n1 F EXP F MD 2 NaN 68.0 \n Index(['i_f_code', 'rept_cod', 'sex', 'occp_cod', 'n_outc', 'wt_lbs',\n 'age_yrs'],\n dtype='object') \n\nStep 1: Train-test set split\n\nX_train\n (182500, 7) \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n108556 F EXP M HP 2 176.369840 75.0\n187688 I EXP M MD 1 190.589658 NaN\ny_train\n (182500, 7) \n [[0 0 0 1 0 1 0]\n [0 1 0 0 0 0 0]]\nX_test\n (78215, 7) \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n227008 I PER M MD 1 NaN 19.0\n144205 I EXP M MD 1 NaN 23.0\ny_test\n (78215, 7) \n [[0 0 0 0 0 1 0]\n [0 1 0 0 0 0 0]] \n\nStep 2: Group features by type for pipeline\n\nnum_features\n ['n_outc', 'wt_lbs', 'age_yrs']\ncat_features\n ['i_f_code', 'rept_cod', 'sex', 'occp_cod']\nfeature_labels\n ['n_outc', 'wt_lbs', 'age_yrs', 'i_f_code', 'rept_cod', 'sex', 'occp_cod'] \n\nStep 3: Column transformers by type for pipeline\n\nnum_transformer\n Pipeline(steps=[('imputer', SimpleImputer(strategy='median')),\n ('scaler', StandardScaler())])\ncat_transformer\n Pipeline(steps=[('1_hot', OneHotEncoder(handle_unknown='ignore'))]) \n\nStep 4: Preprocessor for pipeline\n\npreprocessor\n ColumnTransformer(remainder='passthrough',\n transformers=[('num',\n Pipeline(steps=[('imputer',\n SimpleImputer(strategy='median')),\n ('scaler', StandardScaler())]),\n ['n_outc', 'wt_lbs', 'age_yrs']),\n ('cat',\n Pipeline(steps=[('1_hot',\n OneHotEncoder(handle_unknown='ignore'))]),\n ['i_f_code', 'rept_cod', 'sex', 'occp_cod'])]) \n\ny_pred\n (78215, 7) \n [[1 1 0 ... 0 1 0]\n [0 0 0 ... 0 0 0]\n [0 0 0 ... 0 0 0]\n ...\n [0 0 0 ... 0 0 1]\n [0 0 0 ... 0 1 0]\n [0 0 0 ... 0 0 0]] \n\nMultilabel Classifier: Performance Metrics:\n\naccuracy: 0.14393658505401777\nhamming loss: 0.21646925598852979\njaccard score: 0.3271451442586095\nroc auc score: 0.7566282487401436\naverage precision score: 0.2571796495934411 \n\nRecall, Specificity, Fall Out and Miss Rate for Multilabel Adverse Event Outcomes:\n ['outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO', 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI']\nrecall (true pos rate):\n [0.99861045 0.90686517 0.98722903 0.72756346 0.98526667 0.47602629\n 1. ]\nspecificity (true neg rate):\n [0.02774059 0.29496738 0.07443366 0.71358645 0.20364288 0.77437995\n 0.03251089]\nfall out (false pos rate):\n [0.97225941 0.70503262 0.92556634 0.28641355 0.79635712 0.22562005\n 0.96748911]\nmiss rate (false neg rate):\n [0.00138955 0.09313483 0.01277097 0.27243654 0.01473333 0.52397371\n 0. ] \n\n"
],
[
"# use model object to create predictions on new data\n\n# load fit clf\nml_clf_obj = load('ml_clf_obj.joblib')\ndir(ml_clf_obj)",
"_____no_output_____"
],
[
"# predict multilabel outcomes\n# prep new data\nX, y_arr = df_prep(ddf)\n# predict ml outcomes\ny_arr_pred = ml_clf_obj.predict(X)\nprint('Predicted AEs: RidgeClassifierCV\\n', y_arr_pred.shape, '\\n', y_arr_pred[5:])",
"Step 0: Create ndarray for multilabel targets + select model features \n\ny_arr\n (260715, 7) \n int64 \n [[0 0 0 0 0 1 0]\n [0 0 0 1 0 1 0]] Index(['outc_cod__CA', 'outc_cod__DE', 'outc_cod__DS', 'outc_cod__HO',\n 'outc_cod__LT', 'outc_cod__OT', 'outc_cod__RI'],\n dtype='object') \n\nX\n (260715, 7) \n i_f_code object\nrept_cod object\nsex object\noccp_cod object\nn_outc int64\nwt_lbs float64\nage_yrs float64\ndtype: object \n i_f_code rept_cod sex occp_cod n_outc wt_lbs age_yrs\n0 F EXP F LW 1 178.574463 NaN\n1 F EXP F MD 2 NaN 68.0 \n Index(['i_f_code', 'rept_cod', 'sex', 'occp_cod', 'n_outc', 'wt_lbs',\n 'age_yrs'],\n dtype='object') \n\nPredicted AEs: RidgeClassifierCV\n (260715, 7) \n [[1 0 1 ... 0 1 0]\n [1 1 1 ... 1 1 0]\n [0 0 1 ... 0 1 0]\n ...\n [0 0 1 ... 0 1 0]\n [0 0 0 ... 0 1 0]\n [0 0 1 ... 0 1 0]]\n"
]
],
[
[
"# STOPPED HERE - 3.5.2021\nTODOs: CLEAN UP NB, GITHUB, SLACK",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
]
] |
e7ec62f6153e738b6c57bca4db3d08fa3d8e0df4 | 421,542 | ipynb | Jupyter Notebook | dqn_plus/notebooks/Results.ipynb | hadleyhzy34/reinforcement_learning | 14371756c2ff8225dc800d146452b7956875410c | [
"MIT"
] | null | null | null | dqn_plus/notebooks/Results.ipynb | hadleyhzy34/reinforcement_learning | 14371756c2ff8225dc800d146452b7956875410c | [
"MIT"
] | null | null | null | dqn_plus/notebooks/Results.ipynb | hadleyhzy34/reinforcement_learning | 14371756c2ff8225dc800d146452b7956875410c | [
"MIT"
] | null | null | null | 1,458.622837 | 52,756 | 0.956232 | [
[
[
"import numpy as np\nimport matplotlib.pyplot as plt",
"_____no_output_____"
],
[
"def read_data(n):\n def to_binary(n):\n result = []\n for i in range(3):\n result.append(n % 2)\n n //= 2\n return result\n binary = to_binary(n)\n binary = [True if num else False for num in binary]\n DUEL, DOUBLE, PRIORITIZED = binary\n RAM_ENV_NAME = 'LunarLander-v2'\n data = np.load('./code/lunarlander_results/{}_{}_{}_{}_rewards.npy'.format(RAM_ENV_NAME, DUEL, DOUBLE, PRIORITIZED))\n return data",
"_____no_output_____"
],
[
"# Vanilla DQN\ndata = read_data(0)\nplt.figure(figsize=(17, 8))\nplt.plot(np.mean(data, axis=0))\nplt.plot(np.quantile(data, 0.95, axis=0), alpha=0.2)\nplt.plot(np.quantile(data, 0.05, axis=0), alpha=0.2)\nplt.plot([0, 1000], [200, 200], '--', color='r')\nplt.plot([0, 1000], [250, 250], '--', color='b')\nplt.ylim(-300, 300)\nplt.show()",
"_____no_output_____"
],
[
"# Duel DQN\ndata = read_data(1)\nplt.figure(figsize=(17, 8))\nplt.plot(np.mean(data, axis=0))\nplt.plot(np.quantile(data, 0.95, axis=0), alpha=0.2)\nplt.plot(np.quantile(data, 0.05, axis=0), alpha=0.2)\nplt.plot([0, 1000], [200, 200], '--', color='r')\nplt.plot([0, 1000], [250, 250], '--', color='b')\nplt.ylim(-300, 300)\nplt.show()",
"_____no_output_____"
],
[
"# Double DQN\ndata = read_data(2)\nplt.figure(figsize=(17, 8))\nplt.plot(np.mean(data, axis=0))\nplt.plot(np.quantile(data, 0.95, axis=0), alpha=0.2)\nplt.plot(np.quantile(data, 0.05, axis=0), alpha=0.2)\nplt.plot([0, 1000], [200, 200], '--', color='r')\nplt.plot([0, 1000], [250, 250], '--', color='b')\nplt.ylim(-300, 300)\nplt.show()",
"_____no_output_____"
],
[
"# Prioritized DQN\ndata = read_data(4)\nplt.figure(figsize=(17, 8))\nplt.plot(np.mean(data, axis=0))\nplt.plot(np.quantile(data, 0.95, axis=0), alpha=0.2)\nplt.plot(np.quantile(data, 0.05, axis=0), alpha=0.2)\nplt.plot([0, 1000], [200, 200], '--', color='r')\nplt.plot([0, 1000], [250, 250], '--', color='b')\nplt.ylim(-300, 300)\nplt.show()",
"_____no_output_____"
],
[
"# Duel + Double DQN\ndata = read_data(3)\nplt.figure(figsize=(17, 8))\nplt.plot(np.mean(data, axis=0))\nplt.plot(np.quantile(data, 0.95, axis=0), alpha=0.2)\nplt.plot(np.quantile(data, 0.05, axis=0), alpha=0.2)\nplt.plot([0, 1000], [200, 200], '--', color='r')\nplt.plot([0, 1000], [250, 250], '--', color='b')\nplt.ylim(-300, 300)\nplt.show()",
"_____no_output_____"
],
[
"# Duel + Prioritized DQN\ndata = read_data(5)\nplt.figure(figsize=(17, 8))\nplt.plot(np.mean(data, axis=0))\nplt.plot(np.quantile(data, 0.95, axis=0), alpha=0.2)\nplt.plot(np.quantile(data, 0.05, axis=0), alpha=0.2)\nplt.plot([0, 1000], [200, 200], '--', color='r')\nplt.plot([0, 1000], [250, 250], '--', color='b')\nplt.ylim(-300, 300)\nplt.show()",
"_____no_output_____"
],
[
"# Double + Prioritized DQN\ndata = read_data(6)\nplt.figure(figsize=(17, 8))\nplt.plot(np.mean(data, axis=0))\nplt.plot(np.quantile(data, 0.95, axis=0), alpha=0.2)\nplt.plot(np.quantile(data, 0.05, axis=0), alpha=0.2)\nplt.plot([0, 1000], [200, 200], '--', color='r')\nplt.plot([0, 1000], [250, 250], '--', color='b')\nplt.ylim(-300, 300)\nplt.show()",
"_____no_output_____"
],
[
"# Dule + Double + Prioritized DQN\ndata = read_data(7)\nplt.figure(figsize=(17, 8))\nplt.plot(np.mean(data, axis=0))\nplt.plot(np.quantile(data, 0.95, axis=0), alpha=0.2)\nplt.plot(np.quantile(data, 0.05, axis=0), alpha=0.2)\nplt.plot([0, 1000], [200, 200], '--', color='r')\nplt.plot([0, 1000], [250, 250], '--', color='b')\nplt.ylim(-300, 300)\nplt.show()",
"_____no_output_____"
]
]
] | [
"code"
] | [
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
]
] |
e7ec952bff3bf31d2d90fc9313ba4d5576608f21 | 403,954 | ipynb | Jupyter Notebook | .ipynb_checkpoints/Zoo dataset-Giovanni commit-checkpoint.ipynb | GiovanniDiMasi/Cluster_animali | d066e7e44c72e592af1fa17ce74a0a6fe9f860f5 | [
"MIT"
] | 1 | 2020-12-22T17:32:29.000Z | 2020-12-22T17:32:29.000Z | .ipynb_checkpoints/Zoo dataset-Giovanni commit-checkpoint.ipynb | GiovanniDiMasi/Cluster_animali | d066e7e44c72e592af1fa17ce74a0a6fe9f860f5 | [
"MIT"
] | null | null | null | .ipynb_checkpoints/Zoo dataset-Giovanni commit-checkpoint.ipynb | GiovanniDiMasi/Cluster_animali | d066e7e44c72e592af1fa17ce74a0a6fe9f860f5 | [
"MIT"
] | 1 | 2020-12-31T15:06:49.000Z | 2020-12-31T15:06:49.000Z | 189.383029 | 193,448 | 0.85505 | [
[
[
"import numpy as np\nimport pandas as pd",
"_____no_output_____"
],
[
"p,f = \"class.csv\",\"zoo.csv\"",
"_____no_output_____"
],
[
"data,classi = pd.read_csv(f),pd.read_csv(p)",
"_____no_output_____"
],
[
"y_verita = data['class_type'].to_numpy() ###questo è l'array verità in base al quale confrontare i vari risultati di clustering \nprint(y_verita)\n",
"[1 1 4 1 1 1 1 4 4 1 1 2 4 7 7 7 2 1 4 1 2 2 1 2 6 5 5 1 1 1 6 1 1 2 4 1 1\n 2 4 6 6 2 6 2 1 1 7 1 1 1 1 6 5 7 1 1 2 2 2 2 4 4 3 1 1 1 1 1 1 1 1 2 7 4\n 1 1 3 7 2 2 3 7 4 2 1 7 4 2 6 5 3 3 4 1 1 2 1 6 1 7 2]\n"
]
],
[
[
"# Preprocessing",
"_____no_output_____"
]
],
[
[
"X = data.drop(['animal_name']+['class_type'],axis=1)\n###Eliminiamo dal dataframe i nomi degli animali che sono una variabile categorica e la classe di appartenenza che \n###vogliamo trovare con gli algoritmi di clustering",
"_____no_output_____"
],
[
"from sklearn.preprocessing import MinMaxScaler",
"_____no_output_____"
],
[
"scaler = MinMaxScaler()\nXs = pd.DataFrame(scaler.fit_transform(X), columns=X.columns)\n###come sappiamo applicando uno scaler otteniamo un oggetto numpy, perciò ricreiamo il dataframe utilizzando gli indici di colonna originali",
"_____no_output_____"
],
[
"Xs\n###Come abbiamo visto il nostro dataset non contiene valori nulli e tutti gli attributi hanno valori booleani 1 o 0 \n###a eccezione della colonna legs, perciò ho apllicato MinMaxScaler per scalare i valori in modo che anche legs \n###assuma valori tra 0 e 1.\n###A questo punto i dati sono pronti per applicare gli algoritmi di clustering.",
"_____no_output_____"
]
],
[
[
"# Clustering",
"_____no_output_____"
]
],
[
[
"###come funziona ciascuno di questi algoritmi?????",
"_____no_output_____"
]
],
[
[
"Kmeans",
"_____no_output_____"
]
],
[
[
"from sklearn.cluster import KMeans,AgglomerativeClustering,SpectralClustering,DBSCAN,Birch",
"_____no_output_____"
],
[
"kmeans = KMeans(n_clusters= 7,random_state=0)\n### Random state è un parametro che ci serve per fare in modo che i centroidi di partenza siano determinati a partire \n### da un numero, e non generati casualmente in modo che ripetendo il clustering tutte le volte abbiamo lo stesso \n### risultato\ny_pred_k = kmeans.fit_predict(Xs)",
"_____no_output_____"
]
],
[
[
"Agglomerative clustering",
"_____no_output_____"
]
],
[
[
"aggc = AgglomerativeClustering(n_clusters = 7, affinity = 'euclidean', linkage = 'ward' )\ny_pred_aggc =aggc.fit_predict(Xs)",
"_____no_output_____"
]
],
[
[
"SpectralClustering",
"_____no_output_____"
]
],
[
[
"spc = SpectralClustering(n_clusters=7, assign_labels=\"discretize\", random_state=0)\ny_pred_spc = spc.fit_predict(Xs)",
"_____no_output_____"
]
],
[
[
"DBSCAN",
"_____no_output_____"
]
],
[
[
"dbscan =DBSCAN(eps=0.3,min_samples=4)\ny_pred_dbscan = dbscan.fit_predict(Xv)\n#### Ho verificato che con Dbscan otteniamo un risultato molto migliroe se usiamo il dataset in 2 dimensioni Xv\n#### che a questo punto ci conviene calcolare prima della parte di results visualization ",
"_____no_output_____"
]
],
[
[
"Birch",
"_____no_output_____"
]
],
[
[
"brc = Birch(n_clusters=7, threshold = 0.1)\ny_pred_brc = brc.fit_predict(Xs)",
"_____no_output_____"
]
],
[
[
"# Results visualization",
"_____no_output_____"
]
],
[
[
"import matplotlib.pyplot as plt\nfrom sklearn.decomposition import PCA",
"_____no_output_____"
],
[
"pca = PCA(2)\nXv = pca.fit_transform(Xs)\n### Applichiamo la principal component analysis per comprimere i dati in due dimensioni e poterli visualizzare.",
"_____no_output_____"
],
[
"fig, ax = plt.subplots(figsize=(16,9),ncols=3, nrows=2)\nax[0][0].scatter(Xv[:,0],Xv[:,1], s=110, c=y_verita)\nax[0][1].scatter(Xv[:,0],Xv[:,1], s=110, c=y_pred_k)\nax[0][2].scatter(Xv[:,0],Xv[:,1], s=110, c=y_pred_aggc)\nax[1][0].scatter(Xv[:,0],Xv[:,1], s=110, c=y_pred_spc)\nax[1][1].scatter(Xv[:,0],Xv[:,1], s=110, c=y_pred_dbscan)\nax[1][2].scatter(Xv[:,0],Xv[:,1], s=110, c=y_pred_brc)\n\nax[0][0].set_title('Classificazione reale', fontsize = 22)\nax[0][1].set_title('Kmeans', fontsize = 22)\nax[0][2].set_title('Agglomerative clustering', fontsize = 22)\nax[1][0].set_title('Spectral clustering', fontsize = 22)\nax[1][1].set_title('Dbscan', fontsize = 22)\nax[1][2].set_title('Birch',fontsize = 22)\n\nplt.tight_layout()\nplt.show()\n### Visualizziamo i diversi risultati di clustering sulle coordinate dei nostri animali portate in 2 dimensioni.",
"_____no_output_____"
]
],
[
[
"# Benchmark e interpretazione",
"_____no_output_____"
]
],
[
[
"from sklearn.metrics import adjusted_rand_score, completeness_score\n### Bisogna dare una piccola descrizione di cosa misurano queste due metriche e perchè sono state scelte",
"_____no_output_____"
]
],
[
[
"Utilizziamo due metriche diverse per verificare quanto il risultato ottenuto con gli algoritmi di clustering sia accurato rispetto alla ground truth.",
"_____no_output_____"
],
[
"Kmeans",
"_____no_output_____"
]
],
[
[
"risultati = {}\nk_c= completeness_score(y_verita,y_pred_k)\nk_a = adjusted_rand_score(y_verita,y_pred_k)\nrisultati['Kmeans'] =[k_c,k_a]",
"_____no_output_____"
]
],
[
[
"AgglomerativeClustering",
"_____no_output_____"
]
],
[
[
"aggc_c= completeness_score(y_verita,y_pred_aggc)\naggc_a = adjusted_rand_score(y_verita,y_pred_aggc)\nrisultati['Agglomerative clustering']=[aggc_c,aggc_a]",
"_____no_output_____"
]
],
[
[
"SpectralClustering",
"_____no_output_____"
]
],
[
[
"spc_c= completeness_score(y_verita,y_pred_spc)\nspc_a = adjusted_rand_score(y_verita,y_pred_spc)\nrisultati['Spectral clustering']=[spc_c,spc_a]",
"_____no_output_____"
]
],
[
[
"DBSCAN",
"_____no_output_____"
]
],
[
[
"dbscan_c= completeness_score(y_verita,y_pred_dbscan)\ndbscan_a = adjusted_rand_score(y_verita,y_pred_dbscan)\nrisultati['Dbscan']=[dbscan_c,dbscan_a]",
"_____no_output_____"
]
],
[
[
"Birch",
"_____no_output_____"
]
],
[
[
"brc_c= completeness_score(y_verita,y_pred_brc)\nbrc_a = adjusted_rand_score(y_verita,y_pred_brc)\nrisultati['Birch']=[brc_c,brc_a]",
"_____no_output_____"
],
[
"risultati\n###perchè è il migliore???",
"_____no_output_____"
]
],
[
[
"L'algoritmo migliore si rivela essere lo spectral clustering",
"_____no_output_____"
]
],
[
[
"## funzione per trovare la posizione di ogni membro del cluster nel dataset originale \ndef select_points(X, y_pred, cluster_label):\n pos = [i for i, x in enumerate(y_pred) if x == cluster_label]\n return X.iloc[pos]",
"_____no_output_____"
],
[
"select_points(data,y_pred_spc,3)\n### Tutti animali dello stesso class_type eccetto la tartaruga come ci aspettiamo visto il punteggio molto \n### alto dell'algoritmo spectral clustering",
"_____no_output_____"
],
[
"select_points(data,y_pred_dbscan,3)\n### dbscan invece conferma il punteggio basso mettendo nella stessa classe animali piuttosto diversi",
"_____no_output_____"
],
[
"from scipy.cluster.hierarchy import dendrogram , linkage",
"_____no_output_____"
],
[
"##qui costruisco un dendogramma per un clustering gerarchico\nZ = linkage(X, method = 'complete')\nplt.figure(figsize = (32,40))\ndendro = dendrogram(Z, orientation = \"left\",\n labels=[x for x in data[\"animal_name\"]],\n leaf_font_size=22)\nplt.title(\"Dendrogram\", fontsize = 30, fontweight=\"bold\")\nplt.ylabel('Euclidean distance', fontsize = 22)\nplt.xlabel(\"Animal \", fontsize = 22)\nplt.show()",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
]
] |
e7ec9f4a3a1e09d1781a2807ab861993aca1068a | 34,690 | ipynb | Jupyter Notebook | 04_Apply/Students_Alcohol_Consumption/Solutions.ipynb | alexkataev/pandas_exercises | 60b4a0f8b760eec6fdfae854be76e2481fcade85 | [
"BSD-3-Clause"
] | 8,030 | 2016-07-19T19:55:08.000Z | 2022-03-31T20:59:21.000Z | 04_Apply/Students_Alcohol_Consumption/Solutions.ipynb | alexkataev/pandas_exercises | 60b4a0f8b760eec6fdfae854be76e2481fcade85 | [
"BSD-3-Clause"
] | 82 | 2016-11-07T13:31:53.000Z | 2022-03-21T16:41:50.000Z | 04_Apply/Students_Alcohol_Consumption/Solutions.ipynb | alexkataev/pandas_exercises | 60b4a0f8b760eec6fdfae854be76e2481fcade85 | [
"BSD-3-Clause"
] | 6,897 | 2016-07-19T19:22:00.000Z | 2022-03-31T13:14:05.000Z | 29.448217 | 181 | 0.31228 | [
[
[
"# Student Alcohol Consumption",
"_____no_output_____"
],
[
"### Introduction:\n\nThis time you will download a dataset from the UCI.\n\n### Step 1. Import the necessary libraries",
"_____no_output_____"
],
[
"### Step 2. Import the dataset from this [address](https://raw.githubusercontent.com/guipsamora/pandas_exercises/master/04_Apply/Students_Alcohol_Consumption/student-mat.csv).",
"_____no_output_____"
],
[
"### Step 3. Assign it to a variable called df.",
"_____no_output_____"
],
[
"### Step 4. For the purpose of this exercise slice the dataframe from 'school' until the 'guardian' column",
"_____no_output_____"
],
[
"### Step 5. Create a lambda function that will capitalize strings.",
"_____no_output_____"
],
[
"### Step 6. Capitalize both Mjob and Fjob",
"_____no_output_____"
],
[
"### Step 7. Print the last elements of the data set.",
"_____no_output_____"
],
[
"### Step 8. Did you notice the original dataframe is still lowercase? Why is that? Fix it and capitalize Mjob and Fjob.",
"_____no_output_____"
],
[
"### Step 9. Create a function called majority that returns a boolean value to a new column called legal_drinker (Consider majority as older than 17 years old)",
"_____no_output_____"
],
[
"### Step 10. Multiply every number of the dataset by 10. \n##### I know this makes no sense, don't forget it is just an exercise",
"_____no_output_____"
]
]
] | [
"markdown"
] | [
[
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown",
"markdown"
]
] |
e7ecb0fa3bb8b67a7b54ab508ad58a7b69a0bb40 | 2,542 | ipynb | Jupyter Notebook | notebooks/Standard Build-in Objects/Untitled.ipynb | harolz/learn-js-in-jupyter | 117dce39461baa575fe7e0c122b55f51741306f6 | [
"MIT"
] | null | null | null | notebooks/Standard Build-in Objects/Untitled.ipynb | harolz/learn-js-in-jupyter | 117dce39461baa575fe7e0c122b55f51741306f6 | [
"MIT"
] | null | null | null | notebooks/Standard Build-in Objects/Untitled.ipynb | harolz/learn-js-in-jupyter | 117dce39461baa575fe7e0c122b55f51741306f6 | [
"MIT"
] | null | null | null | 27.630435 | 330 | 0.575924 | [
[
[
"empty"
]
]
] | [
"empty"
] | [
[
"empty"
]
] |
e7ecb7ae641a1cc5982377740d2af405ba388873 | 1,344 | ipynb | Jupyter Notebook | Test1_Mask_RCNN.ipynb | Art-phys/Mask_RCNN | aa80c54df0f5f2099964c140b46d386eff4054c2 | [
"MIT"
] | null | null | null | Test1_Mask_RCNN.ipynb | Art-phys/Mask_RCNN | aa80c54df0f5f2099964c140b46d386eff4054c2 | [
"MIT"
] | null | null | null | Test1_Mask_RCNN.ipynb | Art-phys/Mask_RCNN | aa80c54df0f5f2099964c140b46d386eff4054c2 | [
"MIT"
] | null | null | null | 21.677419 | 232 | 0.476935 | [
[
[
"<a href=\"https://colab.research.google.com/github/Art-phys/Mask_RCNN/blob/master/Test1_Mask_RCNN.ipynb\" target=\"_parent\"><img src=\"https://colab.research.google.com/assets/colab-badge.svg\" alt=\"Open In Colab\"/></a>",
"_____no_output_____"
],
[
"# Новый раздел",
"_____no_output_____"
],
[
"# Установка и импорт \n",
"_____no_output_____"
]
],
[
[
"",
"_____no_output_____"
]
]
] | [
"markdown",
"code"
] | [
[
"markdown",
"markdown",
"markdown"
],
[
"code"
]
] |
e7ecc58504bd68ad88bd89826a2e562d1317df29 | 115,852 | ipynb | Jupyter Notebook | hm-recbole-ifs.ipynb | ManashJKonwar/Kaggle-HM-Recommender | abd6e7f040b6560ded1f7f67fe68dff40aa58ff7 | [
"MIT"
] | null | null | null | hm-recbole-ifs.ipynb | ManashJKonwar/Kaggle-HM-Recommender | abd6e7f040b6560ded1f7f67fe68dff40aa58ff7 | [
"MIT"
] | null | null | null | hm-recbole-ifs.ipynb | ManashJKonwar/Kaggle-HM-Recommender | abd6e7f040b6560ded1f7f67fe68dff40aa58ff7 | [
"MIT"
] | null | null | null | 40.35249 | 336 | 0.510513 | [
[
[
"!pip install recbole",
"Collecting recbole\r\n Downloading recbole-1.0.1-py3-none-any.whl (2.0 MB)\r\n |████████████████████████████████| 2.0 MB 218 kB/s \r\n\u001b[?25hCollecting scipy==1.6.0\r\n Downloading scipy-1.6.0-cp37-cp37m-manylinux1_x86_64.whl (27.4 MB)\r\n |████████████████████████████████| 27.4 MB 16.2 MB/s \r\n\u001b[?25hRequirement already satisfied: torch>=1.7.0 in /opt/conda/lib/python3.7/site-packages (from recbole) (1.9.1)\r\nRequirement already satisfied: scikit-learn>=0.23.2 in /opt/conda/lib/python3.7/site-packages (from recbole) (1.0.1)\r\nRequirement already satisfied: colorama==0.4.4 in /opt/conda/lib/python3.7/site-packages (from recbole) (0.4.4)\r\nRequirement already satisfied: pandas>=1.0.5 in /opt/conda/lib/python3.7/site-packages (from recbole) (1.3.5)\r\nRequirement already satisfied: pyyaml>=5.1.0 in /opt/conda/lib/python3.7/site-packages (from recbole) (6.0)\r\nRequirement already satisfied: tensorboard>=2.5.0 in /opt/conda/lib/python3.7/site-packages (from recbole) (2.6.0)\r\nRequirement already satisfied: tqdm>=4.48.2 in /opt/conda/lib/python3.7/site-packages (from recbole) (4.62.3)\r\nCollecting colorlog==4.7.2\r\n Downloading colorlog-4.7.2-py2.py3-none-any.whl (10 kB)\r\nRequirement already satisfied: numpy>=1.17.2 in /opt/conda/lib/python3.7/site-packages (from recbole) (1.20.3)\r\nRequirement already satisfied: python-dateutil>=2.7.3 in /opt/conda/lib/python3.7/site-packages (from pandas>=1.0.5->recbole) (2.8.2)\r\nRequirement already satisfied: pytz>=2017.3 in /opt/conda/lib/python3.7/site-packages (from pandas>=1.0.5->recbole) (2021.3)\r\nRequirement already satisfied: threadpoolctl>=2.0.0 in /opt/conda/lib/python3.7/site-packages (from scikit-learn>=0.23.2->recbole) (3.0.0)\r\nRequirement already satisfied: joblib>=0.11 in /opt/conda/lib/python3.7/site-packages (from scikit-learn>=0.23.2->recbole) (1.1.0)\r\nRequirement already satisfied: wheel>=0.26 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (0.37.0)\r\nRequirement already satisfied: google-auth-oauthlib<0.5,>=0.4.1 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (0.4.6)\r\nRequirement already satisfied: requests<3,>=2.21.0 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (2.26.0)\r\nRequirement already satisfied: werkzeug>=0.11.15 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (2.0.2)\r\nRequirement already satisfied: grpcio>=1.24.3 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (1.43.0)\r\nRequirement already satisfied: absl-py>=0.4 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (0.15.0)\r\nRequirement already satisfied: setuptools>=41.0.0 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (59.5.0)\r\nRequirement already satisfied: google-auth<2,>=1.6.3 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (1.35.0)\r\nRequirement already satisfied: protobuf>=3.6.0 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (3.19.4)\r\nRequirement already satisfied: tensorboard-plugin-wit>=1.6.0 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (1.8.0)\r\nRequirement already satisfied: tensorboard-data-server<0.7.0,>=0.6.0 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (0.6.1)\r\nRequirement already satisfied: markdown>=2.6.8 in /opt/conda/lib/python3.7/site-packages (from tensorboard>=2.5.0->recbole) (3.3.6)\r\nRequirement already satisfied: typing-extensions in /opt/conda/lib/python3.7/site-packages (from torch>=1.7.0->recbole) (4.1.1)\r\nRequirement already satisfied: six in /opt/conda/lib/python3.7/site-packages (from absl-py>=0.4->tensorboard>=2.5.0->recbole) (1.16.0)\r\nRequirement already satisfied: cachetools<5.0,>=2.0.0 in /opt/conda/lib/python3.7/site-packages (from google-auth<2,>=1.6.3->tensorboard>=2.5.0->recbole) (4.2.4)\r\nRequirement already satisfied: pyasn1-modules>=0.2.1 in /opt/conda/lib/python3.7/site-packages (from google-auth<2,>=1.6.3->tensorboard>=2.5.0->recbole) (0.2.7)\r\nRequirement already satisfied: rsa<5,>=3.1.4 in /opt/conda/lib/python3.7/site-packages (from google-auth<2,>=1.6.3->tensorboard>=2.5.0->recbole) (4.8)\r\nRequirement already satisfied: requests-oauthlib>=0.7.0 in /opt/conda/lib/python3.7/site-packages (from google-auth-oauthlib<0.5,>=0.4.1->tensorboard>=2.5.0->recbole) (1.3.0)\r\nRequirement already satisfied: importlib-metadata>=4.4 in /opt/conda/lib/python3.7/site-packages (from markdown>=2.6.8->tensorboard>=2.5.0->recbole) (4.11.3)\r\nRequirement already satisfied: certifi>=2017.4.17 in /opt/conda/lib/python3.7/site-packages (from requests<3,>=2.21.0->tensorboard>=2.5.0->recbole) (2021.10.8)\r\nRequirement already satisfied: idna<4,>=2.5 in /opt/conda/lib/python3.7/site-packages (from requests<3,>=2.21.0->tensorboard>=2.5.0->recbole) (3.1)\r\nRequirement already satisfied: urllib3<1.27,>=1.21.1 in /opt/conda/lib/python3.7/site-packages (from requests<3,>=2.21.0->tensorboard>=2.5.0->recbole) (1.26.7)\r\nRequirement already satisfied: charset-normalizer~=2.0.0 in /opt/conda/lib/python3.7/site-packages (from requests<3,>=2.21.0->tensorboard>=2.5.0->recbole) (2.0.9)\r\nRequirement already satisfied: zipp>=0.5 in /opt/conda/lib/python3.7/site-packages (from importlib-metadata>=4.4->markdown>=2.6.8->tensorboard>=2.5.0->recbole) (3.6.0)\r\nRequirement already satisfied: pyasn1<0.5.0,>=0.4.6 in /opt/conda/lib/python3.7/site-packages (from pyasn1-modules>=0.2.1->google-auth<2,>=1.6.3->tensorboard>=2.5.0->recbole) (0.4.8)\r\nRequirement already satisfied: oauthlib>=3.0.0 in /opt/conda/lib/python3.7/site-packages (from requests-oauthlib>=0.7.0->google-auth-oauthlib<0.5,>=0.4.1->tensorboard>=2.5.0->recbole) (3.1.1)\r\nInstalling collected packages: scipy, colorlog, recbole\r\n Attempting uninstall: scipy\r\n Found existing installation: scipy 1.7.3\r\n Uninstalling scipy-1.7.3:\r\n Successfully uninstalled scipy-1.7.3\r\n Attempting uninstall: colorlog\r\n Found existing installation: colorlog 6.6.0\r\n Uninstalling colorlog-6.6.0:\r\n Successfully uninstalled colorlog-6.6.0\r\n\u001b[31mERROR: pip's dependency resolver does not currently take into account all the packages that are installed. This behaviour is the source of the following dependency conflicts.\r\npymc3 3.11.5 requires scipy<1.8.0,>=1.7.3, but you have scipy 1.6.0 which is incompatible.\r\npdpbox 0.2.1 requires matplotlib==3.1.1, but you have matplotlib 3.5.1 which is incompatible.\r\nfeaturetools 1.6.0 requires numpy>=1.21.0, but you have numpy 1.20.3 which is incompatible.\r\narviz 0.11.4 requires typing-extensions<4,>=3.7.4.3, but you have typing-extensions 4.1.1 which is incompatible.\u001b[0m\r\nSuccessfully installed colorlog-4.7.2 recbole-1.0.1 scipy-1.6.0\r\n\u001b[33mWARNING: Running pip as the 'root' user can result in broken permissions and conflicting behaviour with the system package manager. It is recommended to use a virtual environment instead: https://pip.pypa.io/warnings/venv\u001b[0m\r\n"
]
],
[
[
"## Importing Libraries",
"_____no_output_____"
]
],
[
[
"import os\nimport gc\nimport numpy as np\nimport pandas as pd",
"_____no_output_____"
],
[
"data_path = r'../input/h-and-m-personalized-fashion-recommendations/transactions_train.csv'\ncustomer_data_path = r'../input/h-and-m-personalized-fashion-recommendations/customers.csv'\narticle_data_path = r'../input/h-and-m-personalized-fashion-recommendations/articles.csv'\nsubmission_data_path = r'../input/h-m-ensembling/submission.csv'",
"_____no_output_____"
],
[
"!mkdir /kaggle/working/recbole_data\nrecbole_data_path = r'/kaggle/working/recbole_data'",
"_____no_output_____"
],
[
"# Data Extraction\ndef create_data(datapath, data_type=None):\n if data_type is None:\n df = pd.read_csv(datapath)\n elif data_type == 'transaction':\n df = pd.read_csv(datapath, dtype={'article_id': str}, parse_dates=['t_dat'])\n elif data_type == 'article':\n df = pd.read_csv(datapath, dtype={'article_id': str})\n return df",
"_____no_output_____"
]
],
[
[
"## Reading Transaction data",
"_____no_output_____"
]
],
[
[
"%%time\n\n# Load all sales data (for 3 years starting from 2018 to 2020)\n# ALso, article_id is treated as a string column otherwise it \n# would drop the leading zeros while reading the specific column values\ntransactions_data=create_data(data_path, data_type='transaction')\nprint(transactions_data.shape)\n\n# # Unique Attributes\nprint(str(len(transactions_data['t_dat'].drop_duplicates())) + \"-total No of unique transactions dates in data sheet\")\nprint(str(len(transactions_data['customer_id'].drop_duplicates())) + \"-total No of unique customers ids in data sheet\")\nprint(str(len(transactions_data['article_id'].drop_duplicates())) + \"-total No of unique article ids courses names in data sheet\")\nprint(str(len(transactions_data['sales_channel_id'].drop_duplicates())) + \"-total No of unique sales channels in data sheet\")\n\ntransactions_data.head()",
"(31788324, 5)\n734-total No of unique transactions dates in data sheet\n1362281-total No of unique customers ids in data sheet\n104547-total No of unique article ids courses names in data sheet\n2-total No of unique sales channels in data sheet\nCPU times: user 55.3 s, sys: 4.24 s, total: 59.5 s\nWall time: 1min 20s\n"
]
],
[
[
"## Postprocessing Transaction data \n\n1. timestamp column is created from transaction dates column \n2. columns are renamed for easy reading",
"_____no_output_____"
]
],
[
[
"transactions_data['timestamp'] = transactions_data.t_dat.values.astype(np.int64)// 10 ** 9\ntransactions_data = transactions_data[transactions_data['timestamp'] > 1585620000]\ntransactions_data = transactions_data[['customer_id','article_id','timestamp']].rename(columns={'customer_id': 'user_id:token', \n 'article_id': 'item_id:token', \n 'timestamp': 'timestamp:float'})\ntransactions_data",
"_____no_output_____"
]
],
[
[
"## Saving transaction data to kaggle based recbole output directory",
"_____no_output_____"
]
],
[
[
"transactions_data.to_csv(os.path.join(recbole_data_path, 'recbole_data.inter'), index=False, sep='\\t')\ndel [[transactions_data]]\ngc.collect()",
"_____no_output_____"
]
],
[
[
"## Reading Article data",
"_____no_output_____"
]
],
[
[
"%%time\n\n# Load all Customers\narticle_data=create_data(article_data_path, data_type='article')\nprint(article_data.shape)\n\nprint(str(len(article_data['article_id'].drop_duplicates())) + \"-total No of unique article ids in article data sheet\")\n\narticle_data.head()",
"(105542, 25)\n105542-total No of unique article ids in article data sheet\nCPU times: user 716 ms, sys: 43.6 ms, total: 760 ms\nWall time: 1.07 s\n"
]
],
[
[
"## Postprocessing Article data \n\n1. drop duplicate columns to avoid multicollinearity\n2. columns are renamed for easy reading",
"_____no_output_____"
]
],
[
[
"article_data = article_data.drop(columns = ['product_type_name', 'graphical_appearance_name', 'colour_group_name', \n 'perceived_colour_value_name', 'perceived_colour_master_name', 'index_name', \n 'index_group_name', 'section_name', 'garment_group_name', \n 'prod_name', 'department_name', 'detail_desc'])\narticle_data = article_data.rename(columns = {'article_id': 'item_id:token', \n 'product_code': 'product_code:token', \n 'product_type_no': 'product_type_no:float',\n 'product_group_name': 'product_group_name:token_seq', \n 'graphical_appearance_no': 'graphical_appearance_no:token', \n 'colour_group_code': 'colour_group_code:token', \n 'perceived_colour_value_id': 'perceived_colour_value_id:token', \n 'perceived_colour_master_id': 'perceived_colour_master_id:token', \n 'department_no': 'department_no:token', \n 'index_code': 'index_code:token', \n 'index_group_no': 'index_group_no:token', \n 'section_no': 'section_no:token', \n 'garment_group_no': 'garment_group_no:token'})\narticle_data",
"_____no_output_____"
]
],
[
[
"## Saving article data to kaggle based recbole output directory",
"_____no_output_____"
]
],
[
[
"article_data.to_csv(os.path.join(recbole_data_path, 'recbole_data.item'), index=False, sep='\\t')\ndel [[article_data]]\ngc.collect()",
"_____no_output_____"
]
],
[
[
"## Setting up Recbole based dataset and configurations",
"_____no_output_____"
]
],
[
[
"import logging\nfrom logging import getLogger\nfrom recbole.config import Config\nfrom recbole.data import create_dataset, data_preparation\nfrom recbole.model.sequential_recommender import GRU4RecF\nfrom recbole.trainer import Trainer\nfrom recbole.utils import init_seed, init_logger",
"_____no_output_____"
],
[
"parameter_dict = {\n 'data_path': '/kaggle/working',\n 'USER_ID_FIELD': 'user_id',\n 'ITEM_ID_FIELD': 'item_id',\n 'TIME_FIELD': 'timestamp',\n 'user_inter_num_interval': \"[40,inf)\",\n 'item_inter_num_interval': \"[40,inf)\",\n 'load_col': {'inter': ['user_id', 'item_id', 'timestamp'],\n 'item': ['item_id', 'product_code', 'product_type_no', 'product_group_name', 'graphical_appearance_no',\n 'colour_group_code', 'perceived_colour_value_id', 'perceived_colour_master_id',\n 'department_no', 'index_code', 'index_group_no', 'section_no', 'garment_group_no']\n },\n 'selected_features': ['product_code', 'product_type_no', 'product_group_name', 'graphical_appearance_no',\n 'colour_group_code', 'perceived_colour_value_id', 'perceived_colour_master_id',\n 'department_no', 'index_code', 'index_group_no', 'section_no', 'garment_group_no'],\n 'neg_sampling': None,\n 'epochs': 100,\n 'eval_args': {\n 'split': {'RS': [10, 0, 0]},\n 'group_by': 'user',\n 'order': 'TO',\n 'mode': 'full'},\n 'topk':[12]\n}",
"_____no_output_____"
],
[
"config = Config(model='GRU4RecF', dataset='recbole_data', config_dict=parameter_dict)\n\n# init random seed\ninit_seed(config['seed'], config['reproducibility'])\n\n# logger initialization\ninit_logger(config)\nlogger = getLogger()\n# Create handlers\nc_handler = logging.StreamHandler()\nc_handler.setLevel(logging.INFO)\nlogger.addHandler(c_handler)\n\n# write config info into log\nlogger.info(config)",
"\nGeneral Hyper Parameters:\ngpu_id = 0\nuse_gpu = True\nseed = 2020\nstate = INFO\nreproducibility = True\ndata_path = /kaggle/working/recbole_data\ncheckpoint_dir = saved\nshow_progress = True\nsave_dataset = False\ndataset_save_path = None\nsave_dataloaders = False\ndataloaders_save_path = None\nlog_wandb = False\n\nTraining Hyper Parameters:\nepochs = 100\ntrain_batch_size = 2048\nlearner = adam\nlearning_rate = 0.001\nneg_sampling = None\neval_step = 1\nstopping_step = 10\nclip_grad_norm = None\nweight_decay = 0.0\nloss_decimal_place = 4\n\nEvaluation Hyper Parameters:\neval_args = {'split': {'RS': [10, 0, 0]}, 'group_by': 'user', 'order': 'TO', 'mode': 'full'}\nrepeatable = True\nmetrics = ['Recall', 'MRR', 'NDCG', 'Hit', 'Precision']\ntopk = [12]\nvalid_metric = MRR@10\nvalid_metric_bigger = True\neval_batch_size = 4096\nmetric_decimal_place = 4\n\nDataset Hyper Parameters:\nfield_separator = \t\nseq_separator = \nUSER_ID_FIELD = user_id\nITEM_ID_FIELD = item_id\nRATING_FIELD = rating\nTIME_FIELD = timestamp\nseq_len = None\nLABEL_FIELD = label\nthreshold = None\nNEG_PREFIX = neg_\nload_col = {'inter': ['user_id', 'item_id', 'timestamp'], 'item': ['item_id', 'product_code', 'product_type_no', 'product_group_name', 'graphical_appearance_no', 'colour_group_code', 'perceived_colour_value_id', 'perceived_colour_master_id', 'department_no', 'index_code', 'index_group_no', 'section_no', 'garment_group_no']}\nunload_col = None\nunused_col = None\nadditional_feat_suffix = None\nrm_dup_inter = None\nval_interval = None\nfilter_inter_by_user_or_item = True\nuser_inter_num_interval = [40,inf)\nitem_inter_num_interval = [40,inf)\nalias_of_user_id = None\nalias_of_item_id = None\nalias_of_entity_id = None\nalias_of_relation_id = None\npreload_weight = None\nnormalize_field = None\nnormalize_all = None\nITEM_LIST_LENGTH_FIELD = item_length\nLIST_SUFFIX = _list\nMAX_ITEM_LIST_LENGTH = 50\nPOSITION_FIELD = position_id\nHEAD_ENTITY_ID_FIELD = head_id\nTAIL_ENTITY_ID_FIELD = tail_id\nRELATION_ID_FIELD = relation_id\nENTITY_ID_FIELD = entity_id\nbenchmark_filename = None\n\nOther Hyper Parameters: \nwandb_project = recbole\nrequire_pow = False\nembedding_size = 64\nhidden_size = 128\nnum_layers = 1\ndropout_prob = 0.3\nselected_features = ['product_code', 'product_type_no', 'product_group_name', 'graphical_appearance_no', 'colour_group_code', 'perceived_colour_value_id', 'perceived_colour_master_id', 'department_no', 'index_code', 'index_group_no', 'section_no', 'garment_group_no']\npooling_mode = sum\nloss_type = CE\nMODEL_TYPE = ModelType.SEQUENTIAL\nMODEL_INPUT_TYPE = InputType.POINTWISE\neval_type = EvaluatorType.RANKING\ndevice = cuda\ntrain_neg_sample_args = {'strategy': 'none'}\neval_neg_sample_args = {'strategy': 'full', 'distribution': 'uniform'}\n\n\n"
],
[
"dataset = create_dataset(config)\nlogger.info(dataset)",
"recbole_data\nThe number of users: 15459\nAverage actions of users: 59.21956268598784\nThe number of items: 7330\nAverage actions of items: 124.9032610178742\nThe number of inters: 915416\nThe sparsity of the dataset: 99.19214553975321%\nRemain Fields: ['user_id', 'item_id', 'timestamp', 'product_code', 'product_type_no', 'product_group_name', 'graphical_appearance_no', 'colour_group_code', 'perceived_colour_value_id', 'perceived_colour_master_id', 'department_no', 'index_code', 'index_group_no', 'section_no', 'garment_group_no']\n"
],
[
"# dataset splitting\ntrain_data, valid_data, test_data = data_preparation(config, dataset)",
"[Training]: train_batch_size = [2048] negative sampling: [None]\n[Evaluation]: eval_batch_size = [4096] eval_args: [{'split': {'RS': [10, 0, 0]}, 'group_by': 'user', 'order': 'TO', 'mode': 'full'}]\n"
],
[
"# model loading and initialization\nmodel = GRU4RecF(config, train_data.dataset).to(config['device'])\nlogger.info(model)\n\n# trainer loading and initialization\ntrainer = Trainer(config, model)\n\n# model training\nbest_valid_score, best_valid_result = trainer.fit(train_data)",
"GRU4RecF(\n (item_embedding): Embedding(7330, 64, padding_idx=0)\n (feature_embed_layer): FeatureSeqEmbLayer(\n (token_embedding_table): ModuleDict(\n (item): FMEmbedding(\n (embedding): Embedding(3935, 64)\n )\n )\n (float_embedding_table): ModuleDict(\n (item): Embedding(1, 64)\n )\n (token_seq_embedding_table): ModuleDict(\n (item): ModuleList(\n (0): Embedding(16, 64)\n )\n )\n )\n (item_gru_layers): GRU(64, 128, bias=False, batch_first=True)\n (feature_gru_layers): GRU(768, 128, bias=False, batch_first=True)\n (dense_layer): Linear(in_features=256, out_features=64, bias=True)\n (dropout): Dropout(p=0.3, inplace=False)\n (loss_fct): CrossEntropyLoss()\n)\nTrainable parameters: 1156288\nepoch 0 training [time: 45.54s, train loss: 3642.3637]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 1 training [time: 43.23s, train loss: 3390.6134]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 2 training [time: 43.00s, train loss: 3250.4472]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 3 training [time: 43.16s, train loss: 3163.3735]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 4 training [time: 42.99s, train loss: 3099.2533]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 5 training [time: 42.99s, train loss: 3044.1074]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 6 training [time: 43.14s, train loss: 2998.5542]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 7 training [time: 42.97s, train loss: 2962.4046]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 8 training [time: 42.91s, train loss: 2932.5592]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 9 training [time: 42.97s, train loss: 2907.5308]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 10 training [time: 42.84s, train loss: 2885.6282]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 11 training [time: 42.92s, train loss: 2867.5368]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 12 training [time: 42.82s, train loss: 2850.5957]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 13 training [time: 42.83s, train loss: 2836.0930]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 14 training [time: 43.10s, train loss: 2822.5238]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 15 training [time: 42.77s, train loss: 2811.0895]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 16 training [time: 42.90s, train loss: 2799.8698]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 17 training [time: 42.98s, train loss: 2790.4907]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 18 training [time: 42.79s, train loss: 2781.8785]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 19 training [time: 42.84s, train loss: 2774.2283]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 20 training [time: 42.86s, train loss: 2766.8078]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 21 training [time: 42.85s, train loss: 2760.0341]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 22 training [time: 43.00s, train loss: 2753.8305]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 23 training [time: 42.64s, train loss: 2748.0924]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 24 training [time: 42.53s, train loss: 2742.5462]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 25 training [time: 42.67s, train loss: 2737.9435]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 26 training [time: 42.88s, train loss: 2732.9869]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 27 training [time: 43.01s, train loss: 2728.7535]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 28 training [time: 42.79s, train loss: 2724.6929]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 29 training [time: 42.88s, train loss: 2720.6401]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 30 training [time: 42.76s, train loss: 2716.7650]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 31 training [time: 42.87s, train loss: 2712.6816]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 32 training [time: 42.76s, train loss: 2709.9449]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 33 training [time: 42.76s, train loss: 2706.3356]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 34 training [time: 42.60s, train loss: 2703.5010]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 35 training [time: 42.95s, train loss: 2700.1212]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 36 training [time: 42.59s, train loss: 2696.9098]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 37 training [time: 42.57s, train loss: 2694.2124]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 38 training [time: 42.80s, train loss: 2691.2175]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 39 training [time: 42.72s, train loss: 2688.2775]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 40 training [time: 42.52s, train loss: 2687.1207]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 41 training [time: 42.78s, train loss: 2683.6108]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 42 training [time: 42.70s, train loss: 2680.3406]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 43 training [time: 42.75s, train loss: 2678.0644]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 44 training [time: 42.91s, train loss: 2676.2267]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 45 training [time: 42.72s, train loss: 2673.2900]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 46 training [time: 42.84s, train loss: 2670.9084]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 47 training [time: 42.59s, train loss: 2668.3615]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 48 training [time: 42.60s, train loss: 2667.0580]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 49 training [time: 42.56s, train loss: 2664.2967]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 50 training [time: 42.78s, train loss: 2662.0451]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 51 training [time: 42.73s, train loss: 2660.1352]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 52 training [time: 42.78s, train loss: 2657.7945]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 53 training [time: 42.61s, train loss: 2656.4686]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 54 training [time: 42.59s, train loss: 2654.0421]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 55 training [time: 42.58s, train loss: 2651.8889]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 56 training [time: 42.60s, train loss: 2650.4294]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 57 training [time: 42.74s, train loss: 2648.3119]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 58 training [time: 42.59s, train loss: 2646.3108]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 59 training [time: 42.52s, train loss: 2644.7120]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 60 training [time: 42.59s, train loss: 2642.6164]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 61 training [time: 42.59s, train loss: 2640.6564]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 62 training [time: 42.57s, train loss: 2639.2032]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 63 training [time: 42.41s, train loss: 2637.3143]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 64 training [time: 42.18s, train loss: 2635.7403]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 65 training [time: 42.17s, train loss: 2634.4346]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 66 training [time: 42.22s, train loss: 2632.6797]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 67 training [time: 42.12s, train loss: 2630.9897]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 68 training [time: 42.24s, train loss: 2629.3250]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 69 training [time: 42.21s, train loss: 2627.6307]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 70 training [time: 42.24s, train loss: 2626.2554]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 71 training [time: 42.09s, train loss: 2624.0660]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 72 training [time: 42.27s, train loss: 2623.2111]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 73 training [time: 42.10s, train loss: 2621.6412]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 74 training [time: 42.08s, train loss: 2623.1531]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 75 training [time: 42.14s, train loss: 2618.6150]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 76 training [time: 42.16s, train loss: 2617.9686]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 77 training [time: 42.02s, train loss: 2615.9039]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 78 training [time: 42.13s, train loss: 2613.9363]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 79 training [time: 42.12s, train loss: 2612.7309]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 80 training [time: 42.09s, train loss: 2610.8800]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 81 training [time: 42.16s, train loss: 2609.9121]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 82 training [time: 42.10s, train loss: 2608.5970]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 83 training [time: 42.08s, train loss: 2607.7214]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 84 training [time: 42.15s, train loss: 2605.9061]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 85 training [time: 42.24s, train loss: 2604.9589]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 86 training [time: 42.12s, train loss: 2603.3150]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 87 training [time: 42.05s, train loss: 2602.3913]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 88 training [time: 42.11s, train loss: 2600.7972]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 89 training [time: 42.12s, train loss: 2599.0803]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 90 training [time: 42.23s, train loss: 2598.2695]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 91 training [time: 42.18s, train loss: 2596.9184]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 92 training [time: 42.15s, train loss: 2596.0503]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 93 training [time: 42.47s, train loss: 2594.9000]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 94 training [time: 42.34s, train loss: 2593.6262]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 95 training [time: 42.17s, train loss: 2592.5283]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 96 training [time: 42.30s, train loss: 2591.2610]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 97 training [time: 42.32s, train loss: 2589.8100]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 98 training [time: 42.29s, train loss: 2589.1635]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\nepoch 99 training [time: 42.22s, train loss: 2588.0752]\nSaving current: saved/GRU4RecF-Apr-29-2022_07-24-58.pth\n"
]
],
[
[
"## Generate trained recommender based predictions ",
"_____no_output_____"
]
],
[
[
"from recbole.utils.case_study import full_sort_topk\nexternal_user_ids = dataset.id2token(\n dataset.uid_field, list(range(dataset.user_num)))[1:]#fist element in array is 'PAD'(default of Recbole) ->remove it ",
"_____no_output_____"
],
[
"import torch\nfrom recbole.data.interaction import Interaction\n\ndef add_last_item(old_interaction, last_item_id, max_len=50):\n new_seq_items = old_interaction['item_id_list'][-1]\n if old_interaction['item_length'][-1].item() < max_len:\n new_seq_items[old_interaction['item_length'][-1].item()] = last_item_id\n else:\n new_seq_items = torch.roll(new_seq_items, -1)\n new_seq_items[-1] = last_item_id\n return new_seq_items.view(1, len(new_seq_items))\n\ndef predict_for_all_item(external_user_id, dataset, model):\n model.eval()\n with torch.no_grad():\n uid_series = dataset.token2id(dataset.uid_field, [external_user_id])\n index = np.isin(dataset.inter_feat[dataset.uid_field].numpy(), uid_series)\n input_interaction = dataset[index]\n test = {\n 'item_id_list': add_last_item(input_interaction, \n input_interaction['item_id'][-1].item(), model.max_seq_length),\n 'item_length': torch.tensor(\n [input_interaction['item_length'][-1].item() + 1\n if input_interaction['item_length'][-1].item() < model.max_seq_length else model.max_seq_length])\n }\n new_inter = Interaction(test)\n new_inter = new_inter.to(config['device'])\n new_scores = model.full_sort_predict(new_inter)\n new_scores = new_scores.view(-1, test_data.dataset.item_num)\n new_scores[:, 0] = -np.inf # set scores of [pad] to -inf\n return torch.topk(new_scores, 12)",
"_____no_output_____"
],
[
"topk_items = []\nfor external_user_id in external_user_ids:\n _, topk_iid_list = predict_for_all_item(external_user_id, dataset, model)\n last_topk_iid_list = topk_iid_list[-1]\n external_item_list = dataset.id2token(dataset.iid_field, last_topk_iid_list.cpu()).tolist()\n topk_items.append(external_item_list)\nprint(len(topk_items))",
"15458\n"
],
[
"external_item_str = [' '.join(x) for x in topk_items]\nresult = pd.DataFrame(external_user_ids, columns=['customer_id'])\nresult['prediction'] = external_item_str\nresult.head()",
"_____no_output_____"
],
[
"del external_item_str\ndel topk_items\ndel external_user_ids\ndel train_data\ndel valid_data\ndel test_data\ndel model\ndel Trainer\ndel logger\ndel dataset\ngc.collect()",
"_____no_output_____"
]
],
[
[
"## Reading Submission data",
"_____no_output_____"
]
],
[
[
"submission_data = pd.read_csv(submission_data_path)\nsubmission_data.shape",
"_____no_output_____"
]
],
[
[
"## Postprocessing submision data \n\n1. Replacing trained customer ids based prediction from recbole based predictions by performing merge \n2. Filling up Nan values for customer ids which were not part of recbole training session \n3. Generating the final prediction column \n4. Dropping redundant columns",
"_____no_output_____"
]
],
[
[
"submission_data = pd.merge(submission_data, result, on='customer_id', how='outer')\nsubmission_data",
"_____no_output_____"
],
[
"submission_data = submission_data.fillna(-1)\nsubmission_data['prediction'] = submission_data.apply(\n lambda x: x['prediction_y'] if x['prediction_y'] != -1 else x['prediction_x'], axis=1)\nsubmission_data",
"_____no_output_____"
],
[
"submission_data = submission_data.drop(columns=['prediction_y', 'prediction_x'])\nsubmission_data",
"_____no_output_____"
]
],
[
[
"## Writing final submission file to kaggle output disk",
"_____no_output_____"
]
],
[
[
"submission_data.to_csv('submission.csv', index=False)",
"_____no_output_____"
]
]
] | [
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code"
] | [
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
]
] |
e7ecd9df78b1ef04c0b9086f6e812a6caa4da56a | 275,552 | ipynb | Jupyter Notebook | module3-quantile-regression/Copy_of_LS_DS1_233_Quantile_Regression.ipynb | quinn-dougherty/DS-Unit-2-Sprint-3-Advanced-Regression | 8c54dc6ee5ad9c2cee20e02647b8cb3ef7629297 | [
"MIT"
] | 1 | 2020-01-06T10:30:39.000Z | 2020-01-06T10:30:39.000Z | module3-quantile-regression/Copy_of_LS_DS1_233_Quantile_Regression.ipynb | quinn-dougherty/DS-Unit-2-Sprint-3-Advanced-Regression | 8c54dc6ee5ad9c2cee20e02647b8cb3ef7629297 | [
"MIT"
] | null | null | null | module3-quantile-regression/Copy_of_LS_DS1_233_Quantile_Regression.ipynb | quinn-dougherty/DS-Unit-2-Sprint-3-Advanced-Regression | 8c54dc6ee5ad9c2cee20e02647b8cb3ef7629297 | [
"MIT"
] | null | null | null | 101.045838 | 136,066 | 0.680841 | [
[
[
"<a href=\"https://colab.research.google.com/github/quinn-dougherty/DS-Unit-2-Sprint-3-Advanced-Regression/blob/master/module3-quantile-regression/Copy_of_LS_DS1_233_Quantile_Regression.ipynb\" target=\"_parent\"><img src=\"https://colab.research.google.com/assets/colab-badge.svg\" alt=\"Open In Colab\"/></a>",
"_____no_output_____"
],
[
"# Lambda School Data Science - Quantile Regression\n\nRegressing towards the median - or any quantile - as a way to mitigate outliers and control risk.",
"_____no_output_____"
],
[
"## Lecture\n\nLet's look at data that has a bit of a skew to it:\n\nhttp://archive.ics.uci.edu/ml/datasets/Beijing+PM2.5+Data",
"_____no_output_____"
]
],
[
[
"import numpy as np\nimport pandas as pd\nfrom sklearn.linear_model import LinearRegression\nimport statsmodels.formula.api as smf\n\n\ndf = pd.read_csv('http://archive.ics.uci.edu/ml/machine-learning-databases/'\n '00381/PRSA_data_2010.1.1-2014.12.31.csv')",
"_____no_output_____"
],
[
"df.head()",
"_____no_output_____"
],
[
"df.describe()",
"_____no_output_____"
],
[
"df['pm2.5'].plot.hist();\n\nnp.log(df['pm2.5'])",
"/usr/local/lib/python3.6/dist-packages/ipykernel_launcher.py:3: RuntimeWarning: divide by zero encountered in log\n This is separate from the ipykernel package so we can avoid doing imports until\n"
],
[
"# How does linear regression handle it?\nfrom sklearn.linear_model import LinearRegression\n\n# Let's drop NAs and limit to numeric values\ndf = df._get_numeric_data().dropna()\nX = df.drop('pm2.5', axis='columns')\ny = df['pm2.5']\n\nlinear_reg = LinearRegression().fit(X, y)\nlinear_reg.score(X, y)",
"_____no_output_____"
],
[
"# Not bad - but what if we wanted to model the distribution more conservatively?\n# Let's try quantile\nimport statsmodels.formula.api as smf\n\n# Different jargon/API in StatsModel documentation\n# \"endogenous\" response var is dependent (y), it is \"inside\"\n# \"exogenous\" variables are independent (X), it is \"outside\"\n# Bonus points - talk about \"exogenous shocks\" and you're a bona fide economist\n\n# ~ style formulas look like what R uses\n# y ~ x1 + x2 + ...\n# they also support * for interaction terms, polynomiasl \n# Also, these formulas break with . in variable name, so lets change that\ndf = df.rename(index=str, columns={'pm2.5': 'pm25'})\n\n# Now let's construct the formula string using all columns\nquant_formula = 'pm25 ~ ' + ' + '.join(df.drop('pm25', axis='columns').columns)\nprint(quant_formula)\n\nquant_mod = smf.quantreg(quant_formula, data=df)\nquant_reg = quant_mod.fit(q=.5)\nquant_reg.summary() # \"summary\" is another very R-thing",
"pm25 ~ No + year + month + day + hour + DEWP + TEMP + PRES + cbwd + Iws + Is + Ir\n"
]
],
[
[
"That fit to the median (q=0.5), also called \"Least Absolute Deviation.\" The pseudo-R^2 isn't really directly comparable to the R^2 from linear regression, but it clearly isn't dramatically improved. Can we make it better?",
"_____no_output_____"
]
],
[
[
"help(quant_mod.fit)",
"Help on method fit in module statsmodels.regression.quantile_regression:\n\nfit(q=0.5, vcov='robust', kernel='epa', bandwidth='hsheather', max_iter=1000, p_tol=1e-06, **kwargs) method of statsmodels.regression.quantile_regression.QuantReg instance\n Solve by Iterative Weighted Least Squares\n \n Parameters\n ----------\n q : float\n Quantile must be between 0 and 1\n vcov : string, method used to calculate the variance-covariance matrix\n of the parameters. Default is ``robust``:\n \n - robust : heteroskedasticity robust standard errors (as suggested\n in Greene 6th edition)\n - iid : iid errors (as in Stata 12)\n \n kernel : string, kernel to use in the kernel density estimation for the\n asymptotic covariance matrix:\n \n - epa: Epanechnikov\n - cos: Cosine\n - gau: Gaussian\n - par: Parzene\n \n bandwidth: string, Bandwidth selection method in kernel density\n estimation for asymptotic covariance estimate (full\n references in QuantReg docstring):\n \n - hsheather: Hall-Sheather (1988)\n - bofinger: Bofinger (1975)\n - chamberlain: Chamberlain (1994)\n\n"
],
[
"quantiles = (.05, .96, .1)\n\nfor quantile in quantiles:\n print(quant_mod.fit(q=quantile).summary())",
" QuantReg Regression Results \n==============================================================================\nDep. Variable: pm25 Pseudo R-squared: 0.04130\nModel: QuantReg Bandwidth: 8.908\nMethod: Least Squares Sparsity: 120.7\nDate: Sun, 20 Jan 2019 No. Observations: 41757\nTime: 23:01:53 Df Residuals: 41745\n Df Model: 11\n==============================================================================\n coef std err t P>|t| [0.025 0.975]\n------------------------------------------------------------------------------\nIntercept 3.072e-05 6.4e-06 4.803 0.000 1.82e-05 4.33e-05\nNo -6.994e-05 9.59e-06 -7.292 0.000 -8.87e-05 -5.11e-05\nyear 0.0998 0.012 8.275 0.000 0.076 0.123\nmonth -0.4536 0.034 -13.419 0.000 -0.520 -0.387\nday 0.1143 0.015 7.862 0.000 0.086 0.143\nhour 0.3777 0.020 19.013 0.000 0.339 0.417\nDEWP 0.7720 0.014 55.266 0.000 0.745 0.799\nTEMP -0.8346 0.020 -41.621 0.000 -0.874 -0.795\nPRES -0.1734 0.024 -7.290 0.000 -0.220 -0.127\nIws -0.0364 0.002 -17.462 0.000 -0.040 -0.032\nIs 1.4573 0.195 7.466 0.000 1.075 1.840\nIr -1.2952 0.071 -18.209 0.000 -1.435 -1.156\n==============================================================================\n\nThe condition number is large, 3.67e+10. This might indicate that there are\nstrong multicollinearity or other numerical problems.\n QuantReg Regression Results \n==============================================================================\nDep. Variable: pm25 Pseudo R-squared: 0.2194\nModel: QuantReg Bandwidth: 10.41\nMethod: Least Squares Sparsity: 1322.\nDate: Sun, 20 Jan 2019 No. Observations: 41757\nTime: 23:01:55 Df Residuals: 41745\n Df Model: 11\n==============================================================================\n coef std err t P>|t| [0.025 0.975]\n------------------------------------------------------------------------------\nIntercept 0.0004 6.87e-05 5.306 0.000 0.000 0.000\nNo 7.821e-05 0.000 0.696 0.486 -0.000 0.000\nyear 1.0580 0.124 8.539 0.000 0.815 1.301\nmonth -3.9661 0.446 -8.895 0.000 -4.840 -3.092\nday 1.0816 0.136 7.936 0.000 0.814 1.349\nhour 2.3661 0.192 12.354 0.000 1.991 2.741\nDEWP 7.5176 0.235 32.004 0.000 7.057 7.978\nTEMP -11.6991 0.302 -38.691 0.000 -12.292 -11.106\nPRES -1.7121 0.244 -7.003 0.000 -2.191 -1.233\nIws -0.4151 0.034 -12.339 0.000 -0.481 -0.349\nIs -5.7267 1.580 -3.624 0.000 -8.824 -2.630\nIr -9.3197 1.457 -6.397 0.000 -12.175 -6.464\n==============================================================================\n\nThe condition number is large, 3.67e+10. This might indicate that there are\nstrong multicollinearity or other numerical problems.\n QuantReg Regression Results \n==============================================================================\nDep. Variable: pm25 Pseudo R-squared: 0.06497\nModel: QuantReg Bandwidth: 8.092\nMethod: Least Squares Sparsity: 104.4\nDate: Sun, 20 Jan 2019 No. Observations: 41757\nTime: 23:01:57 Df Residuals: 41745\n Df Model: 11\n==============================================================================\n coef std err t P>|t| [0.025 0.975]\n------------------------------------------------------------------------------\nIntercept 5.214e-05 7.84e-06 6.650 0.000 3.68e-05 6.75e-05\nNo -9.232e-05 1.17e-05 -7.888 0.000 -0.000 -6.94e-05\nyear 0.1521 0.015 10.386 0.000 0.123 0.181\nmonth -0.5581 0.042 -13.138 0.000 -0.641 -0.475\nday 0.1708 0.017 9.893 0.000 0.137 0.205\nhour 0.4604 0.024 19.350 0.000 0.414 0.507\nDEWP 1.2350 0.017 70.845 0.000 1.201 1.269\nTEMP -1.3088 0.024 -54.101 0.000 -1.356 -1.261\nPRES -0.2652 0.029 -9.183 0.000 -0.322 -0.209\nIws -0.0436 0.003 -16.919 0.000 -0.049 -0.039\nIs 1.0745 0.231 4.653 0.000 0.622 1.527\nIr -1.9619 0.087 -22.504 0.000 -2.133 -1.791\n==============================================================================\n\nThe condition number is large, 3.67e+10. This might indicate that there are\nstrong multicollinearity or other numerical problems.\n"
]
],
[
[
"\"Strong multicollinearity\", eh? In other words - maybe we shouldn't throw every variable in our formula. Let's hand-craft a smaller one, picking the features with the largest magnitude t-statistics for their coefficients. Let's also search for more quantile cutoffs to see what's most effective.",
"_____no_output_____"
]
],
[
[
"quant_formula = 'pm25 ~ DEWP + TEMP + Ir + hour + Iws'\nquant_mod = smf.quantreg(quant_formula, data=df)\nfor quantile in range(50, 100):\n quantile /= 100\n quant_reg = quant_mod.fit(q=quantile)\n print((quantile, quant_reg.prsquared))",
"(0.5, 0.1447879336023583)\n(0.51, 0.1453046499109799)\n(0.52, 0.1457984456251047)\n(0.53, 0.14627848333343263)\n(0.54, 0.1467640446791706)\n(0.55, 0.14733094668790292)\n(0.56, 0.14787821797693512)\n(0.57, 0.14840185621049273)\n(0.58, 0.14892138273908107)\n(0.59, 0.14946793972061212)\n"
],
[
"# Okay, this data seems *extremely* skewed\n# Let's trying logging\nimport numpy as np\n\ndf['pm25'] = np.log(1 + df['pm25'])\nquant_mod = smf.quantreg(quant_formula, data=df)\nquant_reg = quant_mod.fit(q=.25)\nquant_reg.summary() # \"summary\" is another very R-thing",
"_____no_output_____"
]
],
[
[
"Overall - in this case, quantile regression is not *necessarily* superior to linear regression. But it does give us extra flexibility and another thing to tune - what the center of what we're actually fitting in the dependent variable.\n\nThe basic case of `q=0.5` (the median) minimizes the absolute value of residuals, while OLS minimizes the squared value. By selecting `q=0.25`, we're targeting a lower quantile and are effectively saying that we only want to over-estimate at most 25% of the time - we're being *risk averse*.\n\nDepending on the data you're looking at, and the cost of making a false positive versus a false negative, this sort of flexibility can be extremely useful.\n\nLive - let's consider another dataset! Specifically, \"SkillCraft\" (data on competitive StarCraft players): http://archive.ics.uci.edu/ml/datasets/SkillCraft1+Master+Table+Dataset",
"_____no_output_____"
]
],
[
[
"import numpy as np\nimport pandas as pd\nfrom sklearn.linear_model import LinearRegression\nfrom sklearn.linear_model import LogisticRegression\nimport statsmodels.formula.api as smf\nurl = 'http://archive.ics.uci.edu/ml/machine-learning-databases/00272/SkillCraft1_Dataset.csv' \n\ndf = pd.read_csv(url)\n\ndf.head()",
"_____no_output_____"
],
[
"\ndf = df.replace('?', np.nan)\n\n\ndf.shape, df.isna().sum()\n\nhasna = ['Age', 'HoursPerWeek', 'TotalHours']\n\nfor feat in hasna: \n df[feat] = pd.to_numeric(df[feat])\n \ndf[hasna].head()\n\ndf.isna().sum(), df.dtypes, df.shape\n\n\ndf.describe()",
"_____no_output_____"
],
[
"from sklearn.linear_model import LinearRegression \n\ndff = df._get_numeric_data().dropna()\nX = df.drop('APM', axis='columns')\ny = df.APM\n\nlinear_reg = LinearRegression().fit(X,y)\nlinear_reg",
"_____no_output_____"
],
[
"from sklearn.linear_model import LogisticRegression\n\n## what fastest 10% of starcraft players like? \n## wquantile regression with q=0.9\n\nquant_formula = 'APM ~ ' + ' + '.join(df.drop('APM', axis='columns').columns)\n\nquant_mod = smf.quantreg(quant_formula, data=dff) \nquant_reg = quant_mod.fit(q=0.9)\nquant_reg.summary()",
"/usr/local/lib/python3.6/dist-packages/statsmodels/regression/quantile_regression.py:193: IterationLimitWarning: Maximum number of iterations (1000) reached.\n \") reached.\", IterationLimitWarning)\n"
],
[
"# TODO Live!\n# Hint - we may only care about the *top* quantiles here\n# Another hint - there are missing values, but Pandas won't see them right away\n\nnp.sqrt(0.4076)",
"_____no_output_____"
]
],
[
[
"## Assignment - birth weight data\n\nBirth weight is a situation where, while the data itself is actually fairly normal and symmetric, our main goal is actually *not* to model mean weight (via OLS), but rather to identify mothers at risk of having children below a certain \"at-risk\" threshold weight.\n\nQuantile regression gives us just the tool we need. For the data we are using, see: http://people.reed.edu/~jones/141/BirthWgt.html\n\n bwt: baby's weight in ounces at birth\n gestation: duration of pregnancy in days\n parity: parity indicator (first born = 1, later birth = 0)\n age: mother's age in years\n height: mother's height in inches\n weight: mother's weight in pounds (during pregnancy)\n smoke: indicator for whether mother smokes (1=yes, 0=no) \n \nUse this data and `statsmodels` to fit a quantile regression, predicting `bwt` (birth weight) as a function of the other covariates. First, identify an appropriate `q` (quantile) to target a cutoff of 90 ounces - babies above that birth weight are generally healthy/safe, babies below are at-risk.\n\nThen, fit and iterate your model. Be creative! You may want to engineer features. Hint - mother's age likely is not simply linear in its impact, and the other features may interact as well.\n\nAt the end, create at least *2* tables and *1* visualization to summarize your best model. Then (in writing) answer the following questions:\n\n- What characteristics of a mother indicate the highest likelihood of an at-risk (low weight) baby?\n- What can expectant mothers be told to help mitigate this risk?\n\nNote that second question is not exactly a data science question - and that's okay! You're not expected to be a medical expert, but it is a good exercise to do a little bit of digging into a particular domain and offer informal but informed opinions.",
"_____no_output_____"
]
],
[
[
"import numpy as np\nimport pandas as pd\nfrom sklearn.linear_model import LinearRegression\nfrom sklearn.linear_model import LogisticRegression\nimport statsmodels.formula.api as smf\n\nfrom scipy.stats import percentileofscore\nfrom numpy.testing import assert_almost_equal\n\nbwt_df = pd.read_csv('http://people.reed.edu/~jones/141/Bwt.dat')\nbwt_df.head()",
"_____no_output_____"
],
[
"bwt_df.dtypes, bwt_df.isna().sum(), bwt_df.dtypes, bwt_df.isna().sum()\n\nbwt_df.describe()",
"_____no_output_____"
],
[
"#First, identify an appropriate q (quantile) to target a cutoff of 90 ounces - \n# babies above that birth weight are generally healthy/safe, babies below are at-risk.\n\nq_90_test = 0.055 #percentileofscore(bwt_df.bwt, 90)/100\n\nq_90 = np.divide(percentileofscore(bwt_df.bwt, 90), 100)\n\nbwt_df.bwt.quantile(q=q_90)\n\n\n#0.5-distn == q_90\n\n",
"_____no_output_____"
],
[
"\nquant_formula = 'bwt ~ ' + ' + '.join(bwt_df.drop(['bwt'], axis='columns').columns)\n\nquant_mod = smf.quantreg(quant_formula, data=bwt_df) \nquant_reg = quant_mod.fit(q=q_90)\nquant_reg.summary()",
"_____no_output_____"
],
[
"bwt_df['weirdfeat'] = bwt_df.age * (bwt_df.parity - 2)\n\nbwt_df['age_per_weight'] = np.divide(bwt_df.age, bwt_df.weight)\n\nbwt_df['apw_times_parityplus1'] = bwt_df.age_per_weight * (bwt_df.parity+1)\n\nbwt_df['BMI'] = np.divide(bwt_df.weight, bwt_df.height**2) * 703 \n\nbwt_df['weirdishfeat'] = bwt_df.gestation ** (bwt_df.parity+1)\n\nbwt_df['gestation_squrd'] = bwt_df.gestation ** 2\n\n#bwt_df['weirdfeat'] = bwt_df.weirdfeat + 3\n\nquant_formula = 'bwt ~ ' + ' + '.join(bwt_df.drop(['bwt', 'weirdfeat', 'apw_times_parityplus1', 'weirdishfeat'], axis='columns').columns)\n\nquant_mod = smf.quantreg(quant_formula, data=bwt_df) \nquant_reg = quant_mod.fit(q=q_90)\nquant_reg.summary()\n\n#bwt_df.weirdfeat",
"/usr/local/lib/python3.6/dist-packages/statsmodels/regression/quantile_regression.py:193: IterationLimitWarning: Maximum number of iterations (1000) reached.\n \") reached.\", IterationLimitWarning)\n"
],
[
"",
"_____no_output_____"
],
[
"from seaborn import pairplot\n\npairplot(data=bwt_df, \n x_vars=bwt_df.drop(['bwt', 'weirdfeat', 'apw_times_parityplus1', \n 'weirdishfeat'], axis='columns').columns, \n y_vars='bwt');",
"_____no_output_____"
]
],
[
[
"## Gestation is our most reliable feature.\n- it's errors are consistently reasonable, and it's p-value is consistently very small, adding gestation ** 2 improved the model. \n## BMI is better\n- the model is better with BMI than without \n\n## Adding BMI made age_per_weight less effective \n- age_per_weight's error and p-value both went up when BMI was added. \n\n# Obviously, _don't smoke_ ",
"_____no_output_____"
]
],
[
[
"'''\n What characteristics of a mother indicate the highest likelihood of an at-risk (low weight) baby?\n What can expectant mothers be told to help mitigate this risk?\n'''",
"_____no_output_____"
]
],
[
[
"## Resources and stretch goals",
"_____no_output_____"
],
[
"Resources:\n- [statsmodels QuantReg example](http://www.statsmodels.org/dev/examples/notebooks/generated/quantile_regression.html)\n- [How Shopify used Quantile Regression in modeling risk](https://medium.com/data-shopify/how-shopify-capital-uses-quantile-regression-to-help-merchants-succeed-10ee1b36b17d)\n\nStretch goals:\n- Find a dataset where you think quantile regression may be appropriate, and try both it and linear regression - compare/contrast their strengths/weaknesses, and write a summary for which you think is better for the situation and why\n- Check out [deep quantile regression](https://www.kdnuggets.com/2018/07/deep-quantile-regression.html), an approach that uses a custom quantile loss function and Keras to train a quantile model",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown",
"markdown",
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
]
] |
e7ecf186ee6a85ad4ce5243bad11379f66cc7135 | 648,514 | ipynb | Jupyter Notebook | Results/res1.ipynb | Jayshil/csfrd | 9f9e8ccea93f464cfd7cf45c06b05738194d3da5 | [
"MIT"
] | null | null | null | Results/res1.ipynb | Jayshil/csfrd | 9f9e8ccea93f464cfd7cf45c06b05738194d3da5 | [
"MIT"
] | null | null | null | Results/res1.ipynb | Jayshil/csfrd | 9f9e8ccea93f464cfd7cf45c06b05738194d3da5 | [
"MIT"
] | null | null | null | 1,525.915294 | 107,031 | 0.768101 | [
[
[
"# Updating SFRDs: UV data\n\nThanks to the improvements in observational facilities in the past few years, we were able to compute luminosity function more accurately. We now use this updated measurements of luminosity fucntion to update the values of SFRDs. In the present notebook, we focus on UV luminosity functions, which are described by the classical Schechter function (detailed description can be found in [this](https://github.com/Jayshil/csfrd/blob/main/p1.ipynb) notebook). We assumes the correlation between the Schechter function parameters similar to what observed in Bouwens et al. (2021) --- that means, at any redshift, the correlation assumed to be the same.",
"_____no_output_____"
]
],
[
[
"import numpy as np\nimport matplotlib.pyplot as plt\nimport astropy.constants as con\nimport astropy.units as u\nfrom scipy.optimize import minimize as mz\nfrom scipy.optimize import curve_fit as cft\nimport utils as utl\nimport os",
"_____no_output_____"
]
],
[
[
"We have already computed the SFRDs by using [this](https://github.com/Jayshil/csfrd/blob/main/sfrd_all.py) code -- here we only plot the results.",
"_____no_output_____"
]
],
[
[
"ppr_uv = np.array(['Khusanova_et_al._2020', 'Ono_et_al._2017', 'Viironen_et_al._2018', 'Finkelstein_et_al._2015', 'Bouwens_et_al._2021', 'Alavi_et_al._2016', 'Livermore_et_al._2017', 'Atek_et_al._2015', 'Parsa_et_al._2016', 'Hagen_et_al._2015', 'Moutard_et_al._2019', 'Pello_et_al._2018', 'Bhatawdekar_et_al._2018'])\n\ncols = np.array(['cyan', 'deepskyblue', 'steelblue', 'dodgerblue', 'cornflowerblue', 'royalblue', 'navy', 'blue', 'slateblue', 'darkslateblue', 'blueviolet', 'indigo', 'mediumorchid'])\n\n#ppr_uv = np.array(['Khusanova_et_al._2020', 'Ono_et_al._2017', 'Viironen_et_al._2018', 'Finkelstein_et_al._2015', 'Bouwens_et_al._2021', 'Alavi_et_al._2016', 'Livermore_et_al._2017', 'Atek_et_al._2015', 'Parsa_et_al._2016', 'Moutard_et_al._2019', 'Pello_et_al._2018', 'Bhatawdekar_et_al._2018'])\n\n# Loading papers\nppr_uv1 = np.loadtxt('sfrd_uv_new.dat', usecols=0, unpack=True, dtype=str)\nzd_uv, zu_uv, sfrd_uv, sfrd_uv_err = np.loadtxt('sfrd_uv_new.dat', usecols=(1,2,3,4), unpack=True)\nzcen_uv = (zd_uv + zu_uv)/2\nzup, zdo = np.abs(zu_uv - zcen_uv), np.abs(zcen_uv - zd_uv)\nlog_sfrd_uv, log_sfrd_uv_err = utl.log_err(sfrd_uv, sfrd_uv_err)\n\nplt.figure(figsize=(16, 9))\n# Plotting them\nfor i in range(len(ppr_uv)):\n zc_uv, zp, zn, lg_sf, lg_sfe = np.array([]), np.array([]), np.array([]), np.array([]), np.array([])\n for j in range(len(ppr_uv1)):\n if ppr_uv1[j] == ppr_uv[i]:\n zc_uv = np.hstack((zc_uv, zcen_uv[j]))\n lg_sf = np.hstack((lg_sf, log_sfrd_uv[j]))\n lg_sfe = np.hstack((lg_sfe, log_sfrd_uv_err[j]))\n zp = np.hstack((zp, zup[j]))\n zn = np.hstack((zn, zdo[j]))\n if ppr_uv[i] == 'Hagen_et_al._2015':\n continue\n else:\n plt.errorbar(zc_uv, lg_sf, xerr=[zn, zp], yerr=lg_sfe, c=cols[i], label=ppr_uv[i].replace('_',' ') + '; UV LF', fmt='o', mfc='white', mew=2)\n\n#plt.plot(znew, psi2, label='Best fitted function')\nplt.xlabel('Redshift')\nplt.ylabel(r'$\\log{\\psi}$ ($M_\\odot year^{-1} Mpc^{-3}$)')\nplt.grid()\nplt.ylim([-2.4, -1.2])\nplt.xlim([0, 8.5])\nplt.legend(loc='best')",
"_____no_output_____"
]
],
[
[
"Note that, for most of the values, the SFRD is tightly constrained. We again note here that, in this calculation we have assumed that the Schechter function parameters are correlated (except for lower redshifts), and the correlation matrix is according to Bouwens et al. (2021). For lowest redshifts ($z=0$ and $z=1$), we, however, assumed the independency among the Schechter function parameters.\n\nWe can now overplot the best fitted function from Madau & Dickinson (2014) on this plot,\n\n$$ \\psi(z) = 0.015 \\frac{(1+z)^{2.7}}{1 + [(1+z)/2.9]^{5.6}} M_\\odot \\ year^{-1} Mpc^{-3}$$\n\nHere, the symbols have thir usual meanings.",
"_____no_output_____"
]
],
[
[
"# Defining best-fitted SFRD\n\ndef psi_md(z):\n ab = (1+z)**2.7\n cd = ((1+z)/2.9)**5.6\n ef = 0.015*ab/(1+cd)\n return ef\n\n# Calculating psi(z)\nznew = np.linspace(0,9,1000)\npsi1 = psi_md(znew)\npsi2 = np.log10(psi1)",
"_____no_output_____"
],
[
"plt.figure(figsize=(16, 9))\n# Plotting them\nfor i in range(len(ppr_uv)):\n zc_uv, zp, zn, lg_sf, lg_sfe = np.array([]), np.array([]), np.array([]), np.array([]), np.array([])\n for j in range(len(ppr_uv1)):\n if ppr_uv1[j] == ppr_uv[i]:\n zc_uv = np.hstack((zc_uv, zcen_uv[j]))\n lg_sf = np.hstack((lg_sf, log_sfrd_uv[j]))\n lg_sfe = np.hstack((lg_sfe, log_sfrd_uv_err[j]))\n zp = np.hstack((zp, zup[j]))\n zn = np.hstack((zn, zdo[j]))\n if ppr_uv[i] == 'Hagen_et_al._2015':\n continue\n else:\n plt.errorbar(zc_uv, lg_sf, xerr=[zn, zp], yerr=lg_sfe, c=cols[i], label=ppr_uv[i].replace('_',' ') + '; UV LF', fmt='o', mfc='white', mew=2)\n\nplt.plot(znew, psi2, label='Best fitted function', lw=3, c='silver')\nplt.xlabel('Redshift')\nplt.ylabel(r'$\\log{\\psi}$ ($M_\\odot year^{-1} Mpc^{-3}$)')\nplt.grid()\nplt.legend(loc='best')",
"_____no_output_____"
]
],
[
[
"It can readily be observed from the above figure that, the best-fitted function from Madau & Dickinson (2014) does not exactly match with our computation of SFRDs, which shows the need to correct for dust in these calculations. However, in the present work, we are not going to take the dust corrections. We shall compute the SFRDs for UV and IR seperately, and then just add them together. Hence, there is no need to fit the exact function to the data. What we do is to make all of the number variable in the best-fitted function from Madau & Dickinson (2014), and try to fit this to the data. Essentially, the function that we want to fit to the data is following:\n\n$$ \\psi(z) = A \\frac{(1+z)^{B}}{1 + [(1+z)/C]^{D}} M_\\odot \\ year^{-1} Mpc^{-3}$$\n\nhere, $A$, $B$, $C$ and $D$ are variables.\n\nWe use `scipy.optimize.minimize` function to perform this task. The idea is to compute the maximum likelihood function.",
"_____no_output_____"
]
],
[
[
"# New model\ndef psi_new(z, aa, bb, cc, dd):\n ab = (1+z)**bb\n cd = ((1+z)/cc)**dd\n ef = aa*ab/(1+cd)\n return ef\n\n# Negative likelihood function\ndef min_log_likelihood(x):\n model = psi_new(zcen_uv, x[0], x[1], x[2], x[3])\n chi2 = (sfrd_uv - model)/sfrd_uv_err\n chi22 = np.sum(chi2**2)\n yy = 0.5*chi22 + np.sum(np.log(sfrd_uv_err))\n return yy\n\n#xinit, pcov = cft(psi_new, zcen_uv, sfrd_uv, sigma=sfrd_uv_err)\n#xinit = np.array([0.015, 2.7, 2.9, 5.6])\nxinit = np.array([0.01, 3., 3., 6.])\nsoln = mz(min_log_likelihood, xinit, method='L-BFGS-B')\nsoln",
"_____no_output_____"
]
],
[
[
"So, the fitting in converged; that's good! Let's see how this new fitting looks like...",
"_____no_output_____"
]
],
[
[
"best_fit_fun = psi_new(znew, *soln.x)\nlog_best_fit = np.log10(best_fit_fun)\n\nplt.figure(figsize=(16,9))\nplt.errorbar(zcen_uv, log_sfrd_uv, xerr=[zup, zdo], yerr=log_sfrd_uv_err, fmt='o', c='cornflowerblue')\nplt.plot(znew, log_best_fit, label='Best fitted function', lw=2, c='orangered')\nplt.xlabel('Redshift')\nplt.ylabel(r'$\\log{\\psi}$ ($M_\\odot year^{-1} Mpc^{-3}$)')\nplt.grid()",
"_____no_output_____"
]
],
[
[
"That's sounds about right. Here, the fitted function would be,\n\n$$ \\psi(z) = 0.006 \\frac{(1+z)^{1.37}}{1 + [(1+z)/4.95]^{5.22}} M_\\odot \\ year^{-1} Mpc^{-3}$$\n\nWe want to make note here though. There are some points present in the plot which have large errorbars. Those are from the paper Hagen et al. (2015). From the quick look at the paper, it seems that, these large errorbars are there because of the large errorbars in $\\phi_*$. Anyway, in the following I try to remove those points from the data and see if the shape of the best-fitted function changes or not.",
"_____no_output_____"
]
],
[
[
"# Loading new data\nsfrd1, sfrd_err1 = np.array([]), np.array([])\nlog_sfrd1, log_sfrd_err1 = np.array([]), np.array([])\nzcen1, zdo1, zup1 = np.array([]), np.array([]), np.array([])\n\nfor i in range(len(ppr_uv1)):\n if ppr_uv1[i] != 'Hagen_et_al._2015':\n sfrd1 = np.hstack((sfrd1, sfrd_uv[i]))\n sfrd_err1 = np.hstack((sfrd_err1, sfrd_uv_err[i]))\n log_sfrd1 = np.hstack((log_sfrd1, log_sfrd_uv[i]))\n log_sfrd_err1 = np.hstack((log_sfrd_err1, log_sfrd_uv_err[i]))\n zcen1 = np.hstack((zcen1, zcen_uv[i]))\n zdo1 = np.hstack((zdo1, zdo[i]))\n zup1 = np.hstack((zup1, zup[i]))\n\n# Fitting new data\n# Negative likelihood function\ndef min_log_likelihood1(x):\n model = psi_new(zcen1, x[0], x[1], x[2], x[3])\n chi2 = (sfrd1 - model)/sfrd_err1\n chi22 = np.sum(chi2**2)\n yy = 0.5*chi22 + np.sum(np.log(sfrd_err1))\n return yy\n\n#xinit, pcov = cft(psi_new, zcen_uv, sfrd_uv, sigma=sfrd_uv_err)\n#xinit = np.array([0.015, 2.7, 2.9, 5.6])\nxinit1 = np.array([0.01, 3., 3., 6.])\nsoln1 = mz(min_log_likelihood1, xinit1, method='L-BFGS-B')\nsoln1",
"_____no_output_____"
],
[
"best_fit_fun1 = psi_new(znew, *soln1.x)\nlog_best_fit1 = np.log10(best_fit_fun1)\n\nplt.figure(figsize=(16,9))\nplt.plot(znew, log_best_fit1, label='Best fitted function', lw=2, c='silver')\n\n# Plotting Data\nfor i in range(len(ppr_uv)):\n zc_uv, zp, zn, lg_sf, lg_sfe = np.array([]), np.array([]), np.array([]), np.array([]), np.array([])\n for j in range(len(ppr_uv1)):\n if ppr_uv1[j] == ppr_uv[i]:\n zc_uv = np.hstack((zc_uv, zcen_uv[j]))\n lg_sf = np.hstack((lg_sf, log_sfrd_uv[j]))\n lg_sfe = np.hstack((lg_sfe, log_sfrd_uv_err[j]))\n zp = np.hstack((zp, zup[j]))\n zn = np.hstack((zn, zdo[j]))\n if ppr_uv[i] == 'Hagen_et_al._2015':\n continue\n else:\n plt.errorbar(zc_uv, lg_sf, xerr=[zn, zp], yerr=lg_sfe, c=cols[i], label=ppr_uv[i].replace('_',' ') + '; UV LF', fmt='o', mfc='white', mew=2)\n\nplt.xlabel('Redshift')\nplt.ylabel(r'$\\log{\\psi}$ ($M_\\odot year^{-1} Mpc^{-3}$)')\nplt.ylim([-2.4, -1.2])\nplt.xlim([0, 8.5])\nplt.legend(loc='best')\nplt.grid()",
"_____no_output_____"
]
],
[
[
"Well, it seems that, by removing the values with large errorbars, the function doesn't change much. Its new form is,\n$$ \\psi(z) = 0.006 \\frac{(1+z)^{1.37}}{1 + [(1+z)/4.94]^{5.22}} M_\\odot \\ year^{-1} Mpc^{-3}$$",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code"
],
[
"markdown"
],
[
"code",
"code"
],
[
"markdown"
]
] |
e7ecf6a43c0dfc059810bc2da5c78ed513cba652 | 497,567 | ipynb | Jupyter Notebook | SC0x/Unit 1 - Supply Chain Management Overview.ipynb | fhk/MITx_CTL_SCx | 44de3fee8637d2e97385883c9fcaffda4c0f38da | [
"MIT"
] | null | null | null | SC0x/Unit 1 - Supply Chain Management Overview.ipynb | fhk/MITx_CTL_SCx | 44de3fee8637d2e97385883c9fcaffda4c0f38da | [
"MIT"
] | 1 | 2021-08-23T20:42:29.000Z | 2021-08-23T20:42:29.000Z | SC0x/Unit 1 - Supply Chain Management Overview.ipynb | fhk/MITx_CTL_SCx | 44de3fee8637d2e97385883c9fcaffda4c0f38da | [
"MIT"
] | null | null | null | 596.603118 | 462,532 | 0.919267 | [
[
[
"# Why do we care?\n\nSupply chains have grown to span the globe.\n\nMultiple functions are now combined (warehousing, inventory, transportation, demand planning and procurement)\n\nRuns within and across a firm\n\n\"Bridge\" & \"Shockabsorber\"\n\nFrom customers to suppliers.\n\nAdapt and adjust and be flexible.\n\nPrediciting the future is hard.\n\nUsing diesel fuel as an example. \\#2 Diesel used as fuel for <u>truck load</u> and <u>less than truckload</u> transportation.",
"_____no_output_____"
]
],
[
[
"!wget https://www.eia.gov/petroleum/gasdiesel/xls/pswrgvwall.xls -O ./data/pswrgvwall.xls",
"--2020-04-24 19:35:51-- https://www.eia.gov/petroleum/gasdiesel/xls/pswrgvwall.xls\nResolving www.eia.gov (www.eia.gov)... 23.72.183.159, 2600:1406:e800:387::b93, 2600:1406:e800:3a9::b93\nConnecting to www.eia.gov (www.eia.gov)|23.72.183.159|:443... connected.\nHTTP request sent, awaiting response... 200 OK\nLength: 5796864 (5.5M) [application/vnd.ms-excel]\nSaving to: ‘./data/pswrgvwall.xls’\n\n./data/pswrgvwall.x 100%[===================>] 5.53M 5.62MB/s in 1.0s \n\n2020-04-24 19:35:53 (5.62 MB/s) - ‘./data/pswrgvwall.xls’ saved [5796864/5796864]\n\n"
],
[
"import pandas as pd",
"_____no_output_____"
],
[
"diesel = pd.read_excel(\"./data/pswrgvwall.xls\", sheet_name=None)",
"_____no_output_____"
],
[
"diesel.keys()",
"_____no_output_____"
],
[
"diesel['Data 1']",
"_____no_output_____"
],
[
"%matplotlib inline",
"_____no_output_____"
],
[
"no_head = diesel['Data 1']\nno_head.columns = no_head.iloc[1]\nno_head = no_head.iloc[2:]",
"_____no_output_____"
],
[
"no_head['Date'] = pd.to_datetime(no_head['Date'])\nno_head.set_index('Date', inplace=True)",
"/home/plantz/anaconda3/envs/sc0x/lib/python3.7/site-packages/ipykernel_launcher.py:1: SettingWithCopyWarning: \nA value is trying to be set on a copy of a slice from a DataFrame.\nTry using .loc[row_indexer,col_indexer] = value instead\n\nSee the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy\n \"\"\"Entry point for launching an IPython kernel.\n"
],
[
"no_head.plot(title=\"Diesel price $USD over time\", figsize=(20,12))",
"_____no_output_____"
]
],
[
[
"# Question: What is the impact of such variability on a supply chain?\n\nIf price was fixed you could desing a supply chain to last 10 years. But its not so you need to have a \"shock absorber\", these uncertainties/types of factors need to be considered. By using a data drive and metrics based approach organizations can pursue effeciencies that will pay dividends when prices are high and low.",
"_____no_output_____"
],
[
"## Observation: Supply Chain has been gained efficiencies such that the % contribution to GDP has been reduced\n\nFrom the Deloitter report the category is sitting at around 3%.\n\nhttps://www2.deloitte.com/us/en/insights/economy/spotlight/economics-insights-analysis-07-2019.html",
"_____no_output_____"
]
],
[
[
"from IPython.display import Image\nfrom IPython.core.display import HTML \nImage(url= \"https://www2.deloitte.com/content/dam/insights/us/articles/5024_Economics-Spotlight-July2019/figures/5024_Fig2.jpg\")",
"_____no_output_____"
]
],
[
[
"## Challenge: What got us here might not be whats needed for the future. Cost optimization might not always be the priority.",
"_____no_output_____"
],
[
"## Trend: Moving to an integrated organization, a supply chain role becomes about relationships, visibilty, collaboration, and data. Scope regional now becoming national and global",
"_____no_output_____"
]
]
] | [
"markdown",
"code",
"markdown",
"code",
"markdown"
] | [
[
"markdown"
],
[
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code",
"code"
],
[
"markdown",
"markdown"
],
[
"code"
],
[
"markdown",
"markdown"
]
] |
Subsets and Splits
No community queries yet
The top public SQL queries from the community will appear here once available.